このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
胚性幹細胞を生体内でクロマチン蛋白質のダイナミクスを測定するアッセイ(FRAP&FLIP)を光退色
要約
我々は、光退色後蛍光回復(FRAP)や胚性幹(ES)細胞におけるクロマチン蛋白質の動態を監視する(FLIP)光退色で蛍光の損失を含む光退色の方法を説明します。クロマチン可塑性を研究する手段の一つであると考えられているクロマチン蛋白質のダイナミクスは、多能性細胞で強化されています。
要約
光退色後蛍光回復(FRAP)や光退色で蛍光損失(FLIP)が良好な空間分解能と時間分解能を持つ生体細胞内の蛋白質のダイナミクスの研究を可能にします。ここでは、FRAP及びマウス胚性幹(ES)細胞のH1とHP1、を含む、クロマチンタンパク質のFLIPアッセイを、実行する方法について説明します。 FRAP実験で、細胞は緑色蛍光タンパク質(GFP)またはその誘導体(YFP、CFP、チェリー、など)を融合させた目的のタンパク質で、どちらか一過性または安定的に、トランスフェクトされています。トランスフェクト、蛍光細胞では、強烈な焦点レーザービームは、興味の比較的小さい領域(ROI)を漂白剤。レーザーの波長は、融合に使用する蛍光蛋白質に応じて選択されます。レーザー光が不可逆的にROI内の分子の蛍光シグナルを漂白剤と、すぐに漂白以下、漂白地域における蛍光シグナルの回復 - 無漂白の分子と漂白分子の交換によって媒介は - タイムラプスイメージングを使用して監視されます。生成された蛍光回復曲線は、タンパク質の流動性に関する情報を提供しています。蛍光分子が不動であれば、何蛍光回復は観察されません。補完的なアプローチでは、光退色における蛍光損失(FLIP)は、レーザービームは、繰り返し同じ場所を漂白し、信号強度は、他の場所で蛍光を発する細胞で測定されます。 FLIPの実験では、したがって、信号の減衰ではなく、蛍光の回復を測定し、タンパク質の移動度と同様に細胞区画を行き来し蛋白質を決定するのに便利です。一時的な結合は、クロマチン関連タンパク質の一般的なプロパティです。各クロマチン蛋白質の主要な割合が定常状態での任意の時点でクロマチンにバインドされていますが、バインディングは一時的であり、ほとんどのクロマチンタンパク質は、秒の順番で滞留時間で、クロマチン上で高い離職率を持っている。これらのプロパティは、ゲノムの式1に高い可塑性を発生させるために重要です。退色実験は、特にクロマチンタンパク質の動的な交換が(ヘテロクロマチンタンパク質1(HP1)、リンカーヒストンH1とコアヒストンを含む)以上であるES細胞、で、それゆえクロマチン構造蛋白質のGFP融合バージョンを使用してクロマチン可塑性を決定するために特に有用です。 2,3分化細胞に比べて。
プロトコル
1。 ES細胞をめっき
T = 0時間
MEFのメッキ
- コートライブイメージングμ-スライド8ウェル(ibidi、ミュンヘン、ドイツ)ゼラチンまたはチャンバーカバーガラスで(ラボ- Tek社、ロチェスター、ニューヨーク州)とやガラス底培養皿で(マテック、アッシュランド、マサチューセッツ州)。 5〜30分放置し、遊離ゼラチンを離れて吸引除去する。
- シード22000 MEFを/ウェルのDMEMを250μlの総容量で[10%ウシ胎児血清(FBS)を添加した]。細胞は組織培養インキュベーター(37℃、5%CO2)で成長することができます。
T = 6時間
ES細胞のメッキ
- DMEMを吸引除去する。
- ES細胞の培地250μlの総体積で15,000 R1細胞/ウェルでうまく被覆種子各MEFの[10%ESC -グレードウシ胎児血清(FBS)、1mMピルビン酸ナトリウム、0.1mMの非必須アミノ酸、0.1mMのβ-を添加したメルカプトエタノール、および1000 U / mlの白血病抑制因子(LIF)]、翌日30%〜50%コンフルエントに取得する。
2。 ES細胞のトランスフェクション
T = 24時間
一過性トランスフェクション
- 250μL/ウェル、新鮮なES細胞のメディアのES細胞の培地を交換してください。
- 1.5 mlの滅菌試験管に、100μlの無血清培地[のOpti - MEM(Gibco社)]、その後、直接無血清培地に10μlのトランジット- LT1トランスフェクション試薬(Mirus)を追加を追加。穏やかなピペッティングにより混和し、5〜20分間室温でインキュベートする。
- 希薄化後のトランジット- LT1試薬に1.5μgのGFP融合プラスミドDNA(H1E、H1oまたはHP1)を追加します。穏やかなピペッティングにより混和し、15〜30分間室温でインキュベートする。
- トランスフェクション混合物の13.5μl/ウェルを追加。さらに分散性を確保するためにμ-スライド8ウェル渦巻き。 24時間後には、新鮮なES細胞の培地を250μlと古いのES細胞培地を交換してください。
3。 FRAPやFLIPの実行
T = 48〜72時間
- 実験は、任意の共焦点レーザー走査顕微鏡(CLSM)で実行することができますが、通常のFRAP / FLIP実験で以来、多くの連続した画像が取得され、それが買収の速度を可能にしないし、その何を確実に回転するディスク共焦点顕微鏡を、使用することをお勧めします。漂白望ましくないサンプルは、最初の意図的な白化現象は、次の発生します。ここで、我々は、横河電機CSU - X回転するディスクのヘッドを、ディスク共焦点システム(www.Andor.comを)回転革命を使用することをお勧めします。このシステムは、ポイントスキャンシステムで特殊なFRAPPAモジュールを使用して、そして急速に回転するディスクを使用して画像を収集するためにバックライトを切り替えるには光退色に二重の能力を持っています。実験の光退色に使用する3最も一般的な蛍光タンパク質はGFP、YFPとチェリーです。 GFPまたはYFPが使用されている場合は、〜488 nmのレーザーが必要です。チェリーの場合は、〜560nmのレーザーを使用してください。すべてのケースでは、我々は固体レーザを使用することをお勧めします。自動ステージを持つことは有用ですが、必須ではありません。生きた細胞が撮像されているので、それは酸素、湿度、CO 2と温度を制御する、(私たちはLIS、スイスからいずれかを使用)環境室を使用することが不可欠です。 FRAPは、イメージングが(通常10%の面積で、蛍光レベルが十分なとき)に必要な最小限のレーザーパワーで行われている間、最大レーザー強度を使用して実行されます。
適切な波長の蛍光灯の光で細胞を観察し、60X油浸レンズを用いてGFPを発現する細胞を選択する。正しい細胞内分布を確認してください。時折、発現レベルが高すぎるときに、タンパク質の局在化は、核小体などの他の区画に"スピル"があります。このような細胞が選択されるべきではない。 - 今イメージングプロトコルを設定します:ユークロマチンまたはヘテロクロマチン(要約GFP巣として表示)にし、退色前に退色3から5フレームを収集し、250から1000ミリ秒の間隔で、光退色後の90から120フレームを収集してください。H1E - GFP、1000 MS、H1oとHP1 - GFP、250ミリ秒(非常に動的な蛋白質は、短い間隔の時間を必要とする蛋白質のダイナミクスに応じてインターバル時間の変更、)。我々は通常、20〜40μseconds(1-2の繰り返し)のレーザーパルスで光退色に対して80〜100%のレーザー強度を使用しますが、これらの数値は、分析タンパク質と発現レベルに応じて変更することができます。退色が適切な場合、あなたはGFPの蛍光で"ブラックホール"を観察する必要があります。ブラックホールは徐々に蛍光次の回復と再充填されている。回転するディスクは1秒間に60前後の画像(良い蛍光強度を持つとする場合、単一のセルにズームイン)まで得ることができますが、我々は低画質と潜在的な増加光毒性に起因するような高速でシステムを使用することはお勧めしません。
- FLIP実験では、別の撮像プロトコルを設定する:画像を収集中に同じ場所に漂白を繰り返し開始し、漂白の前に3から5フレームを収集する。 H1o - GFP、漂白のためのH1E - GFP、漂白剤ごとに5秒、毎2秒のため、とHP1 - GFP漂白剤毎に1秒のため。漂白剤D実験全体を通して何度も画像を収集する。
- どちらの手法の場合、20〜30細胞にプロセスを繰り返します。統計上の目的のために、できれば別の日に、実験を3回以上繰り返す。均質な集団と正しい設定では、標準偏差は通常低い(<5%)。
FRAPやFLIPの両方に対して、漂白領域の大きさや形状は、回復のダイナミクスに影響を与え、実験内で一定でなければなりません。また、つのセルを比較する場合、同一のプロトコルを使用する必要がありますし、細胞をパワーレーザーと他の条件は変動することがありますし、実験の結果に影響を及ぼす可能性があるため、同じ日に連続して分析する必要があります。
4。 FRAPやFLIPデータ解析
- すべてのFRAPフレームが収集さで、ROI(投資収益率B =漂白エリア)、背景領域(ROI BG)で蛍光強度を測定し、前と漂白後の時間の関数としての無漂白の核領域(ROI NB)。漂白地域はごくわずかであるときに全体の核は、正規化の目的のために選択することができます。
- /(ROI NB - ROI BG) - (ROI BG ROI B)/(pbROI B - pbROI BG)/(pbROI NB - pbROI BG)、鉛は、事前に漂白意味:すべての時間ポイントの場合は、公式にしたがってデータを正規化する。事前に漂白剤の画像の場合は約1の値を取得する必要があります。漂白剤の後の最初のイメージは、ブリーチの深さが示されます。実際のブリーチ深さ値1から値を引く。すべてのセルと各実験から平均20から30のセルに対して繰り返します。
- 収集されたすべてのFLIPのフレームで、(ROI NB =無漂白面積、ROI BG =バックグラウンド)無漂白核領域での蛍光強度、および背景領域を測定する。 FLIPデータの計算は、FRAPの曲線に似てのみ分析したROI(投資収益率nbは )計算に使用されていない実際の漂白地域、異なっている必要があります:(ROI NB - ROI BG)/(pbROI NB - pbROI BG) 。それは正規化の目的のために隣接セル(ROI N =隣接セル)を使用することも可能です:(ROI NB - ROI BG)/(ROI N - ROI BG)/(pbROI NB - pbROI BG)/(pbROI N - pbROI BG )。
データの収集に続いて、それはコンピュータシミュレーションに実験データをフィットすることが可能です。これは良い近接、モバイル分数、不動小数と半値で、計算することができます。ここでは、FRAP解析の数学的および統計的側面 を議論し、他の優れた出版物4-9を参照されたいことはありません。漂白剤の深さが事前に漂白剤(100%)信号と漂白剤の後の最初のイメージとの間の距離(y軸上)を指し、モバイル割合は、漂白剤の深さとの間の距離(y軸上)を指し反応速度がプラトーに達する、と不動小数が復元された信号とプリ漂白剤(100%)の信号の間の距離(y軸上)(図1Bおよび2Bを参照)を参照している。復元された信号この分析を超えて、データに合わせて適切な数学モデルがあります。単一指数、方程式のための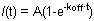
tは時間であるときは、1モバイル割合であり不動割合であり、kはオフ解離定数であり、データに合わせて使用することができ、結合のオフ率の直接推定値(K オフ )することができます得だけでなく、会合速度を計算するために使用できるパラメータ、のために。
5。代表的な結果:
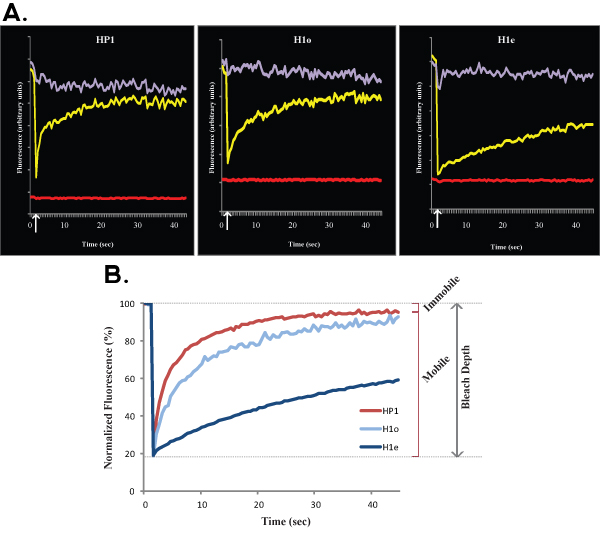
図1及びBは、R1 ES細胞では、HP1(左)の代表的なFRAP曲線を示すH1o(真ん中)とH1E(右)。簡単でわかりやすく図の場合は1Aは、いかなる正規化し、計算の前に単セルの生データを示しています。黄色の曲線は、漂白の領域に対応する、紫の曲線は、無漂白の核領域に相当する(漂白地域は核全体が正規化の目的のために選択することができる無視できる場合)、および赤の線は、バックグラウンドの蛍光に対応し、されているこの場合の最小限の。垂直方向の矢印は、漂白時間を表します。正規化と平均化されたデータは、図1Bに示されています。 HP1(赤)と比較してH1(青)の遅い回復に注意してください。また、H1Eバリアント(ダークブルー)はH1oバリアント(ライトブルー)よりも遅くなります。モバイルと不動の分画とブリーチの深さは、HP1のために示されている。
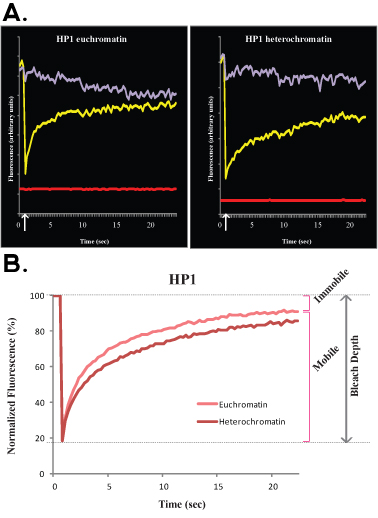
図2及びBは、ユークロマチンを比較する代表的なFRAP曲線を示す(R1 ES細胞におけるHP1のヘテロクロマチン(右)と)左。同様に図1を、図2Aは、単一セルの生データを示し、黄色の曲線は、漂白の領域に対応する、紫の曲線は、無漂白の核領域に相当し、赤の線は、バックグラウンドの蛍光に対応しています。垂直方向の矢印は、漂白時間を表します。正規化と平均化されたデータは、図2Bに示されています。ユークロマチン(明るい赤)と比較してヘテロクロマチン(暗赤色)の遅い回復に注意してください。モバイルと不動の分画とブリーチの深さは、ユークロマチンのために示されている。
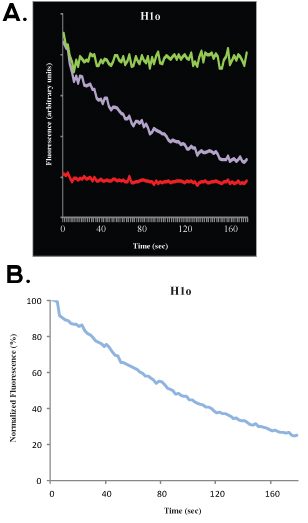
図3。。 H1o R1 ES細胞の典型的なFLIPの実験は、図3A(生、非正規化データ)とB(正規化と平均化されたデータ)に示されています。この実験では紫色の曲線は、無漂白の核領域に対応する、緑色の線は隣接する細胞核に相当し、赤線は、バックグラウンドの蛍光に対応しています。
ディスカッション
細胞集団または固定化細胞、FRAP実験から精製されたクロマチンを含むほとんどの利用できる技術とは異なり、生きた細胞のクロマチン蛋白質のダイナミクスの変化に従ってください。我々はクロマチンの蛋白質のダイナミクスは、クロマチン可塑性の良い指標であることが判明。しかし、それは融合のGFPと目的の遺伝子を必要とするため、蛍光タグを追加するには、タンパク質の機能を妨げ...
開示事項
謝辞
私たちは、批判的なコメントのためにと日常的にトラブルシューティング退色実験のため、特にMeshorer研究室のメンバーは、シャイMelcer、アディAlajem、Edupugantiラグーラム、バディスリランカSailaja、アンナMattoutとアルバBiranに感謝。 EMは、ジョセフH.とベルR.ブラウンライフサイエンスの上級講師であり、イスラエル科学財団(ISF 943/09)、保健イスラエル省(6007)欧州連合(IRG - 206872および238176)でサポートされていますイスラエル癌研究財団、ヘブライ大学、イスラエルの精神生物学研究所の内部実用的な医療補助。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | |
|---|---|---|---|
| DMEM | シグマ | D5671 | |
| ゼラチン | メルク | 1.04078 | |
| のOpti - MEM | ギブコ | 31985 | |
| トランジット- LT1 | Mirus | MIR2300 | |
| μ-スライド8ウェル | ibidi | 80826 |
参考文献
- Phair, R. D. Global nature of dynamic protein-chromatin interactions in vivo: three-dimensional genome scanning and dynamic interaction networks of chromatin proteins. Mol Cell Biol. 24, 6393-6402 (2004).
- Meshorer, E., Girard, L. . Imaging chromatin in embyonic stem cells in StemBook. , (2008).
- Meshorer, E. Hyperdynamic plasticity of chromatin proteins in pluripotent embryonic stem cells. Dev Cell. 10, 105-116 (2006).
- Bancaud, A., Huet, S., Rabut, G., Ellenberg, J. . Fluorescence perturbation techniques to study mobility and molecular dynamics of proteins in live cells: FRAP, photoactivation, photo conversion, and FLIP. , (2009).
- Dundr, M., Misteli, T. Measuring dynamics of nuclear proteins by photobleaching. Curr Protoc Cell Biol. Chapter 13, Unit 13-Unit 13 (2003).
- Ellenberg, J. Nuclear membrane dynamics and reassembly in living cells: targeting of an inner nuclear membrane protein in interphase and mitosis. J Cell Biol. 138, 1193-1206 (1997).
- Lenser, T., Weisshart, K., Ulbricht, T., Klement, K., Hemmerich, P. Fluorescence fluctuation microscopy to reveal 3D architecture and function in the cell nucleus. Methods Cell Biol. 98, 2-33 (2010).
- Mueller, F., Mazza, D., Stasevich, T. J., McNally, J. G. FRAP and kinetic modeling in the analysis of nuclear protein dynamics: what do we really know. Curr Opin Cell Biol. 22, 403-411 (2010).
- Phair, R. D., Misteli, T. Kinetic modelling approaches to in vivo imaging. Nat Rev Mol Cell Biol. 2, 898-907 (2001).
- Poser, I. BAC TransgeneOmics: a high-throughput method for exploration of protein function in mammals. Nat Methods. 5, 409-415 (2008).
- Sigal, A. Generation of a fluorescently labeled endogenous protein library in living human cells. Nat Protoc. 2, 1515-1527 (2007).
- Cohen, A. A. Dynamic proteomics of individual cancer cells in response to a drug. Science. 322, 1511-1516 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved