このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
リジン脱メチル化酵素活性の定量測定のためのMassSQUIRMのアプリケーション
要約
我々は、リジンメチル化の変化を定量化するためにMALDI質量分析法と還元的メチル化化学を使用するための手法を提案する。
要約
最近では、エピジェネティックな調節因子は1-3さまざまな疾患の主要なプレーヤーとして発見されました。その結果、これらの酵素は、小分子の研究や薬剤開発4の主要な標的である。多くのエピジェネティックなレギュレータは、ごく最近発見されていると分類されているプロセスにまだある。これらの酵素の中ではヒストンや他のタンパク質のリジンからメチル基を除去するリジン脱メチル化酵素である。酵素のこのクラスの新たな性質のために、いくつかのアッセイは、彼らの活動を研究するために開発されている。これは、分類とヒストン脱メチル化酵素のハイスループット研究の両方に道路ブロックされています。現在、非常に少数のデメチラーゼアッセイは存在しています。存在することをそれらは自然の中で定性的な傾向にあると同時に、異なるリジンメチル化の状態(非、モノ - 、ジ - およびトリ - )の間に識別することはできません。質量分析は、一般的に脱メチル化酵素活性を測定するために使用されるが、現在の質量分析アッセイはないということです差動メチル化ペプチドが異なってイオン化するかどうかに対処できません。メチル化ペプチドの差イオンは、メチル化状態は難しい(図1A)確かに定量的ではないと比較になります。したがって、利用可能なアッセイは、脱メチル化酵素活性の包括的な分析のために最適化されていません。
ここでは、このように本質的に同じ化学種、それらを作るため、イオン化し、すべてのリジンジ - メチル化を強制的に重水素化されたホルムアルデヒドとアミン基の還元的メチル化に基づいていますMassSQUIRMと呼ばれる方法(同位体の還元的メチル化を用いた質量分析の定量)を記述(図1B)と同じです。還元的メチル化後にのみ化学の違いは、MALDIイオン化効率に影響を与えません水素と重水素である。 MassSQUIRMアッセイは非、モノ - 又はジ - メチルリジンの脱メチル化反応生成物に対して特異的である。アッセイはまた、saを与えリジンメチルトランスフェラーゼにも適用可能である私の反応生成物。ここでは、還元的メチル化化学とLSD1の活性を測定するためのMALDI質量分析、合成ペプチドを基板5上に、ジ-およびモノメチル基を除去することができるリジン脱メチル化酵素の組み合わせを使用します。このアッセイは、ラボやプロテオミクス機能を通じてMALDI質量分析計へのアクセス権を持つすべてのラボにシンプルかつ簡単に従順です。アッセイは、〜8倍のダイナミックレンジを持ち、プレートフォーマット5に容易に拡張することができます。
プロトコル
このプロトコルは、ブレアらから変更されています。6。
1。 LSD1脱メチル化アッセイ
- 20μLの最終的なボリュームでは、脱メチル化酵素緩衝液中で0.25μgのジ-メチルヒストンH3ペプチド(ARTKme2QTARKSTGGKAPRKQLYK-ビオチン)と125 ngの組換えLSD1を組み合わせる(50mMのトリス-Cl pH8.5で、50mMのKCl、5mMのMgCl 2、5%グリセロール)。さらに、酵素なしでペプチド単独で制御を行う。コントロールは、セクション3のためであり、還元的メチル化を必要としません。
- 37℃で2時間インキュベート℃、
- ペプチドを収集するには、各サンプルに8μLPOROS R2の20ミクロンのビーズを追加し、室温で15分間振とうする。メタノールの1ボリュームとビーズの1ボリュームを停止させることによってPOROSビーズを準備し、次いで、5%ギ酸/ 0.2%TFAの10巻を追加します。
- 条件2の先端に0.1%TFAの20μLを吸引し、ピペットで戻ってそれを押すことにより、C 18 ZipTips。 Tは、0.1%で二回この操作を行う0.1%TFAでFA、再び70%アセトニトリル/ 0.1%TFAで4回、4回。
- ZipTipsでC 18樹脂の後ろにビーズスラリーをピペッティングし、ピペッターとを介してボリュームを押すことにより、エアコンC 18 ZipTips上に2つのサンプルを(制御および脱メチル化酵素反応)をロードします。
- 0.1%TFAの20μLで二回ZipTipsを洗ってください。
- 70%アセトニトリル/ 0.1%TFAの40μLで溶出します。
- SpeedVacコンセントレータと完全にそれぞれの溶離液を凍結乾燥。
2。還元的メチル化
プロトコルのこの部分は、ブレアらから変更されています。とレイメントら 6,7。
- 50mMリン酸緩衝液、pH 7.4の100μLの脱メチル化酵素反応を再懸濁する。
- デメチラーゼ反応に、15 mg / mLのジメチルアミンボランの8μLを追加します。ボランジメチルアミン15 mg / mLのストック溶液を与えるために、50mMリン酸緩衝液、pH7.4中に溶解されています。これは新鮮ななされるべきである。
- デメチラーゼ反応に、250mMの重水素化ホルムアルデヒドの16μLを追加します。 250 mMの重水素ホルムアルデヒドはH 2 0の株式を希釈することによって調製されています。これは新鮮ななされるべきである。
- 2時間4℃でインキュベートします。
- 手順2.2、2.3および2.4を繰り返します。
- 手順2.2を繰り返します。
- 4℃でインキュベートC〜15時間。
- デメチラーゼサンプルに、還元的メチル化反応をクエンチするために1Mトリス、pH 7.4の12.5μLを追加します。
3。 MALDIマススペクトロメトリー
- デメチラーゼ反応にPOROS R2 20ミクロンのビーズを追加し、ステップ1.3から1.6で説明されているようZipTipsに収集します。
注:50 mMリン酸緩衝液、pH 7.4の100μLにコントロールを再中断し、他のサンプルでは、ステップ3.1から始まるとして扱う。
- 33%飽和の2μLを用いてMALDIサンプルプレート上にZipTipsから制御および脱メチル化反応のサンプルを溶出させるD 2,5 - ジヒドロキシ安息香酸。 33パーセント2,5 - ジヒドロキシ安息香酸を調製するために、70%アセトニトリル/ 0.1%TFAで飽和33%に希釈し、70%アセトニトリル/ 0.1%TFAで2,5 - ジヒドロキシ安息香酸の飽和溶液を作成します。溶出したサンプルが乾燥し結晶化することができます。
- PerkinElmerSciex MALDI-prOTOF質量分析計または使用可能な高分解能MALDI質量分析計8,9,10を使用してマススペクトルを収集します。
- スペクトルを見ると質量分析計の製造元から提供されてPerkinElmerSciex TOFworksソフトウェアまたはソフトウェアを使用してピーク面積を抽出します。
- タンデム質量分析機能を提供可能な質量分析システムとMS 2で反応生成物を確認します。
4。データ解析
- 重いホルムアルデヒド(図1B)を使用して作成された同位体の重なりを補償するために、ピーク面積の13 C 2と13 C 4同位体relativの()比を決定還元的メチル化(図2A)を受けていないコントロールサンプルのためのモノアイソトピックピークに電子。
式1:13C2 / 12C = R 1
式2:13C4 / 12C = R 2
これはあなたの実験と質量分析計に特有のこれらの同位体の状態で存在するペプチドの比(R 1とR 2)を与えます。 - デメチラーゼ反応サンプルの各変更の状態(図2B)に存在するペプチドの相対量を定量化するには、次の式中のR 1とR 2の値を使用します。
式3:H3K4me2 = 1
式4:H3K4me = A 2 - R 1(A 1)
式5:H3K4 = A 3 - R 2(A 1) - [R 1(2 - R 1(A 1))]
5。代表的な結果
LSD1を用いて、定量化するためMassSQUIRMの有効性を示すリジン脱メチル化。などのプロトコルのテキストのセクションで説明したように、我々は、LSD1の125 ngおよび0.25μgのジ - メチルヒストンH3ペプチド(ARTKme2QTARKSTGGKAPRKQLYK-ビオチン)でデメチラーゼアッセイを行った。制御および脱メチル化酵素反応のサンプルは還元的メチル化とMALDI質量分析に供した。還元的メチル化を受けなかったコントロール反応は、このペプチドの典型的な同位体の包絡線を示し、式1および2(図2A)のピーク面積を提供しました。デメチラーゼ反応の質量スペクトルは、式3-5(図2B)のピーク面積を提供した。制御および脱メチル化酵素反応からのピーク面積を用いて、我々は、LSD1は、以下の反応生成物を与えるためにペプチドを脱メチル化することを決定:33.7%のジ - メチルLys4、42.3パーセントモノメチルLys4、24%非メチル化リジン4。 Blair らを参照してください。フルLSD1脱メチル化酵素活性の分析と同様に阻害剤の研究6に。 、酵素などの反応時間などの変数を変更することに注意してください濃度と基質濃度は、アクティビティの詳細な分析が可能になる。

図1。MassSQUIRMの概要。 (A)ヒストンH3のN末端ペプチドは、非(緑)、モノ(赤)、またはジ - (青)のリジン4でメチル化されているとして表示されます。各ペプチドの化学組成の変化が定量化複合体を作る差動電離につながります。 (B)還元メチル化は、すべてのペプチドは、同様にイオン化させ、ジ - メチル状態にすべてのリジン残基に変換します。還元メチル化反応の重ホルムアルデヒドの使用は、オリジナルのメチル化のアイデンティティの保持を可能にします。黒丸が重いメチル化を示しながら、白丸は光メチル化を示しています。Blairら2011年 6 から変更しました 。
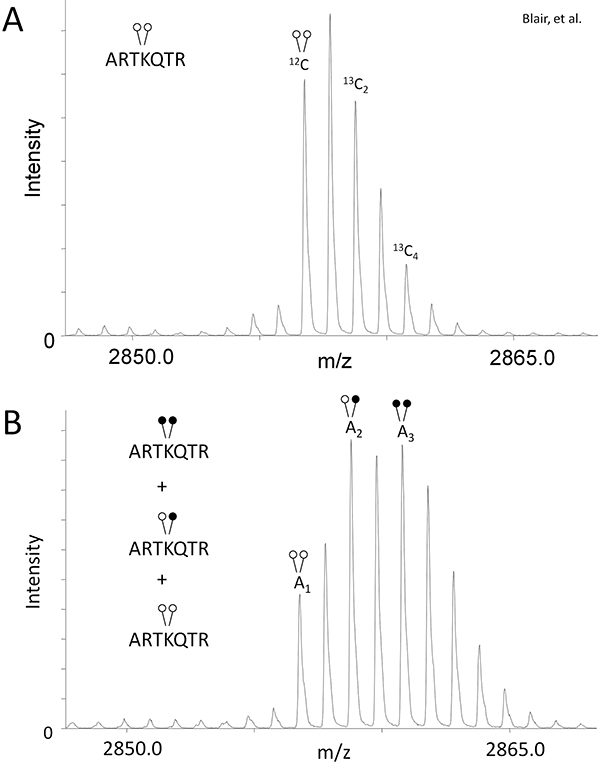
FigurE 2。MassSQUIRMは差動メチル化ペプチドを定量化するために使用することができます。 (A)ジ-メチル合成ヒストンH3ペプチド(ARTKme2QTARKSTGGKAPRKQLYK-ビオチン)は、モノアイソトピックピークの相対質量とのピーク比を用いて解析した式1および2で、R 1およびR 2として認められた。 (B)と同じ合成ペプチドは、37℃で2時間のためにデメチラーゼバッファ125 ngのLSD1とインキュベートした次いで、試料を分析しMassSQUIRMに供した。重複ピークの混合集団では、図1Bに見つの異なるメチル化状態を表しています。モノアイソトピックピーク下の面積は、1、2、3として認められた。ピーク面積は、式3-5に使用されました。黒丸が重いメチル化を示しながら、白丸は光メチル化を示しています。Blairらから変更しました。 2011 6。
ディスカッション
MassSQUIRMは、モノ - 及びジ - メチル化に関与するリジン脱メチル化酵素活性の包括的な分析のための安価で、定量的な方法です。 MassSQUIRMは、反応生成物のだけでなく、中間体だけでなく、定量化を提供しています。このアッセイは、LSD1や他のヒストン脱メチル化のメカニズムを研究する上で強力なツールとして使用することができます。それはまた、PHF8などの多くの新たに発見されたリジ?...
開示事項
我々は、開示することは何もありません。
謝辞
我々は質量分析をサポートするためUAMSプロテオミクス施設に感謝します。このプロジェクトの資金は、NIHの助成金P20RR015569、P20RR016460とR01DA025755によって提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | |
| LSD1 | BPSサイエンス | 50100 | |
| H3K4me2-ビオチンペプチド | 社内で準備 | なし | |
| POROS R2 20ミクロンのビーズ | アプライドバイオシステムズ | 1-1129-06 | |
| C 18 ZipTip | ミリポア | ZTC18M | |
| トリフルオロ酢酸(TFA) | サーモ | 28904 | |
| アセトニトリル | フィッシャー | A996 | |
| 2,5 - ジヒドロキシ安息香酸 | シグマ | 85707 | |
| ボランジメチルアミン | シグマ | 180238 | |
| 同位体重いD 2ホルムアルデヒド | ケンブリッジアイソトープラボラトリーズ | DLM-805から20 | |
| トリス | フィッシャー | BP154 | |
| 塩化カリウム | フィッシャー | BP366 | |
| MgCl 2を | フィッシャー | BP214 | |
| グリセロール | フィッシャー | G33 | |
| ギ酸 | フルカ | 06440 | |
| メタノール | フィッシャー | A452 | |
| リン酸ナトリウム | フィッシャー | BP329 | |
| SpeedVacコンセントレータ | 学者 | DNA110 | |
| MALDI-prOTOF質量分析計とTOFworksソフトウェア | PerkinElmerSciex | なし |
参考文献
- Goldberg, A. D., Allis, C. D., Bernstein, E. Epigenetics: a landscape takes shape. Cell. 128, 635-638 (2007).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128, 693-705 (2007).
- Blair, L. P., Cao, J., Zou, M. R., Sayegh, J., Yan, Q. Epigenetic Regulation by Lysine Demethylase 5 (KDM5) Enzymes in Cancer. Cancers (Basel). 3, 1383-1404 (2011).
- Cole, P. A. Chemical probes for histone-modifying enzymes. Nat. Chem. Biol. 4, 590-597 (2008).
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119, 941-953 (2004).
- Blair, L. P. MassSQUIRM: An assay for quantitative measurement of lysine demethylase activity. Epigenetics. 6, 490-499 (2011).
- Rayment, I. Three-dimensional structure of myosin subfragment-1: a molecular motor. Science. 261, 50-508 (1993).
- Collom, S. L., Jamakhandi, A. P., Tackett, A. J., Radominska-Pandya, A., Miller, G. P. CYP2E1 active site residues in substrate recognition sequence 5 identified by photoaffinity labeling and homology modeling. Arch. Biochem. Biophys. 459, 59-69 (2007).
- Gradolatto, A. Saccharomyces cerevisiae Yta7 Regulates Histone Gene Expression. Genetics. 179, 291-304 (2008).
- Taverna, S. D. Yng1 PHD finger binding to histone H3 trimethylated at lysine 4 promotes NuA3 HAT activity at Lysine 14 of H3 and transcripiton at a subset of targeted ORFs. Mol. Cell. 24, 785-796 (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved