Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Die Anwendung des MassSQUIRM für quantitative Messungen von Lysin-Demethylase
In diesem Artikel
Zusammenfassung
Wir stellen eine Methode für die Verwendung von MALDI-Massenspektrometrie und reduktive Methylierung Chemie auf Veränderungen in Lysinmethylierung quantifizieren.
Zusammenfassung
In jüngster Zeit haben epigenetische Regulatoren als wichtige Akteure in vielen verschiedenen Krankheiten 3.1 entdeckt worden. Als ein Ergebnis sind diese Enzyme Hauptziele für kleine Moleküle Studien und Entwicklung von Arzneimitteln 4. Viele epigenetische Regulatoren haben erst vor kurzem entdeckt und sind noch im Gange klassifiziert. Unter diesen Enzymen sind Lysin Demethylasen die Methylgruppen von Lysine an Histonen und andere Proteine zu entfernen. Durch die neuartige Charakter dieser Klasse von Enzymen, haben einige Tests entwickelt worden, um ihre Aktivität zu studieren. Dies war eine Straßensperre sowohl die Einstufung und hohem Durchsatz Studie Histondemethylasen. Derzeit existieren nur sehr wenige Demethylase Assays. Diejenigen, die es gibt, sind in der Regel in der Natur qualitativen und können nicht gleichzeitig zwischen den verschiedenen Lysin Methylierungszustände zu erkennen (un-, mono-, di und tri-). Die Massenspektrometrie wird häufig verwendet, um Demethylase-Aktivität zu bestimmen, aber die aktuellen massenspektrometrischen Assays tunnicht ansprechen, ob differentiell methylierte Peptide anders zu ionisieren. Differential-Ionisation von Peptiden methylierte macht den Vergleich Methylierung Staaten schwierig und schon gar nicht quantitativ (Abbildung 1A). So verfügbaren Assays sind nicht für die umfassende Analyse der Demethylase optimiert.
Hier beschreiben wir ein Verfahren genannt MassSQUIRM (massenspektrometrische Quantifizierung unter Verwendung Isotopen reduktive Methylierung), die auf der reduktiven Methylierung Amingruppen mit deuteriertem Formaldehyd zu erzwingen, Lysine sein di-methylierten basiert, sodass sie im wesentlichen die gleichen chemischen Spezies und daher ionisieren gleichen (Abbildung 1B). Die einzige chemische Unterschied nach der reduktiven Methylierung ist Wasserstoff und Deuterium, die keinen Einfluss auf MALDI-Ionisierung Wirkungsgrade. Die MassSQUIRM Nachweis ist spezifisch für Demethylase Reaktionsprodukte mit un-, mono-oder di-methylierte Lysine. Der Assay ist auch anwendbar auf Lysin-Methyltransferasen geben die saIch Reaktionsprodukte. Wir verwenden hier eine Kombination aus der reduktiven Methylierung Chemie und MALDI-Massenspektrometrie, um die Aktivität von LSD 1 zu messen, eine Lysin-Demethylase entfernen kann di-und mono-Methylgruppen, einem synthetischen Peptid Substrat 5. Dieser Assay ist einfach und leicht zugänglich einem der Labor mit Zugang zu einem MALDI-Massenspektrometer im Labor oder durch eine Proteomik-Anlage. Der Test hat ~ 8-fach dynamischen Bereich und ist leicht skalierbar, um Platten-Format 5.
Protokoll
Dieses Protokoll wird von Blair et al modifiziert. 6.
1. LSD1 Demethylierung Assay
- In einem Endvolumen von 20 mL, 125 ng rekombinantem kombiniert mit 0,25 ug LSD1 Di-Methyl-Histon H3-Peptid (ARTKme2QTARKSTGGKAPRKQLYK-Biotin) im Demethylase-Puffer (50 mM Tris-Cl pH 8,5, 50 mM KCl, 5 mM MgCl 2, 5% Glycerin). Darüber hinaus führen Sie eine Peptid allein Kontrolle ohne Enzym. Die Steuerung ist für Abschnitt 3 und braucht keine reduktive Methylierung.
- Inkubieren für 2 Stunden bei 37 ° C
- Um die Peptide zu sammeln, fügen 8 ul POROS R2 20-Mikron-Perlen zu jeder Probe und agitieren für 15 Minuten bei Raumtemperatur. Bereiten POROS Perlen durch die Aussetzung ein Volumen von Perlen mit 1 Volumen Methanol, und fügen Sie dann 10 Volumen 5% Ameisensäure / 0,2% TFA.
- Zustand zwei C 18 ZipTips durch Absaugen 20 uL 0,1% TFA in die Spitze und drücken Sie es wieder aus mit einer Pipette. Tun Sie dies zweimal mit 0,1% TFA, viermal mit 70% Acetonitril / 0,1% TFA, und viermal erneut mit 0,1% TFA.
- Legen Sie die zwei Proben (Kontrolle und die Reaktion Demethylase) auf die konditionierte C 18 ZipTips durch Pipettieren die Kügelchenaufschlämmung hinter dem C 18-Harz in den ZipTips und schieben Sie die Lautstärke durch eine Pipette.
- Waschen Sie ZipTips zweimal mit 20 ul 0,1% TFA.
- Man eluiert in 40 ul 70% Acetonitril / 0,1% TFA.
- Lyophilisieren jedes Elutionsmittel vollständig mit einer SpeedVac Konzentrator.
2. Reduktive Methylierung
Dieser Teil des Protokolls wird von Blair et al modifiziert. Und Rayment et al. 6,7.
- Erneut zu suspendieren die Demethylase Reaktion in 100 ul 50 mM Phosphatpuffer, pH 7,4.
- Um die Reaktion Demethylase, fügen 8 &mgr; l 15 mg / ml Borandimethylamin. Boran-Dimethylamin in 50 mM Phosphatpuffer, pH 7,4 gelöst, die 15 mg / ml Stammlösung.Dies sollte frisch zubereitet werden.
- Um die Reaktion Demethylase, fügen Sie 16 ul 250 mM deuteriertem Formaldehyd. Der 250 mm deuteriertem Formaldehyd wird durch Verdünnen der Lager in H 2 0 vorbereitet. Dies sollte frisch zubereitet werden.
- Inkubieren bei 4 ° C für 2 Stunden.
- Wiederholen Sie die Schritte 2.2, 2.3 & 2.4.
- Wiederholen Sie den Schritt 2.2.
- Inkubieren bei 4 ° C für ca. 15 Stunden.
- Zum Demethylase Probe wird mit 12,5 ul 1 M Tris, pH 7,4, um die reduktive Methylierung zu quenchen.
3. MALDI-Massenspektrometrie
- Fügen Sie POROS R2 20-Mikron-Perlen zum Demethylase Reaktion, und sammeln Sie auf ZipTips wie in den Schritten von 1,3 bis 1,6 beschrieben.
Hinweis: Die Re-suspendieren die Kontrolle in 100 ul 50 mM Phosphatpuffer, pH 7,4 und zu behandeln, als die andere Probe ab Schritt 3.1.
- Eluieren die Kontrolle und Demethylase Reaktion Proben aus den ZipTips auf eine MALDI-Platte mit 2 ul von 33% zu sättigend 2,5-Dihydroxybenzoesäure. Zur Herstellung der 33% 2,5-Dihydroxybenzoesäure, machen Sie eine gesättigte Lösung von 2,5-Dihydroxybenzoesäure in 70% Acetonitril / 0,1% TFA verdünnt und dann auf 33% in 70% Acetonitril / 0,1% TFA gesättigt. Lassen Sie die eluierten Proben zu trocknen und kristallisieren.
- Sammle Massenspektren mit einem PerkinElmerSciex MALDI-Massenspektrometer oder prOTOF verfügbar hochauflösende MALDI-Massenspektrometer 8,9,10.
- Alle Spektren extrahieren und Peakflächen mit PerkinElmerSciex TOFworks Software oder Software, die vom Massenspektrometer Herstellers.
- Stellen Sie sicher, Reaktionsprodukte von MS 2 mit einem verfügbaren massenspektrometrischen System, das Tandem-Massenspektrometrie-Fähigkeiten.
4. Datenanalyse
- Um den Isotopen Überlappung mit schweren Formaldehyd (1B) verursacht werden können, bestimmen die Peakfläche (A)-Verhältnis von 13 C 2 und 13 C 4-Isotopen relativee an den monoisotopischen Peak für die Kontrollprobe, die nicht reduktive Methylierung (2A) unterzogen wurde.
Gl 1: Ein 13C2 / A 12C = r 1
Gl 2: Ein 13C4 / A 12C = r 2
Dadurch erhalten Sie das Verhältnis von Peptid bestehenden Zustände in diesen Isotopen (r 1 & r 2) spezifisch für Ihr Experiment und Massenspektrometer. - Mit dem R 1 & R 2-Werte in den folgenden Formeln zur Quantifizierung der relativen Menge an Peptid in den einzelnen Änderungen in der staatlichen Reaktion Demethylase Probe (Abb. 2B):
Gl 3: H3K4me2 = A 1
Gleichung 4: H3K4me = A 2 - r 1 (A 1)
Gleichung 5: H3K4 = A 3 - r 2 (A 1) - [r 1 (A 2 - r 1 (A 1))]
5. Repräsentative Ergebnisse
Mit LSD1, zeigen wir die Wirksamkeit der MassSQUIRM zur QuantifizierungLysin-Demethylierung. Wie in dem Protokoll Text beschrieben, haben wir eine Demethylase-Assay mit 125 ng von LSD 1 und 0,25 ug Di-Methyl-Histon H3-Peptid (ARTKme2QTARKSTGGKAPRKQLYK-Biotin). Die Steuerung und Demethylase Reaktion Proben wurden reduktive Methylierung und MALDI Massenspektrometrie unterzogen. Die Kontroll-Reaktion, die nicht unterzogen wurden reduktive Methylierung zeigten die typische Isotopenverhältnis Umschlag für dieses Peptid und zur Verfügung gestellt Peakflächen für Gleichungen 1 und 2 (Abbildung 2A). Das Massenspektrum für die Reaktion zur Verfügung gestellt Demethylase Peakflächen für Gleichungen 3-5 (Abbildung 2B). Unter Verwendung der Peakflächen von der Steuerung und Demethylase Reaktion haben wir festgestellt, dass das Peptid LSD1 demethyliert, um die folgenden Reaktionsprodukte geben: 33,7% Di-Methyl Lys4, 42,3% mono-Methyl Lys4, und 24% un-methylierter Lys 4. Siehe Blair et al. Für die vollständige Analyse der LSD1 Demethylase sowie Inhibitorstudien 6. Beachten Sie, dass sich ändernden Variablen wie Reaktionszeit, EnzymKonzentrations-und Substrat-Konzentration wird für eine eingehende Analyse der Tätigkeit zu ermöglichen.

Abbildung 1. MassSQUIRM Überblick. (A) ein N-terminales Peptid von Histon H3 wird dargestellt als un-(grün), Mono-(rot), oder Di-(blau) an Lysin 4 methyliert. Die Variation in der chemischen Zusammensetzung von jedem Peptid führt zu-Differential-Ionisation machen Quantifizierung komplex. (B) reduktive Methylierung konvertiert alle Lysinreste an den di-methyl Staat, der alle Peptide zu ionisieren, ähnlich verursacht. Die Verwendung von schweren Formaldehyd in der reduktiven Methylierung ermöglicht die Beibehaltung der Identität des ursprünglichen Methylierung. Offene Kreise zeigen Licht-Methylierung während geschlossene Kreise schweren Methylierung zeigen. Geändert von Blair et al. 2011 6.
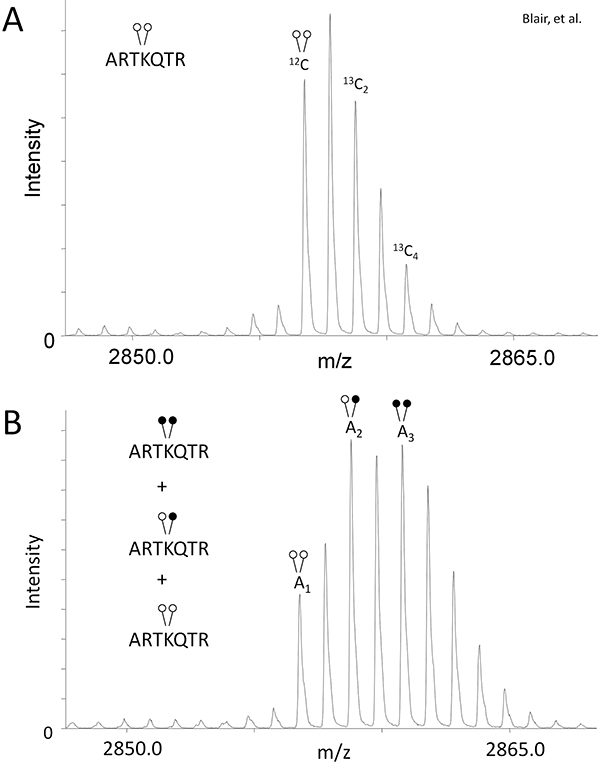
Figure 2. MassSQUIRM kann verwendet werden, um differentiell methylierte Peptide zu quantifizieren. (A)-A-di-methylierten synthetischen Histon H3-Peptid (ARTKme2QTARKSTGGKAPRKQLYK-Biotin) wurde unter Verwendung der Massenspektrometrie und Peak-Verhältnisse relativ zu dem monoisotopischen Peaks wurden als r 1 und r 2 in den Gleichungen 1 und 2 angegeben. (B) Das gleiche synthetische Peptid wurde mit 125 ng LSD1 in Demethylase Puffer für zwei Stunden bei 37 ° C inkubiert Die Proben wurden dann einer Analyse MassSQUIRM. Eine gemischte Bevölkerung von überlappenden Peaks stellt drei unterschiedliche Methylierung Zustände wie in 1B gesehen. Bereiche, die unter monoisotopischen Peaks wurden als A 1, A 2 und A 3 notiert haben. Die Peakflächen wurden in den Gleichungen 3-5 hatten. Offene Kreise zeigen Licht-Methylierung während geschlossene Kreise schweren Methylierung zeigen. Geändert von Blair et al. 2011 6.
Diskussion
MassSQUIRM ist eine preisgünstige und quantitative Methode zur umfassenden Analyse der Aktivität von Lysin Demethylasen in Mono-und Di-Methylierung beteiligt. MassSQUIRM bietet nicht nur die Quantifizierung des Produkts der Reaktion, sondern auch für die Zwischenprodukte. Dieser Test kann als ein mächtiges Werkzeug bei der Untersuchung des Mechanismus von LSD 1 und andere Histondemethylasen verwendet werden. Es sind auch nützlich für die Klassifizierung wie die neu entdeckten Lysin Demethylase Enzyme wie PHF8 und ...
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Wir danken der UAMS Proteomics-Fazilität für die massenspektrometrische Unterstützung. Finanzierung für dieses Projekt wurde durch die NIH Zuschüsse P20RR015569, P20RR016460 und R01DA025755 zur Verfügung gestellt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
| LSD1 | BPS Biosciences | 50100 | |
| H3K4me2-Biotin-Peptid | hergestellt im Haus | keiner | |
| POROS R2 20-Mikron-Perlen | Applied Biosystems | 1-1129-06 | |
| C 18 ZipTip | Millipore | ZTC18M | |
| Trifluoressigsäure (TFA) | Thermo | 28904 | |
| Acetonitril | Fischer | A996 | |
| 2,5-Dihydroxybenzoesäure | Sigma | 85707 | |
| Borandimethylamin | Sigma | 180238 | |
| isotopisch schwere d 2-Formaldehyd- | Cambridge Isotope Laboratories | DLM-805-20 | |
| Tris | Fischer | BP154 | |
| KCl | Fischer | BP366 | |
| MgCl 2 | Fischer | BP214 | |
| Glycerin | Fischer | G33 | |
| Ameisensäure | Fluka | 06440 | |
| Methanol | Fischer | A452 | |
| Na-Phosphat | Fischer | BP329 | |
| SpeedVac Concentrator | Gelehrter | DNA110 | |
| MALDI-Massenspektrometer und prOTOF TOFworks Software | PerkinElmerSciex | keiner |
Referenzen
- Goldberg, A. D., Allis, C. D., Bernstein, E. Epigenetics: a landscape takes shape. Cell. 128, 635-638 (2007).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128, 693-705 (2007).
- Blair, L. P., Cao, J., Zou, M. R., Sayegh, J., Yan, Q. Epigenetic Regulation by Lysine Demethylase 5 (KDM5) Enzymes in Cancer. Cancers (Basel). 3, 1383-1404 (2011).
- Cole, P. A. Chemical probes for histone-modifying enzymes. Nat. Chem. Biol. 4, 590-597 (2008).
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119, 941-953 (2004).
- Blair, L. P. MassSQUIRM: An assay for quantitative measurement of lysine demethylase activity. Epigenetics. 6, 490-499 (2011).
- Rayment, I. Three-dimensional structure of myosin subfragment-1: a molecular motor. Science. 261, 50-508 (1993).
- Collom, S. L., Jamakhandi, A. P., Tackett, A. J., Radominska-Pandya, A., Miller, G. P. CYP2E1 active site residues in substrate recognition sequence 5 identified by photoaffinity labeling and homology modeling. Arch. Biochem. Biophys. 459, 59-69 (2007).
- Gradolatto, A. Saccharomyces cerevisiae Yta7 Regulates Histone Gene Expression. Genetics. 179, 291-304 (2008).
- Taverna, S. D. Yng1 PHD finger binding to histone H3 trimethylated at lysine 4 promotes NuA3 HAT activity at Lysine 14 of H3 and transcripiton at a subset of targeted ORFs. Mol. Cell. 24, 785-796 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten