É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Aplicação de Medidas para MassSQUIRM quantitativa da actividade demetilase Lisina
Neste Artigo
Resumo
Apresentamos um método para o uso de espectrometria de massa MALDI e química metilação redutiva para quantificar as mudanças na metilação da lisina.
Resumo
Recentemente, os reguladores epigenéticos têm sido descobertos como atores-chave em muitos diferentes doenças 1-3. Como resultado, estas enzimas são alvos principais para os estudos de moléculas pequenas e 4 desenvolvimento de drogas. Muitos reguladores epigenéticos têm apenas recentemente descobertos e ainda estão em vias de ser classificado. Entre estas enzimas são demethylases lisina que removem grupos metilo a partir de lisinas na histonas e outras proteínas. Devido à natureza novo desta classe de enzimas, ensaios de poucos têm sido desenvolvidos para estudar a sua actividade. Este tem sido um bloqueio na estrada para tanto a classificação e estudo de alto rendimento de demethylases histonas. Atualmente, ensaios demetilase existem poucos. As que existem tendem a ser de natureza qualitativa e não pode discernir entre simultaneamente os diferentes estados de metilação de lisina (un-, mono-, di-e tri-). Espectrometria de massa é comumente usado para determinar a atividade demetilase mas atuais ensaios de espectrometria de massa donão dirigir se peptídeos diferencialmente metilados ionizar de forma diferente. Ionização diferencial de péptidos metilados faz comparando estados de metilação difícil e certamente não quantitativa (Figura 1A). Assim, os ensaios disponíveis não são otimizados para a análise global da atividade demetilase.
Descrevemos aqui um método chamado MassSQUIRM (quantificação por espectrometria de massa utilizando metilação redutiva isotópica) que é baseado em metilação redutiva de grupos amina com formaldeído deuterado para forçar todos os lisinas ser di-metilado, tornando-as assim essencialmente os mesmos espécies químicas e, portanto, ionizar o mesmo (Figura 1B). A diferença química apenas após a metilação redutiva é hidrogénio e deutério, que não afecta a eficiência de ionização MALDI. O ensaio MassSQUIRM é específico para produtos de reacção desmetilase com lisinas un-, mono-ou di-metilados. O ensaio é também aplicável a metiltransferases lisina dando o same produtos de reacção. Aqui, nós usar uma combinação de química metilação redutiva e espectrometria de massa MALDI para medir a actividade de LSD1, um desmetilase lisina capaz de remover di-e mono-metilo grupos, sobre um substrato de péptido sintético 5. Este ensaio é simples e facilmente acessíveis a qualquer laboratório com acesso a um espectrómetro de massa MALDI em laboratório ou através de um mecanismo proteômica. O ensaio tem ~ gama de 8 vezes dinâmico e é prontamente adaptar ao formato de placa 5.
Protocolo
Este protocolo é modificado a partir de Blair et al. 6.
1. LSD1 Ensaio desmetilação
- Em um volume final de 20 uL, combinar 125 ng LSD1 recombinante com 0,25 ug de di-metil histona H3 péptido (ARTKme2QTARKSTGGKAPRKQLYK-biotina) em tampão de desmetilase (50 mM Tris-Cl pH 8,5, 50 mM de KCl, 5 mM MgCl2, 5% glicerol). Além disso, realizar um péptido de controlo sozinha, sem enzima. O controle é para a secção 3 e não precisa de metilação redutiva.
- Incubar durante 2 horas a 37 ° C.
- Para recolher os péptidos, adicionar 8 uL Poros R2 20 micron grânulos para cada amostra e agitar durante 15 minutos à temperatura ambiente. Preparar grânulos Poros suspendendo 1 volume de esferas com um volume de metanol, e depois adicionar 10 volumes de 5% de ácido fórmico / TFA a 0,2%.
- Condição dois C 18 ZipTips por aspiração de 20 uL de TFA 0,1% na ponta e empurrando-o de volta com uma pipeta. Faça isso duas vezes com T 0,1%FA, quatro vezes com acetonitrilo 70% / TFA a 0,1%, e quatro vezes novamente com TFA a 0,1%.
- Carregar as duas amostras (controle ea reacção desmetilase) sobre os condicionado C 18 ZipTips pipetando a lama grânulo por trás da resina C 18 nas ZipTips e empurrando o volume através com um pipetador.
- Lavar duas vezes com ZipTips 20 uL de TFA a 0,1%.
- Eluir em 40 uL de acetonitrilo 70% / TFA a 0,1%.
- Liofilizar cada eluente completamente com um concentrador Speedvac.
2. Metilação redutiva
Esta porção do protocolo é modificado a partir de Blair et al. E Rayment et al. 6,7.
- Re-suspender a reacção desmetilase em 100 uL de tampão fosfato 50 mM, pH 7,4.
- Para a reacção de desmetilase, adicionar 8 uL de 15 mg / mL de borano dimetilamina. Borano dimetilamina é dissolvido em tampão fosfato 50 mM, pH 7,4, para dar o 15 mg / ml de solução concentrada.Isto deve ser feito fresco.
- Para a reacção de desmetilase, adicionar 16 uL de 250 mM de formaldeído deuterado. O formaldeído 250 mM deuterado é preparada diluindo o estoque em H 2 0. Isto deve ser feito fresco.
- Incubar a 4 ° C durante 2 horas.
- Repita os passos 2.2, 2.3 e 2.4.
- Repita o passo 2.2.
- Incubar a 4 ° C durante ~ 15 horas.
- Para a amostra desmetilase, adicionar 12,5 ul de 1M Tris, pH 7,4 para extinguir a reacção de metilação redutiva.
3. Espectrometria de Massa MALDI
- Adicionar Poros R2 20 micron grânulos para a reacção desmetilase, e recolher em ZipTips como descrito nos passos 1,3-1,6.
Nota: Re-suspende o controle em 100 uL de tampão fosfato 50 mM, pH 7,4 e tratar como a outra amostra de partida no passo 3.1.
- Eluir o controlo e as amostras reaccionais desmetilase a partir dos ZipTips sobre uma placa de amostra MALDI usando 2 uL de 33% saturard ácido 2,5-dihidroxibenzóico. Para preparar a 33% de ácido 2,5-dihidroxibenzoico, fazer uma solução saturada de 2,5-di-hidroxibenzóico em acetonitrilo 70% / TFA a 0,1% e, em seguida, diluir a 33% saturado em acetonitrilo 70% / TFA a 0,1%. Permitir que as amostras eluidas para secar e cristalizar.
- Coletar espectros de massa usando um espectrômetro de massa MALDI PerkinElmerSciex-prOTOF ou disponível de alta resolução espectrômetro de massa MALDI 8,9,10.
- Ver espectros e extrair áreas dos picos usando PerkinElmerSciex software TOFworks ou software fornecido pelo fabricante do espectrômetro de massa.
- Verificar produtos de reacção por MS 2, com um sistema de massa disponível por espectrometria de proporcionar capacidades de espectrometria de massa em tandem.
4. Análise de Dados
- A fim de compensar a sobreposição isotópicos criadas usando formaldeído pesada (Figura 1B), determinar a área do pico (A) proporção de 13 C e 13 C 2 4 isótopos relative para o pico monoisotópico para a amostra de controle, que não foi objecto de metilação redutiva (Fig. 2A).
Eq. 1: Um 13C2 / A 12C = r 1
Eq. 2: Uma 13C4 / A 12C = r 2
Isto lhe dará a relação do peptídeo existentes nesses estados isotópica (r 1 e r 2) específico para o seu experimento e espectrômetro de massa. - Utilizar a r 1 e valores de r 2 nas fórmulas seguintes para quantificar a quantidade relativa de péptido existentes em cada estado modificação na amostra reacção desmetilase (Fig. 2B):
Eq. 3: H3K4me2 = A 1
Eq. 4: H3K4me = A 2 - r 1 (A 1)
Eq. 5: H3K4 = A 3 - r 2 (A 1) - [r 1 (A 2 - r 1 (A 1))]
5. Os resultados representativos
Usando LSD1, mostramos a eficácia do MassSQUIRM para quantificardesmetilação lisina. Como descrito na secção de Texto protocolo, realizou-se um ensaio de desmetilase com 125 ng de LSD1 e 0,25 ug de di-metil histona H3 péptido (ARTKme2QTARKSTGGKAPRKQLYK-biotina). O controlo e as amostras reaccionais desmetilase foram submetidos a metilação redutiva e espectrometria de massa MALDI. A reacção de controlo que não foi submetido a metilação redutiva mostrou o envelope típico isotópica para este péptido, e desde as áreas dos picos para as equações 1 e 2 (Figura 2A). O espectro de massa para a reacção de desmetilase desde áreas de pico para equações 3-5 (Figura 2B). Usando as áreas dos picos do controlo e reacção desmetilase, determinou-se que o péptido LSD1 desmetilado para dar os produtos de reacção seguintes: 33,7% de di-metil Lys4, 42,3% de mono-metil Lys4, e 24% un-metilado Lys 4. Consulte a Blair et al. Para a análise completa de LSD1 atividade demetilase bem como estudos de inibidores 6. Note-se que as variáveis mutação tais como o tempo de reacção da enzima,concentração de concentração e substrato permitirá uma análise aprofundada da atividade.

Figura 1. Resumo MassSQUIRM. (A) um péptido N-terminal a partir de histona H3 é mostrado como sendo un-(verde), mono-(vermelho), ou di-(azul) metilado na lisina 4. A variação na composição química de cada péptido leva a ionização diferencial tornando complexo quantificação. (B) metilação redutiva converte todos os resíduos de lisina para o estado de di-metil que faz com que todos os péptidos para ionizar de forma semelhante. A utilização de formaldeído pesado na reacção de metilação redutiva permite a retenção da identidade do metilação original. Círculos abertos indicam metilação luz, enquanto círculos fechados indicam metilação pesado. Modificado de Blair et al. 2011 6.
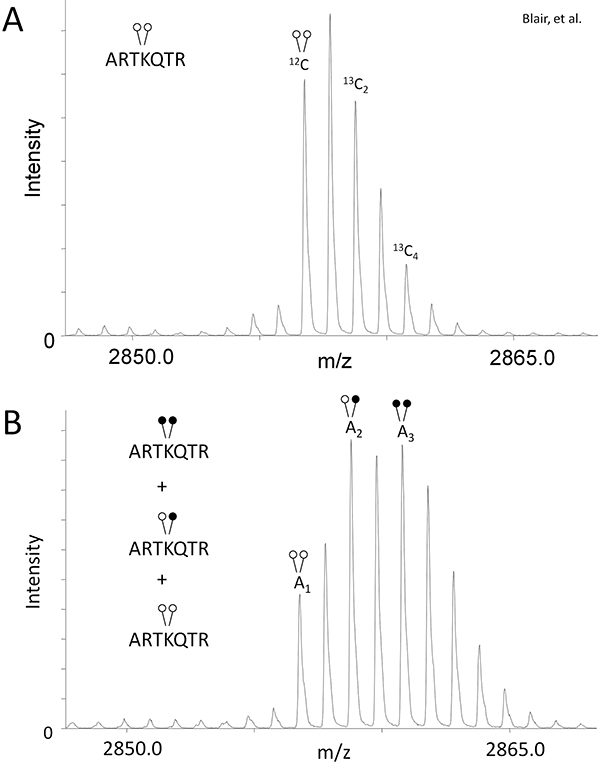
Figur2 e. MassSQUIRM pode ser usado para quantificar os péptidos diferencialmente metilados. (A) Um di-metilado sintético histona H3 péptido (ARTKme2QTARKSTGGKAPRKQLYK-biotina) foi analisado por espectrometria de massa e os rácios dos picos relativos ao pico monoisotópico foram anotados como R1 e R 2 nas equações 1 e 2. (B) O péptido sintético mesmo foi incubado com 125 ng LSD1, em tampão de desmetilase durante duas horas a 37 ° C. As amostras foram então submetidas a análise MassSQUIRM. Uma população mista de sobreposição picos representa três estados diferentes de metilação tal como são vistos na Figura 1B. As áreas sob os picos monoisotópico foram notadas como um 1, A 2, e A 3. Áreas de pico foram utilizados nas equações 3-5. Círculos abertos indicam metilação luz, enquanto círculos fechados indicam metilação pesado. Modificado de Blair et al. 2011 6.
Discussão
MassSQUIRM é um método barato e quantitativa para análise abrangente da atividade de demethylases lisina envolvidas na mono-e di-metilação. MassSQUIRM oferece não só a quantificação do produto da reacção, mas também para os intermediários. Este ensaio pode ser utilizado como uma ferramenta poderosa para estudar o mecanismo de LSD1 e outros demethylases histona. Também irá ser útil para a classificação de muitas enzimas recém-descobertas desmetilase de lisina, tais como PHF8 e poderiam ser usados ?...
Divulgações
Não temos nada a divulgar.
Agradecimentos
Agradecemos ao UAMS Facilidade Proteomics de apoio por espectrometria de massa. O financiamento para este projeto foi fornecido pelo NIH concede P20RR015569, P20RR016460 e R01DA025755.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
| LSD1 | BPS Biosciences | 50100 | |
| H3K4me2-biotina péptido | preparada em casa | nenhum | |
| POROS R2 20 esferas de micro | Applied Biosystems | 1-1129-06 | |
| C 18 ZipTip | Millipore | ZTC18M | |
| Ácido trifluoroacético (TFA) | Thermo | 28904 | |
| acetonitrila | Pescador | A996 | |
| 2,5-di-hidroxibenzóico | Sigma | 85707 | |
| Borano dimetilamina | Sigma | 180238 | |
| isotopicamente pesado d 2-formaldeído | Cambridge Isotope Laboratories | DLM-805-20 | |
| Tris | Pescador | BP154 | |
| KCl | Pescador | BP366 | |
| MgCl 2 | Pescador | BP214 | |
| Glicerina | Pescador | G33 | |
| O ácido fórmico | Fluka | 06440 | |
| Metanol | Pescador | A452 | |
| Na-fosfato | Pescador | BP329 | |
| Concentrador Speedvac | Sábio | DNA110 | |
| MALDI-prOTOF espectrômetro de massa e TOFworks software | PerkinElmerSciex | nenhum |
Referências
- Goldberg, A. D., Allis, C. D., Bernstein, E. Epigenetics: a landscape takes shape. Cell. 128, 635-638 (2007).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128, 693-705 (2007).
- Blair, L. P., Cao, J., Zou, M. R., Sayegh, J., Yan, Q. Epigenetic Regulation by Lysine Demethylase 5 (KDM5) Enzymes in Cancer. Cancers (Basel). 3, 1383-1404 (2011).
- Cole, P. A. Chemical probes for histone-modifying enzymes. Nat. Chem. Biol. 4, 590-597 (2008).
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119, 941-953 (2004).
- Blair, L. P. MassSQUIRM: An assay for quantitative measurement of lysine demethylase activity. Epigenetics. 6, 490-499 (2011).
- Rayment, I. Three-dimensional structure of myosin subfragment-1: a molecular motor. Science. 261, 50-508 (1993).
- Collom, S. L., Jamakhandi, A. P., Tackett, A. J., Radominska-Pandya, A., Miller, G. P. CYP2E1 active site residues in substrate recognition sequence 5 identified by photoaffinity labeling and homology modeling. Arch. Biochem. Biophys. 459, 59-69 (2007).
- Gradolatto, A. Saccharomyces cerevisiae Yta7 Regulates Histone Gene Expression. Genetics. 179, 291-304 (2008).
- Taverna, S. D. Yng1 PHD finger binding to histone H3 trimethylated at lysine 4 promotes NuA3 HAT activity at Lysine 14 of H3 and transcripiton at a subset of targeted ORFs. Mol. Cell. 24, 785-796 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados