このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
単一の卵重亜硫酸塩の突然変異誘発
要約
亜硫酸塩の突然変異誘発は、DNAメチル化を解析するためのゴールドスタンダードです。私たちの変更されたプロトコルは、単一細胞レベルでのDNAメチル化解析を可能にし、特に個々の卵のために設計されました。また、開裂段階の胚に使用することができます。
要約
エピジェネティクスは、すべてがこのように遺伝子のアクセシビリティを変更し、遺伝子の転写1を調整するための主要なメカニズムであることをクロマチンに遺伝性で可逆的改変を包含する。 DNAメチル化は、抑圧的なマークとして主に作用するエピジェネティックな修飾である。 CpGジヌクレオチドのシトシン上のメチル基の共有結合的付加により、それは、クロマチンを凝縮と遺伝子2をサイレンシングに関与するプロセスを開始するために追加の抑圧的なタンパク質やヒストン修飾を募集することができます。それは発達のプログラミング、細胞分化、レトロエレメントの弾圧、X染色体不活性化とゲノムインプリンティングに重要な役割を果たしているDNAのメチル化は正常な発達に不可欠である。
DNAメチル化解析のための最も強力な方法の一つは、亜硫酸水素塩の突然変異誘発である。亜硫酸水素ナトリウムは、ウラシルにシトシンをdeaminates DNAの変異原である。 PCR増幅とseque以下 ncingは、これらの変換イベントはチミンとして検出されます。メチル化シトシンは脱アミノ化から保護するため、個々のヌクレオチドレベル3でDNAメチル化の同定を可能にする、シトシンのままにされています。亜硫酸塩の突然変異誘発アッセイの開発は、当初より高感度と7再現性のあるものに向かって4-6を報告されたものから発展してきた。一つの重要な進歩は、それによって過酷な重亜硫酸塩処理8からDNAを保護し、アガロースビーズにDNAの少量を埋め込 むされました。卵母細胞および胚盤胞段階の胚9のプール上で実行するために、この対応のメチル化解析。これまでで最も洗練された亜硫酸塩の突然変異誘発のプロトコルは、個々の胚盤胞段階の胚は10です。胚盤胞は、平均64個のセル(ゲノムDNAの120から720 pgを含む)を持っているのでしかし、この方法は、個々の卵母細胞または切断ステージ胚におけるメチル化研究のための効果的ではありません。 テント">卵母細胞11を含む分DNA量の埋め込 みをアガロースから手がかりをとると、ここでは卵母細胞を直接検索し、卵母細胞から透明帯を除去した後、直ちにアガロース溶解ソリューションビーズに埋め込 まれているさせる方法を提示します。これは私たちに可能単一の卵重亜硫酸突然変異誘発の2つの主要な課題を回避する:多数のプロトコルステップの間に低下し、その後の損失からのDNAの微量を保護する重要なことは、データが単一の卵母細胞から得られたとして、プール内のPCRバイアスの問題が排除されるさらに。複数のメチル化パターンを持つ任意のサンプルはこのプロトコルは、単一細胞レベルでのDNAメチル化の成功と再現性の解析のための改良方法を提供する。分析12から除外されることがあり、理想的に適していますので、不注意な卵丘細胞の混入は、この方法で検出可能である個々の卵母細胞と同様に開裂段階の胚のために。
プロトコル
1日目
そのようなGIBCO水などの滅菌蒸留水で卵母細胞の収集日に新鮮な以下のソリューションを準備します。 DNAの混入の可能性を減らすために、頻繁に手袋を変更し、フィルターチップを使用しています。オープン離れて角度チューブを保持し、使用しないときは、すべてのチューブをおさらい。我々は解決策は、n +1として行われることをお勧めします。
3パーセントLMPアガロース
30 mgの低融点(LMP)アガロース
まで1ミリリットルGIBCO H 2 Oに
@ 70°Cを溶解
溶菌液
8μlの溶解バッファー
1μlのプロテイナーゼK
1μlの10%IGEPAL
使用直前まで氷上に静置
2時01分アガロース:Lysis Solutionを (個々の卵母細胞あたり10μlの量が3卵母細胞のためのものです)
20μlの3パーセントLMPアガロース
10μlのLysis Solutionを
ミックス@ 70°C
SDSのLyのSISバッファ(個々の卵母細胞あたり501μl)を
| 1X TE pH7.5の | 450μlの |
| 10%SDS | 50μlの |
| プロテイナーゼK | 1μlの |
| 501μlの |
1。卵母細胞コレクション
- M2培地中で解剖し、マウスの卵管を置き、卵丘細胞複合体を抽出するために膨大部を引き裂く。
- M2メディア30μlのドロップに0.3 mg / mlのヒアルロニダーゼ溶液を用いて卵丘細胞複合体から卵母細胞を分離します。だけ長い長い露出がそれらを傷める可能性がありますので、それは卵丘細胞を除去するのにかかるように溶液中で卵母細胞を保持します。定期的に卵丘細胞を除去しM2メディア30μlのドロップ3倍の卵を、洗浄します。
- 酸性タイロード液を用いて透明帯を除去します。最初の解決策の一つ30μlのドロップに卵子を配置し、別の30に転送するすべてのメディアに沿って行わとして液滴は、酸を希釈し、その効率が低下します。のみ限り、それは長い露出がそれらを傷める可能性がありますので、透明帯を除去するのにかかるように溶液中で卵母細胞を保持します。注:人間の透明帯は、マウスより酸性タイロード溶液を用いた治療に対してより抵抗性であるため、酸性タイロード溶液またはプロナーゼの増加濃度は、ヒトサンプルに使用することができます。
- M2メディア30μlのドロップにもう一度卵母細胞を洗浄します。
2。アガロースの埋め込みと溶解
- アガロースの埋め込みを実行するには、70°C heatblockに溶解液を配置します。溶解液:2:1アガロースを生産する溶解液に予熱されたLMPアガロースを追加します。
- 最小限のM2メディアで清浄なガラススライド上に単一の卵母細胞を配置します。ピペットの先端に溶解溶液、(顕微鏡下で)軽くはミニムと混合することができ、スライドガラス上に(〜1μl以下)少量を追放:アガロースの10μlを取るアルメディア。静かにピペットの先端に卵母細胞をピックアップし、ビーズ球を形成するように300μlのミネラルオイルをエッペンドルフチューブにすべての10μlを置く。
注:このプロセスは、アガロースとして、かなり迅速に行わなければなりませんなり硬化温度は、わずか5°C 70℃を下回った場合 - 氷上で10分間チューブをインキュベートします。溶解を実行するには、300μlのミネラルオイルを除去し、SDS溶解バッファー500μlを添加します。 50°Cの水浴中で一晩インキュベートします。
注:溶解液( 表1)も、この目的のために使用されることがあります。
2日目
亜硫酸塩の突然変異誘発の日に新鮮な以下のソリューションを準備します。 DNAの混入の可能性を減らすために、頻繁に手袋を変更し、フィルターチップを使用しています。オープン離れて角度チューブを保持し、使用しないときは、すべてのチューブをおさらい。我々は解決策は、n +1として行われることをお勧めします。
2| 3 M NaOHを | |
| 0.1 M NaOHを | 14.5ミリリットルオートクレーブのddH 2 Oで3Mの0.5ミリリットル |
| 0.3 M NaOHで | 13.5ミリリットルの3M 1.5 mlのオートクレーブのddH 2 O |
2.5 Mの亜硫酸水素塩溶液
- 3.8グラムの亜硫酸水素ナトリウム
5.5ミリリットルGIBCO蒸留H 2 O
1ミリリットル3 M NaOHを
@室温を溶かす - 110 mgのヒドロキノン
1ミリリットルGIBCO蒸留H 2 O
@ 90°C(のみ限り、それが溶解するのにかかるようにするため、定期的に混在させること)を溶解する
完全に溶解したとき、溶液(a)および(b)を混ぜて
*光*から遠ざける
3。亜硫酸塩の突然変異誘発
- 完全に500μlのSDS溶解バッファーを除去し、300μlのミネラルオイル(〜20時間)を追加します。残っている溶解バッファーは、THを希釈します。それが加熱され、ビーズは、後続のステップで溶解の影響を受けやすくなります電子アガロース。亜硫酸直ちに誘発、または-20℃で保存し5日までのを続行します。
- 該当する場合は、冷凍庫から卵母細胞を除去し、(アガロースビーズは比較的透明であるだけになるまで)解凍してみましょう。 10分間氷上でインキュベートした後に、90℃のヒートブロックで2.5分間インキュベートします。
注:ミックスや炒め、2.5分以上延長するか、温度が変動しないようにしてください。 - 変性を実行するには、ミネラルオイルを除去し、各チューブに1ミリリットルの0.1M NaOHを加え、フリック、5-6回転倒。
- 37°Cの水浴、反転毎に3-4分で15分間インキュベートします。ビーズはNaOHにフロートする必要があります。
- 重亜硫酸塩処理を実行するために、穏やかにチューブをスピンし、NaOHを除去し、300μlのミネラルオイルと500μlの亜硫酸水素塩溶液を追加します。 50℃水浴中で3.5時間チューブをインキュベートします。*光*から遠ざける
注:インキュベーションの長さは経験的に、目的の遺伝子のために決定する必要があります。 - 脱スルホン化を実行するには、3分間氷上でインキュベートし、その後、鉱物油と亜硫酸水素塩溶液を除去穏やかに回転し、1ミリリットル0.3 M NaOHを追加します。フリックおよび5-6回転倒。
- 37°Cの水浴、反転毎に3-4分で15分間インキュベートします。ビーズはNaOHにフロートする必要があります。
- サンプルを洗浄し、最初に軽く回転させて、その後NaOHを除去し、1 mlの1X TE pHが7.5を追加します。室温(シェーカー上で)で5〜10分間振とうする。再び静かに回転し、1×TEを削除します。回この洗浄プロセスを繰り返します。
- 1ミリリットルオートクレーブのddH 2 Oを追加する室温(シェーカー上で)で5〜10分間振とうする。静かに回転し、H 2 Oを削除するのddH 2 Oが2回洗浄を繰り返します。
- 上清のpHをチェックして、それはpHが5.0である必要があります。まだあまりにも基本的な場合には、H 2 Oで再び洗浄唯一のアガロースがある残して、すべての上清を除去広告。
4。第1と第2ラウンドのPCR増幅
- **を洗濯しながら第1ラウンドPCRミックス**を準備します。
| 10μMプライマーフォワードアウター | 0.5μL |
| アウター10μMのプライマー、リバース | 0.5μL |
| 240 ng / mlのtRNAの | 1μlの |
| H 2 O | 13μlの |
Illustraレディ·ツー·移動ホットスタートPCRビーズに追加
慎重に (〜10μl)のPCRチューブに固アガロースビーズをスライドさせ
70°C、ミックスへの熱
25μlのミネラルオイルを追加します。
合計:50μlの
- 増幅する
注:マウスSnrpnためのサイクリング条件の例は、94℃で2分間変性である94℃30秒を40サイクル、続い°C、°C、50°Cで1分間、168分°Cおよび68で最後の10分間の伸長工程℃、マウスH19のための第1ラウンドPCRの温度とPeg3アニーリングは50℃です。 - 第2ラウンドのPCRミックスを調製する
| フォワードインナー10μMのプライマー | 0.5μL |
| インナー10μMのプライマー、リバース | 0.5μL |
| H 2 O | 19μlの |
Illustraレディ·ツー·移動ホットスタートPCRビーズに追加
テンプレートとして5μlの第1ラウンドの製品を追加します。 °C 1分間アガロースを和らげるために70に第1ラウンド、製品を加熱する。鉱物油の層の下にピペットしてください。
25μlのミネラルオイルを追加します。
合計:50μlの
注:Snrpn、H19、およびPeg3のネストされたプライマー配列は、市場Velker ら 10で見つけることができます12。
- 増幅する
注:サイクリング条件マウスSnrpnためには、94℃で2分間変性である94℃で30秒間の40サイクル、続いてC、°C、50°Cで1分、68℃1分°C;と最後の10分間の伸長68ステップで10℃。マウスH19およびPeg3は、第2ラウンドのPCRのために50℃アニーリング温度を必要とします。 - 診断テストとして、第二ラウンドのサンプルは、メチル化または株特異的である制限酵素で切断することができます。
| 第2ラウンドの製品 | 4μlの |
| 制限酵素 | 1μlの |
| バッファ | 1μlの |
| H 2 O | 4μlの |
- 8%アクリルアミドゲル上で消化産物を電気泳動させる。すべての異種のバンドは、複数のsequeを表すNCE。
5。 TAクローニングとコロニーPCR
- プロメガのpGEM-Tベクターシステム(フィッシャーサイエンティフィック猫#A1360)を使用してベクターにライゲーションし、℃で1分間アガロースを和らげるために第2ラウンド、製品、70〜最初のヒートのクローンを作成する。
| 第2ラウンドのPCR | 1μlの |
| pGEMT-EASYベクトル | 1μlの |
| リガーゼ | 1μlの |
| H 2 O | 2μlの |
| 2×ライゲーションバッファー | 5μlの |
@ 4℃PCRマシンで一晩インキュベートします。
- 15分間氷上でコンピテント大腸菌細胞 (Zymoリサーチ社のカタログ番号T3009)を解凍します。 8μlの大腸菌と15分間氷冷した後ライゲーションに3μlのライゲーション反応を追加します。
- 42℃水浴中で40秒間熱ショック、そして2分間氷上でインキュベートする。 1時間(シェーカー)で37℃で60μlのSOC培地とインキュベートする。
- 37℃で一晩LB /寒天/ IPTG / Xgal /アンププレートとインキュベートするプレート上で反応混合物のすべてを配置します。
- コロニーPCRミックスを調製する
| 20μMM13フォワードプライマー | 0.7μL |
| 20μMM13リバースプライマー | 0.7μL |
| 5XゴーグリーンのTaqバッファー | 7.0μL |
| を10mMのdNTP | 0.7μL |
| Taq DNAポリメラーゼ | 0.28μL |
| H 2 O | 25.62μL |
| 35μlの合計 |
PCRチューブに35μlのコロニーPCRマスターミックスを追加します。ピペットチップ、PCR反応に渦巻き、それをしてプレートから白い細菌のコロニーを選択します。
- 94℃で10分間変性を増幅する94℃45秒を30サイクル、続いてC、°57のC、30秒℃、72℃1分°Cおよび72最後の10分間の伸長工程℃、 1.5%アガロースゲル上で4μlを電気泳動させる。シークエンシング用のPCR産物の〜30μlを送信します。
注:卵母細胞では、5コロニーPCR産物の塩基配列を決定しています。 - シーケンシングの結果が得られたら、メチル化パターンを読み取ることができます。 CGとして残ったすべてのオリジナルのCGがメチル化され、現在はTGである任意のオリジナルCGがメチル化されました。
6。代表的な結果
我々の仕事では、アッセイは、個々の卵母細胞および胚(図1)でメチル化を刻印。亜硫酸水素塩変換されたプライマーを用いてネステッドPCR増幅に続いて、それはアガロースゲル(図2)で正しいフラグメントサイズを可視化することにより成功した変換を確認することが可能です。個々の卵母細胞1親の対立遺伝子を表しており、理論では、1つのインプリンティング、メチル化パターンを有している。このように、第二ラウンドのPCR産物は、意図しないコンタミネーションのためにテストすることができます。 DNAメチル化(たとえば、HinfIまたはDpnIIなど)に敏感な制限酵素は、それがメチル化または非メチル化アレル(図3)が含まれているかどうかを評価する第二ラウンドのPCR産物を消化するために使用することができます。 Tに変換される非メチル化Cがもはや酵素によって認識されず、ノーカットでいる間に酵素認識配列内のメチル化Cが切断される。それは卵丘細胞の汚染(図3)の指標であるとして、メチル化及び非メチル化対立遺伝子の両方を含む任意のMII卵母細胞サンプルは、破棄する必要があります。ライゲーションおよび形質転換に続いて、成功したコロニーPCR増幅は、適切な製品サイズのサンプルを確保するためにアガロースゲル上で可視化することができるシーケンス(図4)に送信されます。最後に、5つの個々のCLのシーケンスMII卵母細胞からのものは、5つの同一のメチル化パターンと同一のnonCpGのコンバージョン率(図5a)を生成する必要があります。複数のパターンを含む任意の試料(図5b)破棄する必要があります。排卵MII卵母細胞は、2染色体のコピーまたは接続された極体を持っているので、2つの類似配列パターン(図5c)を得るための可能性があります。私たちは、卵丘細胞の汚染が排除できないので、非常に異なるメチル化パターンを持っている卵母細胞からデータを破棄することをお勧めします。

図1:シングル卵重亜硫酸塩の突然変異誘発アッセイの模式図。
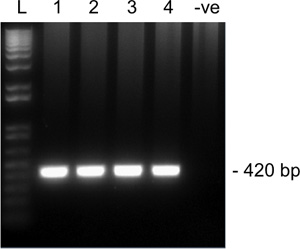
歌うからSnrpnための第2ラウンドの増幅から図2代表的な結果1.5%アガロースゲル上ルのMII卵母細胞。レーン1-4は、4つのシングルMII卵母細胞であり、レーン5は、陰性対照(無卵母細胞)である。 Snrpnの予想アンプリコンサイズは420 bpである。 L、はしご。
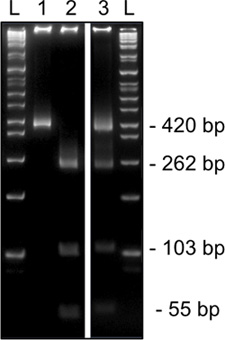
8%アクリルアミドゲル上で単一のMII卵母細胞からSnrpnための第2ラウンドメチル化特異的制限酵素消化から図3代表的な結果。HinfI診断の制限消化(420bp、レーン1)制限酵素サイトを廃止し、Tを抱いて非メチル化DNAを示しています。認識サイト内のC(カット、262、103、54 bpの、レーン2)が含まれているか、メチル化DNA。両方のメチル化及び非メチル化制限酵素サイト(カット&ノーカットバンド、レーン3)を示す消化は卵丘細胞の混入の指標である。 L、はしご。
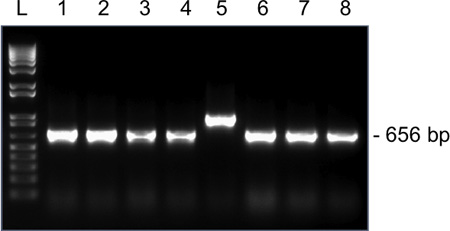
図4。 1.5%アガロースゲル上で単一のMII卵母細胞からSnrpnためのコロニーPCR増幅のための代表的な結果。予想されるアンプリコンサイズをpGEM-T EasyベクターにSnrpnのライゲーションの後、前方M13を使用し、リバースプライマーは、656 bpである。レーン1-8、クローン1-8のアンプリコン。クローン5は、誤ったアンプリコンサイズを持っており、シーケンシングのために送られるべきではありません。

図5単一のMII卵母細胞からSnrpnの代表的なシーケンス結果は。Snrpnは、卵母細胞においてメチル化されている。黒丸は、メチル化CpGがを示しています。白丸は非メチル化CpGがを示しています。 CpGの数及び配置は、B6系統の雌マウスの代表です。 a)単一のMII卵母細胞からSnrpnの結果をシーケンス期待しています。 DNAの唯一の一本鎖は、すべての5つのクローンで増幅する必要があります。単一のメチル化パターンと同一の非CpGに変換パタパタと卵nは、(非CpGの転化率は、総非CpGのシトシンの割合としてチミンに変換され、非CpGのシトシンの数として計算された右側に示されている)の分析に含まれるべきである。卵丘細胞の混入を持つ単一のMII卵母細胞からSnrpnためのb)のシークエンシングの結果。複数鎖の増幅を示すメチル化状態と変換パターン間の相違に注意してください。 c)の染色体のコピーまたは極体の取り込みの両方を持つ単一のMII卵母細胞からSnrpnの結果をシーケンス。
ディスカッション
この単一の卵母細胞アッセイは重要であり、特別なケアを必要とする数多くの手順が含まれています。最初は卵母細胞の洗浄です。それは、できるだけ多くの卵丘細胞のように削除するヒアルロニダーゼ処理後に廃棄し新鮮な培地に各卵母細胞を複数回洗浄することが特に重要である。また、透明帯を除去するため、酸性タイロード液に卵を転送するときは、周囲の媒体が卵丘細胞の明確で?...
開示事項
著者らは、開示することは何もありません。
謝辞
この作品は、ウェスタン·オンタリオ、産科と婦人科の大学によってサポートされていました。とMinistryof研究とイノベーション、初期のリサーチャー賞からの助成金ER06-02-188。 MMDは、再生、早期開発と健康(REDIH)大学院奨学金への影響のCIHRのトレーニングプログラムによってサポートされていました。
資料
特定の試薬 や機器のテーブルです。
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | コメント |
| 卵母細胞コレクション | |||
| ヒアルロニダーゼ | シグマ | H4272 | |
| 酸性タイロード | シグマ | T1788 | |
| プロテイナーゼK | シグマ | P5568 | |
| 10%IGEPAL | Bioshop | NON999.500 | |
| 溶菌液 | |||
| トリス(pH7.5) | Bioshop | TRS001.5 | |
| LiClを | シグマ | L9650 | &n個のBSPは、 |
| EDTA pH8.0で | シグマ | E5134 | |
| LIDS | Bioshop | LDS701.10 | |
| DTT | インビトロジェン | P2325 | |
| SDS溶解バッファー | |||
| TE pH7.5の | Bioshop(トリス) シグマ(EDTA) | TRS001.5 E5134 | |
| 10%SDS | Bioshop | SDS001.500 | |
| Bisulfite変換 | |||
| 水酸化ナトリウム | シグマ | S8045 | |
| 亜硫酸ナトリウム(亜硫酸水素ナトリウム) | シグマ | 243973 | ; |
| ハイドロキノン | シグマ | H9003 | |
| 低融点(LMP)アガロース | シグマ | A9414 | |
| 鉱油 | シグマ | M8410 | |
| M2中 | シグマ | M7167 | |
| GIBCO蒸留水 | インビトロジェン | 15230-196 | |
| オートクレーブ滅菌蒸留(DD)水 | |||
| PCR | |||
| IllustraホットスタートミックスRTG | GEヘルスケア | 28-9006-54 | |
| 240 ng / mlの酵母tRNA | インビトロジェン | 15401-011 | |
| 5倍グリーンGoTaq反応バッファー | プロメガ | M7911 | |
| 内側と外側のネストされたプライマー | シグマ | ||
| ライゲーション | |||
| プロメガのpGEM-T Easyベクター | フィッシャー·サイエンティフィック | A1360 | |
| TAクローニング | |||
| 有能な大腸菌細胞 | Zymoリサーチ(株) | T3009 | |
| 機器 | |||
| 解剖顕微鏡 | |||
| 70°Cと90°Cヒートブロック | |||
| 37°Cと50°C Waterbaths(42°Cの変換用) | |||
| ロッカー | |||
| PCRマシン | |||
参考文献
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. 33, 245-254 (2003).
- Rodenhiser, D., Mann, M. Epigenetics and human disease: translating basic biology into clinical applications. CMAJ. 174, 341-348 (2006).
- Frommer, M. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc. Natl. Acad. Sci. U.S.A. 89, 1827-1831 (1992).
- Clark, S. J., Harrison, J., Paul, C. L., Frommer, M. High sensitivity mapping of methylated cytosines. Nucleic Acids Res. 22, 2990-2997 (1994).
- Feil, R., Charlton, J., Bird, A. P., Walter, J., Reik, W. Methylation analysis on individual chromosomes: improved protocol for bisulphite genomic sequencing. Nucleic Acids Res. 22, 695-696 (1994).
- Raizis, A. M., Schmitt, F., Jost, J. P. A bisulfite method of 5-methylcytosine mapping that minimizes template degradation. Anal. Biochem. 226, 161-166 (1995).
- Patterson, K., Molloy, L., Qu, W., Clark, S. DNA Methylation: Bisulphite Modification and Analysis. J. Vis. Exp. (56), e3170 (2011).
- Olek, A., Oswald, J., Walter, J. A modified and improved method for bisulphite based cytosine methylation analysis. Nucleic Acids Res. 24, 5064-5066 (1996).
- Mann, M. R. Selective loss of imprinting in the placenta following preimplantation development in culture. Development. 131, 3727-3735 (2004).
- Market-Velker, B. A., Zhang, L., Magri, L. S., Bonvissuto, A. C., Mann, M. R. Dual effects of superovulation: loss of maternal and paternal imprinted methylation in a dose-dependent manner. Hum. Mol. Genet. 19, 36-51 (2010).
- Meng, L. H., Xiao, S. Q., Huang, X. F., Zhou, Y., Xu, B. S. A study on bisulfite sequencing method for methylation status of imprinted genes in single human oocytes. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 25, 289-292 (2008).
- Denomme, M. M., Zhang, L., Mann, M. R. Embryonic imprinting perturbations do not originate from superovulation-induced defects in DNA methylation acquisition. Fertil. Steril. 96, 734-738 (2011).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Hajj, N. E. l. Limiting dilution bisulfite (pyro)sequencing reveals parent-specific methylation patterns in single early mouse embryos and bovine oocytes. Epigenetics. 6, 1176-1188 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved