É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Mutagênese Bissulfito Único de ovócitos
Neste Artigo
Resumo
Mutagênese bissulfito é o padrão ouro para a análise de metilação do DNA. Nosso protocolo modificado permite a análise de metilação do DNA ao nível de uma única célula e foi projetado especificamente para oócitos individuais. Também pode ser utilizado para a clivagem do estágio-embriões.
Resumo
Epigenética engloba todas hereditárias e modificações reversíveis de cromatina que alter acessibilidade gene e, portanto, são os principais mecanismos de regulação da transcrição do gene 1. Metilação do DNA é uma modificação epigenética, que atua predominantemente como uma marca repressiva. Através da adição covalente de um grupo metilo em citosinas na dinucleótidos CpG, pode recrutar adicionais proteínas repressores e modificações histona para iniciar processos envolvidos na condensação da cromatina e silenciamento de genes 2. Metilação do DNA é essencial para o desenvolvimento normal, pois desempenha um papel fundamental na programação do desenvolvimento, diferenciação celular, a repressão de elementos retrovirais, inativação do cromossomo X e imprinting genômico.
Um dos métodos mais poderosos para a análise de metilação de DNA é a mutagénese bissulfito. Bissulfito de sódio é um agente mutagénico DNA que deaminates citosinas em uracilos. Após amplificação por PCR e Seque ncing, estes eventos de conversão são detectados como timinas. Citosinas metiladas são protegidos de desaminação e, assim, permanecer como citosinas, permitindo a identificação de metilação de ADN a nível individual de nucleótidos 3. Desenvolvimento da mutagênese bissulfito avançou daqueles originalmente informados 4-6 para aqueles que são mais sensíveis e reprodutíveis 7. Um avanço chave foi a incorporação de pequenas quantidades de DNA em um grânulo de agarose, protegendo assim o DNA a partir do tratamento com bissulfito dura 8. Esta análise de metilação habilitado a ser realizada em piscinas de oócitos e embriões em estágio de blastocisto 9. O protocolo de mutagênese mais sofisticado bissulfito de data é para cada estágio de blastocisto-embriões 10. No entanto, uma vez que os blastocistos têm, em média 64 células (contendo 120-720 pg de DNA genómico), este método não é eficaz para os estudos de metilação em oócitos individuais ou de clivagem-stage embriões. tenda "> Tomar pistas de agarose incorporação de quantidades de DNA minutos, incluindo oócitos 11, aqui apresentamos um método pelo qual os oócitos são diretamente incorporado em um agarose e solução de lise talão de recuperação imediatamente a seguir e remoção da zona pelúcida do ovócito. Isso nos permite ignorar os dois principais desafios da mutagénese único oócito bissulfito:. protectores uma pequena quantidade de DNA a partir de degradação, e subsequente perda durante os passos do protocolo numerosas importante, como os dados são obtidos a partir de oócitos individuais, a questão da PCR viés dentro piscinas é eliminado Além disso. , a contaminação de células do cumulus inadvertida é detectável por este método uma vez que qualquer amostra com mais do que um padrão de metilação pode ser excluídos da análise 12. Este protocolo fornece um método melhorado para análises de sucesso e reprodutível de metilação do DNA ao nível de uma única célula e é idealmente adequado para oócitos individuais, bem como estágios de clivagem dos embriões.
Protocolo
DIA 1
Prepare as seguintes soluções frescas no dia da coleta de oócitos com água destilada estéril como GIBCO água. Para reduzir a chance de contaminação de DNA, mudar frequentemente e usar luvas de pontas com filtro. Manter tubos angular longe quando aberto, e recapitular todos os tubos quando não em uso. Sugerimos que as soluções são feitas como se n +1.
3% de agarose LMP
30 mg baixo ponto de fusão (LMP) Agarose
até 1 ml de H2O GIBCO
dissolver @ 70 ° C
Solução de lise
8 ul de tampão de lise
1 uL de proteinase K
1 uL de 10% IGEPAL
lugar em gelo até estar pronto para utilização
02:01 Agarose: solução de lise (10 uL por oócito indivíduo, a quantidade é de 3 oócitos)
20 ul de 3% de agarose LMP
10 ul da solução de lise
misturar @ 70 ° C
Ly SDSsis tampão (501 ul por oócito individual)
| 1x TE pH 7,5 | 450 ul |
| 10% de SDS | 50 uL |
| Proteinase K | 1 ml |
| 501 ul |
1. Recolha de ovócitos
- Coloque os ovidutos rato dissecadas em mídia M2, e rasgar a ampolas para extrair o complexo de células cumulus.
- Separa-se os oócitos a partir do complexo de células do cumulus usando 0,3 mg / ml em solução de hialuronidase uma queda de 30 uL de meios M2. Manter os oócitos em solução apenas enquanto que leva para remover as células do cumulus, como a exposição prolongada pode danificar. Lave os oócitos em 3x a queda de 30 ul de mídia M2, removendo as células cumulus periodicamente.
- Remover a zona pelúcida utilizando uma solução ácida de Tyrode. Coloque os oócitos em uma queda de 30 ml de solução em primeiro lugar, e depois transferir para outras 30gota ul, como quaisquer meios transportados ao longo irá diluir o ácido e reduzir a sua eficiência. Manter os oócitos em solução apenas enquanto que leva para remover a zona, como a exposição prolongada pode danificar. Nota: um aumento da concentração de solução ácida de Tyrode ou pronase podem ser utilizados para amostras humanas, tal como o ser humano zona pelúcida é mais resistente ao tratamento com solução ácida de Tyrode do que o rato.
- Lavar os oócitos mais uma vez em uma queda de 30 uL de meios M2.
2. Agarose Embedding e Lise
- Para realizar a incorporação de agarose, colocar a solução de lise em um 70 ° C heatblock. Adicionar o pré-aquecido LMP de agarose para a solução de lise, produzindo um agarose 02:01: solução de lise.
- Coloque um único óvulo numa lâmina de vidro limpa no mínimo mídia M2. Tome-se 10 uL da agarose: solução de lise numa ponta de pipeta, e (sob um microscópio) suavemente expelir uma pequena quantidade (~ 1 uL ou menos) para a lâmina de vidro, permitindo que ele se misturar com o minimal mídia. Levante com cuidado o oócito na ponta da pipeta e colocar todos os ul 10 em um tubo Eppendorf com 300 uL de óleo mineral para o talão forma uma esfera.
Nota: este processo tem de ser feito muito rapidamente como a agarose vai endurecer se a temperatura descer tão pouco como 5 ° C abaixo de 70 ° C. - Incubar o tubo em gelo durante 10 minutos. Para realizar a lise, remover o óleo mineral 300 uL e adicionar 500 uL de tampão de lise SDS. Incubar durante uma noite em um banho de água 50 ° C.
Nota: solução de lise (Tabela 1) podem também ser utilizados para esta finalidade.
DIA 2
Preparar as seguintes soluções frescas no dia da mutagénese bissulfito. Para reduzir a chance de contaminação do DNA, mudar frequentemente e usar luvas de pontas com filtro. Manter tubos angular longe quando aberto, e recapitular todos os tubos quando não em uso. Sugerimos que as soluções são feitas como se n +1.
| 3 M de NaOH | 2,4 g de NaOH em 20 ml autoclavado ddH2O |
| 0,1 M de NaOH | 0,5 ml de 3M em 14,5 ml autoclavado ddH2O |
| 0,3 M de NaOH | 1,5 ml de 3M em 13,5 ml autoclavado ddH2O |
Solução 2,5 M Bissulfito
- 3,8 g de bissulfito de sódio
5,5 ml GIBCO H2O destilada
1 ml NaOH 3 M
dissolver @ temperatura ambiente - 110 mg Hidroquinona
1 ml GIBCO H2O destilada
dissolver @ 90 ° C (apenas durante o tempo que leva a dissolver-se, misturar regularmente)
Quando completamente dissolvido, misturar a solução (a) e (b)
* Mantenha longe de luz *
3. Mutagênese Bissulfito
- Remover completamente a 500 uL de tampão de lise de SDS e adicionam-se 300 uL de óleo mineral (~ 20 horas). Qualquer tampão de lise restante irá diluir the agarose quando é aquecido ea esfera será mais susceptível a dissolução nos passos subsequentes. Prosseguir com mutagénese bissulfito imediatamente, ou armazenar a -20 ° C durante até 5 dias.
- Se for o caso, remova os oócitos do congelador e deixe descongelar (somente até agarose grânulo é relativamente translúcido). Incubar durante 2,5 minutos num bloco de aquecimento 90 ° C, após o que Incubar em gelo durante 10 minutos.
Nota: Não misturar ou agitar, estender mais do que 2,5 minutos, ou flutuar temperatura. - Para realizar a desnaturação, remover o óleo mineral e adicionar 1 ml 0,1 M de NaOH para cada tubo de filme, e inverter 5-6 vezes.
- Incubar durante 15 minutos num banho-maria a 37 ° C, invertendo a cada 3-4 minutos. O grânulo deve flutuar no NaOH.
- Para realizar tratamento com bissulfito, girar suavemente o tubo, em seguida, remover o NaOH e adicionar 300 ul de óleo mineral e 500 solução de bissulfito ul. Incubar o tubo para 3,5 horas a 50 ° C em banho. * Mantenha longe de luz *
Nota: Comprimento de incubação pode precisar de ser determinada empiricamente para o gene de interesse. - Para executar dessulfonação, incubar em gelo durante 3 minutos, em seguida remover o óleo mineral e da solução de bissulfito, girar suavemente, e adicionar 1 ml 0,3 M de NaOH. Flick e inverter 5-6 vezes.
- Incubar durante 15 minutos num banho-maria a 37 ° C, invertendo a cada 3-4 minutos. O grânulo deve flutuar no NaOH.
- Lavar as amostras, por fiação primeiro suavemente, em seguida remover o NaOH e adicionar 1 ml 1x TE pH 7,5. Agitar durante 5-10 minutos à temperatura ambiente (num agitador). Gire suavemente novamente, em seguida, remova o TE 1x. Repetir este processo de lavagem duas vezes.
- Adicionar 1 ml autoclavado DDH 2 O. Agitar durante 5-10 minutos à temperatura ambiente (num agitador). Gire suavemente, em seguida, remover o H 2 O. Repita DDH 2 lavagem O duas vezes.
- Verificar o pH do sobrenadante, que deve ser 5,0 pH. Se ainda demasiado básico, lavar novamente com H2O Remova todo o sobrenadante, deixando apenas a ser agaroseanúncio.
4. 1 ª e 2 ª Rodada de amplificação por PCR
- Prepare 1 ª rodada ** PCR mix enquanto lava **
| 10 Primer mM Encaminhar Exterior | 0,5 ul |
| 10 Reverso Primer mM Exterior | 0,5 ul |
| 240 ng / ml tRNA | 1 ml |
| H 2 S | 13 ul |
Adicionar ao Illustra Ready-to-Go Hot contas de início de PCR
Deslize cuidadosamente o sólido agarose esferas dentro do tubo de PCR (~ ul 10)
Aquecer até 70 ° C e misturar
Adicionar 25 ul de óleo mineral
Total: 50 ul
- Amplificar
Nota: Um exemplo de condições de ciclismo para mouse SNRPN é desnaturação durante 2 minutos a 94 ° C, seguido por 40 ciclos de 30 segundos a 94 ° C, 1 minuto a 50 ° C, e 1minutos a 68 ° C, e um passo final de 10 minutos de alongamento a 68 ° C. Recozimento temperatura para 1 º ciclo de PCR para mouse e H19 Peg3 é de 50 ° C. - Prepare 2 ª rodada PCR mix
| 10 Primer mM Encaminhar Interior | 0,5 ul |
| 10 Reverso Primer mM Interior | 0,5 ul |
| H 2 S | 19 ul |
Adicionar ao Illustra Ready-to-Go Hot contas de início de PCR
Adicionar 5 ul 1 produto Rodada st como um modelo. Aquecer o produto 1 r rodada a 70 ° C durante 1 minuto para amolecer a agarose. Certifique-se de pipetar abaixo da camada de óleo mineral.
Adicionar 25 ul de óleo mineral
Total: 50 ul
Nota: sequências de primers aninhados para SNRPN, H19, e Peg3 pode ser encontrado no Mercado Velker et al 10,12.
- Amplificar
Nota: As condições de amplificação para o rato é SNRPN desnaturação durante 2 minutos a 94 ° C, seguido por 40 ciclos de 30 segundos a 94 ° C, 1 minuto a 50 ° C, e 1 minuto a 68 ° C, e um alongamento final de 10 minutos passo a 68 ° C 10. Rato H19 e Peg3 requerem uma a 50 ° C de temperatura para recozimento 2 nd ciclo de PCR. - Como um teste de diagnóstico, a segunda amostras redondas pode ser cortada com uma enzima de restrição que é a metilação ou estirpe específica.
| 2 ª Rodada de produto | 4 ul |
| Enzima de Restrição | 1 ml |
| Tampão | 1 ml |
| H 2 S | 4 ul |
- Electroforese dos produtos de digestão num gel de acrilamida a 8%. Todas as bandas heterogêneos representar mais de um Sequence.
5. TA Clonagem e Colônia PCR
- Para clonar 2 produto nd rodada, o calor primeiro a 70 ° C durante 1 minuto para amolecer a agarose, em seguida, ligadura no vector utilizando o Promega pGEM-T Vector System (Fisher Scientific Cat # A1360).
| 2 ª Rodada de PCR | 1 ml |
| pGEMT-EASY vector | 1 ml |
| Ligase | 1 ml |
| H 2 S | 2 uL |
| Tampão de ligação 2x | 5 ul |
Incubar durante a noite @ 4 ° C em máquina de PCR.
- Descongelar as células competentes de E. coli em gelo durante 15 minutos (Zymo Research Cat # Corp T3009). Adicionar 3 reacção de ligação uL de 8 uL E.coli e ligadura incubar em gelo durante 15 minutos.
- Choque térmico durante 40 segundos em um banho-maria 42 ° C,e incubar em gelo durante 2 minutos. Adicionar 60 ul meio SOC e incubar a 37 ° C durante 1 hora (em agitador).
- Coloque toda a mistura de reacção sobre uma placa de agar LB / / IPTG / Xgal / Amp e placa incubar a 37 ° C durante a noite.
- Prepare mix colônia PCR
| 20 mM primer forward M13 | 0,7 ul |
| 20 uM iniciador reverso de M13 | 0,7 ul |
| 5X Verde Go Taq Buffer de | 7,0 ul |
| 10 mM de dNTP | 0,7 ul |
| Taq DNA polimerase | 0,28 ul |
| H 2 S | 25,62 ul |
| 35 Total ul |
Adicionar 35 ul Colony PCR master mix em um tubo de PCR. Escolha uma colónia branca bacteriana a partir da placa com uma ponta de pipeta, e agite-lo para a reacção de PCR.
- Amplificar com a desnaturação durante 10 minutos a 94 ° C, seguido por 30 ciclos de 45 segundos a 94 ° C, 30 segundos a 57 ° C, e 1 minuto a 72 ° C, e um passo final de alongamento 10 minutos a 72 ° C. Electroforese 4 uL sobre um 1,5% de gel de agarose. Enviar ~ uL 30 do produto de PCR para a sequenciação.
Nota: Para oócitos, 5 produtos de PCR de colônia são seqüenciados. - Uma vez que os resultados de sequenciação são obtidos, padrões de metilação pode ser lido. Qualquer CG original que se manteve como um CG foi metilado, e qualquer CG original, que é agora uma TG foi não metilado.
6. Os resultados representativos
No nosso trabalho, nós ensaio impressa metilação em oócitos individuais e embriões (Figura 1). Após amplificação por PCR aninhada utilizando iniciadores bissulfito convertidos, é possível confirmar uma conversão bem sucedida, visualizando um tamanho do fragmento correcto sobre um gel de agarose (Figura 2). Um oócito indivíduorepresenta um alelo parental, e em teoria, tem um padrão de metilação impresso. Como tal, segundo ciclo de PCR produtos podem ser testados para a contaminação não intencional. Uma enzima de restrição sensíveis à metilação de DNA (como HinfI ou DpnII) pode ser usado para digerir o segundo produto de PCR rodada para avaliar se ele contém um alelo metilado ou não metilado (Figura 3). Um C metilado no interior da sequência de reconhecimento da enzima é clivado, enquanto um C não metilado que é convertido em T não é reconhecido pela enzima e é cortada. Qualquer amostra de oócitos MII contendo ambos os alelos metilados e não metiladas deve ser descartado, uma vez que é indicativo de contaminação cumulus celular (Figura 3). Após ligação e transformação, bem sucedida colónia de amplificação por PCR podem ser visualizados num gel de agarose para assegurar as amostras com o tamanho do produto correcto são enviados para a sequenciação (Figura 4). Finalmente, a seqüência de cinco cl indivíduomais de um ovócito MII deve produzir cinco padrões de metilação idênticos e as taxas de conversão nonCpG idênticos (Figura 5A). Qualquer amostra que contêm mais do que um padrão deve ser eliminada (Figura 5b). Uma vez que os oócitos ovulados MII tem duas cópias do cromossoma ou um corpo em anexo polar, existe uma possibilidade para a obtenção de dois padrões de sequência semelhante (Figura 5C). Recomendamos descartando dados a partir de oócitos que têm padrões de metilação altamente desiguais desde a contaminação de células cumulus não pode ser descartada.

Figura 1. Esquemática do único ensaio de mutagênese Oocyte Bissulfito.
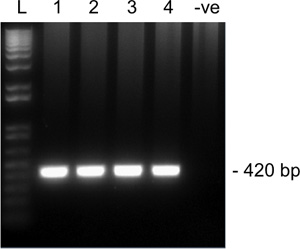
Figura 2. Resultados representativos de 2 ª rodada de amplificação para SNRPN de um cantarle oócito MII em um gel de agarose 1,5%. As pistas 1-4 são quatro oócitos individuais Mii e pista 5 é um controle negativo (sem oócito). Tamanho amplicon esperado para SNRPN é de 420 pb. Escada L,.
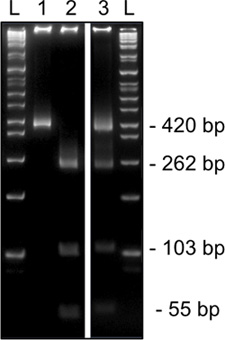
Figura 3. Os resultados representativos de digestão de restrição 2 nd rodada-metilação específico para SNRPN a partir de um oócito MII única em um gel de acrilamida a 8%. HinfI digestão de restrição de diagnóstico mostra ADN não metilado que abriga um T que suprime o local de restrição (420bp, pista 1) ou ADN metilado que contém um local de reconhecimento C dentro de (corte, 262, 103, e 54 pb, pista 2). Digestão mostrando locais de restrição tanto metilados e não metilado enzima de bandas de corte e não cortada, pista 3) são indicativos de contaminação de células do cumulus. Escada L,.
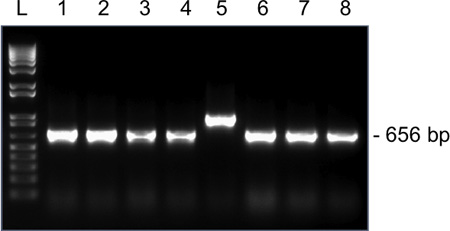
Figura 4. Os resultados representativos para a amplificação por PCR de colónias para SNRPN a partir de um oócito MII única em um 1,5% de gel de agarose. Tamanho amplicon esperado após a ligadura do SNRPN no vetor pGEM-T Easy e usando M13 forward e primers reversos é 656 bp. Pista 1-8, amplicões a partir de clones 1-8. Clone 5 tem um tamanho de fragmento amplificado incorrecta e não deve ser enviada para a sequenciação.

Figura 5. Representativas sequenciação resultados para SNRPN a partir de um oócito MII único. SNRPN é metilado em oócitos. Círculos pretos indicam CpGs metilados. Círculos brancos indicam CpGs não metiladas. Número de CpG e colocação é representativo para um rato B6 estirpe feminina. a) Esperado sequenciação resultados para SNRPN a partir de um oócito MII único. Apenas um cordão único de DNA deve amplificar em todos os cinco clones. Oócitos com um único padrão de metilação e patter conversão idêntica não CpGn devem ser incluídos nas análises (percentagem de conversão de não-CpGs indicado à direita foi calculada como o número de não-CpG citosinas convertidos para timina como uma percentagem do total não-CpG citosinas). b) resultados de sequenciação para SNRPN a partir de um ovócito MII único com a contaminação de células cumulus. Observe a dessemelhança entre os estados de metilação e padrões de conversão, indicando amplificação vertente múltipla. c) Seqüenciamento resultados para SNRPN a partir de um ovócito MII única com ambas as cópias do cromossoma ou inclusão corpo polar.
Discussão
Este ensaio único óvulo contém muitas etapas com um número que são críticas e requerem cuidados especiais. A primeira é a lavagem do oócito. É particularmente importante para lavar cada oócito várias vezes em meio fresco cai após o tratamento hialuronidase para remover as células do cumulus tantas quanto possível. Além disso, quando a transferência de ovócitos a solução ácida de Tyrode para zona pelúcida remoção certifique-se envolvente meio é claro de células do cumulus. O oócito é muito pega...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi financiado pela Universidade de Western Ontario, no Departamento de Obstetrícia e Ginecologia, e uma bolsa ER06-02-188 a partir da Pesquisa MINISTÉRIO e Inovação, Prêmio Pesquisador precoce. MMD foi apoiada por um programa de treinamento CIHR em Reprodução, Desenvolvimento precoce eo Impacto na Saúde Bolsas de Pós-Graduação (REDIH).
Materiais
Tabela de reagentes específicos e equipamentos.
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| Recolha de ovócitos | |||
| Hialuronidase | Sigma | H4272 | |
| Ácida de Tyrode | Sigma | T1788 | |
| Proteinase K | Sigma | P5568 | |
| IGEPAL 10% | Bioshop | NON999.500 | |
| Solução de lise | |||
| Tris pH 7,5 | Bioshop | TRS001.5 | |
| LiCl | Sigma | L9650 | & Nbsp; |
| EDTA pH 8,0 | Sigma | E5134 | |
| LIDS | Bioshop | LDS701.10 | |
| TDT | Invitrogen | P2325 | |
| SDS Tampão de Lise | |||
| TE pH 7,5 | Bioshop (Tris) Sigma (EDTA) | TRS001.5 E5134 | |
| 10% de SDS | Bioshop | SDS001.500 | |
| Conversão Bissulfito | |||
| Hidróxido de Sódio | Sigma | S8045 | |
| Hidrogenossulfito de sódio (bissulfito de sódio) | Sigma | 243973 | ; |
| Hidroquinona | Sigma | H9003 | |
| Baixo ponto de fusão (LMP) Agarose | Sigma | A9414 | |
| Óleo mineral | Sigma | M8410 | |
| Meio M2 | Sigma | M7167 | |
| GIBCO água destilada | Invitrogen | 15230-196 | |
| Autoclavado duas vezes água destilada (dd) | |||
| PCR | |||
| Illustra Iniciar Hot Mix RTG | GE Healthcare | 28-9006-54 | |
| 240 ng / ml de levedura tRNA | Invitrogen | 15401-011 | |
| Tampão de reacção 5x Verde GoTaq | PróMega | M7911 | |
| Interno e externo primers aninhados | Sigma | ||
| Ligadura | |||
| Promega pGEM-T Vector Fácil | Fisher Scientific | A1360 | |
| TA Cloning | |||
| Competentes células de E.coli | Zymo Research Corp | T3009 | |
| Equipamento | |||
| Microscópio de dissecção | |||
| 70 ° C e 90 ° C Calor Blocos | |||
| 37 ° C e 50 ° C Os tanques de imersão (42 ° C para as transformações) | |||
| Cadeira de balanço | |||
| Máquina de PCR | |||
Referências
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. 33, 245-254 (2003).
- Rodenhiser, D., Mann, M. Epigenetics and human disease: translating basic biology into clinical applications. CMAJ. 174, 341-348 (2006).
- Frommer, M. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc. Natl. Acad. Sci. U.S.A. 89, 1827-1831 (1992).
- Clark, S. J., Harrison, J., Paul, C. L., Frommer, M. High sensitivity mapping of methylated cytosines. Nucleic Acids Res. 22, 2990-2997 (1994).
- Feil, R., Charlton, J., Bird, A. P., Walter, J., Reik, W. Methylation analysis on individual chromosomes: improved protocol for bisulphite genomic sequencing. Nucleic Acids Res. 22, 695-696 (1994).
- Raizis, A. M., Schmitt, F., Jost, J. P. A bisulfite method of 5-methylcytosine mapping that minimizes template degradation. Anal. Biochem. 226, 161-166 (1995).
- Patterson, K., Molloy, L., Qu, W., Clark, S. DNA Methylation: Bisulphite Modification and Analysis. J. Vis. Exp. (56), e3170 (2011).
- Olek, A., Oswald, J., Walter, J. A modified and improved method for bisulphite based cytosine methylation analysis. Nucleic Acids Res. 24, 5064-5066 (1996).
- Mann, M. R. Selective loss of imprinting in the placenta following preimplantation development in culture. Development. 131, 3727-3735 (2004).
- Market-Velker, B. A., Zhang, L., Magri, L. S., Bonvissuto, A. C., Mann, M. R. Dual effects of superovulation: loss of maternal and paternal imprinted methylation in a dose-dependent manner. Hum. Mol. Genet. 19, 36-51 (2010).
- Meng, L. H., Xiao, S. Q., Huang, X. F., Zhou, Y., Xu, B. S. A study on bisulfite sequencing method for methylation status of imprinted genes in single human oocytes. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 25, 289-292 (2008).
- Denomme, M. M., Zhang, L., Mann, M. R. Embryonic imprinting perturbations do not originate from superovulation-induced defects in DNA methylation acquisition. Fertil. Steril. 96, 734-738 (2011).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Hajj, N. E. l. Limiting dilution bisulfite (pyro)sequencing reveals parent-specific methylation patterns in single early mouse embryos and bovine oocytes. Epigenetics. 6, 1176-1188 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados