Method Article
CRISPR / CASシステムの酵素のための基質の生成
要約
CRISPR / CASシステムは、バクテリアや古細菌の適応免疫を媒介する。多くのCAタンパク質は様々な長さのcrRNA前駆体に作用するエンドリボヌクレアーゼとして機能するように提案されている。ここでは、CASのエンドヌクレアーゼ活性の生化学的解析のための事前crRNA基質を生成するために3つの異なるアプローチを示しています。
要約
ウイルスとその原核生物宿主の相互作用は、細菌と古細菌の生命の進化を形作った。原核生物は制限修飾、不稔感染とCRISPR / CASシステムを含むウイルスの攻撃を回避するためにいくつかの戦略を開発しました。 cは私がSホルト電話 alindromic R EPEAT(CRISPR)配列とsociated(CAS)遺伝子としてC RISPR数(図1)1-3 nterspaced egularly rを光沢のある、多くの細菌と最も古細菌で見つかったこれらの適応性がある免疫システムが構成されています。 Casの蛋白質とリピートの異なるセットがCRISPR / CASシステム4の少なくとも3つの主要な発散型を定義します。ユニバーサルタンパク質CAS1とCAS2は、ウイルスDNAの取り込みに関与することが提案されていること延長CRISPRクラスタ5の5 '末端に2を繰り返す間に新しいスペーサ要素が生成されます。クラスタ全体をすべてのスペーサと反復配列を含む前駆-crRNAに転写され、その後も小さいcrRNAs 6月8日に多様なCas6ファミリーの酵素によって処理されます。これらcrRNAsは、5 '末端(8ヌクレオチド)および3'反復配列9由来末端タグに挟まれたスペーサー配列で構成されています。新しいcrRNAはウイルスDNAにCASタンパク質複合体(カスケード)監督と塩基相補10を介して、そういうものとしてそれを識別されるように、ウイルスの反復性感染症は現在、ブロックすることができます。最後に、CRISPR / Casの1型システムでは、ヌクレアーゼCAS3は、検出された侵入者のDNA 11,12を破壊してしまいます。
これらのプロセスは、原核生物の適応免疫系としてCRISPR / CASを定義し、関係Casのタンパク質の研究のために魅力的な研究分野を開いた。多くのCAタンパク質の機能は依然としてとらえどころあり、CRISPR / CASシステムの見かけの多様性の原因は照らされていない。ほとんどのCAタンパク質の潜在的な活動は、詳細なコンピュータ分析を介して予測した。 Casのタンパク質の主要な部分は、どちらのエンドヌクレアーゼ4として機能することが示されたり提案されている。
ここでは、CASエンドリボヌクレアーゼの研究のためcrRNAsと前駆-cRNAsを生成するための方法を提示します。異なるエンドヌクレアーゼアッセイは、直接RNAオリゴヌクレオチドまたはランオフ転写体外 T7 RNAポリメラーゼにおける介して生成されなくなりましcrRNAとプリcrRNA配列として合成することができる、短い反復配列を必要とします。この方法論は、内部的に標識された酵素の基質の生成と合成または変異crRNAsの作成のための放射性ヌクレオチドの取り込みを可能にします。 Cas6エンドヌクレアーゼ活性は、5'-hydrとcrRNAsに事前にcrRNAsを成熟するために利用されるオキシル及び2 '、3'-環状リン酸末端。
プロトコル
1。 PCRによる長いプリcrRNA基板の世代
- CRISPRクラスタのスペーサー領域を標的とするPCRプライマーを設計。両方のプライマーにベクターへのクローニングPCR産物を用フォワードプライマーおよび制限部位(pUC19のための例えば、BamHI及びHind IIIで、 図2A)にT7 RNAポリメラーゼ(T7RNAP)プロモーター配列(5 '-taatacgactcactata-3')を追加。
注:T7RNAPが転写の適切な開始のためのグアニジン残基を必要とします。 - PCRによりゲノムDNAから目的のプレcrRNA配列を増幅する。
- アガロースゲル電気泳動でPCR産物を分離し、ゲル所望のバンドを抽出します。粘着末端(例えば、BamHI及びHindIII、 図2(a))を作成するための制限酵素を用いてPCR産物を消化。副産物裂を除去するためにPCR精製キットを使用してPCR産物を精製する。
- 、T4 DNAリガーゼを含んでライゲーション反応を設定し、T4 DNAリガーゼ緩衝液および切断されたPCR産物とそれに対応する粘着末端で脱リン酸化線形pUCベクターを3:1のモル比。 16℃で一晩混合物をインキュベートする。標準プロトコルによって有能な大腸菌 DH5α細胞中にライゲーション混合物を変革し、成功したライゲーションを識別するために、青、白スクリーニングを使用しています。
- プラスミド調製キットを用いた白色コロニーからプラスミドを分離します。プラスミドシングによって陽性クローンを識別します。代わりに、コロニーPCRスクリーニングのために利用される可能性があります。
2。 DNAオリゴヌクレオチドのアニーリングを介して中間プレcrRNA基板の世代
- 前方に設計し、希望のCRISPRリピート/スペーサー配列を有するオリゴヌクレオチドを逆にしてください。オリゴヌクレオチドは、T7 RNAPプロモーターの配列と同様に、端末の制限部位を(pUC19のための例えば、BamHI及びHind IIIで)が含まれています。アニール後のその粘着末端フォームを確保するために、オリゴヌクレオチドを終了する( 図2(b)参照)。
- T4ポリヌクレオチドキナーゼ(PNK)を5μl、T4 PNK 10×緩衝液2μl、2μlのATPを(10mM)を含む別々の20μlの反応中の各オリゴヌクレオチドの5'-リン酸化1nmolの。 37℃で1時間のために、各試料をインキュベート℃に
- 2リン酸化オリゴヌクレオチドをハイブリダイズする。リン酸化オリゴフォワード混合物(2.2〜)、リン酸化オリゴ逆混合物(2.2〜)、10μlの反応でT4 DNAリガーゼ10×緩衝液1μl1μlの1μlを兼ね備えています。 95℃で5分間、サンプルをインキュベートする加熱ブロック上または熱湯でのC、熱源をオフにして、混合物を室温(約2-3時間)まで冷却させます。
注:この重要なステップでは、ゆっくりとした冷却プロセスはそれぞれ単一のオリゴヌクレオチド内の構造体の形成に比べて2オリゴヌクレオチドのアニーリングに有利に働く。 - 消化し、脱リン酸化したpUCベクター(0.1μgの/1μlの、ハイブリダイゼーション混合物を4μlを連結20μlのライゲーション混合物中のT4 DNAリガーゼ、T4 DNAリガーゼ10×緩衝液および10mMのATPを含む)添加します。 16℃で一晩静置して下さい。
- 標準プロトコルによって有能な大腸菌 DH5α細胞中に連結したプラスミドを変革し、青白スクリーニングを利用しています。プラスミドを分離し、消化(所望の大きさのインサートをスクリーニングするために)とそれに続くプラスミドシングによって陽性クローンを識別します。
3。カスタムRNAオリゴヌクレオチドの合成を介してショートCasのRNA基質の生成
デザインショートCasのRNA基質(例えば、単一反復配列、 図2C)とカスタムRNAオリゴヌクレオチド合成の施設を利用しています。
注:RNAオリゴヌクレオチドの指定された位置( 図2C)におけるデオキシリボヌクレオチドの封入は、RNA切断の部位を特定するために使用することができます。
4 インビトロ T7 RNAポリメラーゼ転写
- maxiprepプラスミド精製キットを使用して設計された構築物(1.9または2.7から。)を有するプラスミドを分離します。
- クローン化断片(例えば、HindIIIで)の下流を切断する制限酵素でプラスミドを線形化します。完全な消化を確認してください。
注:RNA転写物の発散定義された3 '末端が望まれる場合には、設計された構造物は、上流のHindIII配列の"流出"は、転写用の追加特定の制限部位を含んでいる必要があります。 - クロロホルム(1:1)で抽出し、エタノール沈殿、フェノールによって線状化プラスミドを精製する。 DEPC処理滅菌水にペレットを再懸濁することにより核酸を回収する。
- 体外 T7 RNAP で 40mMのHEPES / KOH(pH 8)を、22 mMのMgCl 2、5mMジチオスレイトール、1mMのスペルミジン、各ヌクレオシド三リン酸の4mMの(ATP、CTP、GTP、UTPを含む転写混合物をオフに実行する設定)、消化の40から100μg/ mlのDEPC処理水でプラスミドおよび0.1 mg / mlのT7 RNAP。 37℃で3時間インキュベート℃、
- 8 M尿素を変性12%ポリアクリルアミドゲル( 図3A)で得られたRNA転写物を分析します。 RNA転写物はモノQ陰イオン交換クロマトグラフィー13を介して精製し、DEPC処理滅菌水でRNA画分とペレットの再懸濁のエタノール沈殿により回収することができる。将来の使用のために、-80℃でRNAを℃で保存
5。 Cas6ヌクレアーゼアッセイ
- セットアップする 体外 T7 RNAP で 20μlの2 mMのATP量の減少が含まれており、2.5μlで補完された転写物(4.4参照)は、α-[32 Pは]-ATPを(10 mCiの/ mlであり、5000 Ci / mmol)はオフを実行します。変性8M尿素12%ポリアクリルアミドゲルからのゲル抽出を経由して反応生成物を精製する。オートラジオグラフィーによりバンドを可視化。
- 所望の組換えCasのタンパク質を生産し、精製する。この例では、Cクロストリジウム·サーモセラムからAS6は熱降水量とNi-NTAクロマトグラフィーにより精製した。
- ヌクレアーゼアッセイ反応( クロストリジウム·サーモセラム Cas6用などを設定し、反応混合物は、20mMのHepes(KOH PH8)、250のKCl、2mMのMgCl 2、1mMのDTT、12,000 cpmのRNA基質および1μMの酵素が含まれており、インキュベートした37℃で30分間)。
- 8 M尿素、12%ポリアクリルアミドゲル上で反応混合物(+ 10μlのRNAは、95%ホルムアミドを含むバッファをローディング)5μlをロードします。オートラジオグラフィーにより電気泳動後の切断産物を可視化します。
6。代表的な結果
Casのエンドヌクレアーゼ活性を分析するためのRNA基質の例は、図3Aに示されている。 in vitro転写反応における分析100μlの5μlのアリコートをロードしました。 RNA産生の効率が異なる構築物との間で変化することに注意してください。いくつかの要因トン帽子は、得られたRNAの量に影響することが観察され、転写開始に必要な1 g次の(i)の初期シーケンスであり、(ⅱ)RNA構造の転写中に形成や世代のための制限部位の(iii)の選択肢の可能性ランオフ開裂位置の。
RNAエンドヌクレアーゼ活性の調査は、高度に精製された組換えCasのタンパク質( 図3B)および適切なネガティブコントロールの両方が必要です。理想的には、この陰性対照試料は、調査Casのエンドヌクレアーゼ反応からできるだけ少し異なります。これは、CAS式(と同一の精製手順に従う)を使用せずに、反応緩衝液および細胞ライセートを用いたRNAのインキュベーションすることによって達成することができる。理想的なネガティブコントロールは、提案された切断部位リボの追加です。図3Cに、5 '末端標識した反復配列の切断は、 クロストリジウム·サーモセラム Cas6ために示されています。下同一の条件、このリピートはデオキシリボヌクレオチドが位置-9に導入されるもう基板ではありません。また、このメソッドは、切断部位に関する情報を提供します。最後に、長い間、内部でラベルされたプレcrRNAはCas6によって切断され、2つの切断断片が観察される。
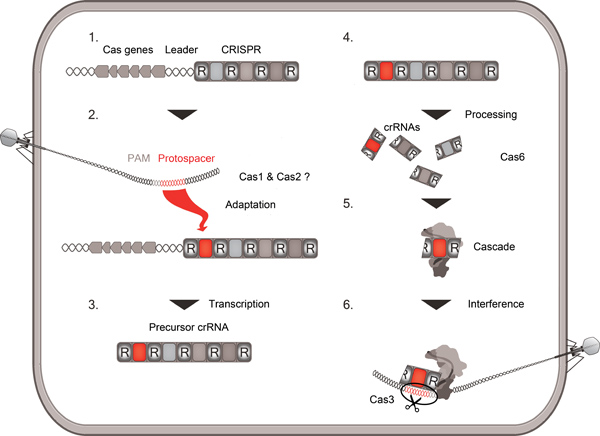
図1 CRISPR / Casの活動の概略概観。概要では、カスケード·コンプレックスとの干渉にcrRNAsの取り込み、Cas6ヌクレアーゼによってCRISPRクラスタ(適応)、小crRNAsにCRISPR配列の転写および処理へのウイルスのDNA配列(protospacer)の挿入を次crRNAとprotospacer間の相補性に基づいてウイルス攻撃を繰り返した。 Protospacer隣接モチーフ(PAM)のマークウイルスprotospacerシーケンス。
igure 2 "SRC =" / files/ftp_upload/4277/4277fig2.jpg "/>
図2 Casのタンパク質に対するRNA基質の生成。スキームは、事前crRNA生産のために()長いプリcrRNA基板、(B)の中間プレcrRNA基板と、(C)短いカスRNA基質を生成するためのワークフローを示しています。例の配列はクロストリジウム·サーモセラムのCRISPR配列のために提示されます。 拡大図を表示するにはここをクリック 。

図3 Cas6ヌクレアーゼアッセイ。カスタム設計されたRNAオリゴヌクレオチド(250 pmol)のおよび in vitro RNA転写物の 2つの (典型的な100μlの反応液5μl)のA.)のトルイジンブルー染色ポリアクリルアミドゲル。 B.)熱後のクロストリジウム·サーモセラムからCas6準備(80ピコモル)のSDS-PAGEゲル50℃降水℃で1時間としたNi-NTAクロマトグラフィー。 in vitro転写産物の 5 '末端標識した反復配列とプレcrRNAためのエンドヌクレアーゼ活性のCas6℃)検出。位置-9におけるdNTPの導入が短いCasのRNA基質(S、 図2C)のCas6切断を廃止しました。 5 '末端8 NTタグは、長いプリcrRNA基質(L、 図2(a))からcrRNAの成熟のために生成されます。バンドが変性8M尿素12%ポリアクリルアミドゲル上で分離し、オートラジオグラフィーによって可視化した。
ディスカッション
提示方法は異なったサイズの範囲のCASエンドヌクレアーゼの基質の生成を有効にして、配列設計の自由度を変化させて。合成RNAオリゴヌクレオチド基質を生成するための最も簡単なアプローチは、より長いRNAオリゴマーを作成する際のコスト上昇と技術的な制限があるため、短いRNAのデザインに限定されています。成功したRNA合成は、100以上のヌクレオチド長の未修飾RNAオリゴマーのために報告されてきたが、カスタムRNA合成のための実用的かつ経済的な最大値は以下の40個のヌクレオチドに位置しています。しかし、任意の所与の配列を合成することができると修飾ヌクレオチドの標的導入(例えばデオキシリボヌクレオチド)が詳細に切断部位を分析するために利用することができます。長いプレcRNAsはin vitro転写を介して生成されるべきである。
T7 RNAポリメラーゼプロモーターを含むオリゴヌクレオチドのアニーリングは中間の長さプリcrRNの製造を可能にとして。日常的にかつ経済的に合成されたDNAオリゴヌクレオチドの最大の長さはちょうど150ヌクレオチド以上であり、この方法で合成プリcrRNAsの最大値を表します。お互いに粘着末端を形成するいくつかのアニールしたDNAオリゴヌクレオチドのペアのアセンブリは、この最大の長さを延長するが、構築物のクローニングの増加の課題を必要とすることができます。この方法の主な利点は、任意の順序で事前crRNAコンストラクト(したがって、エンドヌクレアーゼCasの基質)を生成する機能です。これは合成crRNA設計のテストを可能にします。
最後に、大規模プレcrRNAsは、その全ゲノムCRISPR要素またはその画分のPCR増幅物から得ることができます。プラスミドのin vitro転写鋳型内への変化はプリcrRNAバリアントの生成のための部位特異的変異誘発を介して導入することができます。これらの構築物は、全体の中にグローバルなエンドヌクレアーゼ切断パターンを分析するために使用することができますプリcrRNA。
in vitro転写 T7 RNAポリメラーゼを介した修正されていないRNAの生産のための先駆的な仕事は、トランスファーRNA 14およびブロムモザイクウイルスRNAの15に基づくものであった。コンセンサスT7 RNAポリメラーゼプロモーターが認識ドメイン(-5を通して-17)、必須グアノシン1 16で転写開始とともに開始ドメイン(6を通じて、-4)で構成されています。 1の位置より下流は、ほぼ任意のRNA配列の転写を可能にする変更することができる。 体外ランオフ転写テンプレートで生成するための提示方法は、ゲノムCRISPR領域または合成予備crRNAバリアントを一致完全プレcrRNAsのin vitro合成を可能にします。転写反応は、5 '一リン酸末端14を得るために、GMPにプライミングされていない限り、転写物は5'-末端三リン酸の存在で生成されます。転写物が標識されるなら、そのような末端が必要であるT4ポリヌクレオチドキナーゼおよびγ-[32 P]-ATP。 Cas6とCas6様酵素の切断活性、5'-水酸基と2 '、3'-環状リン酸末端を含むcrRNAを生成します。これらのRNAは、その後、カスケード複合体によって認識について分析することができる。
開示事項
特別な利害関係は宣言されません。
謝辞
著者らは、図1の準備を支援するための組換え型T7 RNAポリメラーゼの準備のためのジャネットSchermulyとノーマンEbeltに感謝したいと思います。この作品は、ドイツ学術協会(DFG FOR1680)とマックス·プランク協会からの資金によって賄われていた。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名称 | 会社 | カタログ番号 | コメント(オプション) |
| 南極ホスファターゼ | NEB | M0289L | |
| in vitro転写グレード純ヌクレオチド三リン酸で | イエナバイオサイエンス | NU-1010 - NU-1013 | |
| 液体クロマトグラフィー、ktapurifier 100 | GEヘルスケア | ||
| DNAオリゴヌクレオチド | MWGオペロン | ||
| RNAオリゴヌクレオチド | MWGオペロン | ||
| ゲル抽出キット | QIAGEN | 28704 | |
| PCR精製キット | QIAGEN | 28104 | |
| Spin Miniprepキット | QIAGEN | 27106 | |
| プラスミドマキシキット | QIAGEN | 12163 | |
| BamHIで | NEB | R0136L | |
| HindIIIで | NEB | R0104L | |
| T4 DNAリガーゼ | NEB | M0202S | |
| T4ポリヌクレオチドキナーゼ | アンビオン | AM2310 | |
| T7 RNAポリメラーゼ | 独自の準備 | ||
| デオキシソリューションミックス | NEB | N0447L | |
| ミニプロティアンテトラ電気泳動システム | バイオ·ラッド | 165-8001 | |
| ストーム840スキャナ | GEヘルスケア | 163723 | |
| ストレージPhosphorscreen | 分子動力学 | 63-0034-81 | |
| モノQ 50分の5 GL | GEヘルスケア | 17-5166-01 | |
| Phusion高忠実度DNAポリメラーゼ | NEB | M0530L | GCのバッファを使用 |
| トルイジンブルー | シグマ | T3260 | |
| pUC19を | NEB | N3041S | |
| γ-[32 P]-ATPは、α-[32 P]-ATP | ハルトマンアナリ | SRP-401は、SRP-307 |
参考文献
- Barrangou, R. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315, 1709-1712 (2007).
- Makarova, K. S., Grishin, N. V., Shabalina, S. A., Wolf, Y. I., Koonin, E. V. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action. Biol. Direct. 1, 7 (2006).
- Lillestøl, R. K., Redder, P., Garrett, R. A., Brugger, K. A putative viral defence mechanism in archaeal cells. Archaea. 2, 59-72 (2006).
- Makarova, K. S. Evolution and classification of the CRISPR-Cas systems. Nat. Rev. Microbiol. 9, 467-477 (2011).
- Brouns, S. J. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321, 960-964 (2008).
- Carte, J., Pfister, N. T., Compton, M. M., Terns, R. M., Terns, M. P. Binding and cleavage of CRISPR RNA by Cas6. RNA. 16, 2181-2188 (2010).
- Haurwitz, R. E., Jinek, M., Wiedenheft, B., Zhou, K., Doudna, J. A. Sequence- and structure-specific RNA processing by a CRISPR endonuclease. Science. 329, 1355-1358 (2010).
- Gesner, E. M., Schellenberg, M. J., Garside, E. L., George, M. M., Macmillan, A. M. Recognition and maturation of effector RNAs in a CRISPR interference pathway. Nat. Struct. Mol. Biol. 18, 688-692 (2011).
- Carte, J., Wang, R., Li, H., Terns, R. M., Terns, M. P. Cas6 is an endoribonuclease that generates guide RNAs for invader defense in prokaryotes. Genes Dev. 22, 3489-3496 (2008).
- Wiedenheft, B. Structures of the RNA-guided surveillance complex from a bacterial immune system. Nature. 477, 486-489 (2011).
- Garneau, J. E. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468, 67-71 (2010).
- Sinkunas, T. Cas3 is a single-stranded DNA nuclease and ATP-dependent helicase in the CRISPR/Cas immune system. EMBO J. 30, 1335-1342 (2011).
- Jahn, M. J., Jahn, D., Kumar, A. M., Söll, D. Mono Q chromatography permits recycling of DNA template and purification of RNA transcripts after T7 RNA polymerase reaction. Nucleic Acids Res. 19, 2786-2787 (1991).
- Sampson, J. R., Uhlenbeck, O. C. Biochemical and physical characterization of an unmodified yeast phenylalanine transfer RNA transcribed in vitro. Proc. Natl. Acad. Sci. USA. 85, 1033-1037 (1988).
- Dreher, T. W., Bujarski, J. J., Hall, T. C. Mutant viral RNAs synthesized in vitro show altered aminoacylation and replicase template activities. Nature. 311, 171-175 (1984).
- McGinness, K. E., Joyce, G. F. Substitution of ribonucleotides in the T7 RNA polymerase promoter element. J. Biol. Chem. 277, 2987-2991 (2002).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved