Method Article
Generazione Substrato per endonucleasi di CRISPR / Cas Sistemi
In questo articolo
Riepilogo
CRISPR / Cas sistemi mediata immunità adattativa in Batteri e Archaea. Molte proteine Cas si propongono di agire come agiscono sul endoribonucleases crRNA precursori di varia lunghezza. Qui si illustrano tre diversi approcci per la generazione pre-crRNA substrati per l'analisi biochimica di attività endonucleasi Cas.
Abstract
L'interazione di virus e loro ospiti procariotici plasmato l'evoluzione della vita batterica e archaeal. Procarioti sviluppato diverse strategie per evitare gli attacchi virali che includono la modifica restrizione, l'infezione abortiva e CRISPR / Cas sistemi. Questi sistemi immunitarie che si trovano in molti batteri e la maggior parte sono costituiti da Archaea c lustered r egularly i nterspaced s hort p alindromic r EPEAT (CRISPR) sequenze e un certo numero di C RISPR come sociated (CAS) geni (Fig. 1) 1-3. Gli insiemi differenti di proteine Cas e le ripetizioni definire almeno tre principali tipi divergenti di CRISPR / Cas sistemi 4. Le proteine universali CAS1 e CAS2 sono proposti per essere coinvolti nella assunzione di DNA virale chegenererà un nuovo elemento distanziatore tra due ripetizioni al 5 'terminale di un cluster CRISPR estendentesi 5. L'intero cluster viene trascritto in un precursore-crRNA contenente tutte distanziale e sequenze ripetute e viene successivamente elaborato da un enzima del diverso Cas6 famiglia in piccole crRNAs 6-8. Questi crRNAs consistono nella sequenza spacer fiancheggiato da una 5 'terminale (8 nucleotidi) e 3' terminale tag derivata dalla sequenza di ripetizione 9. Una infezione ripetuta del virus può essere bloccato come il nuovo crRNA sarà diretto da un complesso proteico Cas (Cascade) al DNA virale e identificarlo come tale attraverso la complementarità base 10. Infine, per CRISPR / Cas sistemi di tipo 1, il CAS3 nucleasi distruggerà l'invasore rilevato DNA 11,12.
Questi processi definiscono CRISPR / Cas come un sistema immunitario adattativo di procarioti e aperto un affascinante campo di ricerca per lo studio delle proteine coinvolte Cas.La funzione di molte proteine Cas è ancora sfuggente e le cause per la diversità apparente dei CRISPR / Cas sistemi restano da illuminare. Attività potenziali della maggior parte delle proteine Cas sono stati previsti attraverso dettagliate analisi computazionali. Una frazione importante di proteine Cas sono mostrate o proposti per funzionare come endonucleasi 4.
Qui, presentiamo metodi per generare crRNAs e precursori-cRNA per lo studio di endoribonucleases Cas. Saggi di endonucleasi diversi richiedono sequenze ripetute sia brevi che possono direttamente essere sintetizzati come oligonucleotidi di RNA o sequenze più crRNA e pre-crRNA che vengono generati tramite in vitro T7 RNA polimerasi ballottaggio trascrizione. Questa metodologia permette l'incorporazione di nucleotidi radioattivi per la generazione di substrati endonucleasi internamente etichettati e la creazione di crRNAs sintetici o mutante. Cas6 attività endonucleasica è utilizzato per maturare pre-crRNAs in crRNAs con 5'-hydroxyl e 2 ', 3'-ciclici termini fosfato.
Protocollo
1. Generazione di lunga pre-crRNA substrati mediante PCR
- Disegno primer PCR mirati alle regioni spaziatrici di un cluster CRISPR. Aggiungere l'RNA polimerasi T7 (T7RNAP) sequenza promotore (5 '-taatacgactcactata-3') per il primer forward e siti di restrizione per il clonaggio del prodotto di PCR in un vettore di entrambi i primers (es BamHI e Hind III per pUC19, Fig. 2A) .
Nota: Il T7RNAP richiede un residuo guanidina per iniziazione della trascrizione. - Amplifica il vostro pre-crRNA sequenza di interesse da DNA genomico mediante PCR.
- Separare i prodotti di PCR mediante elettroforesi su gel di agarosio e gel estrarre la banda desiderata. Digerire il prodotto di PCR con gli enzimi di restrizione per creare estremità coesive (ad es BamHI e HindIII, Fig. 2A). Purificare il prodotto di PCR con un kit di purificazione PCR per eliminare clivaggio sottoprodotti.
- Impostare una reazione di ligazione che contiene DNA ligasi T4, T4 DNA ligasi e tamponeun rapporto molare di 3:1 del prodotto spaccati PCR e il vettore lineare pUC defosforilato con corrispondenti estremità appiccicose. Incubare la miscela a 16 ° C per una notte. Trasforma la miscela di ligazione in cellule di Escherichia coli competenti DH5a di protocolli standard e utilizzare il blu di screening per identificare bianco legatura successo.
- Isolare plasmidi da colonie bianche utilizzando un kit di preparazione di plasmidi. Identificare cloni positivi mediante sequenziamento plasmide. In alternativa, colonia PCR potrebbe essere utilizzata per lo screening.
2. Generazione di pre-intermedio crRNA substrati mediante ricottura di oligonucleotidi di DNA
- Progettare avanti e indietro oligonucleotidi con il desiderato CRISPR ripetizione / distanziale sequenza. Gli oligonucleotidi contengono la sequenza di un promotore T7 RNAP nonché siti di restrizione BamHI terminali (ad esempio e Hind III per pUC19). Terminate le oligonucleotidi per garantire che forma appiccicoso dopo ricottura estremità (vedi fig. 2B ).
- 5'-fosforilare 1 nmol di ciascun oligonucleotide in una reazione separata 20μl contenente 5 ml di T4 polinucleotide chinasi (PNK), 2 microlitri di tampone di T4 PNK 10x, 2 microlitri ATP (10 mM). Incubare ciascun campione per 1 ora a 37 ° C.
- Ibridare due oligonucleotidi fosforilati. Combinare 1ml del fosforilata miscela avanti oligo (da 2,2.), 1 ml di miscela fosforilata inversa oligo (da 2,2.), 1 ml di tampone di T4 DNA ligasi 10x in una reazione di 10 microlitri. Incubare i campioni per 5 min a 95 ° C su una piastra riscaldante o in acqua bollente, spegnere la fonte di calore e lasciare raffreddare la miscela a temperatura ambiente (~ 2-3 ore).
Nota: In questa fase critica, il processo di raffreddamento lento favorisce l'appaiamento dei due oligonucleotidi rispetto alla formazione di strutture all'interno di ogni singolo oligonucleotide. - Legare 4 microlitri della miscela di ibridazione, 1ml di digerito e defosforilato vettore pUC (0,1 mg /microlitri) con DNA ligasi T4, T4 DNA ligasi tampone 10x e 10 mM ATP in una miscela di ligazione 20 microlitri. Incubare il campione a 16 ° C per una notte.
- Trasformare i plasmidi competenti legato in cellule di Escherichia coli DH5a da protocolli standard e di utilizzare lo screening blu bianco. Plasmidi isolare e identificare cloni positivi mediante digestione (per lo screening per inserti della dimensione desiderata) e successivo sequenziamento plasmide.
3. Generazione di breve Cas RNA substrati tramite sintesi personalizzata oligonucleotidi di RNA
Progettazione breve Cas RNA substrati (ad esempio, sequenze ripetute singole, fig. 2C) e utilizzare servizi personalizzati RNA di sintesi di oligonucleotidi.
Nota: L'inclusione di un deossiribonucleotide in una posizione specificata di un oligonucleotide RNA (Fig. 2C) può essere utilizzata per individuare il sito di scissione RNA.
4. In vitro T7 RNA polimerasi della trascrizione
- Isolare plasmidi con il costrutto progettato (da 1.9. O 2.7.) Utilizzando un kit di purificazione plasmide maxiprep.
- Linearizzare il plasmide con l'enzima di restrizione che fende valle del frammento clonato (es. HindIII). Garantire la completa digestione.
Nota: Se un divergente 3 definito 'terminale del trascritto di RNA è desiderato, il costrutto progettato dovrebbe contenere un sito aggiuntivo restrizione specifica per trascrizione "run-off" a monte della sequenza HindIII. - Purificare il plasmide linearizzato con fenolo: cloroformio (1:1) estrazione e precipitazione con etanolo. Recuperare gli acidi nucleici dal risospendere pellet in acqua DEPC trattata sterile.
- Impostare un in vitro T7 RNAP scappare miscela di trascrizione che contiene 40 mM Hepes / KOH (pH 8), 22 mM MgCl2, 5 mM DTT, 1 spermidina mm, 4 mm di ogni nucleoside trifosfato (ATP, CTP, GTP, UTP ), 40-100 ug / ml di digeritoplasmide e 0,1 mg / ml in acqua T7 RNAP trattata DEPC. Incubare per 3 ore a 37 ° C.
- Analizzare i trascritti di RNA ottenuti su una denaturazione urea 8 M gel di poliacrilammide al 12% (Fig. 3A). I trascritti di RNA può essere purificato tramite anionico Mono Q cromatografia a scambio 13 e recuperato per precipitazione in etanolo delle frazioni di RNA e risospensione del pellet in acqua trattata con DEPC sterile. Per uso futuro, memorizzare l'RNA a -80 ° C.
5. Cas6 endonucleasi test
- Impostare un 20 pl in vitro T7 RNAP scappare miscela di trascrizione (vedi 4.4) che contiene una quantità ridotta di 2 mM ATP ed è integrata da 2,5 microlitri α-[32 P]-ATP (10 mCi / ml, 5000 Ci / mmol). Purificare prodotti di reazione mediante estrazione da un gel denaturante gel urea 8 M 12% di poliacrilammide. Visualizza bande mediante autoradiografia.
- Produrre e purificare le proteine ricombinanti desiderate Cas. In questo esempio, CAS6 da Clostridium thermocellum stato purificato tramite precipitazione calore e Ni-NTA cromatografia.
- Impostare una reazione saggio endonucleasi (ad esempio per Clostridium thermocellum Cas6, la miscela di reazione contenente 20 mM di Hepes (KOH pH8), 250 mM KCl, 2 mM MgCl2, 1 mM DTT, 12.000 cpm RNA substrato e 1 pM enzima ed è stato incubato a 37 ° C per 30 min).
- Carico 5 pl di miscela di reazione (+ 10 microlitri tampone contenente RNA carico 95% formammide) su un gel di poliacrilammide urea 8 M 12%. Visualizzare i prodotti di taglio dopo elettroforesi mediante autoradiografia.
6. Risultati rappresentativi
Un esempio di RNA substrati per l'analisi dell'attività endonucleasi Cas è mostrata in Figura 3A. Un'aliquota di 5 microlitri di una analitica 100 pl in reazione di trascrizione in vitro sono stati caricati. Notare che l'efficienza della produzione di RNA varia tra diversi costrutti. Alcuni fattori di tcappello sono stati osservati per influenzare la quantità di RNA ottenuti sono (i) la sequenza iniziale dopo il 1 G richiesto per l'inizio della trascrizione, (ii) la possibilità di formazione di RNA struttura durante la trascrizione e (iii) la scelta del sito di restrizione per la generazione del run-off posizione di scissione.
L'indagine di attività endonucleasica RNA richiede sia altamente purificata ricombinante proteine Cas (Fig. 3B) e adeguati controlli negativi. Idealmente, questo campione di controllo negativo si differenzia il meno possibile dalla reazione indagato endonucleasi Cas. Ciò può essere ottenuto mediante incubazione del RNA con tampone di reazione e lisato cellulare senza Cas espressione (e seguendo la procedura di purificazione identica). Un controllo ideale negativo è l'aggiunta di un deossiribonucleotide nel sito di clivaggio proposto. In figura 3C, la scissione di un 5 'terminale sequenza di ripetizione etichettata viene mostrato per Clostridium thermocellum Cas6. Sotto condizioni identiche, questa ripetizione non è un substrato più quando un deossiribonucleotide è introdotto nella posizione -9. Questo metodo fornisce anche informazioni sul sito di taglio. Infine, una lunga internamente pre-etichettato crRNA è tagliata da Cas6 e due frammenti di scissione sono osservati.
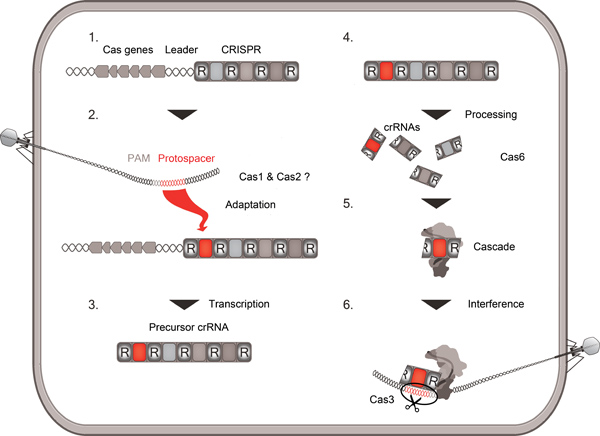
Figura 1. Schema di CRISPR / Cas attività. La rassegna segue l'inserimento di una sequenza di DNA virale (protospacer) nel cluster CRISPR (adattamento), la trascrizione e la lavorazione della matrice CRISPR in crRNAs piccoli da una endonucleasi Cas6, l'assorbimento di crRNAs nel complesso Cascade e l'interferenza di un ripetuto attacco virale basato sulla complementarità tra crRNA e protospacer. Motivi Protospacer adiacenti (PAM) sequenze protospacer marchio virali.
igura 2 "src =" / files/ftp_upload/4277/4277fig2.jpg "/>
Figura 2. Generazione di RNA substrati per proteine Cas. Lo schema mostra il flusso di lavoro per la generazione di (A) a lungo pre-crRNA substrati, (B) intermedio pre-crRNA substrati e (C) breve Cas RNA substrati per la pre-produzione crRNA. Sequenze di esempio sono presentati per la matrice CRISPR di Clostridium thermocellum. Clicca qui per ingrandire la figura .

Figura 3. Cas6 dosaggio endonucleasi. A.) toluidina poliacrilammide gel blu macchiato di una custom-designed oligonucleotide RNA (250 pmol) e due in vitro di RNA trascritti (5 pl di una tipica reazione di 100 pl). B.) SDS-PAGE gel di un preparato Cas6 (80 pmol) da Clostridium thermocellum dopo caloreprecipitazione a 50 ° C per 1 ora e Ni-NTA cromatografia. C.) il rilevamento di endonucleolitico Cas6 attività per 5 'terminale sequenze ripetute in etichetta e alle pre-crRNA trascritti in vitro. L'introduzione di un dNTP in posizione -9 abolisce Cas6 clivaggio per un breve Cas RNA substrato (S, fig. 2C). A 5 'terminale 8 tag nt è generata anche per la maturazione crRNA da tempo pre-crRNA substrati (L, fig. 2A). Le bande sono stati separati su un gel denaturante 8 M urea poliacrilammide al 12% e visualizzati mediante autoradiografia.
Discussione
I metodi presentati consentono la generazione di Cas substrati endonucleasi di forcelle di dimensioni diverse e con vari libertà nel design sequenza. L'approccio più straight-forward per la generazione di oligonucleotidi sintetici substrati di RNA è limitata a brevi disegni RNA causa dei costi crescenti e limitazioni tecniche nella creazione di più oligomeri RNA. Mentre riuscita sintesi di RNA è stato riportato per non modificati oligomeri RNA lunghezza di oltre 100 nucleotidi, la massima pratica ed economica per sintesi su RNA giace sotto 40 nucleotidi. Tuttavia, ogni data sequenza può essere sintetizzato e l'introduzione mirata di nucleotidi modificati (ad es desossiribonucleotidi) può essere utilizzata per analizzare siti di taglio in dettaglio. Più pre-cRNA deve essere generato tramite trascrizione in vitro.
La ricottura di oligonucleotidi che contengono un promotore T7 RNA polimerasi permette la produzione di lunghezza intermedia pre-CRRNCome. La lunghezza massima di routine ed economicamente oligonucleotidi di DNA sintetizzato è appena sopra 150 nucleotidi e rappresenta il massimo di sintesi pre-crRNAs tramite questo metodo. L'assemblaggio di diverse coppie di DNA ricotti oligonucleotidiche che formano estremità appiccicose con l'altro può estendere questa lunghezza massima ma richiede crescenti sfide nella clonazione del costrutto. Il vantaggio principale di questo metodo è la capacità di generare pre-crRNA costrutti (e quindi Cas endonucleasi substrati) con qualsiasi sequenza desiderata. Questo consente di testare i disegni crRNA sintetici.
Infine, più pre-crRNAs possono essere ottenuti da PCR amplificati di elementi interi CRISPR genomiche o in frazioni. Modifica del modello in vitro trascrizione plasmide può essere introdotto tramite mutagenesi sito-diretta per la generazione di pre-crRNA varianti. Questi costrutti possono essere utilizzati per analizzare il pattern globale cleavage endonucleolitica all'interno di un interopre-crRNA.
Il lavoro pionieristico per la produzione di RNA non modificato tramite polimerasi T7 RNA trascrizione in vitro è stata basata su RNA di trasferimento 14 e bromo mosaic virus RNA 15. Il consenso promotore T7 RNA polimerasi è costituito da un dominio di riconoscimento (-17 attraverso -5) e un dominio di iniziazione (-4 attraverso +6) con inizio della trascrizione in una guanosina essenziale +1 16. A valle di posizioni 1 può essere variata che consente la trascrizione di qualsiasi sequenza desiderata RNA. I metodi presentati per generare in vitro deflusso modelli trascrizione consentire sintesi in vitro di completo pre-crRNAs che corrispondono CRISPR regioni genomiche o sintetici pre-crRNA varianti. Le trascrizioni sono generati con un 5'-trifosfato terminale presente a meno che la reazione di trascrizione è innescato con le GMP per ottenere 5 'monofosfato termini 14. Tali termini sono necessarie se le trascrizioni devono essere etichettaticon T4 polinucleotide chinasi e γ-[32 P]-ATP. Attività di scissione Cas6 e Cas6 enzimi simili genera crRNA contenenti 5'-idrossile e 2 ', 3'-ciclici termini fosfato. Questi RNA possono poi essere analizzate per il riconoscimento da parte del complesso Cascade.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Gli autori desiderano ringraziare Jeanette Schermuly per la preparazione di ricombinante T7 RNA polimerasi e Ebelt Norman per l'assistenza nella preparazione di Figura 1. Questo lavoro è stato finanziato con fondi della Deutsche Forschungsgemeinschaft (DFG FOR1680) e del Max Planck Society.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti (opzionale) |
| Antartico Fosfatasi | NEB | M0289L | |
| trifosfati in grado di trascrizione in vitro ultrapuri Nucleotide | Bioscience Jena | NU-1010 - NU-1013 | |
| Cromatografia liquida, 100 ktapurifier | GE Healthcare | ||
| DNA oligonucleotidi | MWG Operon | ||
| RNA oligonucleotidi | MWG Operon | ||
| Gel Extraction Kit | QIAGEN | 28704 | |
| PCR Purification Kit | QIAGEN | 28104 | |
| Spin Miniprep Kit | QIAGEN | 27106 | |
| Maxi Kit plasmide | QIAGEN | 12163 | |
| BamHI | NEB | R0136L | |
| HindIII | NEB | R0104L | |
| DNA ligasi T4 | NEB | M0202S | |
| Polinucleotide Kinase T4 | Ambion | AM2310 | |
| T7 RNA polimerasi | propria preparazione | ||
| Soluzione Mix deossinucleotidi | NEB | N0447L | |
| Mini-PROTEAN Tetra elettroforesi sistema | Bio-Rad | 165-8001 | |
| Tempesta 840 Scanner | GE Healthcare | 163723 | |
| Deposito Phosphorscreen | Dinamica Molecolare | 63-0034-81 | |
| Mono Q 5/50 GL | GE Healthcare | 17-5166-01 | |
| Phusion ad alta fedeltà DNA polimerasi | NEB | M0530L | utilizzare il buffer GC |
| Blu di toluidina | Sigma | T3260 | |
| pUC19 | NEB | N3041S | |
| γ-[32 P]-ATP, α-[32 P]-ATP | Hartmann analitica | SRP-401, SRP-307 |
Riferimenti
- Barrangou, R. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315, 1709-1712 (2007).
- Makarova, K. S., Grishin, N. V., Shabalina, S. A., Wolf, Y. I., Koonin, E. V. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action. Biol. Direct. 1, 7 (2006).
- Lillestøl, R. K., Redder, P., Garrett, R. A., Brugger, K. A putative viral defence mechanism in archaeal cells. Archaea. 2, 59-72 (2006).
- Makarova, K. S. Evolution and classification of the CRISPR-Cas systems. Nat. Rev. Microbiol. 9, 467-477 (2011).
- Brouns, S. J. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321, 960-964 (2008).
- Carte, J., Pfister, N. T., Compton, M. M., Terns, R. M., Terns, M. P. Binding and cleavage of CRISPR RNA by Cas6. RNA. 16, 2181-2188 (2010).
- Haurwitz, R. E., Jinek, M., Wiedenheft, B., Zhou, K., Doudna, J. A. Sequence- and structure-specific RNA processing by a CRISPR endonuclease. Science. 329, 1355-1358 (2010).
- Gesner, E. M., Schellenberg, M. J., Garside, E. L., George, M. M., Macmillan, A. M. Recognition and maturation of effector RNAs in a CRISPR interference pathway. Nat. Struct. Mol. Biol. 18, 688-692 (2011).
- Carte, J., Wang, R., Li, H., Terns, R. M., Terns, M. P. Cas6 is an endoribonuclease that generates guide RNAs for invader defense in prokaryotes. Genes Dev. 22, 3489-3496 (2008).
- Wiedenheft, B. Structures of the RNA-guided surveillance complex from a bacterial immune system. Nature. 477, 486-489 (2011).
- Garneau, J. E. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468, 67-71 (2010).
- Sinkunas, T. Cas3 is a single-stranded DNA nuclease and ATP-dependent helicase in the CRISPR/Cas immune system. EMBO J. 30, 1335-1342 (2011).
- Jahn, M. J., Jahn, D., Kumar, A. M., Söll, D. Mono Q chromatography permits recycling of DNA template and purification of RNA transcripts after T7 RNA polymerase reaction. Nucleic Acids Res. 19, 2786-2787 (1991).
- Sampson, J. R., Uhlenbeck, O. C. Biochemical and physical characterization of an unmodified yeast phenylalanine transfer RNA transcribed in vitro. Proc. Natl. Acad. Sci. USA. 85, 1033-1037 (1988).
- Dreher, T. W., Bujarski, J. J., Hall, T. C. Mutant viral RNAs synthesized in vitro show altered aminoacylation and replicase template activities. Nature. 311, 171-175 (1984).
- McGinness, K. E., Joyce, G. F. Substitution of ribonucleotides in the T7 RNA polymerase promoter element. J. Biol. Chem. 277, 2987-2991 (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon