Method Article
Substrat Generation für Endonukleasen der CRISPR / Cas-Systeme
In diesem Artikel
Zusammenfassung
CRISPR / Cas-Systeme vermitteln adaptive Immunität in Bakterien und Archaea. Viele Cas Proteine vorgeschlagen, als Endoribonukleasen die auf crRNA Vorläufer von unterschiedlicher Länge handeln. Hier erläutern wir drei verschiedene Ansätze vor, crRNA Substrate für die biochemische Analyse von Cas Endonucleaseaktivität generieren.
Zusammenfassung
Das Zusammenspiel von Viren und deren prokaryotischen Wirten prägte die Entwicklung der Bakterien und Archaeen Leben. Prokaryoten entwickelt verschiedene Strategien, um Viren-Attacken, die Restriktions-Modifikations-, abortive Infektion und CRISPR / Cas-Systeme gehören zu entziehen. Diese adaptive Immunsystem in vielen Bakterien und am meisten Archaea gefunden bestehen aus c glänzendes r i egularly nterspaced s p hort alindromic r iederholen (CRISPR) Sequenzen und eine Anzahl von C RISPR als sociated (Cas) Gene (Abb. 1) 1-3. Verschiedene Sätze von Cas Proteine und Wiederholungen definieren mindestens drei große divergierenden Arten von CRISPR / Cas-Systeme 4. Die universellen Proteine und CAS1 CAS2 werden vorgeschlagen, um bei der Aufnahme der viralen DNA eingebunden werden, dasseine neue Abstandselement zwischen zwei Wiederholungen am 5'-Terminus eines erstreckenden CRISPR Cluster 5 erzeugen. Der gesamte Cluster wird in einem Vorläufer-crRNA die alle Abstandhalter und Repeat-Sequenzen transkribiert wird und anschließend durch ein Enzym der Familie vielfältigen Cas6 in kleinere crRNAs 6-8 verarbeitet. Diese bestehen aus der crRNAs Spacersequenz durch eine 5'-Terminal (8 Nucleotide) und einem 3'-terminalen-Tag aus der Repeatsequenz 9 abgeleitet flankiert. Eine wiederholte Infektion des Virus kann nun blockiert, da der neue crRNA wird von einem Proteinkomplex Cas (Cascade) an die virale DNA gerichtet werden und als solche zu kennzeichnen über Basenkomplementarität 10 unterzogen. Schließlich, für CRISPR / Cas-Typ-1-Systeme, wird die Nuklease CAS3 zerstören erkannt invader DNA 11,12.
Diese Prozesse zu definieren CRISPR / Cas als adaptive Immunsystem Prokaryoten und eröffnet einen faszinierenden Forschungsgebiet für das Studium der beteiligten Cas Proteine.Die Funktion vieler Cas Proteine ist noch schwer zu fassen und die Ursachen für die scheinbare Vielfalt der CRISPR / Cas Systeme bleiben beleuchtet werden. Mögliche Aktivitäten der meisten Cas Proteine wurden durch detaillierte rechnerische Analysen vorhergesagt. Ein großer Teil der Cas Proteine werden entweder angezeigt oder vorgeschlagen zu funktionieren, wie Restriktionsendonukleasen 4.
Hier präsentieren wir Methoden, um crRNAs und Vorläufer-cRNAs für das Studium der Cas Endoribonukleasen generieren. Endonuklease verschiedenen Assays erfordern entweder kurzen Wiederholungssequenzen, die direkt als RNA-Oligonukleotide oder länger crRNA und pre-crRNA Sequenzen, die über in vitro RNA-Polymerase T7 run-off-Transkription erzeugt werden, synthetisiert werden können. Diese Methodik erlaubt den Einbau von radioaktiven Nukleotiden zur Erzeugung von intern markiert Endonuklease Substrate und die Schaffung synthetischer oder Mutante crRNAs. Cas6 Endonucleaseaktivität wird verwendet, um pre-crRNAs in crRNAs mit 5'-hydr reifenOxyl und a 2 ', 3'-cyclisches Phosphat-Termini.
Protokoll
Ein. Generation langes Pre-crRNA Substrate mittels PCR
- PCR-Primer-Design gezielt die Spacerbereiche eines CRISPR Cluster. Hinzufügen des T7-RNA-Polymerase (T7RNAP) Promotorsequenz (5'-taatacgactcactata-3 ') mit dem Vorwärts-Primer und Restriktionsschnittstellen zur Klonierung des PCR-Produkt in einen Vektor für beide Primer (zB BamHI und HindIII für pUC19, Fig. 2A) .
Hinweis: Die T7RNAP erfordert eine Guanidinrest für die ordnungsgemäße Initiation der Transkription. - Amplifizieren pre-Sequenz von Interesse crRNA aus genomischer DNA durch PCR.
- Trennen Sie die PCR-Produkte durch Agarosegelelektrophorese und Gel extrahiert das gewünschte Band. Verdauen das PCR-Produkt mit den Restriktionsenzymen zu klebrigen Enden (zB BamHI und HindIII, Fig. 2A) zu schaffen. Entschlacken Sie Ihren PCR Produkt mit einer PCR-Reinigungskit zur Spaltung Nebenprodukte zu beseitigen.
- Einrichten einer Ligationsreaktion die T4 DNA Ligase enthält, T4-DNA-Ligase-Puffer undein 3:1 Molverhältnis des gespaltenen PCR-Produkt und der dephosphorylierte linearen pUC Vektor mit entsprechenden kohäsiven Enden. Inkubieren der Mischung bei 16 ° C über Nacht. Wandeln Sie die Ligationsgemisch in kompetente Escherichia coli DH5 Zellen durch Standard-Protokolle und verwenden blau weiß Screening erfolgreiche Ligation zu identifizieren.
- Isolieren Plasmiden aus weißen Kolonien mit einem Plasmid Vorbereitung Kit. Identifizieren positive Klone durch Plasmid-Sequenzierung. Alternativ könnte Kolonie-PCR für das Screening verwendet werden.
2. Erzeugung von Zwischen-pre-crRNA Substrate durch Temperung von DNA-Oligonukleotiden
- Gestalten Sie vorwärts und rückwärts Oligonukleotide mit der gewünschten CRISPR repeat / Spacer-Sequenz. Die Oligonukleotide enthalten die Sequenz eines T7 RNAP Promotor sowie Terminal Restriktionsstellen (zB BamHI und Hind III für pUC19). Beenden Sie die Oligonukleotide, dass sticky ends Form nach dem Glühen zu gewährleisten (siehe Abb. 2B ).
- 5'-phosphorylieren 1 nmol jedes Oligonukleotids in einer separaten Reaktion mit 20 ul 5 ul T4-Polynukleotid-Kinase (PNK), 2 ul T4 PNK 10X Puffer, 2 ul ATP (10 mM). Inkubieren jeder Probe für 1 Stunde bei 37 ° C.
- Hybridisieren die beiden phosphorylierten Oligonukleotiden. Kombinieren Sie 1 ul des phosphorylierten Vorwärts Oligo-Mischung (vom 2.2.), 1 ul des phosphorylierten umgekehrter Oligo-Mischung (vom 2.2.), 1 ul T4-DNA-Ligase 10x Puffer in einem 10 ul Reaktion. Inkubieren Sie die Proben für 5 min bei 95 ° C auf einem Heizblock oder in kochendes Wasser, schalten Sie die Wärmequelle und lassen Sie die Mischung abkühlen auf Raumtemperatur (~ 2-3 Stunden).
Anmerkung: In diesem kritischen Schritt begünstigt die langsame Abkühlung das Annealing der beiden Oligonukleotide gegenüber der Bildung von Strukturen innerhalb jeder einzelnen Oligonucleotid. - Ligieren 4 ul der Hybridisierung mix, 1 ul verdaut und dephosphoryliert pUC-Vektor (0,1 ug /ul) mit T4 DNA-Ligase, T4-DNA-Ligase 10x Puffer und 10 mM ATP in einem 20 ul Ligationsgemisch. Inkubieren Sie die Probe bei 16 ° C über Nacht.
- Verwandeln Sie die ligierten Plasmide in kompetente Escherichia coli DH5 Zellen durch Standard-Protokolle und nutzen blau weiß-Screening. Isolieren und zu identifizieren Plasmide positive Klone durch Verdauung (für Einsätze mit der gewünschten Größe zu screenen) und anschließende Sequenzierung Plasmid.
3. Erzeugung von kurzen Cas RNA-Substrate über benutzerdefinierte RNA-Oligonukleotid-Synthese
Design kurzen Cas RNA-Substrate (zB Single Repeat-Sequenzen, Fig. 2C) und nutzen eigene RNA-Oligonukleotid-Synthese Einrichtungen.
Anmerkung: Die Aufnahme eines Desoxyribonucleotids an einer festgelegten Position eines RNA-Oligonukleotid (2C) verwendet werden, um den Ort der RNA-Spaltung auszumachen ist.
4. In vitro T7 RNA-Polymerase-Transkription
- Isolieren Plasmide mit Ihrem konzipiert Konstrukt (ab 1.9. Oder 2.7.) Mit einem Maxipräp Plasmid Purification Kit.
- Linearisierung des Plasmids mit dem Restriktionsenzym, das stromabwärts des klonierten Fragments (z. B. HindIII) spaltet. Eine vollständige Verdauung.
Anmerkung: Wenn ein divergenter definierten 3'-Terminus des RNA-Transkript wird gewünscht, die ausgelegt Konstrukt sollte eine zusätzliche spezifische Restriktionsstelle für "Run-off" Transkription stromaufwärts der HindIII-Sequenz enthalten. - Reinigen das linearisierte Plasmid durch Phenol: Chloroform (1:1) und Ethanolfällung. Wiederherstellen der Nukleinsäuren durch Resuspendieren des Pellets in DEPC behandeltem steriles Wasser.
- Richten Sie ein in vitro T7 RNAP ablaufen Transkription Mischung, die 40 mM Hepes / KOH (pH 8), 22 mM MgCl 2, 5 mM Dithiothreitol, 1 mM Spermidin, 4 mM jedes Nukleosid-Triphosphat (ATP, CTP, GTP, UTP enthält ), 40-100 pg / ml verdautPlasmid und 0,1 mg / ml T7 RNAP in DEPC behandeltem Wasser. Einer Inkubationszeit von 3 Stunden bei 37 ° C.
- Analysieren der erhaltenen RNA-Transkripte auf einem denaturierenden 8 M Harnstoff 12% Polyacrylamid-Gel (Abb. 3A). Die RNA-Transkripte können über Mono Q Anionenaustauschchromatographie 13 gereinigt und durch Ethanolfällung gewonnen der RNA-Fraktionen und Resuspension des Pellets in DEPC behandeltem steriles Wasser. Für die zukünftige Verwendung speichern die RNA bei -80 ° C.
5. Cas6 Endonuklease Assay
- Einrichten eines 20 ul in vitro T7 RNAP ablaufen Transkription Mischung (siehe 4.4), die einen reduzierten Betrag von 2 mM ATP enthält, und wird ergänzt 2,5 ul α-[32 P]-ATP (10 mCi / ml, 5000 Ci / mmol). Läutern Reaktionsprodukte über Gel Extraktion aus einem denaturierenden 8 M Harnstoff 12% Polyacrylamidgel. Visualisieren Banden durch Autoradiographie.
- Herstellen und reinigen Sie die gewünschten rekombinanten Cas Proteine. In diesem Beispiel Cas6 aus Clostridium thermocellum wurde über Hitzefällung und Ni-NTA-Chromatographie gereinigt.
- Einrichten einer Endonuklease Assayreaktion (zB für Clostridium thermocellum Cas6 enthält das Reaktionsgemisch 20 mM Hepes (KOH pH 8), 250 mM KCl, 2 mM MgCl 2, 1 mM DTT, 12.000 cpm RNA Substrat und Enzym und 1 pM wurde bei inkubiert 37 ° C für 30 min).
- Beladung 5 ul der Reaktionsmischung (10 ul + RNA Ladepuffer, der 95% Formamid) auf einer 8 M Harnstoff 12% Polyacrylamidgel. Visualisieren Sie die Spaltprodukte nach der Elektrophorese durch Autoradiographie.
6. Repräsentative Ergebnisse
Ein Beispiel für RNA-Substrate für die Analyse von Cas Endonucleaseaktivität ist in 3A gezeigt. Ein Aliquot von 5 ul einer analytischen 100 ul in vitro Transkription geladen wurden. Bitte beachten Sie, dass die Effizienz der RNA-Produktion zwischen verschiedenen Konstrukten variiert. Einige Faktoren, tHut wurde beobachtet, dass die Menge der erhaltenen RNA sind (i) der anfänglichen Folge im Anschluss an die ein G für Transkriptionsinitiation erforderlich, (ii) die Möglichkeit der RNA Strukturbildung während der Transkription und (iii) die Auswahl der Restriktionsstelle für die Erzeugung beeinflussen der Run-off Spaltstelle.
Die Untersuchung von RNA Endonucleaseaktivität erfordert sowohl hochgereinigten rekombinanten Proteinen Cas (3B) und die ordnungsgemäße Negativkontrollen. Idealerweise unterscheidet sich diese negative Kontrollprobe so wenig wie möglich von der untersuchten Cas Endonuklease Reaktion. Dies kann durch Inkubation der RNA mit Reaktionspuffer und Zell-Lysat ohne Cas Ausdruck (und nach dem identischen Reinigungsprozedur) erreicht werden. Eine ideale Negativkontrolle ist der Zusatz eines Desoxyribonucleotid in der vorgeschlagenen Spaltstelle. In 3C wird die Spaltung eines 5'-terminalen Repeat-Sequenz markiert für Clostridium thermocellum Cas6 gezeigt. Unter identischen Bedingungen, ist diese Wiederholung kein Substrat mehr, wenn ein Desoxyribonukleotid an Position -9 eingeführt. Diese Methode liefert auch Informationen über die Spaltstelle. Schließlich wird eine lange intern markiert pre-crRNA durch Cas6 gespalten und zwei Spaltungsfragmente beobachtet.
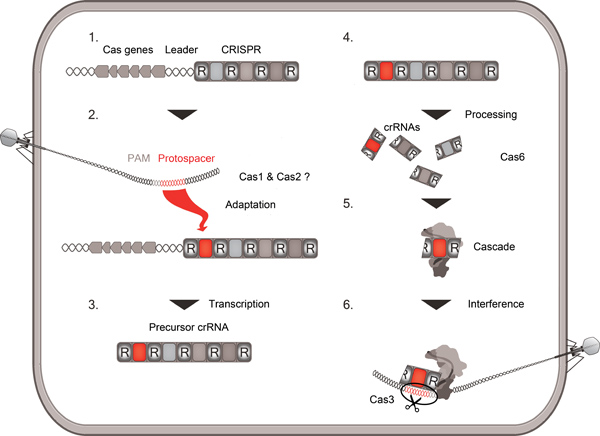
Abbildung 1. Schematische Übersicht CRISPR / Cas Aktivität. Die Übersicht folgt das Einführen eines viralen DNA-Sequenz (protospacer) in die CRISPR-Cluster (Adaption) die Transkription und die Verarbeitung des CRISPR-Array in kleine crRNAs durch eine Cas6 Endonuklease, die Verbreitung von crRNAs in die Kaskade komplexer und die Interferenz ein wiederholten viralen Angriff auf die Komplementarität zwischen crRNA und protospacer basiert. Protospacer angrenzenden Motive (PAM) mark viralen protospacer Sequenzen.
BBILDUNG 2 "src =" / files/ftp_upload/4277/4277fig2.jpg "/>
Abbildung 2. Erzeugung von RNA-Substrate für Cas Proteine. Das Schema zeigt den Workflow zur Erzeugung von (A) lange Vor-crRNA Substraten, (B) Zwischen pre-crRNA Substrate und (C) kurz Cas RNA-Substrate für Pre-crRNA Produktion. Beispiel Sequenzen für die CRISPR Reihe von Clostridium thermocellum vorgestellt. Klicken Sie hier für eine größere Abbildung zu sehen .

Abbildung 3. Cas6 Endonuklease Assay. A.) Toluidinblau gefärbten Polyacrylamidgel einer maßgeschneiderten RNA-Oligonukleotid (250 pmol) und zwei in vitro RNA-Transkripte (5 ul einer typischen 100 ul-Reaktion). B.) SDS-PAGE-Gel von einem Cas6 Zubereitung (80 pmol) aus Clostridium thermocellum nach ThermischerFällung bei 50 ° C für 1 Stunde und Ni-NTA-Chromatographie. C) Detektion von endonukleolytische Cas6 Aktivität für 5 'Anschluss mit der Bezeichnung Repeatsequenzen und Pre-crRNA in vitro Transkripte. Die Einführung eines dNTP an Position -9 abschafft Cas6 Spaltung für eine kurze Cas RNA-Substrat (S, Abb. 2C). A 5 'Klemme 8 nt tag ist auch für crRNA Reifung von langen pre-crRNA Substrate (L, Fig. 2A) erzeugt. Die Bänder wurden auf einem denaturierenden 8 M Harnstoff 12% Polyacrylamid-Gel aufgetrennt und durch Autoradiographie visualisiert.
Diskussion
Die vorgestellten Verfahren ermöglichen die Erzeugung von Cas Endonuklease Substraten unterschiedlicher Größenbereiche und mit unterschiedlicher Sequenz in Freiheit Design. Die geradlinigen Ansatz zur Erzeugung von synthetischen RNA-Oligonukleotid Substrate auf kurze RNA Designs aufgrund steigenden Kosten und technischer Beschränkungen bei der Schaffung längere RNA-Oligomeren begrenzt. Während erfolgreiche RNA-Synthese wurde für unmodifizierte RNA-Oligomere von über 100 Nukleotiden Länge berichtet, liegt die praktische und wirtschaftliche Maximum für benutzerdefinierte RNA-Synthese unter 40 Nukleotiden. Jedoch kann jede gegebene Sequenz synthetisiert und die gezielte Einführung von modifizierten Nukleotiden (zB Desoxyribonukleotiden) kann verwendet werden, um Spaltstellen im Detail analysiert werden. Mehr pre-cRNAs sollte über in vitro Transkription erzeugt werden.
Das Glühen von Oligonucleotiden, die ein T7-RNA-Polymerase-Promotor enthalten können zur Herstellung von Pre-Zwischenlänge CRRNAs. Die maximale Länge der routinemäßig und wirtschaftlich synthetisierte DNA-Oligonukleotiden ist nur oberhalb von 150 Nukleotiden und stellt die maximale synthetischer pre-crRNAs mit dieser Methode. Die Anordnung von mehreren DNA geglühten Oligonukleotidpaare, die klebrigen Enden miteinander bilden erstrecken kann diese maximale Länge erfordert aber immer größeren Schwierigkeiten, die Klonierung des Konstrukts. Der Hauptvorteil dieses Verfahrens ist die Möglichkeit vor, eine crRNA Konstrukte (und daher Cas Endonuklease Substrate) mit einer beliebigen gewünschten Sequenz zu erzeugen. Dies ermöglicht das Testen von synthetischen crRNA Designs.
Schließlich können größere Vor-crRNAs aus PCR-Amplifikate der gesamten genomischen CRISPR Elemente oder Fraktionen davon erhalten werden. Änderungen an den in vitro Transkriptions-Vorlage Plasmid kann über ortsspezifische Mutagenese zur Erzeugung von Vor-crRNA Varianten eingeführt werden. Diese Konstrukte können verwendet werden, um die globale endonukleolytische Spaltung Muster innerhalb eines gesamten analysierenPre-crRNA.
Die Pionierarbeit für die Herstellung von unmodifizierten RNA mittels T7-RNA-Polymerase in vitro-Transkription wurde am Transfer-RNAs 14 und Trespenmosaik-Virus-RNA 15 basiert. Die Konsensus-T7-RNA-Polymerase-Promotor aus einem Erkennungs-Domäne (-17 bis -5) und einer Initiationsstelle Domäne (-4 bis 6) mit Transkriptionsinitiationsregion in einer entscheidenden Guanosin +1 16. Positionen stromabwärts 1 kann variiert werden, die die Transkription von fast jeder gewünschten RNA-Sequenz ist. Die vorgestellten Verfahren zur Erzeugung von in-vitro-run-off-Transkription Vorlagen ermöglichen zur in vitro Synthese von vollständigen Vor-crRNAs die genomische Regionen CRISPR oder synthetische Varianten vor crRNA übereinstimmen. Die Transkripte sind mit einem 5'-Triphosphat-terminalen vorliegenden erzeugt, wenn der Transkriptionsreaktion mit GMP grundiert wird auf 5 'Termini Monophosphat 14 zu erhalten. Solche Termini sind erforderlich, wenn die Transkripte beschriftet werdenmit T4 Polynukleotidkinase und γ-[32 P]-ATP. Spaltungsaktivität Cas6 und Cas6-ähnliche Enzyme erzeugt crRNA, die 5'-Hydroxy-und a 2 ', 3'-cyclisches Phosphat-Enden enthalten. Diese RNAs können dann für die Anerkennung durch die Cascade komplexen analysiert werden.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Autoren bedanken sich bei Jeanette Schermuly danken für die Herstellung von rekombinantem T7-RNA-Polymerase und Norman Ebelt für die Unterstützung bei der Vorbereitung Abbildung 1. Diese Arbeit wurde vom Fonds der Deutschen Forschungsgemeinschaft (DFG FOR1680) und dem Max-Planck-Gesellschaft finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare (optional) |
| Antarctic Phosphatase | NEB | M0289L | |
| in vitro Transcription Grade Ultrapure Nukleotidtriphosphate | Jena Bioscience | NU-1010 - NU-1013 | |
| Flüssigchromatographie, ktapurifier 100 | GE Healthcare | ||
| DNA-Oligonukleotide | MWG Operon | ||
| RNA-Oligonukleotiden | MWG Operon | ||
| Gel Extraction Kit | QIAGEN | 28704 | |
| PCR Purification Kit | QIAGEN | 28104 | |
| Spin Miniprep Kit | QIAGEN | 27106 | |
| Plasmid Maxi Kit | QIAGEN | 12163 | |
| BamHI | NEB | R0136L | |
| HindIII | NEB | R0104L | |
| T4-DNA-Ligase | NEB | M0202S | |
| T4 Polynukleotidkinase | Ambion | AM2310 | |
| T7-RNA-Polymerase | eigene Vorbereitung | ||
| Desoxynukleotid Lösung Mix | NEB | N0447L | |
| Mini-PROTEAN Tetra Elektrophorese-System | Bio-Rad | 165-8001 | |
| Sturm 840 Scanner | GE Healthcare | 163723 | |
| Lagerung Phosphorscreen | Molecular Dynamics | 63-0034-81 | |
| Mono Q 5/50 GL | GE Healthcare | 17-5166-01 | |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | verwenden Sie den GC-Puffer |
| Toluidinblau | Sigma | T3260 | |
| pUC19 | NEB | N3041S | |
| γ-[32 P]-ATP, α-[32 P]-ATP | Hartmann Analytic | SRP-401, SRP-307 |
Referenzen
- Barrangou, R. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315, 1709-1712 (2007).
- Makarova, K. S., Grishin, N. V., Shabalina, S. A., Wolf, Y. I., Koonin, E. V. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action. Biol. Direct. 1, 7 (2006).
- Lillestøl, R. K., Redder, P., Garrett, R. A., Brugger, K. A putative viral defence mechanism in archaeal cells. Archaea. 2, 59-72 (2006).
- Makarova, K. S. Evolution and classification of the CRISPR-Cas systems. Nat. Rev. Microbiol. 9, 467-477 (2011).
- Brouns, S. J. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321, 960-964 (2008).
- Carte, J., Pfister, N. T., Compton, M. M., Terns, R. M., Terns, M. P. Binding and cleavage of CRISPR RNA by Cas6. RNA. 16, 2181-2188 (2010).
- Haurwitz, R. E., Jinek, M., Wiedenheft, B., Zhou, K., Doudna, J. A. Sequence- and structure-specific RNA processing by a CRISPR endonuclease. Science. 329, 1355-1358 (2010).
- Gesner, E. M., Schellenberg, M. J., Garside, E. L., George, M. M., Macmillan, A. M. Recognition and maturation of effector RNAs in a CRISPR interference pathway. Nat. Struct. Mol. Biol. 18, 688-692 (2011).
- Carte, J., Wang, R., Li, H., Terns, R. M., Terns, M. P. Cas6 is an endoribonuclease that generates guide RNAs for invader defense in prokaryotes. Genes Dev. 22, 3489-3496 (2008).
- Wiedenheft, B. Structures of the RNA-guided surveillance complex from a bacterial immune system. Nature. 477, 486-489 (2011).
- Garneau, J. E. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468, 67-71 (2010).
- Sinkunas, T. Cas3 is a single-stranded DNA nuclease and ATP-dependent helicase in the CRISPR/Cas immune system. EMBO J. 30, 1335-1342 (2011).
- Jahn, M. J., Jahn, D., Kumar, A. M., Söll, D. Mono Q chromatography permits recycling of DNA template and purification of RNA transcripts after T7 RNA polymerase reaction. Nucleic Acids Res. 19, 2786-2787 (1991).
- Sampson, J. R., Uhlenbeck, O. C. Biochemical and physical characterization of an unmodified yeast phenylalanine transfer RNA transcribed in vitro. Proc. Natl. Acad. Sci. USA. 85, 1033-1037 (1988).
- Dreher, T. W., Bujarski, J. J., Hall, T. C. Mutant viral RNAs synthesized in vitro show altered aminoacylation and replicase template activities. Nature. 311, 171-175 (1984).
- McGinness, K. E., Joyce, G. F. Substitution of ribonucleotides in the T7 RNA polymerase promoter element. J. Biol. Chem. 277, 2987-2991 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten