Method Article
生細胞中のタンパク質凝集の4Dイメージング
要約
細胞の生存率は、タンパク質のミスフォールディングのタイムリーかつ効率的な管理に依存します。包有物中のフォールディング、劣化、または隔離:ここでは、ミスフォールドタンパク質の異なる潜在的運命を可視化するための方法を説明します。我々はUbc9、折り畳みセンサーの使用方法を示してい TS、監視proteostasisと4D顕微鏡を用いた生細胞における凝集の品質管理のため。
要約
One of the key tasks of any living cell is maintaining the proper folding of newly synthesized proteins in the face of ever-changing environmental conditions and an intracellular environment that is tightly packed, sticky, and hazardous to protein stability1. The ability to dynamically balance protein production, folding and degradation demands highly-specialized quality control machinery, whose absolute necessity is observed best when it malfunctions. Diseases such as ALS, Alzheimer's, Parkinson's, and certain forms of Cystic Fibrosis have a direct link to protein folding quality control components2, and therefore future therapeutic development requires a basic understanding of underlying processes. Our experimental challenge is to understand how cells integrate damage signals and mount responses that are tailored to diverse circumstances.
The primary reason why protein misfolding represents an existential threat to the cell is the propensity of incorrectly folded proteins to aggregate, thus causing a global perturbation of the crowded and delicate intracellular folding environment1. The folding health, or "proteostasis," of the cellular proteome is maintained, even under the duress of aging, stress and oxidative damage, by the coordinated action of different mechanistic units in an elaborate quality control system3,4. A specialized machinery of molecular chaperones can bind non-native polypeptides and promote their folding into the native state1, target them for degradation by the ubiquitin-proteasome system5, or direct them to protective aggregation inclusions6-9.
In eukaryotes, the cytosolic aggregation quality control load is partitioned between two compartments8-10: the juxtanuclear quality control compartment (JUNQ) and the insoluble protein deposit (IPOD) (Figure 1 - model). Proteins that are ubiquitinated by the protein folding quality control machinery are delivered to the JUNQ, where they are processed for degradation by the proteasome. Misfolded proteins that are not ubiquitinated are diverted to the IPOD, where they are actively aggregated in a protective compartment.
Up until this point, the methodological paradigm of live-cell fluorescence microscopy has largely been to label proteins and track their locations in the cell at specific time-points and usually in two dimensions. As new technologies have begun to grant experimenters unprecedented access to the submicron scale in living cells, the dynamic architecture of the cytosol has come into view as a challenging new frontier for experimental characterization. We present a method for rapidly monitoring the 3D spatial distributions of multiple fluorescently labeled proteins in the yeast cytosol over time. 3D timelapse (4D imaging) is not merely a technical challenge; rather, it also facilitates a dramatic shift in the conceptual framework used to analyze cellular structure.
We utilize a cytosolic folding sensor protein in live yeast to visualize distinct fates for misfolded proteins in cellular aggregation quality control, using rapid 4D fluorescent imaging. The temperature sensitive mutant of the Ubc9 protein10-12 (Ubc9ts) is extremely effective both as a sensor of cellular proteostasis, and a physiological model for tracking aggregation quality control. As with most ts proteins, Ubc9ts is fully folded and functional at permissive temperatures due to active cellular chaperones. Above 30 °C, or when the cell faces misfolding stress, Ubc9ts misfolds and follows the fate of a native globular protein that has been misfolded due to mutation, heat denaturation, or oxidative damage. By fusing it to GFP or other fluorophores, it can be tracked in 3D as it forms Stress Foci, or is directed to JUNQ or IPOD.
プロトコル
1。酵母の準備
- GAL1-GFP Y68L(UBC9 ts)のカセットを有するプラスミドで酵母株を形質転換する。
- 16時間またはO / Nのために24時間、2%のラフィノースを含む合成培地で酵母を育て、背中2パーセントガラクトース含有培地に希釈200rpmで振とうしながら30℃で細胞をインキュベートします。
- 翌朝、OD 600 = 0.2にクエリひずみを薄める。文化は= 0.8から1.0のOD 600に到達するまで30℃(200 rpm)で4から6時間振とうする。
- 変更メディアイメージングの前に2%グルコース(SD)の30分を補った合成培地に、(ようにのみ、GFP-Ubc9 tsの翻訳済みと折り畳まプールを監視するために)発現を抑制する。
- 治療 - ヒートショックを37℃で20分間細胞℃のミスフォールディングを誘導する、あるいは連続的に熱ショック条件下でタンパク質凝集を追跡する顕微鏡インキュベーターインチ注 - あなたは、RT上の任意の温度で顕微鏡を使用している場合は、予熱FO(下記参照)顕微鏡や目標の体に沿って温度を平衡化するために2時間ほどR。
2。プレート\スライドの準備
- 適切な板を選択してください。主な考慮事項は、画像取得時にフォーカスを維持する能力である。以下の点は、2D時間経過または3Dイメージングのための4Dイメージングのために重要とされません。
- 複数のウェルプレートの異なるウェルの底部( すなわち、井戸は客観に対して異なる高さになります)均質でないかもしれません。これは、それが困難にかかわらず使用されているオートフォーカス技術の、XYポイントと時間ポイント間のフォーカスを維持するようになります。
- スライド材質:ガラス対プラスチック。ガラス底のスライドはよりz軸均質ウェル間であるが、より高価である。
- プレートの底部の厚さ:私たちは通常、カバースリップ底プレート指数1.5を使用していますが、インデックス1は、目的に応じても構わない。
- COV10分間のConA(0.25 mg / ml)を有する板のER底が\スライド。 ConAでは、経時的に、単一のセルに続く可能スライドに細胞を接着させるために使用されています。
- ConAをを削除し、その過剰のConAが蒸発できるようになるので、化学フードでスライドをインキュベートする。
- よくConAedに酵母サンプルのプレートを200μl(OD 600 = 0.5)。
- 細胞がプレートの表面に付着し有効にすると、15分間インキュベートする。
- 媒体を抽出し、細胞の1つの層を得るために新しい培地で3回洗浄する。注意:長い時間の経過が計画されている場合、種子は、細胞がまばらに、新しい芽が記入し、関心領域を中断しないこと。
3。顕微鏡の準備
- 我々はいくつかの非標準的な修正を加えてニコンA1Rsi共焦点顕微鏡を使用しています。酵母イメージングのために、我々は4レーザー(; 457から514 nmであり、65 mWのアルゴンイオンレーザー、561 nmの、50mWのサファイアレーザー、405 nmの、50mWのCUBEのレーザー波長は640nm、40 mWのCUBEレーザー)まで使用すると、最大を装備した4光電子増倍管フィルター。私たちの撮影のほとんどは、EGFPの緑色フィルタセットとmCherryとtdTomato用赤フィルターセットを用いて行われる。 GFPとmCherryとそこにほとんどないbleedthroughはありませんが、そのためには、多くの場合、同時スキャンを使用。しかし、ラインスキャンはまた、それが発生したときにbleedthroughを防止するために使用することができます。私たちの焦点も毎秒2月3日Z-スタック(30-50節)を有効に、zに3ミリ秒のステップを行うことができますPInanoピエゾステージ(MCL)は、装備されています。ガルバノスキャナとレゾナントスキャナー - システムはまた、スペクトル検出器、パーフェクトフォーカス(レーザーオフセット)、および2つのスキャナを持っています。
- 適切な目標を選択してください。主な検討事項:
- 水/油/空気目的:主な考慮事項は、サンプルの媒体対イメージング媒体の屈折率である。
- 油浸対物の利点:
- 油の目標は、より高い開口数(オイル改水よりも軽いため、より客観的な光子に戻って)を持つことができます。オイル目標は持つことができますNAは1.49、水の1.27と比較しています。 (ただし、これは実際には解像度に大きな違いはありません)。
- オイルの屈折率はガラスの屈折率に一致し、したがって全く光子が目標に、ガラス越しに、サンプルから行く失われることはありません。 (注 - あなたの目的とあなたのカバースリップのための適切な屈折率を有する液浸油を選んだ)。
- それが蒸発しないので、オイルは手間のかからない長い時間が経過するようになります。
- 光が水性媒体(細胞など)を通過しなければならない場合、油の目標の高NA化では有効になって解像度が高くなるほど、急速に低下します。
- 画像は通常、より球面収差を持っており、3Dの効果 "を伸ばして"。
- オイルはきたない、貼り付けられ、目標に危険。
- 水客観的利点
- セル自体は水性である、従ってzにおける少ない歪みがあるND解像度は水性試料に高深く残っています。
- クリーンかつユーザーフレンドリー。水客観的短所:客観的に連続的に水を汲み上げるために行われている特別な準備なしで長い映画のために迅速に、それに適していない蒸発する。
- エア客観的利点:1。材料は、複数のポイント取得や長いムービーの途中で/蒸発し消失しません。 2。クリーンかつユーザーフレンドリー。デメリット:低解像度、低信号感度。
- 油浸対物の利点:
- ルームやインキュベーター温度:温度変化は物質の性質に影響を与え、繊細なシステムにフォーカスを変更します。温度は、買収のためのポイントを選択する前に調整する必要があります。買収時の温度を変更すると、画像が中断され、フォーカスが失われます。私たちは、顕微鏡システムは、撮像前に所望の温度で2時間平衡化することができます。
- 補正カラー:多くの目標が有効に補正リングが付属してい目的は、与えられたカバースリップの厚さに合わせて調整することができます。我々は、点広がり関数を可視化する蛍光ビーズを使用しながら、補正環を調整することをお勧めします。これは、すべてのサンプルのための正しい設定を確実にするでしょう。
- 安定したサンプルホルダー:我々は市販されているほとんどのサンプルホルダーは、顕微鏡の最も弱い部分であることがわかる。彼らは頻繁にグラグラとストレートではありません。これは高解像度、マルチポイント4Dイメージングのための災害です。我々はまっすぐである私たち自身のサンプルホルダを設計し、ステージ上にしっかりとフィット。必要なときに彼らはまたダウンしてボルトで固定することができます。
- マルチポイント集録:私たちの撮像システムは、基本的には、レーザーベースのオートフォーカス機構であるニコン "パーフェクトフォーカス"システムが装備されている。しかし、オートフォーカスシステムは多くのことをドリフトしない場合は特に、4Dイメージングとは必ずしも必要ではありません。
- 水/油/空気目的:主な考慮事項は、サンプルの媒体対イメージング媒体の屈折率である。
4。イメージング
- システムの電源をオンにします:レーザー、ステージ、コントローラ、カメラとコンピューター側TERソフトウェア。
- ステージホルダーの上に置いてプレート、適切に固定され、安定した。
- 酵母の位置と向きを決定するために、目のポートを使用します。
- 明を使用して、形状や質感に応じて細胞の健康と生存率を評価します。
- 関連するフルオロフォア( 例えば GFPのFITCフィルター)によると落射蛍光ライトをオンにして、自分の研究の対象となる表現型を示す細胞に焦点を当てています。複数の波長で高い蛍光性である細胞が死んだので、自家かもしれません。我々は、SV40 NLSシグナル(NLS-TFP)に融合tdTomatoの蛍光色素で核を可視化する。 TFPは倍GFPのサイズであり、核の拡散限界を上回ることである、したがって、それは核マーカーとして非常にうまく機能します。また、GFPと同時に緑色(488 nm)のレーザーでTFPを励起するが、スペクトル分解能のために2つの別々の光電子増倍管に緑と赤の排出量を収集することができます。
- ノイズを最小限に抑えるため、以下の設定を調整すると過飽和:
- レーザパワー - 画像の退色と光毒性対明るさを考慮すべきである。
- ゲイン:カメラの感度に影響を与え、従って、信号対雑音比。
- ピンホール径は - 短い波長のレーザーに合わせて調整してください。ピンホールの直径は、光学部の厚さ(高さ)が結像されるかを決定します。このように、ピンホールを開くと(より多くの光子のlet)より多くの光を収集しますが、Z(少ない共焦点)で解像度が低下します。
- 便利なヒント:
- 異なる蛍光が合同波長を放出した場合、仮想フィルタの選択を可能にするスペクトル検出機能を使用します。スペクトル検出器は定期的な光電子増倍管よりも感度が低いことに留意してください。
- 異なる蛍光体は、同じレーザーによって励起されている場合は、ラインスキャン機能を使用します。
- ガルバノスキャナはもっと感度が可能となりますが、退色のための高いリスクを持っています。レゾナントスキャナは速い可能cquisition、こうして退色のリスクを低下させるが、あまり敏感である。
- 2〜16枚の画像を平均化すること、それは、信号対雑音比が向上しますが、取得が遅くなりますと、もちろん、レーザーへの試料のより多くの露出を伴う。
- の時間生物学的な問題( 例えば JUNQとiPodの形成が〜2時間かかります)での買収は、異なります。我々は、最大30時間までの時間経過を3Dでの共振スキャナで画像化された酵母を持っています。
- タイムラプスインターバル·短い間隔はより一貫性のムービーを作成しますが、退色の原因となるので、信号の損失可能性があります。
結果
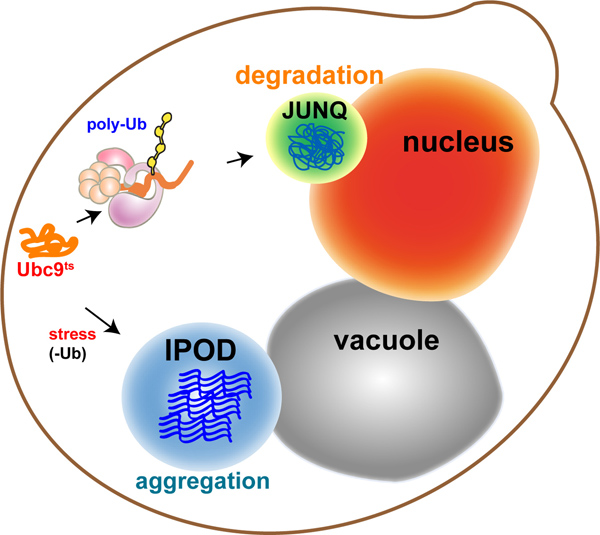
図1。 。品質管理機構が異なる機能を持つ異なるコンパートメントにミスフォールドタンパク質を指示ミスフォールドタンパク質の細胞内区画化:モデル可溶性タンパク質、分解の標的ポリユビキチン化を受け、 チュXTA-N uclear のQ uality制御コンパートメント(JUNQに送信されます)。ユビキチン化することができない不溶性のタンパク質は、それらがアクティブな集約を受ける液胞に隣接私 nsoluble PR otein D eposit(IPOD)に保護隔離のために送信されます。
es.jpgは "src =" / files/ftp_upload/50083/50083fig2.jpg "/>
図2。 GFP-Ubc9 TSとモデリング、タンパク質のミスフォールディング 。 通常の条件下では、GFP-Ubc9 TS(緑)がネイティブに折り畳まれており、核と細胞質にびまんローカライズされています。核は、NLS-TFP(赤)で標識されている。 Ubc9 TSの発現は全ての実験において、イメージングの前に2%グルコースを添加することによって遮断された。 拡大図を表示するには、ここをクリックしてください 。
- 37〜温度変化に応じ℃、GFP-Ubc9 TS(緑)がミスフォールドおよびサイトゾルストレス病巣を形成している。核は、NLS-TFP(赤)で標識されている。
- 23℃の熱ショックからの回復時には、熱的に変性させたGFP-Ubc9 TSは減少蛍光レベルによって示されるように、劣化している。
- 37℃および80 mm MG132でプロテアソーム阻害、GFP-Uへの温度変化に応じて BC9 tsは JUNQとiPod包有物にミスフォールドされ、処理されます。核は、NLS-TFP(赤)で標識されている。
- 37〜温度シフト℃、ユビキチン化阻害すると、GFP-Ubc9 tsは IPODのインクルージョンにミスフォールドされ、処理されます。核は、NLS-TFP(赤)で標識されている。ユビキチンプロテアーゼ4(Ubp4)はUbc9 tsのユビキチン化をブロックするために過剰発現している。
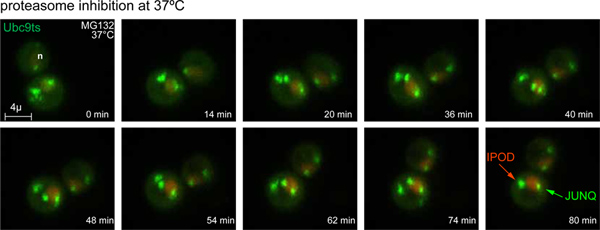
図3。 JUNQとiPod形成の時間経過図 37に温度シフト80mmのMG132で°Cおよびプロテアソーム阻害すると、GFP-Ubc9 tsのストレス病巣はJUNQとiPod包有物に加工される。核は、NLS-TFP(赤)で標識されている。 3D映像は4分間隔で取得した。 (また、 ムービー1を参照)。com/files/ftp_upload/50083/50083fig3large.jpg "ターゲット=" _blank ">拡大図を表示するには、ここをクリックしてください。
ディスカッション
生化学的プロセスについての我々の直感は、反応物と生成物のよく混合溶液をビーカーに平衡に到達することを許可されているベンチトップの実験から派生しています。このような設定では、与えられた化学種の濃度は、巨視的なボリュームへの分子のモル量の比であり、単一の番号、のように表すことができる。細胞の全集団のホモジネートからの抽出で行っウェスタンブロット、遠心分離し、分光光度計:はるかに我々はタンパク質の構造と機能について知っているのは、古典的な、バルク反応像を反映するメソッドを使用してから派生しています。
我々は倍率で細胞を見てするために使用する技術として飛躍的に向上し、それはほとんどの生化学反応が生体内で行われる条件は、古典的なベンチトップのシナリオのものにのみわずかな似ても似つかないことをこれまでより明確になります。セルの内部だけではありませんラが密集クラウディング効果が実質的に種々の反応の活性を変化させている環境を、パックされた、それはまた、よく混合の全く逆です。 in vitroおよび複雑な高分子反応の広い範囲の in vivo効率の間の頻繁な格差を占めています。
どこにも、in vivoでのフォールディング、ミスフォールディング、タンパク質の凝集においてに関する質問のようにミスリードしやすい試験管内の生化学的実験では古典から生じる直感はありません。バルク反応におけるタンパク質化学の研究は単純なyesまたはnoの質問のように与えられたタンパク質の折り畳みの問題を扱うことができるのに対し、生細胞内の高分子の全体の集団のダイナミクスを追跡しようとすると、可能性の全体の分布に敏感でなければなりませんポリペプチド鎖のコンフォメーションが利用できる成果、ミスフォールディングと凝集の危険性に関するものである。たとえば、我々はバルクセルLyを調べるかもしれませんウェスタンブロッティングによりタンパク質の凝集を十分に満足させると、タンパク質はほとんど不溶性とユビキチン化ではないと判断。しかし、多くのセルを上に平均化したことを検出するのが難しい、蛋白質の亜集団離散生きている細胞では、水溶性と種の局所濃度が非常に高い特定の区画内にユビキチン化されることがあります。後者のシナリオでは、容量の大きいバルク·サブ集団よりも細胞の生存のためのより多くの重要な結果をもたらす可能性があります。シャペロンは、多面的な行動や、in vitroで種々の機能を表示するのに対し、さらに、それが細胞内でそれらの離散関数が空間的にも時間的に限られていることが明らかになってきています。
生化学を理解するための新興パラダイムでは、濃度は、細胞内の特定のナノ環境変数のプロパティとなり、生物学的プロセスの根底にある分子のイベントは時間ではなく、SPACだけでなく、測定しなければならない電子。それは他の生物学的プロセスと、それらがどのように空間、時間、環境条件が次のように変更に規制されている任意の数を研究するために使用することができますが、ここで提示4Dイメージングのアプローチは、生細胞内でタンパク質の誤った折り畳みの敏感なモデリングを可能にします。本稿では、効果的に細胞質内タンパク質凝集の発生に対処するためのステージとオプションを示していUbc9 TSフォールディングセンサーを使用しています。集約品質管理の細胞生物学を示すことに加えて、このアプローチは、proteostasis(例えばUbc9 tsは酸化に応じてタンパク質の折り畳みストレスを測定するために使用することができます上の特定の摂動または遺伝的変異の効果を解読するための強力なツールとして使用できます品質管理経路の毒性凝集体、または突然変異の発現)。
4Dイメージングは、正確に二つの異なるタンパク質間のタンパク質の局在や局在を決定するためにも不可欠であるsであり、多分一時的な現象だが重要なことを検出する。例えば、特に酵母などの小さな球状の生物では、4Dイメージングは、これは単に検査の角度のアーティファクトであることを明らかにするかもしれない一方で、構造や集約がjuxtanuclear局在を持っているケースのように見えることがあります。
ここで、本実施例の実験では、細胞質ゾルにおける時間と空間を介して集約品質管理に従うようにタンパク質、Ubc9 tsを 、ミスフォールモデルの使用例を示します。許容温度で、Ubc9 tsは折り畳まれており、核と細胞質内に拡散。熱によるミスフォールディングの際には、最初にプロテアソームによる分解のために処理され、急速に拡散する小さな集合ストレス病巣を形成している。プロテアソームは、部分的に阻害されている場合、これらのストレス病巣は約2時間のコースでJUNQとiPod包有に変換されます。ユビキチン介在性分解は、品質制御オプション、UBとして利用できない場合c9のtsは直ちに保護集約のためのiPodインクルージョンに再ルーティングされます。これらのツールは、集約の品質管理に関与する新規な遺伝因子を発見すると、セル内の空間的·時間的規制を探索する信じられないほどの機会を提供しています。
開示事項
特別な利害関係は宣言されません。
資料
| Name | Company | Catalog Number | Comments |
| MG132 | Mercury | mbs474790 | |
| con A | Sigma | C2010 | |
| Glass bottom plates | ibidi | ibd81158 | |
| 4D Fluorescence Imaging of Protein Aggregation Confocal 3D movies were acquired using a Nikon A1R-si microscope equipped with a PInano Piezo stage (MCL), using a 60x water objective NA 1.27, 0.3 micron slices, 0.5% laser power (from 65 mW 488 laser and 50 mW 561 laser). z-stacks were acquired every 5 min for 90 min. Each z-series was acquired with 0.5 micron step size and 30 total steps. Image processing was performed using NIS-Elements software. | |||
参考文献
- Gershenson, A., Gierasch, L. M. Protein folding in the cell: challenges and progress. Current opinion in structural biology. 21, 32-41 (2011).
- Aguzzi, A., Calella, A. M. Prions: protein aggregation and infectious diseases. Physiological reviews. 89, 1105-1152 (2009).
- Morimoto, R. I. Proteotoxic stress and inducible chaperone networks in neurodegenerative disease and aging. Genes & development. 22, 1427-14 (2008).
- Cohen, E., Dillin, A. The insulin paradox: aging, proteotoxicity and neurodegeneration. Nature reviews. Neuroscience. , (2008).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annual review of biochemistry. 67, (1998).
- Tyedmers, J., Mogk, A., Bukau, B. Cellular strategies for controlling protein aggregation. Nature reviews. Molecular cell biology. 11, 777-788 (2010).
- Treusch, S., Cyr, D. M., Lindquist, S. Amyloid deposits: Protection against toxic protein species?. Cell cycle (Georgetown, Tex.). 8, 1668-1674 (2009).
- Spokoini, R., et al. Confinement to Organelle-Associated Inclusion Structures Mediates Asymmetric Inheritance of Aggregated Protein in Budding Yeast. Cell Rep. , (2012).
- Weisberg, S. J., et al. Compartmentalization of superoxide dismutase 1 (SOD1G93A) aggregates determines their toxicity. Proc Natl Acad Sci U S A. 109, 15811-15816 (2012).
- Kaganovich, D., Kopito, R., Frydman, J. Misfolded proteins partition between two distinct quality control compartments. Nature. 454, 1088 (2008).
- Betting, J., Seufert, W. A yeast Ubc9 mutant protein with temperature-sensitive in vivo function is subject to conditional proteolysis by a ubiquitin- and proteasome-dependent pathway. The Journal of biological chemistry. 271, 25790 (1996).
- Tongaonkar, P., Beck, K., Shinde, U. P., Madura, K. Characterization of a temperature-sensitive mutant of a ubiquitin-conjugating enzyme and its use as a heat-inducible degradation signal. Analytical biochemistry. 272, 263 (1999).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved