このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
質量分析による3つの DNA 病変の定量化と、周囲の微細粒子状物質に曝露したマウスの組織におけるそのレベルの評価
* これらの著者は同等に貢献しました
要約
ここでは、病変の高感度かつ正確な定量のための方法 8-オキソ-7, 8-ジヒドロ-2'-デオキシグアノシン (8-oxodGuo), 1,n6-etheno-2'-デオキシアデノシン (1,n6-茶道) および 1,n2-etheno-2'-デオキシグアノシン (1,N2-dGuo) DNA で.この方法は、曝露された A/J マウスの組織 (肺、肝臓および腎臓) における周囲微細粒子状物質 (PM2.5) の影響の評価に適用した。
要約
Dna 付加物および酸化 DNA 塩基は、electrophilic された物質の毒性評価のために有用なバイオマーカーである DNA 病変の例であり、生体内変換時に反応性毒物を発生させ、または酸化ストレスを誘導する。酸化核酸塩基のうち、最も研究されたものは 8-オキソ-7、8-dihydroguanine (8-oxoGua) または 8-オキソ-7, 8-ジヒドロ-2'-デオキシグアノシン (8-oxodGuo) であり、DNA における酸化的誘発塩基損傷のバイオマーカーである。脂質過酸化プロセスに起因するアルデヒドおよび epoxyaldehydes は、etheno 付加体1、n2-etheno-2' のような変異原性 exocyclic DNA 付加体を形成することができる electrophilic 分子である (1,n2-εdGuo) および 1,n6-etheno-2'-デオキシアデノシン (1,n6-εdAdo) は、炎症の病態生理学における潜在的なバイオマーカーとして示唆されている。DNA における選択的で高感度な方法は、細胞の突然変異率や慢性疾患 (例えば、がん、神経変性疾患) の発症を遅らせる予防戦略の開発に必要である。その検出のために利用可能な高感度の方法の中でも (電気化学的またはタンデム質量分析検出器に結合された高性能液体クロマトグラフィー、彗星アッセイ、イムノアッセイ、 32P-postlabeling)、最も選択的なものであるタンデム質量分析 (HPLC ESI-MS/MS) に結合された高性能液体クロマトグラフィー。複雑な生体試料や、DNA、尿、血漿、唾液などの生物学的マトリックス中の修飾ヌクレオシドを定量化するためのゴールドスタンダードとして進化した HPLC-MS/MS は、選択性が重要な利点です。同位体ラベルの内部標準を使用することにより、DNA 加水分解および検体濃縮ステップにおける分子損失の補正、およびサンプル間の分析種イオン化の違いによる利点が加わります。また、複数のピークが存在する場合に、正しいクロマトグラフィーピークの識別にも役立ちます。
ここでは、A/J マウスの肺、肝臓および腎臓 DNA における 8-oxodGuo、1、n6-茶道および 1,2-dGuo の定量化に首尾よく適用された、高感度、正確かつ正確な HPLC − ms/ms 法を提示する。アンビエント PM2.5暴露の影響の評価。
概要
一部の活性酸素種 (ROS) は、DNA 塩基の炭素二重結合とデオキシリボース部分の炭素を酸化させ、酸化された塩基を生成し、DNA 鎖が1を壊すことがある。窒素と酸素原子が豊富な負の荷電分子として、DNA は、nucleophilic 部位 (窒素と酸素) と共有的に反応する electrophilic グループの標的でもあり、DNA 付加物2と呼ばれる製品を与えている。そこで、DNA 付加物および酸化 DNA 塩基は、electrophilic された物質の毒性評価のために有用なバイオマーカーである DNA 病変の例であり、生体内変換に対して反応性毒物を発生させ、または酸化ストレス1を誘導し、 2修飾された DNA 塩基は塩基切除修復 (BER または NER) によって DNA から除去されるが、dna 病変の生成と除去との間の不均衡の誘導は、DNA 残業におけるそれらのレベルの正味増加をもたらす。/c5 >。結果は、DNA 突然変異率の増加、遺伝子発現の減少、およびタンパク質活性の低下 (2、4、5、6、7) と密接に関連している効果病気の開発。DNA の変異は、細胞のシグナル伝達、細胞周期、ゲノムの完全性、テロメアの安定性、エピゲノム、クロマチン構造、RNA のスプライシング、タンパク質の恒常性、代謝、アポトーシス、細胞分化などの多様な細胞機能に影響を与える可能性があります8 、9。細胞突然変異率および慢性疾患の発症を遅らせるための戦略 (例えば、癌、神経変性疾患) は、変異源の知識を通過し、それらのうち、DNA 病変およびそれらの原因に関する。
過剰に内因的に発生した ROS は、汚染物質の曝露に起因して、持続性の炎症、疾患の病態生理学 (例えば、糖尿病) など、DNA および脂質損傷1を含む生体分子損傷の重要な原因である。一例として、遷移金属イオン (Fe2o3+、Cu+) による h2o-2 から形成される高反応性ヒドロキシルラジカル (OH) は、dna 塩基の酸化、dna 糖部分および多価不飽和脂肪酸を拡散制御レート10.80はすでに酸化核酸塩基3を特徴付けられているが、最も研究したものは 8-オキソ-7, 8-dihydroguanine (8-oxoGua) または 8-オキソ-7, 8-ジヒドロ-2'-デオキシグアノシン (8-OxodGuo,図 1), GT transversions を誘導することができる病変である哺乳類細胞10、11。グアニンのモノラル電子酸化によって、または DNA1におけるグアニンのヒドロキシルラジカルまたは一重項酸素攻撃によって形成される。多価不飽和脂肪酸は、高反応性酸化剤の他の重要な標的である•OH、脂質過酸化のプロセスを開始する1,12。それは、マロンジアルデヒド、4-ヒドロキシ-2-ノネナール、2、4-decadienal、4、5-エポキシ-(2E)-decenal、ヘキセナール、アクロレイン、crotonaldehyde などの electrophilic アルデヒドおよび epoxyaldehydes に分解することができる脂肪酸ヒドロペルオキシドを生じさせるマロンジアルデヒド-、propano、または etheno 付加体1、12、13などの変異原性 exocyclic DNA 付加を形成することができる。Etheno 付加体 1,n2-etheno-2'-デオキシグアノシン (1,n2-εdGuo,図 1) および 1,n6-etheno-2'-デオキシアデノシン (1,n6-εdAdo,図1) は、炎症14,15の病態生理学における潜在的なバイオマーカーとして示唆されている。
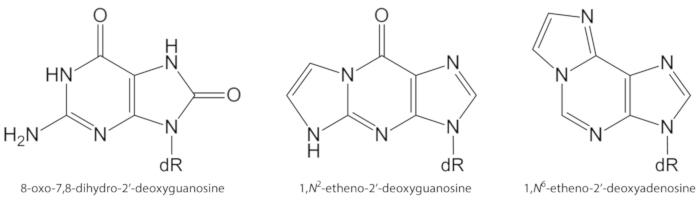
図 1.DNA 病変の化学構造は、本研究において定量化した。dR = 2 ́-デオキシリボース。この図は、オリベイラ et al.34から変更されています。この図の大規模なバージョンを表示するには、ここをクリックしてください。
1980年代初頭に実施された研究は、電気化学的検出 (HPLC ECD) に結合した高性能液体クロマトグラフィーによる 8-oxodGuo の高感度検出を可能にした。いくつかの生物学的システムにおける HPLC − oxodGuo の定量化は酸化条件に供され、DNA1,16における酸化的誘発塩基損傷のバイオマーカーとしての8− oxodGuo の認識につながった。堅牢で、低 fmol 範囲17で 8-oxodGuo の定量化を可能にするが、HPLC ECD 測定は検体同定のための検体保持時間の精度と、干渉を避けるためのクロマトグラフィー分解能に依存します。他のサンプル成分。電気化学検出では、移動相において塩 (例えば、リン酸カリウム、酢酸ナトリウム) を使用する必要があるため、適切な分析条件の維持には、ルーチンカラムおよび機器の洗浄時間が必要です。
あるいは、formamidopyrimidine DNA glycosylase (FPG) およびその後、ヒト 8-oxoguanine glycosylase 1 (hOGG1) の細菌 DNA 修復酵素の使用は、DNA からの 8-oxoGua の検出および除去のための、DNA アルカリ不安定の誘導のための方法として浮上したサイト。アルカリ不安定部位は、DNA 鎖切断に変換され、アルカリ単細胞ゲル電気泳動 (「彗星アッセイ」) による 8-oxoGua の非常に高感度な間接定量を可能にします。細胞 DNA の抽出の必要性のない高い感受性そして分析の達成はこのタイプの試金の主な利点である。これは、DNA において 8-oxoGua の最低定常状態レベルを与え、典型的には、HPLC に基づくシステム法によって得られたレベルよりも低い7-10 倍である。しかし、それは 8-oxoGua の間接測定であり、いくつかの欠点は、1,16,18使用される修復酵素の特異性または未知の効率の欠如である。
免疫アッセイは、1、n6-茶道および1、n2-dGuo12のような 8-oxoGua1および exocyclic DNA 付加物の検出のために使用される方法の他のセットである。感受性にもかかわらず、dna 病変の検出のための抗体の使用の欠点は、正常な dna 塩基1,12を含む生物学的試料の他の成分に対する交差反応性による特異性の欠如である。Exocyclic DNA 付加体としては、1、n6-茶道および 1,n2-dGuo を含む、高感度の32P − postlabeling アッセイ12によって検出および定量することもできる。32P-postlabeling の高感度は、1010通常塩基あたり約1付加体の検出のための非常に少量の DNA (例えば、10μ g) の使用を可能にします。しかし、ラジオ-化学物質の使用は、化学的特異性および低精度の欠如は、いくつかの欠点19,20である。
上記で引用した方法の共有制限は、所望の分子の検出のための低い選択性または特異性である。このシナリオで、エレクトロスプレーイオン化タンデム質量分析法 (HPLC-ESI-MS/MS および HPLC-ms3) に結合された hplc は、DNA、尿、血漿および唾液のような生物学的マトリクスにおける修飾ヌクレオシドの定量化のための金本位として進化した1,19,20. HPLC − MS/ms 法の利点は、感度 (典型的には低 fmol 範囲) と i によって提供される特異性が高いことである) クロマトグラフ分離、ii) 質量内部の分子断片化の特徴と既知のパターン分光器衝突チャンバ、および iii) は、多重反応監視モード1,19において選択された質量を電荷比 (m/z) で正確に測定する。同位体ラベルの内部標準を使用することにより、DNA 加水分解および検体濃縮ステップにおける分子損失の補正、およびサンプル間の分析種イオン化の違いによる利点が加わります。また、1、12、19、20の複数のピークが存在する場合に、正しいクロマトグラフィーピークの同定にも役立ちます。
さまざまな生物学的サンプルから抽出された DNA において、oxodGuo、1、N6-茶道および 1,n2-dGuo の定量には、HPLC − ms/ms に基づくいくつかの方法が用いられる12,15,20 、21、22、23、24、25、26、27、28、29 日.微粒子 (PM2.5) は、多環芳香族炭化水素 (PAHs)、ニトロ-PAHs、アルデヒド、ケトン、カルボン酸、キノリン、金属、水溶性イオンなどの有機および無機化学物質を運び、炎症を誘発する可能性があり酸化ストレスは、生体分子損傷および疾患の発生を優先する条件30、31、32、33である。ここでは、oxodGuo、1、n6-茶道および 1,2-dGuo の肺、肝臓および腎臓 DNA における定量化のために首尾よく適用された検証済みの HPLC ESI/ms 法を提示します。アンビエント PM2.5暴露34の影響
プロトコル
4週齢の雄 A/J マウスは、特定の病原体を遊離し、Fundação オズワルドクルス (FIOCRUZ)、リオデジャネイロ、ブラジルの実験動物の育種センターから得られ、そしてそれに応じて医学部の倫理委員会に扱われたサンパウロ (プロトコル no 1310/09) の。
1. マウスの組織のコレクション
- キシラジンとケタミンで動物を麻酔。体重が 30 g のマウスの場合、2.63 を含む溶液 (2 mL 以下) を注射し、腹腔内のケタミンおよび0.38 のキシラジンの mg を注入する。
- 補足の分析 (例えば、抗酸化酵素の活動、マロンジアルデヒドのレベル) のための血 (0.5-1.5 mL) を集めなさい。
- 骨盤から剣状突起のプロセスに腹部の毛を剃る。ヘアレスエリアの縦の中央線に切開をします。腹部の臓器を露出させるために、水平方向の横線に切開を行います.
- 腹部大動脈を切断して exsanguination を促進し、動物を euthanize する。
- 目的の組織を削除します (この場合, 肝臓, 腎臓と肺).
- 肝臓を除去するには、不良カバ静脈および門脈肝静脈を切断する。
- 腎臓を削除するには, セクション腎静脈と動脈.
- 肺を除去するには、横隔膜の四肢と胸部の壁に近い円周の切開を行います。胸腔の内部にはさみを開くことによって、鎖骨を破ります.剣状突起プロセスから気管に向かって、extern ボーンをカットし、肺と心臓を露出させるために。
- 鉗子で肺を持ち、気管と肺の周りの靭帯をセクションする。ブロック肺プラスの心を慎重に削除します。ブロックの外に肺を除去するには、鉗子で心臓を保持し、そのベース内のすべての容器を切断します。
- 分離した組織を低温食塩水 (0.9% NaCl) で直ちに洗浄し、低温管に移し、直ちにチューブを液体窒素に浸します。作業が完了したら、-80 ° c でチューブを保管してください。
注意: 皮膚、粘膜または眼に直接接触している液体窒素は、火傷を引き起こす。接触を避けるために適切な個々の保護を使用しなさい。液体窒素蒸気による窒息を避けるために、換気のある実験室で作業してください。
2. DNA 抽出
注: DNA 抽出方法は、ここで研究した病変の分析を可能にするために Loureiro et al. (2009)35から改変された。
- 組織を含むチューブをドライアイスに移します。
- 氷の上に置かれた培養プレートをベースにして、メスで組織片を切断します。即時使用のための重量 1 g。残りの組織は、-80 ° c で貯蔵庫に戻るまで、ドライアイスに保管する必要があります。
注: 分析の繰り返しが必要な場合、アーティファクトの形成を防ぐために残りの組織の解凍を避けることが重要です。 - 50 mL の蓋をしたチューブに 1 g の組織をそれぞれ、0.5 mM デフェロキサミンを含む市販の細胞溶解溶液 10 mL を追加し、氷上で維持します。
注: すぐに使用するために、ソリューションのボリュームにデフェロキサミンを追加します。100 mL の溶液ごとに、デフェロキサミンメシル酸塩の 0.0328 g を加える。 - 組織断片を含まない均質な溶液が得られるまで、組織ホモジナイザーを用いて組織を均質化する。均質化の際にチューブを低温 (氷上) にしてください。加熱を避けるために、低速を使用してください。
- 各均質化されたサンプルにプロテイナーゼ K 溶液 (20mg/mL) の150μ l を加えます。反転によってチューブを振ると一晩室温でそれらを維持します。
- 40μ l の溶液 (リボヌクレアーゼ/mL) を加え、逆転によって振盪し、チューブを室温で2時間保持する。
注: 沈殿を避けるために、酢酸ナトリウム緩衝液 10 mM、pH 5.2 で溶液リボヌクレアーゼを調製してください。溶液を100° c で15分間加熱してから、デオキシリボヌクレアーゼから無料で溶液を得てください。 - 市販のタンパク質沈殿溶液の5ml を添加し、激しくボルテックスし、2000 x g、4° c で10分間遠心分離する。
- 上清を 10 mL の冷 isopropanol を含む 50 mL のキャップ付きチューブに移します。沈殿した DNA の観察まで、チューブを何度も優しく反転させます。
注: このプロトコルはここで一時停止し、チューブを-20 ° c に保つことができます。 - パスツールピペットを用いて沈殿した DNA を収集し、最後に閉じた。10 mM トリスバッファー、1mm デフェロキサミン、pH 7.0 の 4 mL を含むチューブに移します。
- DNA が完全に上記の溶液に溶解した後 (ボルテックスしない)、isoamyl アルコールの 4% を含むクロロホルム溶液を 4 mL 加える。
- 均質化のためにチューブを10回反転し、2000 x g、4° c で遠心分離し、2つの相を分離するために10分間、新しいチューブに上相を移す。
- ステップ2.10 と2.11 をさらに2回繰り返します。
- 8 mL の絶対エタノールと 5 M NaCl 溶液の 0.4 mL を加えて、DNA を沈殿させます。
- 沈殿した DNA を再度収集し、70% のエタノールを 3 mL に移します。この手順をもう一度繰り返します。
- エタノール溶液を慎重に廃棄し、吸収性紙の上に沈殿した DNA を含むチューブを反転させ、溶液の過剰を除去する。
- 200μ l の 0.1 mM デフェロキサミン溶液を加えて、DNA を溶解させます。DNA が完全に復元されるまで (一晩) 4 ° c でチューブを維持します。
- 260 nm の吸光度および純度を 260/280 nm の吸光度比で測定することにより、DNA 濃度を決定します。
注: DNA 濃度を測定するには、10μ l の DNA 溶液を990μ l の超純水 (100x 希釈) のアリコートに移します。吸光度を 260 nm (1 以下にする必要があります) 50 (50 μ g/mL は、260 nm の1cm パス長溶液の吸光度が 1) と希釈量 (100x) によって DNA 濃度をμ g/mL で得ることにより、2本鎖 DNA の濃度です。260 nm の吸光度が1を上回っている場合は、追加の希釈が必要です。所望の DNA 純度については 260/280 nm の吸光度比が1.8 以上である必要がありますが、1.6 あたりの比率は許容されます。
3. DNA 酵素加水分解
- 分析レシピ
- 1,n6-ΕdAdo および 1,n2-εdGuo 分析: DNA の150μ g を含有するアリコートに、200 mM トリス/MgCl2バッファー (pH 7.4) の7.5 μ l を添加し、1.4 fmol/μ l を含む内部標準液の250μ l を [15N5] 1,n6-εdAdo と [15N5] 1,n2-εdGuo, 15 単位のデオキシリボヌクレアーゼ私は、超純水で200μ l の最終量を調整し、ステップ3.2.1 で使用する酵素の量を差し引きます。
- 8-oxodGuo 分析: DNA の80μ g を含有するアリコートに、200 mM トリス/MgCl2バッファー (pH 7.4) の3.8 μ l を加え、1000 Fmol/μ l を含む内部標準液の2μ l [15N5] 8-oxodGuo、および8単位のデオキシリボヌクレアーゼ I を添加する。超純水で最終量を100μ l に調整し、ステップ3.2.2 に使用する酵素の量を差し引きます。
注: 内部標準 [15n5] 1,n6-εdAdo, [15n5] 1,n2-εdGuo および [15n5] 8 oxodGuo は synthetized として特徴付けられ35、36、37に記載されている。注入されたサンプル・ボリューム内の内部標準の数量は、注入したキャリブレーション・カーブのボリュームと同じにする必要があります。
- 試料を37° c で1時間インキュベートします。
- ステップ3.1 からのサンプル: 0.006 のCrotalus の atroxを加え、ウシ腸管粘膜からのアルカリホスファターゼの15単位を添加します。
- ステップ3.2 からのサンプル: 0.0032 のCrotalus の atroxを加え、ウシ腸管粘膜からのアルカリホスファターゼの8単位を添加します。
- 試料を37° c で1時間インキュベートします。
- サンプルを 14000 x gで10分間遠心します。
- ステップ3.2.1 からのサンプル: deoxynucleosides (茶道、dGuo) を HPLC/お父によって定量化するための各サンプルの別々の10μ l (ステップ 9)。残留体積を固相抽出に対象とする (ステップ 4)。
- ステップ3.2.2 からのサンプル:80 μ l の上澄み液を50μ l の注射用バイアル (1000 fmol の [15N5] 8-oxodGuo) に HPLC-ESI/ms システムで転写する。HPLC/dGuo の定量化のために残りの20μ l を予約してください (ステップ 9)。
4. 1, n6-ΕdAdo および 1, n2-εdGuo の分析のための固体段階の抽出
- 次の一連の溶液を1ml のカートリッジ (SPE-C18、30 mg/mL、33μ m、1 mL) で負荷します: 100% メタノール、脱イオン水、DNA サンプルの加水分解、脱イオン水、10% メタノール、15% メタノール、および 100% メタノール (回収)。
注: 異なるソリューションのアプリケーション間でカートリッジを乾いたままにしないでください。前のソリューションがカートリッジに完全に入った直後に、次のソリューションを追加します。 - 真空乾燥は、付加体を含有する最後の溶出画分 (100% メタノール) を含む。
- 再懸濁/MS 分析の直前に83.1 μ l の超純水中の乾燥試料を採取し、各試料の50μ l に 200 fmol の各内部標準を得た。
5. 較正曲線の作成
- それぞれの点において、1000 fmol の固定量 (oxodGuo) 8-oxodGuo の間隔で、少なくとも5つの点6000を300の fmol の間に準備してください。注入された体積でこれらの量を考慮してください。
- 1, n 6-εdAdo と 1, n 2-εdGuo の40間隔に少なくとも5つの点を用意し、固定量 200 fmol の [fmol] 1, n 6-εdAdo と [15 n 5] 1,N2-各ポイントでεdGuo。注入された体積でこれらの量を考慮してください。
- DGuo と茶道の nmol の0.05 間隔に少なくとも5つのポイントを準備します。注入された体積でこれらの量を考慮してください。
6. メソッド検証のための DNA サンプルの調製
- 1,n6-ΕdAdo と 1,n2-ΕdGuo 分析: さまざまな量 1,n6-εdAdo と 1,n2-εdGuo (例えば, 1.75, 8.75, 17.5, 35 fmol) と固定量 [15N5] 1,N6-εdAdo および [15N5] 1,N2-εdGuo (350 fmol) を仔ウシ胸腺 DNA の100μ g に、ステップ3に記載されているように酵素加水分解を行う。2つの異なる日に quadruplicate でサンプルを処理します。サンプルを使用して、メソッドの精度と精度を評価します。
注: DNA 加水分解物の最終体積は200μ l (ステップ 3) であり、そこから10μ l は HPLC/deoxynucleosides によっての定量化のために分離される (ステップ 9)。残りの溶液 (190 μ l) を固相抽出に供して (ステップ 4)、乾燥画分を83.1 μ l に再懸濁させ (ステップ 4.3)、そこから50μ l を HPLC − ESI/MS システムに注入する。1, n 6-εdAdo と 1, n 2-εdGuo 注入の量は 1, 5, 10, 20 fmol 200 で、[15 n 5] 1, n 6-εdAdo と [15 n 5] 1, n の fmol各サンプルに2-εdGuo。 - 8-oxodGuo 分析: oxodGuo (例えば、734、1468、2938、4408 fmol) のさまざまな量を加え、一定量の [15N5] 8-oxodGuo (2000 fmol) を仔ウシ胸腺 DNA の100μ g に添加し、ステップ3で説明されているように酵素加水分解を行う。2つの異なる日に quadruplicate でサンプルを処理します。サンプルを使用して、メソッドの精度と精度を評価します。
注: DNA 加水分解物の最終体積は100μ l (ステップ 3) となり、そこから50μ l は HPLC − ESI/MS システムに注入されることになる。8-oxodGuo の注入量は367、734、1469、2204 fmol であり、各サンプルでは [15N5] 8-oxodGuo の 1000 fmol があります。 - 1, n 6-εdAdo (注入量で 7.5 fmol を得るため) および 1, n 2-εdGuo の 35 fmol (注入100量で 20 fmol を得るため) の 13.125 fmol を加えます。
- 内部標準 [15n5] 1,n6-εdAdo および [15n5] 1,n2-εdGuo (200 fmol) を4つのサンプルに加える。すべてのサンプルの DNA 加水分解と固相抽出を続行します。
- 内部標準 [15n5] 1,n6-εdAdo および [15n5] 1,n2-εdGuo (200 fmol) を他の4つのサンプルに追加します。
- 試料を使用して、固相抽出から付加体の回収率を計算する。
7. 8-oxodGuo の HPLC-ESI/MS 分析
- OxodGuo 標準を機器に注入し、多重反応モニタリング (MRM): m/z 284 [m + h] + → m/z 168 [m-2 '-デオキシリボース + h]+によってそのフラグメンテーションパターンを最適に検出するための ESI/ms パラメータを設定します。
- [15N5] 8-oxodGuo: m/z 289 [m + h]+m/z → 173 [m-2 '-デオキシリボース + h]+の検出にも同じパラメータを使用します。
注: この作業で使用する装置と同等以上の装置を使用してください (資料表を参照してください)。ESI/MS パラメーターは表 1で説明されているように設定しました。
- [15N5] 8-oxodGuo: m/z 289 [m + h]+m/z → 173 [m-2 '-デオキシリボース + h]+の検出にも同じパラメータを使用します。
- フィルター (0.22 μ m 多孔質膜を使用) 及び degasify (sonicator を用いた) は、すべての水性 HPLC 溶媒に基づく。
- 分析には以下のクロマトグラフィー条件を使用して、図 2に示すようにシステムを取り付けます。
注: カラム A は、バイナリポンプに接続されています。この溶離液は、図 2aに示すように、クロマトグラフィーの最初の16分間および32から46分の間、UV 検出と廃棄に向けられています。これは、サンプルが注射直後に溶出するカラムです。カラム B は、アイソクラティックポンプと質量分析計に接続されています。これは、バルブが図 2bに示された位置に切り替わったとき、16-32 分間隔でのみカラム A の溶離液を受けます。弁の切換えはバイナリポンプ勾配によって溶出される2つのコラム間の関係を可能にする。図 2bに示す構成は、さらなるピーク分離および狭小化を可能にする。さらに、対象となるクロマトグラフのみが質量分析計に到達し、感度と選択性が向上します。- 50 x 2.0 mm の内径2.5 μ m、C18 カラム (図 2のカラム a) は、3.0% ギ酸 (溶媒 a) の勾配と 0.1% ギ酸 (溶媒 B) を含むメタノールを0.1 μ l/min の流量で、C18 セキュリティガードカートリッジ (4.0 x 150 mm の内径) に結合し、25° c。
- バイナリポンプのための次の勾配プログラムを使用してください: 0 から25分、溶媒 B の 0-15% から。25 ~ 28 分、溶媒 B の 15-80%;28 ~ 31 分、80% の溶媒 B;31〜33分、80-0% の溶媒 B;33に46分、溶媒 B の 0%。
- スイッチングバルブを使用して、溶離液の最初の16分を廃棄物に、16-32 の分画分を2番目のカラム (150 x 2.0 mm、3.0 μ m、C18、図 2のカラム B) に接続し、ESI のソースに結合し、アイソクラティックポンプによって 15% のソリューションで条件付けthanol は、0.1% ギ酸 (150 μ l/min) を含む水に含まれる。
注: ステップ7.3.1.2 のスイッチングバルブプログラムを使用する前に、8-oxodGuo 標準溶出16分後に最初のカラムからのものであるかどうかを確認してください。2番目のカラムから oxodGuo を溶出するためにバイナリポンプの勾配を使用し、鋭いクロマトグラフィーピークを得るには、32分でバルブを閉じることが重要です。病変部 8-oxodGuo 溶出は、2列目から約36分で、使用するカラムや機器によっては、検体の保持時間のばらつきが生じることがある。HPLC 溶媒勾配プログラムの適応が必要な場合があります。
- 50 x 2.0 mm の内径2.5 μ m、C18 カラム (図 2のカラム a) は、3.0% ギ酸 (溶媒 a) の勾配と 0.1% ギ酸 (溶媒 B) を含むメタノールを0.1 μ l/min の流量で、C18 セキュリティガードカートリッジ (4.0 x 150 mm の内径) に結合し、25° c。
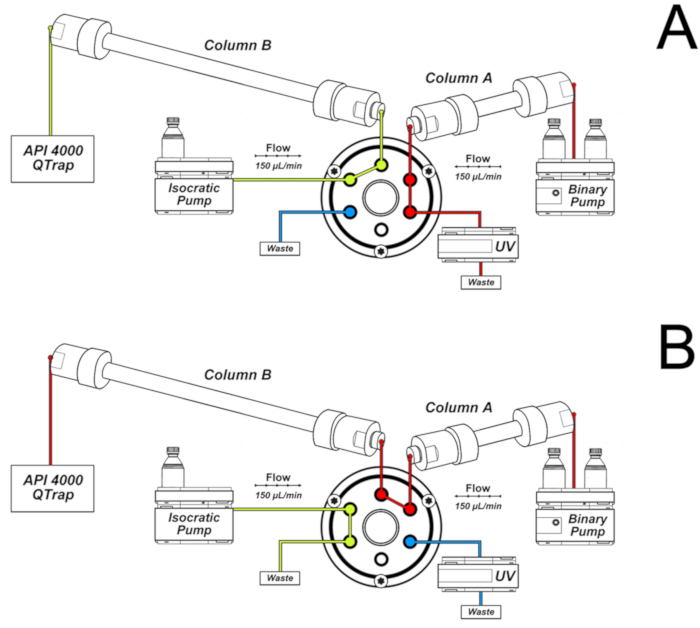
図 2.8-オキソ-7, 8-ジヒドロ-2'-デオキシグアノシン (8-oxodGuo) の分析に使用される2つの列のシステム。A) 最初の16分間および32から46分のクロマトグラフィーで使用される構成。B) 間隔 16-32 分で使用される構成は、質量分析計の ESI 源への溶出前のカラム B におけるさらなる分離およびピーク狭小化を可能にする。この図は、オリベイラ et al.34から再公開されました。この図の大規模なバージョンを表示するには、ここをクリックしてください。
8. 1, n 6 -ΕdAdo および 1, n 2 -εdGuo の HPLC − ms/ms 分析
- 装置に 1,nΕdAdo および 1,n2-εdGuo の標準を注入して、多重反作用の監視によってそれらの断片化パターンの最もよい検出のための ESI/ms 変数を置く (MRM): m/z276 [m + H]+ → m/z 160 [m 2 '-デオキシリボース + h]+ 1,N6-εdAdo とM/z 292 [m + H]+ → m/z 176 [m-2 '-デオキシリボース + h の検出用]+ 1,N2-εdGuo の検出のために.
- [15n5] 1,N6-εdAdo (m/z 281 [m + h]+m/z → 165 [m-2 '-デオキシリボース + h]+) および [15nの検出に同じパラメータを使用します。5] 1,N2-εdGuo (m/z 297 [m + H]+ → m/z 181 [m-2 '-デオキシリボース + H]+)。ESI/MS パラメーターを表 1に示すように設定します。
| ESI/MS パラメータ | 8-oxodGuo | Etheno 付加体 |
| カーテンガス | 20 psi | 20 psi |
| 噴霧ガス | 55 | 50 |
| イオン源ガス | 50 psi | 40 psi |
| 衝突誘発解離ガス | 媒体 | 媒体 |
| イオン噴霧電圧 | 5000 | 4500 |
| ESI プローブ温度 | 450 | 450 |
| Declustering の可能性 | 31 V、8-oxodGuo | 41 V, 1, N6-εdAdo |
| 31 V、[15N5] 8-oxodGuo | 41 V, [15N5] 1, N6-εdAdo | |
| 45 V, 1, N2-εdGuo | ||
| 45V, [15N5] 1, N2-εdGuo | ||
| 衝突エネルギー | 23 eV, 8-oxodGuo | 25 eV, 1, N6-εdAdo |
| 23 eV, [15N5] 8-oxodGuo | 25 eV, [15N5] 1, N6-εdAdo | |
| 27 eV, 1, N2-εdGuo | ||
| 27 eV, [15N5] 1, N2-εdGuo | ||
| コリジョンセル出口の可能性 | 16 V、8-oxodGuo、 | 8 V, 1, N6-εdAdo |
| 16 V、[15N5] 8-oxodGuo | 8 V、[15N5] 1、N6-εdAdo | |
| 16 V, 1, N2-εdGuo | ||
| 16 V、[15N5] 1、N2-εdGuo | ||
| エントランスポテンシャル | 10 V | 10 V |
表 1.DNA 病変の検出のための ESI/MS 装置で使用されるパラメータ。このテーブルは、オリベイラ et al.34から変更されています。
- フィルター (0.22 μ m 多孔質膜を使用) 及び degasify (sonicator を用いた) は、すべての水性 HPLC 溶媒に基づく。
- 分析には以下のクロマトグラフィー条件を使用してください。
- 150 x 2.0 mm の内径3.0 μ m、c18 カラムを C18 セキュリティー・ガード・カートリッジ (4.0 x 3.0 mm 内径) に結合し、5 mM 酢酸アンモニウムの勾配、pH 6.6 (溶媒 A) およびアセトニトリル (溶媒 B) を130μ l/min および25° c の流量で溶出します。
- バイナリポンプのための次の勾配プログラムを使用してください: 0 から10分、溶媒 B の 0%。10〜39分、溶媒 B の 0-20%;39に41分、20-75% の溶媒 B;41に46分、75% の溶媒 B;46に47分, 75-溶媒 B の 0%;47に60分、溶媒 B の 0%。
- スイッチングバルブを使用して、溶離液の最初の35分を廃棄物に、35-42 min 分画を ESI ソースに向けるようにしてください。付加物標準が設定された間隔 (35-42 min) の列から溶出することを確かめてください。必要に応じて調整してください。
- 150 x 2.0 mm の内径3.0 μ m、c18 カラムを C18 セキュリティー・ガード・カートリッジ (4.0 x 3.0 mm 内径) に結合し、5 mM 酢酸アンモニウムの勾配、pH 6.6 (溶媒 A) およびアセトニトリル (溶媒 B) を130μ l/min および25° c の流量で溶出します。
9. 通常の 2 '-deoxyribonucleosides を HPLC-UV による定量化
- この作業で使用する装置と同様の装置を使用します (資料の表を参照してください)。
- 250 mm x 4.6 mm の内径、C18 ・セキュリティ・ガード・カートリッジ (4.0 x 3.0 mm の内径) に取り付けられた、5μ m、C18 カラムを 0.1% のギ酸とメタノールの勾配で溶出します。
- 次のグラデーションプログラムを使用してください: 0 から25分、0から 18% メタノール;25から27分、18から 0% のメタノール;1 mL/min および30° c の流速で27から37分、0% メタノール)。
- 2 '-deoxynucleosides の定量化のために予約された各サンプルの5μ l を注入する。
- DGuo と茶道のピークの統合のための 260 nm のお父さんの探知器を設定します。
10. DNA 病変の定量化
- 8-oxodGuo、[15n5] 8-OxodGuo、1、n6-εdAdo、[15n5] 1、n6-εdAdo、1、n2-εdGuo、および [15n5] 1、n2 のピークを統合します。-εdGuo/ms 分析から。
- 8-oxodGuo/[15n5] 8-OxodGuo、1、n6-εdAdo/[15n5] 1、n6-εdAdo、および 1,n2-εdGuo/[15n 5] の面積比を計算します。] 1,N2-キャリブレーションカーブとサンプルのεdGuo。
- ステップ10.1.1 で得られた面積比を y 軸に、x 軸の各点に存在する検体の量を使用して、較正曲線をプロットします。
- ステップ10.1.1 およびステップ10.1.2 の較正曲線で計算された比率を使用して、各注入サンプルの病変の量 (fmol) を計算します。
- DGuo と茶道のピークを HPLC UV 分析から統合します。
- ステップ10.2 で得られた領域を y 軸に、x 軸の各点に存在する検体の量を使用して、較正曲線をプロットします。
- ステップ10.2 で得られた領域とステップ10.2.1 の較正曲線を使用して、各注入されたサンプルの dGuo および茶道の量 (nmol) を計算します。
- ステップ10.2.2 で計算された量が5μ l のサンプル容量に存在することを考慮して、HPLC − nmol/MS システムに注入した各サンプル中に存在する dGuo および茶道の量 () を計算し、一方、50μ l を HPLC − ESI/MS システムに注入した。
注: oxodGuo 解析に使用されるサンプルの dGuo の量を計算するには、ステップ10.2.2 で取得した量 (nmol/μ l) を50で乗算します。1,n6-ΕdAdo および 1,n2-εdGuo の分析に使用されるサンプル中の茶道および dGuo の量を計算するために、固相抽出後の濃度ステップを考慮する。HPLC − ESI/MS システムに注入された50μ l の体積は、元のサンプルの114.32 μ l に相当する。ステップ10.2.2 で得られた量 (nmol/μ l) には、正しい値を得るために114.32 を掛ける必要があります。 - モル画分を計算します 8-oxodGuo/dGuo, 1,n6-εdAdo/茶道, 1,n2-εdGuo/dGuo.比 (fmol 病変/nmol 正常 deoxynucleoside) は、106正常 dGuo または茶道あたりの病変の数を与える。
結果
マウスの肝臓から得られた平均 DNA 濃度 (± SD)、0.2 肺 (~ 1g の組織) と腎臓 (~ 0.4 g の組織) は、それぞれ5068± 2615, 4369 ± 1021, 3223 ±723μ g/mL の最終的な容量を200μ l とした。精製 DNA の HPLC −パパによって得られた代表的なクロマトグラムを図 3に示す。4つの 2 '-deoxynucleosides の存在は、対応する 2 '-deoxynucleosides の直前に溶出する RNA ribonucleosides から?...
ディスカッション
HPLC 法による 8-oxodGuo 解析で見られる主要な問題は、dna 抽出の workup 手順、dna 加水分解、および dna 加水分解物22,38の濃度におけるその形成の可能な誘導である。8-oxodGuo artifactual 形成の問題を最小限に抑えるために、すべての DNA 抽出、貯蔵および加水分解溶液へのデフェロキサミンの添加、DNA 抽出におけるヨウ化ナトリウムカオトロピック法の使?...
開示事項
作者は何も開示することはありません。
謝辞
FAPESP (Fundação ・デ・アンパロà Pesquisa はメヒコ・デ・サンパウロ、2012/22190-3 および 2012/08616-8)、CNPq (Proc. 454214/2014-6 および 429184/2016-6)、ケープ、PRPUSP (Pró・デ・ Reitoria ・ダ・ Pesquisa ・デ・サンパウロ)、セアラ INCT (MCT/INAIRA/CNPQ/岬/FAPEMIG/FAPERJ/FAPESP;Proc. 573813/2008-6)、INCT Redoxoma (FAPESP/CNPq/岬;573530/2008-4)、NAP Redoxoma (PRPUSP;2011.1.9352.1.8) と CEPID Redoxoma (FAPESP;Proc. 2013/07937-8)。F. オリベイラは、FAPESP (Proc. 2012/21636-8, 2011/09891-0, 2012/08617-4) とケープ (Coordenação ・デ・ Aperfeiçoamento ・デ・ Pessoal ・デ・ Nível ・スーペリア) から奨学金を受けました。M. h. g. メデイロス、p. ディ Mascio、p. h. Saldiva、および a. p. m. Loureiro は CNPq からのフェローシップを受け取りました。
この作品に含まれるいくつかの図および表は、ブラジルのサンパウロ市からの濃縮された周囲微細粒子状物質 (PM2.5) に曝露したマウスにおけるオリベイラ A.A.F. et Al. 遺伝毒性および epigenotoxic 効果において最初に出版した。粒子および繊維毒性学。15、40 (2018)。
資料
| Name | Company | Catalog Number | Comments |
| [15N5]-2’-deoxyadenosine | Cambridge Isotope Laboratories | NLM-3895-25 | |
| [15N5]-2’-deoxyguanosine | Cambridge Isotope Laboratories | NLM-3899-CA-10 | |
| acetonitrile | Carlo Erba Reagents | 412413000 | |
| alkaline phosphatase from bovine intestinal mucosa | Sigma | P5521 | |
| ammonium acetate | Merck | 101116 | |
| calf thymus DNA | Sigma | D1501 | |
| cell lysis solution | QIAGEN | 158908 | |
| chloroform | Carlo Erba Reagents | 412653 | |
| deferoxamine | Sigma | D9533 | |
| deoxyribonuclease I (DNase I) | Bio Basic Inc | DD0649 | |
| ethanol | Carlo Erba Reagents | 414542 | |
| formic acid | Sigma-Aldrich | F0507 | |
| HPLC-ESI-MS/MS system | HPLC: Agilent 1200 series ESI-MS/MS: Applied Biosystems/MDS Sciex Instruments | HPLC: binary pump (G1312B), isocratic pump (G1310A), column oven with a column switching valve (G1316B), diode array detector (G1315C), auto sampler (G1367C). ESI-MS/MS: Linear Quadrupole Ion Trap mass spectrometer, Model 4000 QTRAP. | |
| HPLC/DAD system | Shimadzu | Two pumps (LC-20AT), photo diode array detector (DAD-20AV), auto-injector (Proeminence SIL-20AC), column oven (CTO-10AS/VP) | |
| HPLC column (50 x 2.0 mm i.d., 2.5 µm, C18) | Phenomenex | 00B-4446-B0 | |
| HPLC column (150 x 2.0 mm i.d., 3.0 µm, C18) | Phenomenex | 00F-4251-B0 | |
| HPLC column (250 x 4.6 mm i.d., 5.0 µm, C18) | Phenomenex | 00G-4252-E0 | |
| HPLC C18 security guard cartridge (4.0 x 3.0 mm i.d.) | Phenomenex | AJO-4287 | |
| isoamyl alcohol | Sigma-Aldrich | M32658 | |
| isopropyl alcohol (isopropanol) | Carlo Erba Reagents | A412790010 | |
| ketamine | Ceva | Commercial name: Dopalen | |
| magnesium chloride | Carlo Erba Reagents | 349377 | |
| magnesium chloride | Sigma | M2393 | |
| methanol | Carlo Erba Reagents | L022909K7 | |
| phosphodiesterase I from Crotalus atrox | Sigma | P4506 | |
| protein precipitation solution | QIAGEN | 158912 | |
| proteinase K | Sigma-Aldrich | P2308 | |
| ribonuclease A | Sigma | R5000 | |
| sodium chloride | Sigma-Aldrich | S9625 | |
| SPE-C18 (Strata-X) | Phenomenex | 8B-S100-TAK | |
| tris(hydroxymethyl)-aminomethane | Carlo Erba Reagents | 489983 | |
| xylazine | Syntec do Brasil | Commercial name: Xilazin |
参考文献
- Cadet, J., Davies, K. J. A., Medeiros, M. H. G., Di Mascio, P., Wagner, J. R. Formation and repair of oxidatively generated damage in cellular DNA. Free Radical Biology and Medicine. 107, 13-34 (2017).
- Barnes, J. L., Zubair, M., John, K., Poirier, M. C., Martin, F. L. Carcinogens and DNA damage. Biochemical Society Transactions. 46, 1213-1224 (2018).
- Cadet, J., Davies, K. J. A. Oxidative DNA damage & repair: An introduction. Free Radical Biology and Medicine. 107, 2-12 (2017).
- Cao, H., Jiang, Y., Wang, Y. Stereospecific synthesis and characterization of oligodeoxyribonucleotides containing an N2-(1-carboxyethyl)-2'-deoxyguanosine. Journal of the American Chemical Society. 129, 12123-12130 (2007).
- Breyer, V., et al. Analysis and biological relevance of advanced glycation end-products of DNA in eukaryotic cells. The FEBS Journal. 275, 914-925 (2008).
- Tamae, D., Lim, P., Wuenschell, G. E., Termini, J. Mutagenesis and repair induced by the DNA advanced glycation end product N2-1-(carboxyethyl)-2'-deoxyguanosine in human cells. Biochemistry. 50, 2321-2329 (2011).
- Hecht, S. S. Lung carcinogenesis by tobacco smoke. International Journal of Cancer. 131, 2724-2732 (2012).
- Garraway, L. A., Lander, E. S. Lessons from the cancer genome. Cell. 153, 17-37 (2013).
- Ong, T. P., Loureiro, A. P. M. Nutritional interventions in age-related genetic and epigenetic instability and cancer. Anti-ageing nutrients: Evidence-based prevention of age-associated diseases. , (2015).
- Evans, M. D., Dizdaroglu, M., Cooke, M. S. Oxidative DNA damage and disease: induction, repair and significance. Mutation Research. 567, 1-61 (2004).
- Moriya, M. Single-stranded shuttle phagemid for mutagenesis studies in mammalian cells: 8-oxoguanine in DNA induces targeted GC → TA transversions in simian kidney cells. Proceedings of the National Academy of Sciences of the United States of America. 90, 1122-1126 (1993).
- Medeiros, M. H. G. Exocyclic DNA adducts as biomarkers of lipid oxidation and predictors of disease. Challenges in developing sensitive and specific methods for clinical studies. Chemical Research in Toxicology. 22, 419-425 (2009).
- Guéraud, F. 4-Hydroxynonenal metabolites and adducts in pre-carcinogenic conditions and cancer. Free Radical Biology and Medicine. 111, 196-208 (2017).
- Nair, U., Bartsch, H., Nair, J. Lipid peroxidation-induced DNA damage in cancer-prone inflammatory diseases: A review of published adduct types and levels in humans. Free Radical Biology and Medicine. 43, 1109-1120 (2007).
- Pang, B., et al. Lipid peroxidation dominates the chemistry of DNA adduct formation in a mouse model of inflammation. Carcinogenesis. 28, 1807-1813 (2007).
- Møller, P., et al. Harmonising measurements of 8-oxo-7,8-dihydro-2'-deoxyguanosine in cellular DNA and urine. Free Radical Research. 46, 541-553 (2012).
- Hofer, T., Moller, L. Optimization of the workup procedure for the analysis of 8-oxo-7,8-dihydro-2'-deoxyguanosine with electrochemicaldetection. Chemical Research in Toxicology. 15, 426-432 (2002).
- Collins, A., El Yamani, N., Dusinska, M. Sensitive detection of DNA oxidation damage induced by nanomaterials. Free Radical Biology and Medicine. , 69-76 (2017).
- Zubel, T., Buerkle, A., Mangerich, A. Mass spectrometric analysis of sulfur mustard-induced biomolecular adducts: Are DNA adducts suitable biomarkers of exposure?. Toxicology Letters. 293, 21-30 (2018).
- Tretyakova, N., Goggin, M., Sangaraju, D., Janis, G. Quantitation of DNA adducts by stable isotope dilution mass spectrometry. Chemical Research in Toxicology. 25, 2007-2035 (2012).
- Churchwell, M. I., Beland, F. A., Doerge, D. R. Quantification of multiple DNA adducts formed through oxidative stress using liquid chromatography and electrospray tandem mass spectrometry. Chemical Research in Toxicology. 15, 1295-1301 (2002).
- Chao, M. R., Yen, C. C., Hu, C. W. Prevention of artifactual oxidation in determination of cellular 8-oxo-7,8-dihydro-2'-deoxyguanosine by isotope-dilution LC-MS/MS with automated solid-phase extraction. Free Radical Biology and Medicine. 44, 464-473 (2008).
- Danielsen, P. H., et al. Oxidative stress, inflammation, and DNA damage in rats after intratracheal instillation or oral exposure to ambient air and wood smoke particulate matter. Toxicological Sciences. 118, 574-585 (2010).
- Danielsen, P. H., et al. Oxidative stress, DNA damage, and inflammation induced by ambient air and wood smoke particulate matter in human A549 and THP-1 cell lines. Chemical Research in Toxicology. 24, 168-184 (2011).
- Garcia, C. C. M., et al. [13C2]-Acetaldehyde promotes unequivocal formation of 1,N2-propano-2'-deoxyguanosine in human cells. Journal of the American Chemical Society. 133, 9140-9143 (2011).
- Angeli, J. P. F., et al. Lipid hydroperoxide-induced and hemoglobin-enhanced oxidative damage to colon cancer cells. Free Radical Biology and Medicine. 51, 503-515 (2011).
- Yu, Y., et al. Comprehensive assessment of oxidatively induced modifications of DNA in a rat model of human Wilson's disease. Molecular and Cellular Proteomics. 15, 810-817 (2016).
- Torres-Cuevas, I., Aupi, M., Asensi, M. A., Vento, M., Ortega, &. #. 1. 9. 3. ;., Escobar, J. 7,8-Hydroxy-2'-deoxyguanosine/2'-deoxiguanosine ratio determined in hydrolysates of brain DNA by ultrachromatrography coupled to tandem mass spectrometry. Talanta. 170, 97-102 (2017).
- Wu, D., et al. Detection of 8-hydroxydeoxyguanosine (8-OHdG) as a biomarker of oxidative damage in peripheral leukocyte DNA by UHPLC-MS/MS. Journal of Chromatography B. 1064, 1-6 (2017).
- IARC. . Monographs on the Evaluation of Carcinogenic Risks to Humans: Outdoor Air Pollution. 109, (2016).
- De Martinis, B. S., Kado, N. Y., Carvalho, L. R. F., Okamoto, R. A., Gundel, L. A. Genotoxicity of fractionated organic material in airborne particles from São. Mutation Research. 446, 83-94 (1999).
- Karlsson, H. L., Nygren, J., Möller, L. Genotoxicity of airborne particulate matter: The role of cell-particle interaction and of substances with adduct-forming and oxidizing capacity. Mutation Research. 565, 1-10 (2004).
- Bell, M. L., Dominici, F., Ebisu, K., Zeger, S. L., Samet, J. M. Spatial and temporal variation in PM2.5 chemical composition in the United States for health effects studies. Environmental Health Perspectives. 115, 989-995 (2007).
- Oliveira, A. A. F., et al. Genotoxic and epigenotoxic effects in mice exposed to concentrated ambient fine particulate matter (PM2.5) from São Paulo city, Brazil. Particle and Fibre Toxicology. 15, 40 (2018).
- Loureiro, A. P. M., Zhang, W., Kassie, F., Zhang, S., Villalta, P. W., Wang, M., Hecht, S. S. Mass spectrometric analysis of a cyclic 7,8-butanoguanine adduct of N-nitrosopyrrolidine: comparison to other N-nitrosopyrrolidine adducts in rat hepatic DNA. Chemical Research in Toxicology. 22, 1728-1735 (2009).
- Loureiro, A. P. M., Marques, S. A., Garcia, C. C. M., Di Mascio, P., Medeiros, M. H. G. Development of an on-line liquid chromatography-electrospray tandem mass spectrometry assay to quantitatively determine 1,N2-etheno-2'-deoxyguanosine in DNA. Chemical Research in Toxicology. 15, 1302-1308 (2002).
- Mangal, D., et al. Analysis of 7,8-dihydro-8-oxo-2′-deoxyguanosine in cellular DNA during oxidative stress. Chemical Research in Toxicology. 22, 788-797 (2009).
- ESCODD (European Standards Committee on Oxidative DNA Damage). Comparative analysis of baseline 8-oxo-7,8-dihydroguanine in mammalian cell DNA, by different methods in different laboratories: an approach to consensus. Carcinogenesis. 23, 2129-2133 (2002).
- Helbock, H. J., et al. DNA oxidation matters: The HPLC-electrochemical detection assay of 8-oxo-deoxyguanosine and 8-oxo-guanine. Proceedings of the National Academy of Sciences of the United States of America. 95, 288-293 (1998).
- Risom, L., et al. Oxidative DNA damage and defence gene expression in the mouse lung after short-term exposure to diesel exhaust particles by inhalation. Carcinogenesis. 24, 1847-1852 (2003).
- Risom, L., et al. Repeated inhalations of diesel exhaust particles and oxidatively damaged DNA in young oxoguanine DNA glycosylase (OGG1) deficient mice. Free Radical Research. 41, 172-181 (2007).
- Tsurudome, Y., et al. Changes in levels of 8-hydroxyguanine in DNA, its repair and OGG1 mRNA in rat lungs after intratracheal administration of diesel exhaust particles. Carcinogenesis. 20, 1573-1576 (1999).
- Marie-Desvergne, C., Maître, A., Bouchard, M., Ravanat, J. L., Viau, C. Evaluation of DNA adducts, DNA and RNA oxidative lesions, and 3-hydroxybenzo(a)pyrene as biomarkers of DNA damage in lung following intravenous injection of the parent compound in rats. Chemical Research in Toxicology. 23, 1207-1214 (2010).
- Iwai, K., et al. Early oxidative DNA damages and late development of lung cancer in diesel exhaust-exposed rats. Environmental Research. 84, 255-264 (2000).
- Ichinose, T., et al. Lung carcinogenesis and formation of 8-hydroxy-deoxyguanosine in mice by diesel exhaust particles. Carcinogenesis. 18, 185-192 (1997).
- Schmerold, I., Niedermu, H. Levels of 8-hydroxy-2'-deoxyguanosine in cellular DNA from 12 tissues of young and old Sprague Dawley rats. Experimental Gerontology. 36, 1375-1386 (2001).
- Garcia, C. C. M., Freitas, F. P., Di Mascio, P., Medeiros, M. H. G. Ultrasensitive simultaneous quantification of 1,N2-etheno-2'-deoxyguanosine and 1,N2-propano-2'-deoxyguanosine in DNA by an online liquid chromatography-electrospray tandem mass spectrometry assay. Chemical Research in Toxicology. 23, 1245-1255 (2010).
- Godshalk, R., et al. Comparison of multiple DNA adduct types in tumor adjacent human lung tissue: effect of cigarette smoking. Carcinogenesis. 23, 2081-2086 (2002).
- Dechakhamphu, S., et al. Lipid peroxidation and etheno DNA adducts in white blood cells of liver fluke-infected patients: protection by plasma alpha-tocopherol and praziquantel. Cancer Epidemiology Biomarkers and Prevention. 19, 310-318 (2010).
- Arab, K., et al. Typical signature of DNA damage in white blood cells: a pilot study on etheno adducts in Danish mother-newborn child pairs. Carcinogenesis. 30, 282-285 (2009).
- Nair, J., et al. High dietary omega-6 polyunsaturated fatty acids drastically increase the formation of etheno-DNA base adducts in white blood cells of female subjects. Cancer Epidemiology Biomarkers and Prevention. 6, 597-601 (1997).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved