È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Quantificazione di tre lesioni del DNA mediante spettrometria di massa e valutazione dei loro livelli nei tessuti di topi esposti a particolato fine ambientale
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Descriviamo qui metodi per la quantificazione sensibile e accurata delle lesioni 8-oxo-7, 8-diidro-2'-deossiguanosina (8-oxodGuo), 1,n6-etheno-2'-deossiadenosina (1,n6-dAdo) e 1,n2- etheno-2a '-deossiguanosina (1,N2-DGUO) nel DNA. I metodi sono stati applicati alla valutazione degli effetti del particolato fine ambientale (PM2,5) nei tessuti (polmone, fegato e rene) dei topi a/J esposti.
Abstract
Gli addotti del DNA e le basi di DNA ossidato sono esempi di lesioni del DNA che sono biomarcatori utili per la valutazione della tossicità di sostanze che sono elettrofiliche, generano elettrofili reattivi sulla biotrasformazione o inducono lo stress ossidativo. Tra i nucleobasi ossidati, il più studiato è 8-oxo-7, 8-diidroguanina (8-oxoGua) o 8-oxo-7, 8-diidro-2'-deossiguanosina (8-oxodGuo), un biomarcatore di danno di base ossidativamente indotto nel DNA. Aldeidi ed epoxyaldeidi risultanti dal processo di perossidazione lipidica sono molecole elettrofiliche in grado di formare addotti mutageni del DNA esociclico, come gli addotti addotti 1,n2-addotti-2'-deoxyguanosina (1,n2- εdGuo) e 1,n6-etheno-2'-deossiadenosina (1,n6-εdado), che sono stati suggeriti come potenziali biomarcatori nella fisiopatologia dell'infiammazione. Metodi selettivi e sensibili per la loro quantificazione nel DNA sono necessari per lo sviluppo di strategie preventive per rallentare i tassi di mutazione cellulare e lo sviluppo di malattie croniche (ad esempio, cancro, malattie neurodegenerative). Tra i metodi sensibili disponibili per la loro rilevazione (cromatografia liquida ad alte prestazioni accoppiata ai rivelatori di spettrometria di massa elettrochimica o tandem, test Comet, immunodosaggi, 32P-postetichettatura), le più selettive sono quelle basate su cromatografia liquida ad alte prestazioni accoppiata alla spettrometria di massa tandem (HPLC-ESI-MS/MS). La selettività è un vantaggio essenziale nell'analisi di campioni biologici complessi e l'HPLC-ESI-MS/MS si è evoluto come lo standard Gold per la quantificazione dei nucleosidi modificati nelle matrici biologiche, come il DNA, l'urina, il plasma e la saliva. L'uso di standard interni con etichetta isotopica aggiunge il vantaggio delle correzioni per le perdite di molecola durante le fasi di arricchimento dell'idrolisi e dell'analita del DNA, nonché per le differenze di ionizzazione dell'analita tra i campioni. Aiuta anche a identificare il picco cromatografico corretto quando è presente più di un picco.
Qui vi presentiamo i metodi HPLC-ESI-MS/MS, sensibili, accurati e precisi, che sono stati applicati con successo per la quantificazione di 8-oxodGuo, 1,n6-dado e 1,n2-DGUO nel polmone, nel fegato e nel DNA dei reni dei topi a/J per valutazione degli effetti dell'esposizione ambientale PM2,5 .
Introduzione
Alcune specie reattive dell'ossigeno (ROS) sono in grado di ossidare doppi legami di carbonio di basi di DNA e alcuni carboni nella frazione desossribosio, generando basi ossidate e rottura del filamento del DNA1. Come una molecola carica negativamente ricca di atomi di azoto e ossigeno, il DNA è anche un bersaglio per i gruppi elettrofilici che covalentemente reagiscono con i siti nucleofili (azoto e ossigeno), dando prodotti che sono chiamati addotti del DNA2. Quindi, gli addotti del DNA e le basi del DNA ossidato sono esempi di lesioni del DNA che sono biomarcatori utili per la valutazione della tossicità di sostanze che sono elettrofiliche, generano elettrofili reattivi sulla biotrasformazione, o inducono lo stress ossidativo1, 2. il Anche se le basi di DNA modificate possono essere rimosse dal DNA per riparazione di escissione di base o nucleotide (BER o NER), l'induzione di uno squilibrio tra la generazione e la rimozione delle lesioni del DNA a favore della prima porta ad un aumento netto dei loro livelli nel lavoro straordinario del DNA3 < /C5 >. I risultati sono l'aumento dei tassi di mutazione del DNA, la riduzione dell'espressione genica e la diminuita attività proteica2,4,5,6,7, effetti che sono strettamente correlati alla sviluppo di malattie. Le mutazioni del DNA possono influenzare diverse funzioni cellulari, come la segnalazione cellulare, il ciclo cellulare, l'integrità del genoma, la stabilità dei telomeri, l'epigenoma, la struttura della cromatina, lo splicing dell'RNA, l'omeostasi proteica, il metabolismo, l'apoptosi e la differenziazione cellulare8 ,9. Le strategie per rallentare i tassi di mutazione cellulare e lo sviluppo di malattie croniche (ad esempio, cancro, malattie neurodegenerative) passano attraverso la conoscenza delle fonti di mutazione, tra cui le lesioni del DNA e le loro cause.
ROS ha generato in eccesso in modo endogeno, a causa di esposizione di inquinanti, infiammazione persistente, patofisiologia della malattia (ad esempio, diabete), ecc., sono cause importanti di danni biomolecola, tra cui il danno del DNA e del lipido1. Ad esempio, il radicale idrossile altamente reattivo (OH) formato da H2O2 riduzione degli ioni metallici di transizione (Fe2 +, cu+) ossida le basi del DNA, la porzione di zucchero del DNA e gli acidi grassi polinsaturi a diffusione controllata tassi di cambio10. Tra i 80 già caratterizzati nucleobasi ossidate3, l'uno più studiato è 8-oxo-7, 8-diidroguanina (8-oxoGua) o 8-oxo-7, 8-dihydro-2'-deossiguanosina (8-oxodGuo, Figura 1), una lesione che è in grado di indurre le trasversioni gt in cellule di mammifero10,11. È formata dall'ossidazione mono-elettronica della guanina, o da un attacco di ossigeno radicale idrossile o singollet di guanina nel DNA1. Gli acidi grassi polinsaturi sono altri importanti bersagli di ossidanti altamente reattivi, quali •Oh, che avviano il processo di perossidazione lipidica1,12. Dà origine a idroperossidi di acidi grassi che possono decomporre a aldeidi elettrofiliche ed epoxyaldeidi, come malondialdeide, 4-idrossi-2-nonenal, 2, 4-decadienal, 4,5-epossidico-(2E)-decenale, hexenal, Acrolein, crotonaldeide, che sono in grado di formare addotti mutageni del DNA esociclico, come malondialdeide-, propano-, o addotti di addotti1,12,13. L'addotti addotti 1,n2-addotti-2'-deoxyguanosine (1,n2-εdguo, Figura 1) e 1,n6-addotti-2'-deossiadenosina (1,n6-εdado, Figura 1 ) sono stati suggeriti come potenziali biomarcatori nella patofisiologia dell'infiammazione14,15.
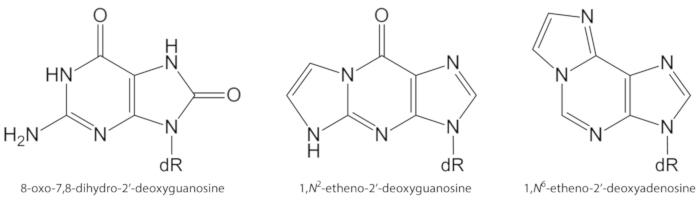
Figura 1. Strutture chimiche delle lesioni del DNA quantificate nel presente studio. dR = 2 ́-desossribosio. Questa cifra è stata modificata da Oliveira et al.34. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
Gli studi condotti nei primi anni ottanta hanno permesso la rilevazione sensibile di 8-oxodGuo mediante cromatografia liquida ad alte prestazioni accoppiata al rilevamento elettrochimico (HPLC-ECD). Quantificazione di 8-oxodGuo da HPLC-ECD in diversi sistemi biologici sottoposti a condizioni ossidanti ha portato al riconoscimento di 8-oxodGuo come un biomarcatore di danno base ossidativamente indotto in DNA1,16. Sebbene robusto e che consenta la quantificazione di 8-oxodGuo nella gamma bassa fmol17, le misurazioni HPLC-ECD si basano sull'accuratezza del tempo di ritenzione dell'analita per l'identificazione dell'analita e sulla risoluzione della cromatografia per evitare interferenze di altri costituenti campione. Poiché la rilevazione elettrochimica richiede l'uso di sale (ad esempio fosfato di potassio, acetato di sodio) nella fase mobile, il mantenimento di condizioni analitiche adeguate necessita di routine di pulizia delle colonne e delle attrezzature.
In alternativa, l'uso dell'enzima di riparazione del DNA batterico formamidopirimidina DNA glicosilasi (FPG) e, successivamente, umana 8-oxoguanina glicosilasi 1 (hOGG1), per il rilevamento e la rimozione di 8-oxoGua dal DNA, è emerso come un modo per l'induzione del DNA alcalino labile Siti. I siti alcalini labili vengono convertiti in rotture del filamento del DNA e consentono la quantificazione indiretta molto elevata sensibile di 8-oxoGua mediante elettroforesi di gel monocellulare alcalina ("saggio Comet"). L'alta sensibilità e la realizzazione delle analisi senza la necessità di estrazione del DNA cellulare sono i principali vantaggi di questo tipo di saggio. Fornisce i livelli più bassi di stato stazionario di 8-oxoGua nel DNA, tipicamente 7-10 volte inferiori ai livelli ottenuti dai metodi bioanalitici basati su HPLC. Tuttavia, è una misura indiretta di 8-oxogua e alcuni inconvenienti sono la mancanza di specificità o l'efficienza sconosciuta degli enzimi di riparazione utilizzati1,16,18.
Gli immunodosaggi sono altri metodi utilizzati per la rilevazione di addotti di 8-oxoGua1 e di DNA esociclico, come 1,n6-dAdo e 1,n2-dGuo12. Nonostante la sensibilità, una carenza dell'uso di anticorpi per il rilevamento delle lesioni del DNA è la mancanza di specificità a causa della reattività incrociata ad altri componenti di campioni biologici, comprese le normali basi di DNA1,12. Gli addotti del DNA exociclico, compresi 1, n6-dado e 1, n2-dGuo, possono essere rilevati e quantificati da 32P-saggi di postetichettatura12. L'elevata sensibilità di 32P-postetichettatura consente l'uso di piccolissime quantità di DNA (ad es. 10 μg) per il rilevamento di circa 1 addotto per 1010 basi normali19. Tuttavia, l'uso di radio-chimiche, mancanza di specificità chimica e bassa precisione sono alcuni svantaggi19,20.
Una limitazione condivisa dei metodi citati sopra è la bassa selettività o specificità per la rilevazione delle molecole desiderate. In questo scenario, l'HPLC accoppiato alla spettrometria di massa tandem a ionizzazione elettrospray (HPLC-ESI-MS/MS e HPLC-MS3) si è evoluto come lo standard Gold per la quantificazione dei nucleosidi modificati nelle matrici biologiche, come il DNA, l'urina, il plasma e la saliva 1 il , 19 anni di , 20. i vantaggi dei metodi HPLC-ESI-MS/MS sono la sensibilità (tipicamente nella gamma bassa fmol) e l'elevata specificità fornita da i) la separazione cromatografica, II) il modello caratteristico e noto della frammentazione molecolare all'interno della massa Camera di collisione dello spettrometro, e III) la misurazione accurata del rapporto massa/carica selezionato (m/z) in modalità di monitoraggio a reazione multipla1,19. L'uso di standard interni con etichetta isotopica aggiunge il vantaggio delle correzioni per le perdite di molecola durante le fasi di arricchimento dell'idrolisi e dell'analita del DNA, nonché per le differenze di ionizzazione dell'analita tra i campioni. Aiuta anche a identificare il picco cromatografico corretto quando più di un picco è presente1,12,19,20.
Diversi metodi basati su HPLC-ESI-MS/MS sono stati utilizzati per la quantificazione di 8-oxodGuo, 1,N6-dAdo e 1,n2-dGuo nel DNA Estratto da diversi campioni biologici12,15,20 ,21,22,23,24,25,26,27,28,29 . Le particelle sottili (PM2,5) trasportano sostanze chimiche organiche e inorganiche, quali idrocarburi policiclici aromatici (IPA), Nitro-IPA, aldeidi, chetoni, acidi carbossilici, chinolini, metalli e ioni solubili in acqua, che possono indurre infiammazione e stress ossidativo, condizioni che favoriscono il verificarsi di biomolecola danni e malattia30,31,32,33. Qui presentiamo i metodi HPLC-ESI-MS/MS convalidati che sono stati applicati con successo per la quantificazione di 8-oxodGuo, 1,n6-dado e 1,n2-dGuo in polmone, fegato e DNA renale di un/J topi per la valutazione del effetti dell'esposizione ambientale PM 2,534.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Quattro settimana vecchio maschio A/J topi, patogeno specifico libero, sono stati ottenuti dal centro di allevamento di animali da laboratorio di Fundação Oswaldo Cruz (FIOCRUZ), Rio de Janeiro, Brasile, e sono stati trattati di conseguenza al comitato etico della facoltà di medicina, Università di San Paolo (protocollo no 1310/09).
1. raccolta dei tessuti dei topi
- Anestetizzare l'animale con xylazina e chetamina. Per un topo con 30 g di peso corporeo, iniettare una soluzione (non più di 2 mL) contenente 2,63 mg di ketamina e 0,38 mg di Xylazine, intraperitoneale.
- Raccogliere il sangue (0,5-1,5 mL) per analisi complementari (ad es. attività enzimatica antiossidante, livelli di malondialdeide).
- Radersi i peli addominali dal bacino al processo xifoideo. Fare un'incisione in una linea mediana verticale nell'area senza peli. Fare incisioni in linee laterali orizzontali al fine di esporre gli organi addominali.
- Tagliare l'aorta addominale per promuovere l'espirazione e per eutanizzare l'animale.
- Rimuovere i tessuti di interesse (in questo caso, fegato, reni e polmoni).
- Per rimuovere il fegato, tagliare la vena di cava inferiore e la vena epatica del portale.
- Per rimuovere i reni, sezione le vene renali e le arterie.
- Per rimuovere i polmoni, fare un'incisione nelle estremità del diaframma e circonferenza vicino alla parete toracica. Rompere le clavicole aprendo una forche all'interno della cavità toracica. Tagliare l'osso extern dal processo xifoideo verso la trachea, al fine di esporre i polmoni e il cuore.
- Tenere il polmone con una pinza, sezione la trachea e i legamenti intorno ai polmoni. Rimuovere accuratamente i polmoni blocco più cuore. Per rimuovere i polmoni dal blocco, tenere il cuore con una pinza e tagliare tutti i vasi nella sua base.
- Lavare immediatamente i tessuti isolati in soluzione salina fredda (0,9% NaCl), trasferire a tubi criogenici e immergere immediatamente i tubi in azoto liquido. Dopo aver completato il lavoro, conservare i tubi a-80 ° c.
Attenzione: l'azoto liquido a contatto diretto con la pelle, la mucosa o gli occhi provoca ustioni. Utilizzare una protezione individuale adeguata per evitare il contatto. Lavorare in un laboratorio ventilato per evitare l'asfissia a causa del vapore di azoto liquido.
2. estrazione del DNA
Nota: il metodo di estrazione del DNA è stato modificato da Loureiro et al. (2009)35 per consentire l'analisi delle lesioni studiate qui.
- Trasferire i tubi contenenti i tessuti per asciugare il ghiaccio.
- Utilizzare una piastra di coltura posta sul ghiaccio come base per tagliare un pezzo di tessuto con un bisturi. Peso 1 g per l'uso immediato. Il tessuto rimanente deve essere mantenuto sul ghiaccio asciutto fino a quando non ritorna in deposito a-80 ° c.
Nota: è importante evitare lo scongelamento del tessuto rimanente per prevenire la formazione di artefatti se sono necessarie ripetizioni delle analisi. - Per ogni 1 g di tessuto in 50 mL di tubi con tappo, aggiungere 10 mL della soluzione di lisi cellulare commerciale contenente 0,5 mM di deferossamina e mantenere il ghiaccio.
Nota: aggiungere deferoxamina al volume di soluzione per l'uso immediato. Per ogni 100 mL di soluzione, aggiungere 0,0328 g di sale mesilato di deferossamina. - Omogeneizzare i tessuti utilizzando un omogeneizzatore tissutale fino a ottenere una soluzione omogenea senza frammenti di tessuto. Mantenere il tubo freddo (sul ghiaccio) durante l'omogeneizzazione. Utilizzare una bassa velocità per evitare il riscaldamento.
- Aggiungere 150 μL di soluzione di proteinasi K (20 mg/mL) ad ogni campione omogeneizzato. Agitare i tubi per inversione e tenerli a temperatura ambiente durante la notte.
- Aggiungere 40 μL di ribonucleasi una soluzione (15 mg/mL), agitare per inversione e tenere i tubi a temperatura ambiente per 2 h.
Nota: preparare la ribonucleasi soluzione in tampone di acetato di sodio 10 mM, pH 5,2 per evitare precipitazioni. Riscaldare la soluzione a 100 ° c per 15 minuti prima dell'uso per ottenere una soluzione priva di deoxyribonuclease. - Aggiungere 5 mL della soluzione di precipitazione proteica commerciale, agitare vigorosamente e centrifugare a 2.000 x g, 4 ° c, per 10 minuti.
- Trasferire i supernatanti a 50 mL di tubi con tappo contenente 10 mL di isopropanolo freddo. Invertire i tubi delicatamente più volte fino a quando l'osservazione del DNA precipitato.
Nota: il protocollo può essere messo in pausa, mantenendo i tubi a-20 ° c. - Raccogli il DNA precipitato usando una pipetta Pasteur chiusa alla fine. Trasferirlo a provette contenenti 4 mL di tampone Tris da 10 mM, 1 mM di deferossamina, pH 7,0.
- Dopo che il DNA è completamente disciolto nella soluzione di cui sopra (non agitare), aggiungere 4 mL di una soluzione di cloroformio contenente il 4% di alcol isoamilico.
- Invertire i tubi 10 volte per l'omogeneizzazione, centrifugare a 2.000 x g, 4 ° c, per 10 minuti per separare le due fasi, e trasferire la fase superiore ad un nuovo tubo.
- Ripetere i passaggi 2,10 e 2,11 altre due volte.
- Aggiungere 8 mL di etanolo assoluto e 0,4 mL di soluzione di NaCl da 5 M per precipitato il DNA.
- Raccogliere di nuovo il DNA precipitato e trasferirlo a 3 mL di 70% etanolo. Ripeti questo passaggio ancora una volta.
- Gettare la soluzione di etanolo con cautela e invertire i tubi contenenti il DNA precipitato su carta assorbente per rimuovere l'eccesso della soluzione.
- Aggiungere 200 μL di soluzione di deferossamina 0,1 mM per sciogliere il DNA. Mantenere i tubi a 4 ° c fino a quando il DNA è completamente reidratato (durante la notte).
- Determinare la concentrazione di DNA misurando l'assorbanza a 260 Nm e la sua purezza con il rapporto di assorbanza 260/280 Nm.
Nota: per determinare la concentrazione del DNA, trasferire un'aliquota di 10 μL della soluzione di DNA a 990 μL di acqua ultrapura (diluizione 100x). Moltiplicare l'assorbanza a 260 Nm (dovrebbe essere inferiore a 1) per 50 (50 μg/mL è la concentrazione di DNA a doppio filamento quando l'assorbanza di una soluzione di lunghezza di percorso di 1 cm a 260 Nm è 1) e la diluizione utilizzata (100x) per ottenere la concentrazione di DNA in μg/mL. Se l'assorbanza a 260 Nm è superiore a 1, sono necessarie ulteriori diluizioni. Il rapporto di assorbanza 260/280 nm deve essere uguale o superiore a 1,8 per la purezza del DNA desiderata, ma i rapporti intorno 1,6 sono accettabili.
3. idrolisi enzimatica del DNA
- Analisi ricetta
- 1,n6-εdado e 1,n2-εdguo analisi: a un'aliquota contenente 150 μg di DNA, aggiungere 7,5 μl di 200 mm di Tris/mgcl2 tampone (pH 7,4), 1,4 μL della soluzione standard interna contenente 250 fmol/μl di [15N5 ] 1,n6-εdado e [15N5] 1,n2-εdguo e 15 unità di deossiribonucleasi i. regolare il volume finale a 200 μl con acqua ultrapura, sottraendo i volumi di enzimi da utilizzare nel passaggio 3.2.1.
- analisi 8-oxodGuo: per un'aliquota contenente 80 μg di DNA, aggiungere 3,8 μl di tampone Tris/MgCl2 da 200 mm (pH 7,4), 2 μl della soluzione standard interna contenente 1.000 fmol/μl di [15N5] 8-oxodGuo e 8 unità di deossiribonucleasi I. Regolare il volume finale a 100 μL con acqua ultrapura, sottraendo i volumi di enzimi da utilizzare nel passaggio 3.2.2.
Nota: gli standard interni [15n5] 1,n6-εdado, [15 n5] 1,n2-εdguo e [15n5] 8-oxodGuo possono essere sintetizzati e caratterizzati come descritto35,36,37. Le quantità degli standard interni nei volumi dei campioni iniettati devono essere uguali a quelle dei volumi delle curve di calibrazione iniettate.
- Incubare i campioni a 37 ° c per 1 ora.
- Campioni dal passo 3,1: aggiungere 0,006 unità di fosfodiesterasi I da Crotalus atrox e 15 unità di fosfatasi alcalina dalla mucosa intestinale bovina.
- Campioni dal passo 3,2: aggiungere 0,0032 unità di fosfodiesterasi I da Crotalus atrox e 8 unità di fosfatasi alcalina dalla mucosa intestinale bovina.
- Incubare i campioni a 37 ° c per 1 ora.
- Centrifugare i campioni a 14.000 x g per 10 minuti.
- Campioni dal punto 3.2.1: separare 10 μL di ciascun campione per la quantificazione dei deossinucleosidi (dAdo, dGuo) da HPLC/DAD (passaggio 9). Sottoporre il volume residuo all'estrazione in fase solida (fase 4).
- Campioni dal punto 3.2.2: trasferire 80 μL di supernatante ai flaconcini per preparazioni iniettabili di 50 μL (1.000 fmol di [15N5] 8-oxodGuo) nel sistema HPLC-ESI-MS/MS. Riservare i restanti 20 μL per la quantificazione di dGuo da HPLC/DAD (passaggio 9).
4. estrazione in fase solida per analisi di 1, n6-εdado e 1, n2-εdguo
- Caricare le cartucce (SPE-C18, 30 mg/mL, 33 μm, 1 mL) con 1 mL della seguente sequenza di soluzioni: 100% metanolo, acqua deionizzata, campione di DNA idrolizzato, acqua deionizzata, 10% metanolo, 15% metanolo e 100% metanolo (da raccogliere).
Nota: non lasciare le cartucce asciutte tra le applicazioni delle diverse soluzioni. Aggiungere la soluzione successiva immediatamente dopo che la soluzione precedente entra completamente nella cartuccia. - Asciugare sottovuoto l'ultima frazione di eluizione (100% metanolo) contenente gli addotti.
- Risospendere i campioni essiccati in 83,1 μL di acqua ultrapura immediatamente prima dell'analisi HPLC-ESI-MS/MS, per ottenere 200 fmol di ogni norma interna in 50 μL di ciascun campione.
5. preparazione delle curve di calibrazione
- Preparare almeno cinque punti nell'intervallo di 300 a 6.000 fmol di standard 8-oxodGuo, con la quantità fissa di 1.000 fmol di [15N5] 8-oxodGuo in ogni punto. Considerare questi importi nel volume iniettato.
- Preparare almeno cinque punti nell'intervallo da 1 a 40 fmol di 1,n6-εdado e 1,n2-εdguo, con importi fissi di 200 fmol di [15n5] 1,n6-εdado e [15n5] 1, N 2-εdguo in ogni punto. Considerare questi importi nel volume iniettato.
- Preparare almeno cinque punti nell'intervallo di 0,05-1 nmol di dGuo e dAdo. Considerare questi importi nel volume iniettato.
6. preparazione di campioni di DNA per la validazione del metodo
- analisi 1, n6-Εdado e 1,n2-Εdguo: aggiungere quantità variabili di 1,n6-εdado e 1,n2-εdguo (ad esempio, 1,75, 8,75, 17,5 e 35 fmol) e importi fissi di [15N5] 1, N6-εdado e [15N5] 1,n2-ΕDGUO (350 fmol) a 100 μg di DNA di timo di vitello ed eseguire l'idrolisi enzimatica come descritto al punto 3. Elabora i campioni in quadruplicato in due giorni diversi. Utilizzare i campioni per la precisione del metodo e la valutazione di precisione.
Nota: il volume finale degli idrosati di DNA sarà di 200 μL (fase 3), da cui 10 μL saranno separati per la quantificazione dei deossinucleosidi da parte di HPLC/DAD (passaggio 9). La soluzione rimanente (190 μL) sarà sottoposta ad estrazione in fase solida (fase 4), la frazione essiccata sarà risospesa in 83,1 μL (Step 4,3), da cui saranno iniettati 50 μL nel sistema HPLC-ESI-MS/MS. Gli importi di 1,n6-Εdado e 1,n2-εdguo iniettati saranno 1, 5, 10 e 20 fmol, con 200 fmol di [15n5] 1,n6-εdado e [15n5] 1,n 2-εdguo in ogni campione. - analisi 8-oxodGuo: aggiungere una quantità variabile di 8-oxodGuo (ad esempio, 734, 1.468, 2.938 e 4.408 fmol) e una quantità fissa di [15N5] 8-oxodGuo (2.000 fmol) a 100 μg di DNA di timo di vitello ed eseguire l'idrolisi enzimatica come descritto al punto 3. Elabora i campioni in quadruplicato in due giorni diversi. Utilizzare i campioni per la precisione del metodo e la valutazione di precisione.
Nota: il volume finale degli idrosati di DNA sarà 100 μL (fase 3), da cui 50 μL saranno iniettate nel sistema HPLC-ESI-MS/MS. Gli importi di 8-oxodGuo iniettato saranno 367, 734, 1469, e 2204 fmol, con 1.000 fmol di [15N5] 8-oxodGuo in ogni campione. - Aggiungere 13,125 fmol di 1,N6-εdado (per ottenere 7,5 fmol nel volume di iniezione) e 35 fmol di 1,n2-εdguo (per ottenere 20 fmol nel volume di iniezione) a otto campioni di 100 μg di DNA di timo di vitello.
- Aggiungere gli standard interni [15n5] 1,n6-εdado e [15n5] 1,n2-εdguo (200 fmol) a quattro dei campioni. Procedere con l'idrolisi del DNA e l'estrazione in fase solida di tutti i campioni.
- Aggiungere gli standard interni [15n5] 1,n6-εdado e [15n5] 1,n2-εdguo (200 fmol) agli altri quattro campioni.
- Utilizzare i campioni per calcolare il recupero degli addotti da estrazione in fase solida.
7. HPLC-ESI-MS/MS analisi di 8-oxodGuo
- Utilizzando lo standard 8-oxodGuo nell'apparecchiatura, impostare i parametri ESI-MS/MS per la migliore rilevazione del suo modello di frammentazione mediante il monitoraggio multiplo delle reazioni (MRM): m/z 284 [m + h] + → m/z 168 [m-2'-deoxyribose + h]+.
- Utilizzare gli stessi parametri per il rilevamento di[15N5] 8-oxodGuo : m/z 289 [m + H]+m/z → 173 [m-2'-deoxyribose + H]+.
Nota: utilizzare un equipaggiamento equivalente o migliore rispetto all'apparecchiatura utilizzata in questo lavoro (vedere la tabella dei materiali). I parametri ESI-MS/MS sono stati impostati come descritto nella tabella 1.
- Utilizzare gli stessi parametri per il rilevamento di[15N5] 8-oxodGuo : m/z 289 [m + H]+m/z → 173 [m-2'-deoxyribose + H]+.
- Filtro (utilizzando membrane porose 0,22 μm) e degassano (utilizzando un sonicatore) tutti i solventi HPLC a base acquosa.
- Utilizzare le seguenti condizioni di cromatografia per le analisi, montando il sistema come mostrato in Figura 2.
Nota: la colonna A è collegata alla pompa binaria. Il suo eluente è diretto alla rilevazione UV e ai rifiuti nei primi 16 min e da 32 a 46 min della cromatografia, come mostrato nella Figura 2a. Questa è la colonna attraverso la quale il campione viene eluito immediatamente dopo l'iniezione. La colonna B è collegata alla pompa isocratica e allo spettrometro di massa. Riceve l'eluente della colonna A solo nell'intervallo di 16-32 min, quando la valvola passa alla posizione mostrata nella Figura 2B. La commutazione della valvola consente il collegamento tra le due colonne, che sono eluse dal gradiente della pompa binaria. La configurazione mostrata nella Figura 2B consente un'ulteriore separazione di picco e restringimento. Inoltre, solo la frazione cromatografica di interesse raggiunge lo spettrometro di massa, migliorando la sensibilità e la selettività.- Elute un 50 x 2,0 mm d.i., 2,5 μm, C18 colonna (colonna A della Figura 2) accoppiato a una cartuccia di protezione c18 (4,0 x 3,0 mm d.i.) con un gradiente di 0,1% acido formico (solvente a) e metanolo contenente 0,1% acido formico (solvente B) ad una portata di 150 μl/min e a 25 ° c.
- Utilizzare il seguente programma di gradiente per la pompa binaria: da 0 a 25 min, 0-15% di solvente B; da 25 a 28 min, 15-80% di solvente B; 28 a 31 min, 80% di solvente B; da 31 a 33 min, 80-0% di solvente B; 33 a 46 min, 0% di solvente B.
- Utilizzare la valvola di commutazione per dirigere i primi 16 min di eluente ai rifiuti e la frazione 16-32 min ad una seconda colonna (150 x 2,0 mm d.i., 3,0 μm, C18, colonna B della Figura 2) collegato alla sorgente ESI e condizionato dalla pompa isocratica con una soluzione di 15% me etanolo in acqua contenente 0,1% di acido formico (150 μL/min).
Nota: prima di utilizzare il programma della valvola di commutazione del passo 7.3.1.2, controllare se le 8-oxodGuo standard durante dalla prima colonna dopo 16 min. È importante chiudere la valvola a 32 minuti per utilizzare il gradiente della pompa binaria per eluto 8-oxodGuo dalla seconda colonna e ottenere un picco cromatografico acuto. La lesione 8-oxodGuo eluti dalla seconda colonna a circa 36 min. variazioni del tempo di ritenzione dell'analita possono verificarsi a seconda della colonna e delle attrezzature utilizzate. Possono essere necessari adattamenti del programma di gradiente di solvente HPLC.
- Elute un 50 x 2,0 mm d.i., 2,5 μm, C18 colonna (colonna A della Figura 2) accoppiato a una cartuccia di protezione c18 (4,0 x 3,0 mm d.i.) con un gradiente di 0,1% acido formico (solvente a) e metanolo contenente 0,1% acido formico (solvente B) ad una portata di 150 μl/min e a 25 ° c.
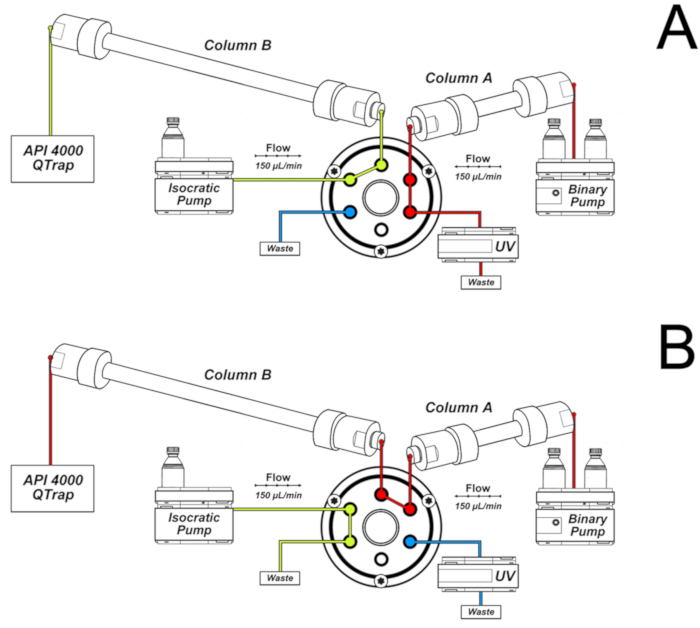
Figura 2. Sistema di due colonne utilizzate per le analisi 8-oxo-7, 8-dihydro-2'-deoxyguanosine (8-oxodGuo). A) configurazione utilizzata nei primi 16 min e da 32 a 46 min della cromatografia; B) configurazione utilizzata nell'intervallo 16-32 min, permettendo ulteriore separazione e restringimento del picco nella colonna B prima dell'eluizione alla fonte ESI dello spettrometro di massa. Questa cifra è stata ripubblicata da Oliveira et al.34. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
8. HPLC-ESI-MS/MS analisi di 1, n 6 -εdado e 1, n 2 -εdguo
- Inseguendo gli standard 1, n6-εdado e 1, n2-ΕDGUO nell'apparecchiatura, impostare i parametri ESI-MS/MS per la migliore rilevazione dei loro modelli di frammentazione mediante il monitoraggio multiplo delle reazioni (MRM): m/z 276 [m + h]+ → m/z 160 [m 2'-deoxyribose + h]+ per il rilevamento di 1,N6-εdado e m/z 292 [m + h]+ → m/z 176 [m-2'-deoxyribose + h ]+ per il rilevamento di 1,N2-εdguo.
- Utilizzare gli stessi parametri per il rilevamento di [15n5] 1, n6-εdado (m/z 281 [m + h]+m/z → 165 [m-2'-deoxyribose + h]+) e [15n 5] 1,N2-εdguo (m/z 297 [m + H]+ → m/z 181 [m-2'-deoxyribose + H]+). Impostare i parametri ESI-MS/MS come descritto nella tabella 1.
| Parametri ESI-MS/MS | 8-oxodGuo | Addotti di etheno |
| Tenda gas | a 20 psi | a 20 psi |
| Nebulizzazione gas | 55 a | 50 a |
| Gas di origine ionica | 50 psi | 40 psi |
| Gas di dissociazione indotto da collisioni | medio | medio |
| Tensione a spruzzo ionica | 5000 a | 4500 a |
| Temperatura sonda ESI | 450 a | 450 a |
| Potenziale di Declustering | 31 V, 8-oxodGuo | 41 V, 1, N6-εdado |
| 31 V, [15N5] 8-oxodGuo | 41 V, [15N5] 1, n6-εdado | |
| 45 V, 1, N2-εdguo | ||
| 45V, [15N5] 1, n2-εdguo | ||
| Energia di collisione | 23 eV, 8-oxodGuo | 25 eV, 1, N6-εdado |
| 23 eV, [15N5] 8-oxodGuo | 25 eV, [15N5] 1, n6-εdado | |
| 27 eV, 1, N2-εdguo | ||
| 27 eV, [15N5] 1, n2-εdguo | ||
| Potenziale di uscita delle celle di collisione | 16 V, 8-oxodGuo, | 8 V, 1, N6-εdado |
| 16 V, [15N5] 8-oxodGuo | 8 V, [15N5] 1, n6-εdado | |
| 16 V, 1, N2-εdguo | ||
| 16 V, [15N5] 1, n2-εdguo | ||
| Potenziale d'ingresso | a 10 V | a 10 V |
Tabella 1. Parametri utilizzati nelle apparecchiature ESI-MS/MS per il rilevamento delle lesioni del DNA. Questa tabella è stata modificata da Oliveira et al.34.
- Filtro (utilizzando membrane porose 0,22 μm) e degassano (utilizzando un sonicatore) tutti i solventi HPLC a base acquosa.
- Utilizzare le seguenti condizioni di cromatografia per le analisi.
- Elute un 150 x 2,0 mm d.i., 3,0 μm, C18 colonna accoppiato a una cartuccia di protezione C18 (4,0 x 3,0 mm d.i.) con un gradiente di 5 mM acetato di ammonio, pH 6,6 (solvente A) e acetonitrile (solvente B) ad una portata di 130 μL/min e 25 ° c.
- Utilizzare il seguente programma di gradiente per la pompa binaria: da 0 a 10 min, 0% di solvente B; da 10 a 39 min, 0-20% di solvente B; 39 a 41 min, 20-75% di solvente B; 41 a 46 min, 75% di solvente B; 46 a 47 min, 75-0% del solvente B; 47 a 60 min, 0% di solvente B.
- Utilizzare la valvola di commutazione per dirigere il primo 35 min di eluente per i rifiuti e la frazione 35-42 min alla fonte ESI. Accertarsi che gli standard addotti eluire dalla colonna nell'intervallo impostato (35-42 min). Se necessario, effettuare le regolazioni.
- Elute un 150 x 2,0 mm d.i., 3,0 μm, C18 colonna accoppiato a una cartuccia di protezione C18 (4,0 x 3,0 mm d.i.) con un gradiente di 5 mM acetato di ammonio, pH 6,6 (solvente A) e acetonitrile (solvente B) ad una portata di 130 μL/min e 25 ° c.
9. quantificazione dei normali 2'-desossiribonucleosidi da HPLC-UV
- Utilizzare un'apparecchiatura simile all'apparecchiatura utilizzata in questo lavoro (vedere la tabella dei materiali).
- Elute un 250 mm x 4,6 mm d.i., 5 μm, C18 colonna fissata a una cartuccia di protezione C18 (4,0 x 3,0 mm d.i.) con un gradiente di 0,1% acido formico e metanolo.
- Utilizzare il seguente programma gradiente: da 0 a 25 min, da 0 a 18% metanolo; da 25 a 27 min, 18 a 0% metanolo; da 27 a 37 min, 0% metanolo) ad una portata di 1 mL/min e 30 ° c.
- Iniettare 5 μL di ciascun campione riservato alla quantificazione di 2'-deossinucleosidi.
- Impostare il rilevatore DAD a 260 Nm per l'integrazione dei picchi dGuo e dAdo.
10. quantificazione delle lesioni del DNA
- Integrare le cime di 8-oxodGuo, [15n5] 8-oxodGuo, 1,n6-εdado, [15n5] 1,n6-εdado, 1,n2-εdguo, e [15n5] 1,n2 -εdguo dalle analisi HPLC-ESI-MS/MS.
- Calcolare i rapporti di superficie di 8-oxodGuo/[15n5] 8-oxodGuo, 1, n6-εdado/[15n5] 1,n6-εdado, e 1,N2-εdguo/[15n5 ] 1,N2-εdguo per le curve di calibrazione e i campioni.
- Tracciare le curve di calibrazione utilizzando i rapporti di area ottenuti nel passaggio 10.1.1 nell'asse y e le quantità di analiti presenti in ogni punto dell'asse x.
- Calcolare gli importi (fmol) delle lesioni in ogni campione iniettato utilizzando i rapporti calcolati nel passaggio 10.1.1 e le curve di calibrazione del passo 10.1.2.
- Integra i picchi di dGuo e dAdo dalle analisi HPLC-UV.
- Tracciare le curve di calibrazione utilizzando le aree ottenute nel passaggio 10,2 nell'asse y e le quantità di analiti presenti in ogni punto dell'asse x.
- Calcolare gli importi (nmol) di dGuo e dAdo in ogni campione iniettato utilizzando le aree ottenute al passo 10,2 e le curve di calibrazione del passo 10.2.1.
- Calcolare gli importi (nmol) di dGuo e dAdo presenti in ciascun campione iniettato nel sistema HPLC-ESI-MS/MS, considerando che gli importi calcolati al punto 10.2.2 sono presenti nel volume campione di 5 μL, mentre 50 μL sono stati iniettati nel sistema HPLC-ESI-MS/MS.
Nota: per calcolare la quantità di dGuo nei campioni utilizzati per l'analisi di 8-oxodGuo, basta moltiplicare la quantità (nmol/μL) ottenuta al punto 10.2.2 per 50. Per calcolare la quantità di dAdo e dGuo nei campioni utilizzati per le analisi di 1,n6-εdado e 1,n2-εdguo, considerare la fase di concentrazione dopo l'estrazione di fase solida. Il volume di 50 μL iniettati nel sistema HPLC-ESI-MS/MS corrisponde a 114,32 μL del campione originale. Gli importi (nmol/μL) ottenuti al punto 10.2.2 devono essere moltiplicati per 114,32 per ottenere i valori corretti. - Calcolare le frazioni molari 8-oxodGuo/dGuo, 1,n6-εdado/dado, 1,n2-εdguo/dGuo. I rapporti (fmol lesion/nmol Normal deoxynucleoside) danno il numero di lesioni per 106 dGuo normale o dAdo.
Access restricted. Please log in or start a trial to view this content.
Risultati
Le concentrazioni medie di DNA (± DS) ottenute da topi epatici (~ 1 g di tessuto), polmone (~ 0,2 g di tessuto) e rene (~ 0,4 g di tessuto) erano, rispettivamente, 5.068 ± 2.615, 4.369 ± 1.021, e 3.223 ± 723 μg/mL nel volume finale di 200 μL. Un cromatogramma rappresentativo ottenuto da HPLC-DAD del DNA purificato è mostrato in Figura 3. La presenza dei quattro 2'-deossinucleosidi, esenti dai ribonucleosidi dell'RNA, che eluto immediatamente prima dei ...
Access restricted. Please log in or start a trial to view this content.
Discussione
Un problema importante riscontrato nelle analisi 8-oxodGuo da HPLC Metodi è la possibile induzione della sua formazione durante le procedure di esame di estrazione del DNA, idrolisi del DNA, e la concentrazione di DNA idrolizzati22,38. Al fine di minimizzare il problema della formazione di 8-oxodGuo smontava, si raccomanda l'aggiunta di deferossamina a tutte le soluzioni di estrazione, stoccaggio e idrolisi del DNA, l'uso del metodo chaotropico ioduro di sodio e...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
FAPESP (Fundação de Amparo à pesquisa do Estado de São Paulo, proc. 2012/22190-3 e 2012/08616-8), CNPq (proc. 454214/2014-6 e 429184/2016-6), Mantes, PRPUSP (PRÓ-Reitoria de Pesquisa da Universidade de São Paulo), into INAIRA (MCT/CNPq/FNDCT/CAPES/ FAPEMIG/FAPERJ/FAPESP; Proc. 573813/2008-6), Redoxoma INCT (FAPESP/CNPq/Mantes; Proc. 573530/2008-4), Redoxoma di NAP (PRPUSP; Proc. 2011.1.9352.1.8) e CEPID Redoxoma (FAPESP; Proc. 2013/07937-8). T. F. Oliveira e A. A. F. Oliveira hanno ricevuto borse di studio da FAPESP (proc. 2012/21636-8, 2011/09891-0, 2012/08617-4) e CAPES (coordenação de aperfeiçoamento de pessoal de nível Superior). M. H. G. Medeiros, P. di mascio, P. H. N. Saldiva e A. P. M. Loureiro hanno ricevuto borse di stato da CNPq.
Alcune figure e tavole presenti in questo lavoro sono state originariamente pubblicate in Oliveira A.A.F. et al. effetti genotossici ed epigenotossici nei topi esposti a particolato fine ambiente concentrato (PM2,5) da San Paolo, Brasile. Tossicologia delle particelle e delle fibre. 15, 40 (2018).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| [15N5]-2’-deoxyadenosine | Cambridge Isotope Laboratories | NLM-3895-25 | |
| [15N5]-2’-deoxyguanosine | Cambridge Isotope Laboratories | NLM-3899-CA-10 | |
| acetonitrile | Carlo Erba Reagents | 412413000 | |
| alkaline phosphatase from bovine intestinal mucosa | Sigma | P5521 | |
| ammonium acetate | Merck | 101116 | |
| calf thymus DNA | Sigma | D1501 | |
| cell lysis solution | QIAGEN | 158908 | |
| chloroform | Carlo Erba Reagents | 412653 | |
| deferoxamine | Sigma | D9533 | |
| deoxyribonuclease I (DNase I) | Bio Basic Inc | DD0649 | |
| ethanol | Carlo Erba Reagents | 414542 | |
| formic acid | Sigma-Aldrich | F0507 | |
| HPLC-ESI-MS/MS system | HPLC: Agilent 1200 series ESI-MS/MS: Applied Biosystems/MDS Sciex Instruments | HPLC: binary pump (G1312B), isocratic pump (G1310A), column oven with a column switching valve (G1316B), diode array detector (G1315C), auto sampler (G1367C). ESI-MS/MS: Linear Quadrupole Ion Trap mass spectrometer, Model 4000 QTRAP. | |
| HPLC/DAD system | Shimadzu | Two pumps (LC-20AT), photo diode array detector (DAD-20AV), auto-injector (Proeminence SIL-20AC), column oven (CTO-10AS/VP) | |
| HPLC column (50 x 2.0 mm i.d., 2.5 µm, C18) | Phenomenex | 00B-4446-B0 | |
| HPLC column (150 x 2.0 mm i.d., 3.0 µm, C18) | Phenomenex | 00F-4251-B0 | |
| HPLC column (250 x 4.6 mm i.d., 5.0 µm, C18) | Phenomenex | 00G-4252-E0 | |
| HPLC C18 security guard cartridge (4.0 x 3.0 mm i.d.) | Phenomenex | AJO-4287 | |
| isoamyl alcohol | Sigma-Aldrich | M32658 | |
| isopropyl alcohol (isopropanol) | Carlo Erba Reagents | A412790010 | |
| ketamine | Ceva | Commercial name: Dopalen | |
| magnesium chloride | Carlo Erba Reagents | 349377 | |
| magnesium chloride | Sigma | M2393 | |
| methanol | Carlo Erba Reagents | L022909K7 | |
| phosphodiesterase I from Crotalus atrox | Sigma | P4506 | |
| protein precipitation solution | QIAGEN | 158912 | |
| proteinase K | Sigma-Aldrich | P2308 | |
| ribonuclease A | Sigma | R5000 | |
| sodium chloride | Sigma-Aldrich | S9625 | |
| SPE-C18 (Strata-X) | Phenomenex | 8B-S100-TAK | |
| tris(hydroxymethyl)-aminomethane | Carlo Erba Reagents | 489983 | |
| xylazine | Syntec do Brasil | Commercial name: Xilazin |
Riferimenti
- Cadet, J., Davies, K. J. A., Medeiros, M. H. G., Di Mascio, P., Wagner, J. R. Formation and repair of oxidatively generated damage in cellular DNA. Free Radical Biology and Medicine. 107, 13-34 (2017).
- Barnes, J. L., Zubair, M., John, K., Poirier, M. C., Martin, F. L. Carcinogens and DNA damage. Biochemical Society Transactions. 46, 1213-1224 (2018).
- Cadet, J., Davies, K. J. A. Oxidative DNA damage & repair: An introduction. Free Radical Biology and Medicine. 107, 2-12 (2017).
- Cao, H., Jiang, Y., Wang, Y. Stereospecific synthesis and characterization of oligodeoxyribonucleotides containing an N2-(1-carboxyethyl)-2'-deoxyguanosine. Journal of the American Chemical Society. 129, 12123-12130 (2007).
- Breyer, V., et al. Analysis and biological relevance of advanced glycation end-products of DNA in eukaryotic cells. The FEBS Journal. 275, 914-925 (2008).
- Tamae, D., Lim, P., Wuenschell, G. E., Termini, J. Mutagenesis and repair induced by the DNA advanced glycation end product N2-1-(carboxyethyl)-2'-deoxyguanosine in human cells. Biochemistry. 50, 2321-2329 (2011).
- Hecht, S. S. Lung carcinogenesis by tobacco smoke. International Journal of Cancer. 131, 2724-2732 (2012).
- Garraway, L. A., Lander, E. S. Lessons from the cancer genome. Cell. 153, 17-37 (2013).
- Ong, T. P., Loureiro, A. P. M. Nutritional interventions in age-related genetic and epigenetic instability and cancer. Anti-ageing nutrients: Evidence-based prevention of age-associated diseases. , John Wiley & Sons. UK. (2015).
- Evans, M. D., Dizdaroglu, M., Cooke, M. S. Oxidative DNA damage and disease: induction, repair and significance. Mutation Research. 567, 1-61 (2004).
- Moriya, M. Single-stranded shuttle phagemid for mutagenesis studies in mammalian cells: 8-oxoguanine in DNA induces targeted GC → TA transversions in simian kidney cells. Proceedings of the National Academy of Sciences of the United States of America. 90, 1122-1126 (1993).
- Medeiros, M. H. G. Exocyclic DNA adducts as biomarkers of lipid oxidation and predictors of disease. Challenges in developing sensitive and specific methods for clinical studies. Chemical Research in Toxicology. 22, 419-425 (2009).
- Guéraud, F. 4-Hydroxynonenal metabolites and adducts in pre-carcinogenic conditions and cancer. Free Radical Biology and Medicine. 111, 196-208 (2017).
- Nair, U., Bartsch, H., Nair, J. Lipid peroxidation-induced DNA damage in cancer-prone inflammatory diseases: A review of published adduct types and levels in humans. Free Radical Biology and Medicine. 43, 1109-1120 (2007).
- Pang, B., et al. Lipid peroxidation dominates the chemistry of DNA adduct formation in a mouse model of inflammation. Carcinogenesis. 28, 1807-1813 (2007).
- Møller, P., et al. Harmonising measurements of 8-oxo-7,8-dihydro-2'-deoxyguanosine in cellular DNA and urine. Free Radical Research. 46, 541-553 (2012).
- Hofer, T., Moller, L. Optimization of the workup procedure for the analysis of 8-oxo-7,8-dihydro-2'-deoxyguanosine with electrochemicaldetection. Chemical Research in Toxicology. 15, 426-432 (2002).
- Collins, A., El Yamani, N., Dusinska, M. Sensitive detection of DNA oxidation damage induced by nanomaterials. Free Radical Biology and Medicine. , 69-76 (2017).
- Zubel, T., Buerkle, A., Mangerich, A. Mass spectrometric analysis of sulfur mustard-induced biomolecular adducts: Are DNA adducts suitable biomarkers of exposure? Toxicology Letters. 293, 21-30 (2018).
- Tretyakova, N., Goggin, M., Sangaraju, D., Janis, G. Quantitation of DNA adducts by stable isotope dilution mass spectrometry. Chemical Research in Toxicology. 25, 2007-2035 (2012).
- Churchwell, M. I., Beland, F. A., Doerge, D. R. Quantification of multiple DNA adducts formed through oxidative stress using liquid chromatography and electrospray tandem mass spectrometry. Chemical Research in Toxicology. 15, 1295-1301 (2002).
- Chao, M. R., Yen, C. C., Hu, C. W. Prevention of artifactual oxidation in determination of cellular 8-oxo-7,8-dihydro-2'-deoxyguanosine by isotope-dilution LC-MS/MS with automated solid-phase extraction. Free Radical Biology and Medicine. 44, 464-473 (2008).
- Danielsen, P. H., et al. Oxidative stress, inflammation, and DNA damage in rats after intratracheal instillation or oral exposure to ambient air and wood smoke particulate matter. Toxicological Sciences. 118, 574-585 (2010).
- Danielsen, P. H., et al. Oxidative stress, DNA damage, and inflammation induced by ambient air and wood smoke particulate matter in human A549 and THP-1 cell lines. Chemical Research in Toxicology. 24, 168-184 (2011).
- Garcia, C. C. M., et al. [13C2]-Acetaldehyde promotes unequivocal formation of 1,N2-propano-2'-deoxyguanosine in human cells. Journal of the American Chemical Society. 133, 9140-9143 (2011).
- Angeli, J. P. F., et al. Lipid hydroperoxide-induced and hemoglobin-enhanced oxidative damage to colon cancer cells. Free Radical Biology and Medicine. 51, 503-515 (2011).
- Yu, Y., et al. Comprehensive assessment of oxidatively induced modifications of DNA in a rat model of human Wilson's disease. Molecular and Cellular Proteomics. 15, 810-817 (2016).
- Torres-Cuevas, I., Aupi, M., Asensi, M. A., Vento, M., Ortega, Á, Escobar, J. 7,8-Hydroxy-2'-deoxyguanosine/2'-deoxiguanosine ratio determined in hydrolysates of brain DNA by ultrachromatrography coupled to tandem mass spectrometry. Talanta. 170, 97-102 (2017).
- Wu, D., et al. Detection of 8-hydroxydeoxyguanosine (8-OHdG) as a biomarker of oxidative damage in peripheral leukocyte DNA by UHPLC-MS/MS. Journal of Chromatography B. 1064, 1-6 (2017).
- IARC. Monographs on the Evaluation of Carcinogenic Risks to Humans: Outdoor Air Pollution. 109, IARC. Lyon, France. (2016).
- De Martinis, B. S., Kado, N. Y., Carvalho, L. R. F., Okamoto, R. A., Gundel, L. A. Genotoxicity of fractionated organic material in airborne particles from São. Mutation Research. 446, 83-94 (1999).
- Karlsson, H. L., Nygren, J., Möller, L. Genotoxicity of airborne particulate matter: The role of cell-particle interaction and of substances with adduct-forming and oxidizing capacity. Mutation Research. 565, 1-10 (2004).
- Bell, M. L., Dominici, F., Ebisu, K., Zeger, S. L., Samet, J. M. Spatial and temporal variation in PM2.5 chemical composition in the United States for health effects studies. Environmental Health Perspectives. 115, 989-995 (2007).
- Oliveira, A. A. F., et al. Genotoxic and epigenotoxic effects in mice exposed to concentrated ambient fine particulate matter (PM2.5) from São Paulo city, Brazil. Particle and Fibre Toxicology. 15, 40(2018).
- Loureiro, A. P. M., Zhang, W., Kassie, F., Zhang, S., Villalta, P. W., Wang, M., Hecht, S. S. Mass spectrometric analysis of a cyclic 7,8-butanoguanine adduct of N-nitrosopyrrolidine: comparison to other N-nitrosopyrrolidine adducts in rat hepatic DNA. Chemical Research in Toxicology. 22, 1728-1735 (2009).
- Loureiro, A. P. M., Marques, S. A., Garcia, C. C. M., Di Mascio, P., Medeiros, M. H. G. Development of an on-line liquid chromatography-electrospray tandem mass spectrometry assay to quantitatively determine 1,N2-etheno-2'-deoxyguanosine in DNA. Chemical Research in Toxicology. 15, 1302-1308 (2002).
- Mangal, D., et al. Analysis of 7,8-dihydro-8-oxo-2′-deoxyguanosine in cellular DNA during oxidative stress. Chemical Research in Toxicology. 22, 788-797 (2009).
- ESCODD (European Standards Committee on Oxidative DNA Damage). Comparative analysis of baseline 8-oxo-7,8-dihydroguanine in mammalian cell DNA, by different methods in different laboratories: an approach to consensus. Carcinogenesis. 23, 2129-2133 (2002).
- Helbock, H. J., et al. DNA oxidation matters: The HPLC-electrochemical detection assay of 8-oxo-deoxyguanosine and 8-oxo-guanine. Proceedings of the National Academy of Sciences of the United States of America. 95, 288-293 (1998).
- Risom, L., et al. Oxidative DNA damage and defence gene expression in the mouse lung after short-term exposure to diesel exhaust particles by inhalation. Carcinogenesis. 24, 1847-1852 (2003).
- Risom, L., et al. Repeated inhalations of diesel exhaust particles and oxidatively damaged DNA in young oxoguanine DNA glycosylase (OGG1) deficient mice. Free Radical Research. 41, 172-181 (2007).
- Tsurudome, Y., et al. Changes in levels of 8-hydroxyguanine in DNA, its repair and OGG1 mRNA in rat lungs after intratracheal administration of diesel exhaust particles. Carcinogenesis. 20, 1573-1576 (1999).
- Marie-Desvergne, C., Maître, A., Bouchard, M., Ravanat, J. L., Viau, C. Evaluation of DNA adducts, DNA and RNA oxidative lesions, and 3-hydroxybenzo(a)pyrene as biomarkers of DNA damage in lung following intravenous injection of the parent compound in rats. Chemical Research in Toxicology. 23, 1207-1214 (2010).
- Iwai, K., et al. Early oxidative DNA damages and late development of lung cancer in diesel exhaust-exposed rats. Environmental Research. 84, 255-264 (2000).
- Ichinose, T., et al. Lung carcinogenesis and formation of 8-hydroxy-deoxyguanosine in mice by diesel exhaust particles. Carcinogenesis. 18, 185-192 (1997).
- Schmerold, I., Niedermu, H. Levels of 8-hydroxy-2'-deoxyguanosine in cellular DNA from 12 tissues of young and old Sprague Dawley rats. Experimental Gerontology. 36, 1375-1386 (2001).
- Garcia, C. C. M., Freitas, F. P., Di Mascio, P., Medeiros, M. H. G. Ultrasensitive simultaneous quantification of 1,N2-etheno-2'-deoxyguanosine and 1,N2-propano-2'-deoxyguanosine in DNA by an online liquid chromatography-electrospray tandem mass spectrometry assay. Chemical Research in Toxicology. 23, 1245-1255 (2010).
- Godshalk, R., et al. Comparison of multiple DNA adduct types in tumor adjacent human lung tissue: effect of cigarette smoking. Carcinogenesis. 23, 2081-2086 (2002).
- Dechakhamphu, S., et al. Lipid peroxidation and etheno DNA adducts in white blood cells of liver fluke-infected patients: protection by plasma alpha-tocopherol and praziquantel. Cancer Epidemiology Biomarkers and Prevention. 19, 310-318 (2010).
- Arab, K., et al. Typical signature of DNA damage in white blood cells: a pilot study on etheno adducts in Danish mother-newborn child pairs. Carcinogenesis. 30, 282-285 (2009).
- Nair, J., et al. High dietary omega-6 polyunsaturated fatty acids drastically increase the formation of etheno-DNA base adducts in white blood cells of female subjects. Cancer Epidemiology Biomarkers and Prevention. 6, 597-601 (1997).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon