Method Article
CorExplorer Web ポータルを用した腫瘍遺伝子発現因子の解析
要約
機械学習アルゴリズムCorEx(相関説明)によって見つかった腫瘍RNAシーケンシング因子を探索するためのリソースであるCorExplorerウェブポータルを紹介し、生存に対する因子の分析方法、データベースアニテーション、タンパク質とタンパク質の相互作用、および腫瘍生物学と治療介入に関する洞察を得るために互いに。
要約
微分遺伝子発現解析は、疾患状態を理解するための重要な手法です。機械学習アルゴリズムCorExは、腫瘍RNA-seqの遺伝子群の差動発現を、精密腫瘍学の進歩に役立つ可能性のある方法で解析する上で有用性を示した。しかし、CorEx は、既存の理解を分析して接続するのが難しい多くの要因を生み出します。このような接続を容易にするために、ユーザーがインタラクティブにデータを探索し、その分析に関連する一般的な質問に答えることを可能にするウェブサイトCorExplorerを構築しました。卵巣、肺、黒色腫、大腸の4種類の腫瘍タイプについて、RNA-seq遺伝子発現データについてCorExをトレーニングしました。その後、対応する生存、タンパク質とタンパク質の相互作用、遺伝子オントロジー(GO)、京都百科事典(KEGG)経路濃縮を組み込み、因子グラフの可視化と関連するヒートマップをウェブサイトに組み込みました。ここでは、この外部データのコンテキストで学習された腫瘍因子の重要性を理解するためのデータベースの使用を説明する例のプロトコルを使用します。
概要
ちょうど10年前に導入されて以来、RNA-seqは遺伝子発現を測定するためのユビキタスツールとなっています1.これは、サンプルのトランスクリプトーム全体の迅速かつ安価なデノボプロファイリングを可能にするためです。しかし、RNA-seq腫瘍データは、本質的に複雑で、しばしばサンプリング不足の基礎生物学を反映していますが、データ自体は高次元でノイズが多い。これは、信頼性の高い信号を抽出するための重要な課題を提示します。CorEx アルゴリズムは、多変量相互情報を利用して、このような状況で微妙なパターンを見つけます2,3 .この技術は、以前に癌ゲノムアトラス(TCGA)から卵巣腫瘍RNA-seqサンプルを分析するために適応され、この文脈では、より一般的に使用される分析方法4よりも有意な利点を有するように見えた。
RNA-seqの使用は、腫瘍学を含む研究アプリケーションで非常に広く普及しているが、これらの努力は、臨床介入の目的のために広範な利用につながっていない5.この理由の一部は、これらの特定の問題を対象としたユーザーフレンドリーなアルゴリズムとソフトウェアの欠如です。このギャップを埋めるために、CorExplorerウェブポータルを設計し、CorEx機械学習アルゴリズムで見つかった腫瘍RNA-seqサンプルの遺伝子発現因子を様々なバックグラウンドの研究者が研究できるようにしました。CorExplorer ポータルは、肺、結腸、黒色腫、卵巣6 、7、8、9など、いくつかの異なる腫瘍タイプからの要因のインタラクティブな視覚化とクエリをサポートします。10は、研究者がデータ相関をふるい、治療目的で患者を階層化するための候補経路を特定するのを助けることを目的としています。
CorExplorer ポータルは、複数の種類のユーザーにとって役立つ可能性があります。ポータルは、公共データベースにおける腫瘍遺伝子発現の違いを駆動する広範な要因を理解し、おそらく同様の腫瘍のコンテキストで個々の遺伝子発現プロファイルを配置したいユーザーを念頭に置いて設計されました特性。ここで説明する代表的なプロトコルに加えて、CorExplorer の調査は、さらなるテストのための仮説を示唆し、CorExplorer 外のデータセットに関する CorEx の調査結果を比較対照し、接続するための出発点となる可能性があります。個々の腫瘍内の1つまたは少数の遺伝子の病理学的発現シグネチャを、協調的に影響を受ける可能性のあるより大きなグループに対する。最後に、この分野で始める人のためのRNA-seqへの機械学習の応用に関するユーザーフレンドリーな紹介として役立つかもしれません。
プロトコル
1. 目的遺伝子を含む因子の探索
- Web ブラウザを開き、Http://corex.isi.edu CorExplorer のホーム ページに移動します。
- クイックリンクの右側にある[+展開]ボタンをクリックすると、卵巣(TCGA-OV)の横にあるボタンをクリックすると、TCGA卵巣癌データでトレーニングされたCorEx因子グラフの概要が表示されます(図1参照)。必要に応じて、他のユーザーをクリックして比較します。
- 因子グラフの検査が完了したら、肺 (TCGA-LUAD)肺癌RNA-seqのCorExplorerページにアクセスします。
- CorExplorer の 「因子グラフ」ウィンドウを使用して、対象遺伝子の CorEx 因子グラフを調べます。
- 係数グラフ表示ウィンドウの上にマウス カーソルを移動します。マウススクロールホイールまたはトラックパッドを使用して因子グラフをズームインすると、各因子で最も重要な遺伝子や異なるレイヤーのノード間の接続などのグラフの詳細が表示されます。または、クリックしてドラッグして、ビューエリアまたは任意のノードを移動します。
- ターゲット遺伝子を見つけるには(ここではBRCA1を使用します)、因子グラフウィンドウの上部にある遺伝子ドロップダウンメニューをクリックします。ドロップダウンリストで選択する「BRCA1」と入力し、「戻る」を押してビューを26倍にすると、BRCA1が最も強く相関している因子を考慮します。
- グラフ表示の上にマウスを置き直し、スクロールして縮小すると、レベル 2 ノード、L2_8、および係数 26 に近い因子が表示されます。[Min リンクウェイトスライダ] に示されたしきい値を超える重みを持つ遺伝子のみが表示されます。
- 因子に関連するすべての遺伝子を表示するには、L1_26ノードをクリックし、ポップアップウィンドウで追加の遺伝子をロードすることを選択します。「完了」という単語が表示されたら、ポップアップ ウィンドウを閉じます。
- 次に、係数グラフ ウィンドウの上のヘッダー セクションに戻り、[Min リンクウェイトモディファイヤ] を取得してドラッグします。これで、リンクウェイト スライダが 0.05 に下がると、BRCA2 を含む因子 L1_26 の他の遺伝子が重量順に表示されます。必要に応じて、配置を改善するために、取得およびドラッグしてノードの位置を変更します。
- 因子に関する患者の階層化が生存ウィンドウでクエリを実行して生存に与える影響を決定します。
- サバイバルウィンドウで、p-valで並べ替えをオフにし、[単一因子]ドロップダウンメニューで因子26を選択して、因子26の生存曲線を表示します。
- 生存グラフを下にスクロールして、X 軸に沿って危険にさらされている患者の数を表示します。
- [アニテーション] ウィンドウ内でクエリを実行して、生物学的関数との関連付けを検索します。
- 注釈ウィンドウで、因子ドロップダウンメニューを偽検出率(FDR)ではなく因子数でソートするには、FDR ソートをオフにします。
- スクロールしてクリックして、アノテーション ウィンドウドロップダウンで因子 26 を選択して、因子のエンリッチメント アノテーションを表示します。
- DNA修復が表示されるまでアノテーションリストを下にスクロールし、それをクリックすると、グラフ表示で黄色で強調表示された関連遺伝子がすぐに表示されます。図 2の中央パネルを参照してください。
- 選択された注釈を持つ遺伝子に対して濃縮されているかどうかに応じて、異なるGO項が選択されるように見えるか、または異なるGO項として現れる要因(例えば「DNA損傷に応じて固有のアポトーシスシグナル伝達経路」)に従って、因子が消失するか、または現れることに注意してください。
- 異なる機能を持つウィンドウを追加して、さらに要因を調べます。
- 上部のメニューバーから、[ウィンドウの追加]ドロップダウンからPPIを選択してタンパク質タンパク質相互作用ネットワーク(PPI)ウィンドウを追加し、[追加]ボタンをクリックしてPPIグラフウィンドウを表示領域に追加します。PPI グラフ ウィンドウで、タンパク質とタンパク質の相互作用を示す因子 'Layer1: 26' を選択します。接続の密度に注意してください。
- PPIの代わりにトップ メニュー バーから[ウィンドウの追加]ドロップダウンからヒートマップを選択し、[追加]ボタンをクリックして表示領域にヒートマップ ウィンドウを追加します。ヒートマップ ウィンドウで、因子 'Layer1: 26' を選択して、遺伝子発現パターンを表示します。
- サバイバル ウィンドウも表示できるように、ヒートマップ ウィンドウを取得して再配置します。ヒートマップの上部に沿って、オレンジ/ブルー/グレーの色のバーが生存グラフ上の患者リスク層にどのように対応しているかを観察します。結果は図 2 の下部に示されています。
- CorExplorer の 「因子グラフ」ウィンドウを使用して、対象遺伝子の CorEx 因子グラフを調べます。
2. 遺伝子重量、生存、ア釈データを用いてCorEx因子をフィルタリング・解釈
- 生存とクラスター品質を使用して、関心のある要因をフィルター処理します。
- 上部の [データセット] ドロップダウン メニューから、[TCGA_OVCA]を選択して、TCGA 卵巣癌 RNA-seq の CorExplorer ページに移動します。
- ページが読み込まれると、サバイバル ウィンドウから、異なる層の最大の生存差を持つ因子が 114 であることを示します。
- 因子グラフウィンドウの上部で、因子ドロップダウンから「レイヤー1:114」を選択します。
- マウスでリンクウェイトスライダをつかみ、0.5まで移動します。因子114(1609)の多数の遺伝子は、重量>0.35を持たないが、比較的弱いクラスタリングを示す。
- 次に、生存ウィンドウ内の因子のリストを展開し、生存ウィンドウドロップダウンの次の最適因子(因子39)を選択して、関連する生存曲線を表示します。
- アニテーション ウィンドウで因子 39 を選択します。重要な GO および KEGG アノテーションが表示されます。
- 因子39における遺伝子の生物学的役割をより良く理解するために、近傍アテーション情報を用いて因子を以下のように解釈する。
- 因子グラフウィンドウの上部で、因子ドロップダウンで因子「レイヤ1:39」を選択します。次に、ファクター グラフ ウィンドウの上にマウスを移動し、縮小して L2_14 クラスター全体を 6 つの因子 (14、32、39、42、52、82)で明らかにします (図 3を参照)。
- L2_14 ノードにリンクされている因子の相対的な有意性を理解するには、まず L2_14 因子ごとに生存差を表示します。サバイバルウィンドウでp-valで並べ替えをオフにし、各因子数を連続してクリックします。これを行うと、因子14、32、および39のみが生存関連を示す点に注意してください。
- トップメニューバーから、[ウィンドウの追加]ドロップダウンからPPIをもう一度選択します。[追加]を押して、PPI グラフ ウィンドウを表示領域に追加します。PPIグラフウィンドウで、有意なタンパク質相互作用を示すために因子「Layer1:52」を選択します。この時点でのウィンドウのレイアウトの例を図 3 に示します。
- PPI ウィンドウの下部にある[StringDB で表示]リンクをクリックして、StringDB オンライン データベースにリンクします。最初の画面から[続行]をクリックし、以前と同様にネットワークグラフの下の[分析]タブを選択して、PPIネットワーク遺伝子のオンラインGO解析を取得します。細胞成分のトップは「MHCクラスIIタンパク質複合体」です。
- [CorExplorer] タブと PPI ウィンドウに戻り、ファクタードロップダウンから今度は因子 32 を選択します。StringDBのリンク ビューをクリックして StringDB 分析に送信します。トップ細胞成分は、前のステップの因子52のクラスIIとは対照的に、「MHCクラスIタンパク質複合体」です!
- 最後に、PPI ウィンドウに戻り、上部の因子ドロップダウンメニューから「Layer1: 39」を選択します。StringDBのリンク ビューをクリックして、StringDB 分析にリンクします。
- 最初の画面から[続行]をクリックし、ネットワークグラフの下の[分析]タブを選択して、PPIネットワーク遺伝子のオンラインGO解析を取得します。最上位の分子機能が'CXCR3ケモカイン受容体結合'であることを観察してください。
3. 生存とデータベースのアニテーションを使用して、有望な治療の組み合わせを探す
- [データセット]ドロップダウン メニューからTCGA_SKCM を選択して、TCGA黒色腫 CorExplorer に切り替えます。
- 最大の生存差を持つ因子は因子171である。スクロールして因子171の注釈を調べ、「免疫応答」と「サイトカイン媒介シグナル伝達経路」が上部付近にあることに注意してください(上の卵巣因子の場合と同様)。
- 相補的な因子を見つけるには、上位の生存関連因子と上位のア釈項を調べます。これを行うには、トップメニューバーの[データセットの概要]リンクをクリックして、データセット処理の詳細を含むテーブルと、生存差のp値に従ったトップファクターの概要を含む別のタブを開きます。なお、最初の非免疫因子は88である。
- [TCGA_SKCM]ブラウザー タブに戻ります。
- 生存ウィンドウ、ア釈ウィンドウ、グラフ ウィンドウで因子 88 を選択します。上位いくつかのGO用語は「rRNA処理」と「ミトコンドリオン組織」に関連しており、免疫関連因子とは異なることを確認しています。
- サバイバルウィンドウで、ペアの因子ドロップダウンで「88_171」を選択して、171と88の発現因子を組み合わせた中間層の患者の生存率がどのように改善されるかを確認します。アヌテーションと生存の比較を図 4 に示します。
4. 検索ページを使用して、腫瘍タイプ間の遺伝子発現変動の共通点と相違点を見つける
- CorExplorerの見出しをクリックして、フロント ページに戻ります。
- CorExplorer サイト上のすべてのデータセットを検索できるページに移動するには、[トップ メニュー バーで検索]をクリックします。
- ジーン検索ボックスに「FLT1」(VEGFR1)と入力し、[戻る]または [検索]を押します。FLT1は、OVCA- 76、LUAD - 162、SKCM - 195およびSKCM-184、ならびにCOAD-112およびCOAD-74の比較的高い重量を持つ。
- または、すべてのデータセットで関連する GO 用語を検索します。「血管新生」と入力し、Returnキーを押すか、検索を押して、[GO 検索] ボックスでこれを試してみてください。SKCM-195を除くすべてのFLT1因子は、「血管新生」遺伝子に対して統計的に濃縮されたものとしてリストされており、因子195は、実際には、ア釈を持っていますが、デフォルトの10-8閾値を下回っています。この検索結果と前の手順を図 5 に示します。
- さらなる例として、GO検索ボックスにおいて、第1型「表皮成長因子受容体」を入力する。この用語ではLUADのみが濃縮され、肺癌のよく知られた層化因子である。次に、検索ボックスに「間葉」と入力します。この用語は、OVCAの遺伝子発現群に富み、そこではよく研究された層化因子である。
結果
肺癌データセット内の遺伝子「BRCA1」を検索すると、CorEx因子26と最も強く関連していることが明らかになりました(図2)。この因子のGO用語濃縮は非常に高く、DNA修復はわずか1 x10-19のFDRを示す。この選択は、子として 6 つの密接に関連する要因を持つ第 2 レベルクラスター L2_8 にも注目されます。GO用語アニテーションまたは因子グラフのGO濃縮ドロップダウンのいずれかで「DNA修復」を選択すると、各因子に関連する遺伝子が強調表示され、因子26は予想通り11を上回る。タンパク質とタンパク質相互作用ネットワークは強く結びつい、因子26における遺伝子の密接に連結された機能性をさらに支持する。関連する生存グラフは、患者の生存との関連性を示唆していますが、これはより大きなデータセットで確認する必要があります。
生存から始めることは、特定の遺伝子発現群に関連する生存率の改善の理由の解剖を可能にすることができる。一例として、卵巣癌の生存に影響を与える最上位因子は39番目と見られ、免疫系に関連する遺伝子に対して強く濃縮されている(図3)。同じレベル2ノードに関連する他の5つの因子も免疫関連であることが示されていますが、生存の影響は強く変動しているように見え、39が最も高く、52が最も低い。因子にタンパク質とタンパク質の相互作用ウィンドウを追加すると、即時相互作用ネットワークが示され、StringDB12ウェブサイトへのリンクを使用してPPIネットワーク遺伝子の様々なエンリッチメントを照会することができます。L2_14 因子のそれぞれについてこれを行うことで、PPIネットワーク遺伝子のStringDBエンリッチメントが生存との関連について以下の考えられる説明を示唆していることがわかります。因子32は、細胞傷害性Tリンパ球によって認識される主要組織適合性複合体(MHC)クラスIタンパク質複合体を構成する遺伝子を含有する。因子39は、CD8+Tリンパ球に関連するサイトカインシグナル伝達およびCXCR3受容体結合に対応する。これらの因子の両方は、対応する遺伝子の比較的高い発現を示す患者に有意な生存優位性を与えるように見える。細胞傷害性CD8+Tリンパ球は、主に抗腫瘍免疫を担当する。一方、因子52は、細胞傷害性Tリンパ球によって直接ではなく、主にCD4+Tヘルパー細胞によって認識されるMHCクラスII複合体におけるタンパク質をコードする遺伝子から構成される。残りのL2_14因子は、2種類のリンパ球集団を区別しない一般化された免疫系活性化を反映する。MCHクラスI細胞抗原の細胞傷害性Tリンパ球認識に特異的な生存関連は、一般的に、および黒色腫などの他の癌からの抗腫瘍免疫の我々の理解と一致している13,14。
ウェブポータルは、効果的な腫瘍特異的併用療法を示唆する相補的な機能を有する因子のペアの発見をサポートする。データセットの概要は、生存との相関関係を示すが、明確なGOエンリッチメントを持つ因子をスキャンすることができます。黒色腫(TCGA_SKCM;図4)は、トップ生存因子171が免疫関連であることが分かり、一方、リストの下の因子88は、ミトコンドリオン組織に関連する遺伝子の濃縮を示す。実際、これは黒色腫15の標的として示唆されている。CorExplorer ページにサバイバル ウィンドウを追加すると、因子ペアを使用した層化を各因子の階層化を個別に比較することができ、両グループの良好な遺伝子発現パターンがどちらよりも生存率の傾向を示すことを示します。単独で係数。最上部の層は改善されていないように見えるが、免疫療法だけが一部の患者にとって最良の選択肢かもしれないことを示唆している。
腫瘍間の共通点と相違点は、遺伝子やGO用語のデータセット間で検索することで見ることができます(図5)。一例として、FLT1(別名VEGFR1)は、よく研究されたプロ血管新生マーカー16、17である。検索バーに入れると、すべての腫瘍にFLT1が主要な役割を果たす要因があります。逆に、GO用語「血管新生」が検索ページに入力されると、FLT1グループの6つのうち5がそのエンリッチメントとともに現れる。SKCM-195を除くすべてのFLT1因子は、「血管新生」遺伝子に対して統計的に濃縮されたものとしてリストされている。6 番目の係数は、実際にはア釈を持ちますが、既定の 10- 8 しきい値を下回っています。因子リスト内の重み付けが代替濃縮電卓(例えば、遺伝子セット濃縮分析(GSEA)18で利用されると、第6因子は「血管新生」遺伝子についても有意に濃縮されることがわかった。
遺伝子発現パターンが生物学的解釈をサポートするのに十分な品質であることを確認するために、ヒートマップをチェックすることが重要です。強い明確な変化を示すヒートマップは、低から高い、またはより複雑なパターンに及ぶ因子遺伝子の協調発現のいずれかを示し、低発現を有する遺伝子と高い相関関係を有する遺伝子を示す可能性がある(図6)。高品質のグループ化の重要なマーカーは、因子スコアの関数としての発現の滑らかな変化を持ついくつかの遺伝子の存在です。因子ヒートマップは、因子スコアに従って順序付けされたサンプルを示すので、滑らかな勾配が左から右に移動する必要があります。ただし、これは少なくとも 2 つの異なる方法で発生する可能性があります。最も一般的には、相関関係は非常に騒々しい(図5C)、生存および/または生物学的機能に関する推論の堅牢性と有用性に疑問を投げかける。また、少数のサンプルでのみ発生するパターンは、CorEx アルゴリズムによって想定される 3 つの式状態のモデルに適合しない可能性があり、結果としてサンプルの誤解を招く分類が生じます (図 5Dの右側)。
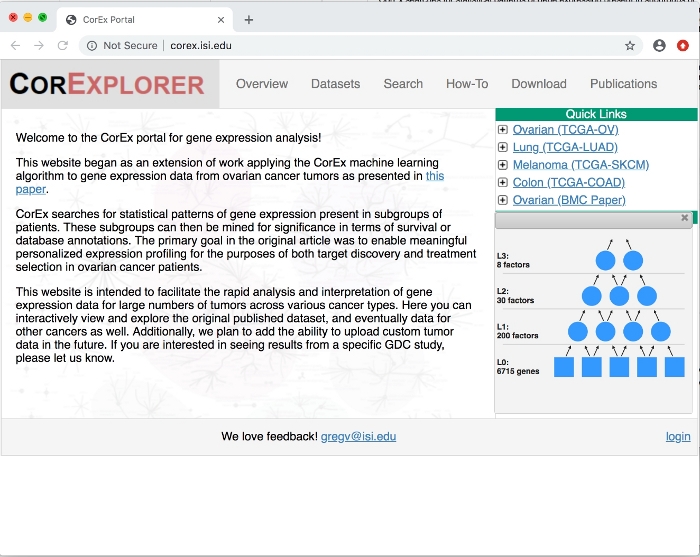
図 1: CorExplorer のフロント ページ。クイックリンクの下の卵巣癌の横に+をクリックすると、因子グラフの詳細が表示されます。CorEx 階層モデルは、最下層の入力変数(この場合は遺伝子発現)で構成され、上位層の潜在因子が推測されます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
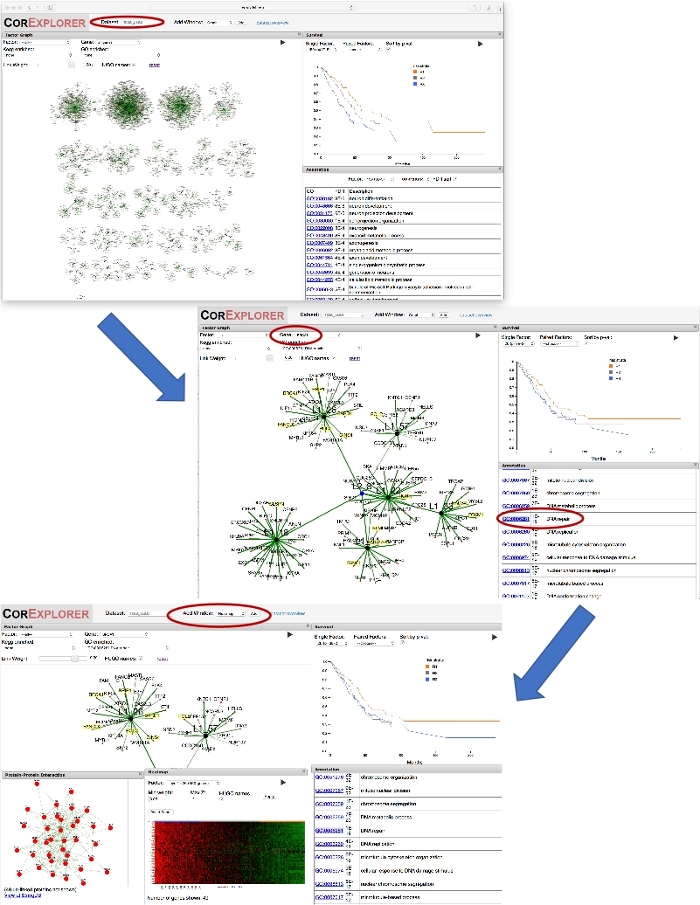
図2:遺伝子名を使用して探査を導く。図は、BRCA1に強く関連するCorEx肺癌因子の探索を示す一連のスクリーンショットを示しています。まず、因子グラフの遺伝子ドロップダウンボックスで「BRCA1」を選択すると、グラフビューでBRCA1の重量が最も大きい因子にズームインします。ビットフレームを縮小すると、その係数を他の関連ノードに接続するレイヤ 2 ノード L2_8 がフレーム化されます。生存とアテーションを比較することができます:GO用語DNA修復をクリックすると、NOTNOT遺伝子が強調表示されます。PPI ウィンドウが追加され、因子内の遺伝子のネットワーク相互作用が表示されます。[ウィンドウの追加]ボタンを使用してヒートマップを追加すると、発現パターンと生存との関連が示され、DNA修復遺伝子の発現の増加が生存率の低下に関連している可能性が示唆される。この図のより大きなバージョンを表示するには、ここをクリックしてください。
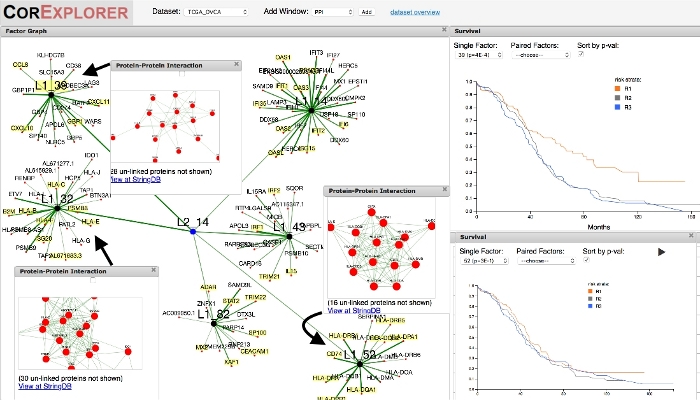
図3:臨床データ(生存)を用いて探査を導く。卵巣癌のトップ生存関連因子(39)を探索すると、近隣の要因間の興味深い関係が明らかになります。因子グラフで因子39を選択し、少しズームアウトした後、因子39にリンクされたレイヤー2因子には、他の5つの関連因子があるが表示されます。追加の生存ウィンドウは、関連する生存差異を直接比較することができます。因子39と32はいずれも正の生存相関を示し、因子52とは対照的に、そうでない。タンパク質とタンパク質の相互作用ネットワークはすべて明確に定義されています。StringDBへのリンクはGOアノテーションの比較を可能にする(図示せず):因子39は細胞傷害性CD8+Tリンパ球活性化に関連するサイトカインシグナル伝達ネットワークに関連し、因子32はMHCクラスI抗原提示タンパク質によって支配される。このようなリンパ球によるトリガー認識;しかし、隣接する因子は、CD4+ヘルパーT細胞などの他の免疫系成分によって支配され、生存相関を示さない。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図4:トップ生存因子の探索は、潜在的な治療的組み合わせを示唆する。ホーム ページ メニュー バーの 「データセット」リンクは、p 値で順序付けた生存因子の簡潔な表と、上位 GO アノテーション (図示せず) につながります。この情報を黒色腫に対して使用すると、ミトコンドリオン組織に対する因子88と免疫機能に対する因子171の組み合わせが相補的に現れる。次の図は、各因子のアヌテーション ウィンドウを対比に示しています。2つの因子によって個別にまたは一緒に階層化された患者の生存曲線は、組み合わせがいずれかの因子単独と比較して生存差を増加させることを示す。この図のより大きなバージョンを表示するには、ここをクリックしてください。
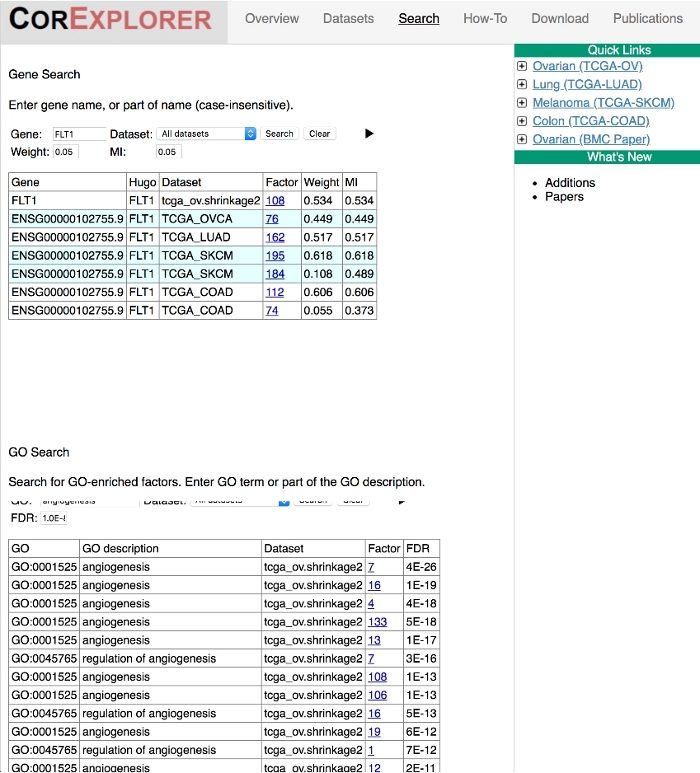
図 5: 検索ページを使用すると、汎癌の分析が容易になります。遺伝子またはGO生物学的プロセス用語は、ホームページからの検索リンクを使用して、すべてのデータセットにわたって検索することができます。図は、遺伝子FLT1およびGO用語「血管新生」の検索結果を示しています。結果は、癌全体で「血管新生」という用語に付加された因子におけるFLT1の存在を示す。この図のより大きなバージョンを表示するには、ここをクリックしてください。
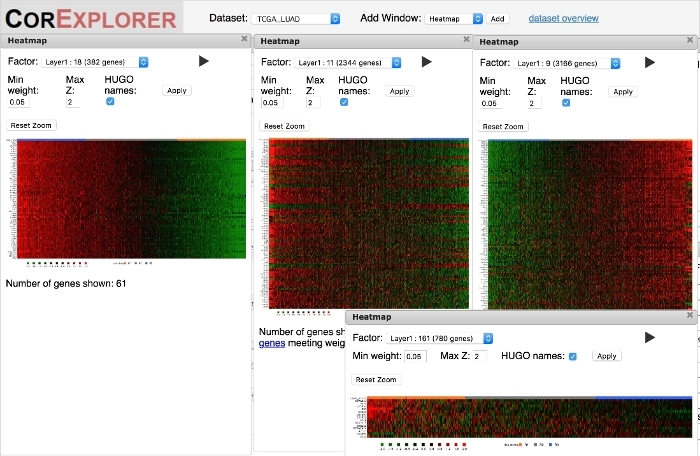
図6:ヒートマップを使用して、因子スコアに従って遺伝子とサンプル間の相関関係を定性的に評価することができます。患者がヒートマップの因子スコアで注文されるとき、高品質の遺伝子発現関係は滑らかなグラデーションによって示される。因子18の左端のヒートマップは、その一例です。パターンには、係数 11 の中央の大きなヒートマップのように、上下の表現の複雑なシグネチャが含まれる場合もあります。低品質パターンは、右下の因子161ヒートマップのように、右側の因子9ヒートマップまたは単純な非常にノイズの多い相関のように、患者のサブグループの発現の急激な変化を示すことがあります。この図のより大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
CorExplorerサイトは、CorExアルゴリズムによって腫瘍RNA-seqから学習された最大相関性遺伝子発現因子のインタラクティブな探索のための公的にアクセス可能なウェブサーバであるCorExplorerサイトを発表しました。腫瘍遺伝子発現に応じて患者を階層化するためにウェブサイトをどのように使用し、そのような層化が生物学的機能と生存にどのように対応するかを示した。
RNA-seq分析用の他のウェブサーバが構築されています。腫瘍の差動および共発現分析は、cbioPortal19,20の他のデータ型と調べ、統合することができる。サーバー GenePattern21、Mev22、および Morpheus 23 には、主成分解析 (PCA)、kmeans、または自己組織化マップ (SOM) などの確立されたクラスタリング手法が組み込まれています。より革新的な取り組みには、自動化されたルール生成分類器に基づく CamurWeb24と、ランダムフォレスト分類器と投げ縄を実装する TACCO25が含まれます。ここで使用される CorEx アルゴリズムは、データのパターンを説明する因子の階層を見つけるために、多変量情報を最適化します。非線形および階層因子の学習は、PCA4を介して見つかった線形グローバル因子に対する改善された解釈性をもたらすようです。さらに、この技術のサンプル信号の細かい解析により、より一般的に使用される広範なサブタイプに対する正確な腫瘍比較が可能になります。重複因子解析と階層因子解析の組み合わせは、CorExplorer を他のほとんどのアプローチと区別し、視覚化と要約のための新しいツールを必要とします。
CorExplorer 因子解析の重要な部分は、複数の因子だけでなく、重複する階層内に配置された有益な遺伝子パターンを持つ 100 以上の因子を探索する機能です。CorExplorerは生物学的および臨床関連のためのこれらの無数の要因の採掘を促進し、個々の腫瘍の非常に詳細な特徴付けを可能にする。このような多数の要因の教師なし学習は、すべてが病気の生物学に関連するわけではないことを意味します。このような場合、関心のある因子を引き出すか、生存などの臨床データに関連する因子を検索するために、アヌテーションまたは既知の遺伝子を使用することが不可欠です。したがって、CorExplorer を使用すると、この非常に重要なフィルタリング手順を実装できます。腫瘍における因子遺伝子パターンの存在は、パーソナライズされた腫瘍学治療へのアプローチを示唆するかもしれない。さらに、潜在的に有用な治療的組み合わせの発見を可能にする各腫瘍に対する因子スコアの多重性。
生存と相関性の高い因子に対して有意なGOアノテーションが表示されない場合があります。これは、ノイズが多いか、サンプリングされたデータの下で発生する可能性がありますが、クラスターサイズが小さすぎて有意な濃縮スコアを登録できないか、グループが一貫性のない多様な経路からの単一遺伝子の「バスケット」であるなど、他の考えられる原因があります。協会。さらに、KEGGおよびGO生物学的プロセスとは異なるアノテーションのカテゴリは、例えば細胞コンパートメントが適切であり、適切でありうる。これらは、プロトコルで示されているように StringDB にリンクすることでアクセスできます。CorExplorerサイトの遺伝子オントロジー濃縮分析は、現在、因子における遺伝子の重み付けを考慮していませんが、近い将来に改善される可能性が高いです。遺伝子リストオプションは、外部ツールでさらなる分析のために完全な因子遺伝子リストをダウンロードすることを可能にする「ウィンドウを追加」の下で利用可能です。
ウェブサイトの目的のために、CorExは各データセットで5回実行され、全体的な相関全体が最も大きい結果を生み出した実行は保持されました。複数の実行の結果を統計的に表現することは、より有益であり、将来の作業の目標です。さらに、サーバー上で利用可能な腫瘍タイプのセットはかなり小さいですが、ユーザーの関心に応じて時間の経過とともに拡大すると予想されます。
前述したように、CorExplorer は CorEx RNA-seq 因子関係と臨床およびデータベース情報を可視化するため、さまざまな異なる尋問モードを可能にします。このツールが、RNA-seq分析の力を腫瘍学における発見と臨床応用に活用するさらなる研究につながることを期待しています。
開示事項
著者らは、彼らが競合する金銭的利益を持っていないと宣言します。
謝辞
GVはDARPA賞W911NF-16-0575によってサポートされました。
資料
| Name | Company | Catalog Number | Comments |
| Public server for CorExplorer website | USC | http://corex.isi.edu | Intel Xeon E5-2690 4-core 2.6 GHz, 8GB RAM. Backend architecture is LAMP: Linux, Apache, MySQL, PHP. |
| Web browser | Google/Apple | Chrome/Safari | Verified web browsers. |
参考文献
- Petryszak, R., et al. The RNASeq-er API-a gateway to systematically updated analysis of public RNA-seq data. Bioinformatics. 33, 2218-2220 (2017).
- Steeg, G. V., Galstyan, A. Maximally Informative Hierarchical Representations of High-Dimensional Data. Proceedings of the Eighteenth International Conference on Artificial Intelligence and Statistics (AISTATS). , (2015).
- Ver Steeg, G., Galstyan, A. Discovering structure in high-dimensional data through correlation explanation. Advances in Neural Information Processing Systems. , (2014).
- Pepke, S., Ver Steeg, G. Comprehensive discovery of subsample gene expression components by information explanation: therapeutic implications in cancer. BMC medical Genomics. 10, 12 (2017).
- Byron, S. A., Van Keuren-Jensen, K. R., Engelthaler, D. M., Carpten, J. D., Craig, D. W. Translating RNA sequencing into clinical diagnostics: opportunities and challenges. Nature Reviews Genetics. 17, 257 (2016).
- Cancer Genome Atlas Research Network. Comprehensive molecular profiling of lung adenocarcinoma. Nature. 511, 543 (2014).
- Cancer Genome Atlas Network. Comprehensive molecular characterization of human colon and rectal cancer. Nature. 487, 330 (2012).
- Akbani, R., et al. Genomic classification of cutaneous melanoma. Cell. 161, 1681-1696 (2015).
- Cancer Genome Atlas Research Network. Integrated genomic analyses of ovarian carcinoma. Nature. 474, 609 (2011).
- Grossman, R. L., et al. Toward a shared vision for cancer genomic data. New England Journal of Medicine. 375, 1109-1112 (2016).
- Moynahan, M. E., Chiu, J. W., Koller, B. H., Jasin, M. Brca1 controls homology-directed DNA repair. Molecular Cell. 4, 511-518 (1999).
- Szklarczyk, D., et al. STRING v11: protein–protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets. Nucleic Acids Research. 47, 607-613 (2018).
- Durgeau, A., Virk, Y., Corgnac, S., Mami-Chouaib, F. Recent advances in targeting CD8 T-cell immunity for more effective cancer immunotherapy. Frontiers in Immunology. 9, 14 (2018).
- Sato, E., et al. Intraepithelial CD8+ tumor-infiltrating lymphocytes and a high CD8+/regulatory T cell ratio are associated with favorable prognosis in ovarian cancer. Proceedings of the National Academy of Sciences of the United States of America. 102, 18538-18543 (2005).
- De Moura, M. B., et al. Mitochondrial respiration-an important therapeutic target in melanoma. PLoS One. 7, 40690 (2012).
- Folkman, J., Merler, E., Abernathy, C., Williams, G. Isolation of a tumor factor responsible for angiogenesis. Journal of Experimental Medicine. 133, 275-288 (1971).
- Takahashi, S. Vascular endothelial growth factor (VEGF), VEGF receptors and their inhibitors for antiangiogenic tumor therapy. Biological and Pharmaceutical Bulletin. 34, 1785-1788 (2011).
- Subramanian, A., et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proceedings of the National Academy of Sciences of the United States of America. 102, 15545-15550 (2005).
- Cerami, E., et al. The cBio Cancer Genomics Portal: An Open Platform for Exploring Multidimensional Cancer Genomics Data. Cancer Discovery. 2, 401-404 (2012).
- Gao, J., et al. Integrative Analysis of Complex Cancer Genomics and Clinical Profiles Using the cBioPortal. Science Signalling. 6, 1 (2013).
- Reich, M., et al. GenePattern 2.0. Nature Genetics. 38, 500 (2006).
- Wang, Y. E., Kutnetsov, L., Partensky, A., Farid, J., Quackenbush, J. WebMeV: A Cloud Platform for Analyzing and Visualizing Cancer Genomic Data. Cancer Research. 77, 11-14 (2017).
- . Morpheus Available from: https://software.broadinstitute.org/morpheus (2019)
- Weitschek, E., Lauro, S. D., Cappelli, E., Bertolazzi, P., Felici, G. CamurWeb: a classification software and a large knowledge base for gene expression data of cancer. BMC Bioinformatics. 19, 354 (2018).
- Chou, P. -. H., et al. tACCo, a Database Connecting transcriptome Alterations, pathway Alterations and Clinical outcomes in Cancers. Scientific Reports. 9, 3877 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved