このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
自動冷凍工場におけるクライオグラインによる細胞抽出物の調製
要約
タンパク質の分解や変性を最小限に抑える極低温冷凍工場を用いて、酵母や他の細胞から全細胞抽出物を調製するための信頼できる方法を説明します。細胞抽出物は、機能性タンパク質複合体の精製、プロテオミクス分析、共免疫沈降試験、不安定タンパク質修飾の検出に適しています。
要約
遺伝子操作の容易さと出芽酵母 サッカロミセスセレビシエ における真核細胞機械の強力な進化的保全は、それを著名な遺伝モデル生物にしました。しかし、効率的なタンパク質の単離は細胞の最適な破壊に依存するため、細胞タンパク質の生化学的分析のための酵母の使用は、酵素的に消化するのに高価な細胞壁(リチケースまたはジモリアーゼを使用)によって妨げられ、サンプルの加熱を引き起こすことなく機械的に破壊することは困難です(伝統的なビーズビーター、フランスのプレスまたはコーヒー粉砕機を使用して)。乳鉢と害虫を使用した液体窒素(LN2)下の酵母細胞の手動粉砕はサンプルの過熱を避けますが、作業集約的であり、オペレータ間の細胞のリシスの変動を受けます。長年にわたり、自動冷凍工場で細胞の凍結粉砕を用いた高品質酵母エキスの調製に成功してきました。LN2 を用いて達成した-196 °Cの温度は、タンパク質、核酸および他の高分子の検索を可能にする、プロテアーゼおよびヌクレアーゼによる分解から生物学的材料を保護する。ここでは、LN2 への液滴添加を通じてライシスバッファー内の細胞の懸濁液を最初に凍結し、「ポップコーン」と呼ばれる細胞の凍結液滴を生成することを含む出芽酵母細胞について詳細に説明する。このポップコーンは、冷凍工場でLN2 の下で粉砕され、ゆっくりと解凍され、難解な破片を除去するために遠心分離によって明確化された凍結した「粉末」抽出物を生成します。得られた抽出物は、タンパク質または核酸の精製、プロテオミクス分析、または共免疫沈降試験などの下流の用途に対応できます。この技術は、様々な微生物、植物および動物組織、サンゴを含む海洋標本、ならびに法医学および永久凍土化石標本からDNA/RNAを単離する細胞抽出物に広く適用される。
概要
酵母は、研究者が利用できる遺伝的および生化学的ツールが豊富にある単純な真核生物であるため、タンパク質研究に人気のモデル生物です。その頑丈な細胞壁のために、研究者が直面する1つの課題は、細胞の内容物を損なうことなく細胞を効率的にlysすることです。酵素リシス(zymolyase)2、3、化学液化4、凍結融解による物理的な液化5、圧力ベース(フレンチプレス)6、7、機械(ガラスビーズ、コーヒーグラインダー)8、超音波処理ベース10および低温2、11を含む酵母細胞の破壊を介してタンパク質抽出物を得るために利用可能な異なる方法が利用可能である。細胞のリシスの効率およびタンパク質収量は、使用される技術によってかなり異なり、したがって、ライセートの所望の下流アプリケーションの最終的な結果または適合性に影響を与える。不安定なタンパク質、翻訳後修飾のタイミング、または温度に敏感なタンパク質を研究する場合、調製中のサンプル損失や分解を最小限に抑える方法を使用することが特に重要です。
| 抽出準備技術 | 詳細 | 利点 | 欠点 | ダウンストリーム解析 | 参照 |
| フランスのプレス:ザイモリャーゼを使用した酵素処理による高圧ホモジナイザー(別名マイクロフルダイザー) | ザイモリアーゼ-20T、マイクロ流体化用高圧ホモジナイザー。ディスラプターは、空気駆動式高圧ポンプ(比1:250;必要な空気圧0.6-l MPa)と、追加のバックプレッシャーユニットを備えた特別な破壊室で構成されています。処理には、最小サンプルサイズ20 mLが必要です。 | 結合されたプロトコルを使用して得られた最終的な全破壊は、Zymolyaseなしで均質化のみを使用して95 MPaで4パスで32%の破壊と比較して、95 MPaの圧力で4パスで100%に近づいた。 | 小規模アプリケーションには適していません。酵素は、大規模な準備のために高価になる可能性があります。 | タンパク質精製 | 6 |
| ビーズビーター:Zymolyase処理された細胞は、高速準備器具でガラスビーズで固着 | ほぼ同量の冷たい乾燥した酸洗浄0.5mmガラスビーズが、リシスバッファー内の所定の量の細胞ペレットに添加され、細胞は激しい手動攪拌によって破壊される。 | タンパク質精製のためではなく、アッサンの目的のために、多くの異なる小さな酵母培養物からの抽出物を作る場合に特に有用である。 | ガラスビーズの手順の間に、タンパク質は、タンパク質の変性につながる広範な発泡を引き起こす過酷な処理されます。細胞破断の量はさまざまですが、タンパク質の変質は機械的破損時に4°C以上の抽出物を加熱した結果として生じる可能性があります。 | 主にDNAとRNAの分析だけでなく、ウェスタンブロッティングの有無にかかわらず、ゲル電気フェオレシスを変性することによってタンパク質分析も行います。 | 8 |
| 続いて、浸透性ショックとDounce均質化の組み合わせを用いたリシスによるジモリアーゼ治療 | 細胞壁の酵素消化後、球状体は、Dounceホモジナイザーで15〜20ストロークの密着性害虫(クリアランス1〜3μm)でリセ化されます。 | BJ926やEJ101などのプロテアーゼ欠損株を用いることが有利である。これは、酵母細胞を破壊する最も穏やかな方法であり、したがって、複雑な酵素機能(例えば、翻訳、転写、DNA複製)を行うことができる抽出物を調製し、高分子構造(例えば、リボソーム、スプライスソーム)の完全性を維持する必要がある。クロマチン研究(ブルームとカーボン、1982)や核タンパク質抽出物(LueとKornberg、1987)に使用できる無傷の核を単離するのにも有用です。 | スフェロプラストのライシス手順の主な欠点は、特に大規模な調製物(>10リットル)では比較的面倒で高価であり、長いインキュベーション期間はタンパク質分解またはタンパク質修飾につながる可能性がある。 クロマチン製剤の場合、それらは(ヌクレオソームはしごの完全性に基づいて)差動遠心によって生成されるものよりも変化するか、または低い品質であるように見える。 | クロマチン研究のために無傷核を分離し、複雑な酵素機能を実行できる抽出物、完全性を必要とする抽出物 高分子構造、核タンパク質抽出物 | 2 |
| 乳鉢/乳棒またはブレンダーを使用して液体窒素を粉砕することにより、フラッシュ凍結細胞の細胞破壊 | 細胞は液体窒素で直ちに凍結し、次いで、乳鉢で手動で粉砕するか、液体窒素の存在下でワーリングブレンダーを使用して溶菌する。 | プロトコルは迅速かつ簡単です。それは非常に大きい培養物を含む酵母細胞の様々な量を収容できる。その主な利点は、細胞が積極的に成長している状態から液体窒素(−−196°C)にすぐに取られ、プロテアーゼやヌクレアーゼなどの分解酵素活性が低下し、タンパク質(例えば、ホスファターゼおよびキナーゼ)を修飾する活性である。これは、大規模なタンパク質精製のための単一の酵母培養物から全細胞抽出物を作るのに特に適しています。 | 不注意な捜査官にとって少し乱雑で潜在的に危険です。小さいサンプル(すなわち、10〜100ml酵母培養)は、ブレンダーで効果的に破壊するのに十分な凍結細胞塊の塊の質量がないため、容易に処理されない。個々のサンプルを処理し、使用間に機器をきれいにすることは時間がかかります。 | 大規模なタンパク質精製のための単一の酵母培養物からの全細胞抽出物。 | 2 |
| オートリシス、ビーズミル | pH 5.0、 50 °C、 24 時間、 200 rpm / Ø 0.5 mm、 5 × 3 分/3 分 | 迅速かつ効率的なリシス、特に小規模な抽出物の調製 | 熱の生成は、高分子の変性と分解につながります。ビーズの打ち装置が必要です。 | 小規模分析。 | 10 |
| 自動起き、超音波処理 | pH 5.0,50°C,24時間,200rpm,4×5分/2分,パルサー80%,パワー80% | 超音波処理装置は、通常、ほとんどの機関で利用可能です。 | 熱の生成は、高分子の変性と分解につながります。超音波処理装置が必要です。遅いリシスは24時間以上かかることがあります。 | 酵母細胞壁製剤。 | |
| 沸騰および凍結解凍プロセス | 標準の冷凍庫と暖房ブロックまたは温水風呂以外に特別な設備は必要ありません。 | 効率的、再現性、シンプルで安価。 | 熱の生成は、高分子の変性と分解につながります。 | PCRによるDNA分析。 | 5 |
表1:酵母エキスの調製に利用可能な方法の比較。
極低温粉砕(別名極低温粉砕/極低温粉砕)は、定量的または定性的な分析のために信頼性の高い方法で温度に敏感なサンプルから核酸、タンパク質または化学物質を取り出すために一般的に使用されます。バイオテクノロジー、毒物学、法医学12、13、環境科学、植物生物学14、食品科学など、さまざまな分野での複数の用途に成功しています。無傷の生体高分子の単離は、通常、温度に重大に依存する。非常に低い温度は、プロテアーゼとヌクレアーゼが不活性のままであることを保証し、その後の分析のために無傷のタンパク質、核酸および他の高分子の信頼できる単離をもたらす。実際、冷凍工場は通常、-196 °C(LN2の沸点)のサンプル温度を維持し、DNA/RNAまたはタンパク質の変性および分解を最小限に抑えます。
冷凍工場は、ステンレス鋼のエンドプラグ間で粉砕されるサンプルを含むバイアル内で固体金属バーまたはシリンダーを迅速に前後に移動する電磁粉砕室を採用しています。この装置は、粉砕チャンバー内の磁場を作成し、迅速に反転させます。磁場が前後にシフトすると、磁石はプラグに対してサンプルを押しつぶし、「クライオグラインディング」とポップコーンの粉砕を達成します。フリーザーミルは乳鉢と害虫を置き換え、複数のサンプル(または最大4個の小さいサンプルを同時に)の順次処理を可能にし、手動研削に伴うユーザー間の変動を回避します。サンプルが処理されると、セル抽出物をさまざまなダウンストリーム アプリケーションに使用できます。
プロトコル
1. 酵母ポップコーンの調製
- YPD培地の0.5 Lで酵母細胞を1 x 107 細胞/mLの密度に成長させます。コールターカウンターまたはその他の手段を使用してセルをカウントします。
- 遠心分離細胞を2,400gおよび4°Cで10分間用いた。
- 500mLの氷冷脱イオン18メガオームミリQ水で各サンプルを1回洗います。
- 氷冷リシスバッファーの15 mLでペレットを再懸濁する [20 mM HEPES-KOH pH 7.5, 110 mM KCl, 0.1%ツイーン、10%グリセロール、10mM βメルカプトエタノール、プロテアーゼ阻害剤カクテル、10μMプロテアソーム阻害剤MG-132、1mMデアセチラーゼ阻害剤ナトリウムブチレートおよびホスファターゼ阻害剤(1 mMバナテン酸ナトリウム、50mMナトリウムフッ化物、50mm β)を添加した。詳細については、資料一覧を参照してください。サンプルが次のステップに向けて準備ができるまで、再懸濁した細胞を氷の上に置いておきます。
注:多数の非イオン性洗剤を含む多種多様なリシスバッファーを、下流の用途に基づいてサンプルおよびそれに含まれる特定の阻害剤と共に使用することができます。プロテアーゼおよびプロテアソーム阻害剤の含有は、タンパク質分解を防ぐために重要です, 特に抽出物が解凍された後. - 冷やされた血清学的ピペットを使用して、ドライアイスに保たれ、リムのすぐ下までLN2 で満たされた1つまたは複数の50 mL遠心分離管に細胞懸濁液を一度に1滴ずつゆっくりと加えることで、スナップ冷凍酵母ポップコーンを作ります。急速な蒸発による損失を補うために頻繁に管の液体窒素を上げる。窒素窒息に伴う危険を避けるために、換気の良い場所で作業してください。
- 50 mLチューブの代わりに、ポップコーン調製のために、50mLチューブを複数使用し(または、極低温に耐えることができる大きなプラスチック遠心分離器ボトルなど、大きな口を持つ任意の容器)を使用します(図1A)。容器とその開口部が大きければ大きいほど、ポップコーンの塊が大きな凝集体を形成するのを防ぐため、ポップコーンの準備が容易になります。ポップコーンの0.3-0.5 cmのサイズ範囲は、冷凍工場を使用して適切な粉砕/粉砕を達成するのに理想的です(図1B)。
- LN2 が-80°Cで保管する前に、ポップコーンを含むチューブから完全に蒸発していることを確認してください。 酵母ポップコーンを調製した後、-80°Cで維持すると数年間安定して停止させることができる。

図1:酵母ポップコーン調製(A)酵母「ポップコーン」は、LN2における細胞懸濁液の滴下凍結によって作られる。ゴムバンドと一緒に保持し、ドライアイスで満たされたアイスバケツに入れた1〜3本の50 mLチューブを使用しています。チューブは、リムのすぐ下までLN2で満たされ、すべての細胞懸濁液がポップコーン(B)に作られるまで、液体窒素を頻繁にトッピングして、ほとんど満杯に保ちます(B)ポップコーンの大きさは、最適な研削効率の重要な決定要因です。ポップコーンのサイズ範囲は直径0.3~0.5cmの範囲にする必要があります。この図の大きなバージョンを表示するには、ここをクリックしてください。
2. クライオグラインド
- 開始するには、冷凍工場を冷却し、バイアル、供給を粉砕し、すべてのサンプルの機械を操作するのに十分なLN2 があることを確認してください。5つ未満のサンプルでは、LN2 の30〜35 Lで十分です。充填ラインまでLN2 で冷凍工場の部屋を充填します。
警告:LN2 を含むすべてのステップは、換気の良い領域で行われるべきであり、冷凍工場自体は窒息の危険を避けるために、そのような領域に配置する必要があります。適切な履物、ラボコート、安全メガネ、ニトリル手袋のベース層、熱手袋、続いてニトリル手袋の別のペアを含む個人的な保護具を着用してください。LN 2 を扱う際には細心の注意を払ってください。 - 冷凍庫の製粉所の蓋をゆっくりと閉じて、LN2 の飛沫を避け、機械が冷めるまで数分間待たします。次のステップに進む前に、蒸発による損失を補うために、チャンバにLN2 を補充する必要があるかもしれません。
注: 必要に応じて、充填ラインまでだけ LN2 を補充します。これは、危険と機械に有害な両方であることが可能なように部屋をオーバーフィルしないでください。自動補充システムは、冷凍工場に接続された貯蔵タンクから直接、必要に応じて蒸発LN2を補充することができる冷凍工場のために利用できる。 - 大きな研削バイアルと磁気インシクターバーをLN2 でダンクして、別の小さなデュワーに保管して冷やします。LN2 がバブリングを停止したときに、すべての LN2 をデスキャンします。次に、サンプル/酵母ポップコーンを粉砕バイアルに加え、2つのステンレススチールエンドプラグでしっかりと密封します。
- 粉砕バイアルの3分の1以上をサンプルで満たさないで、粉砕の効率を低下させることができます。代わりに、より大きなサンプルを2つ以上のバイアルに分割し、それらを順次に粉砕することによって処理することができます。より小さい粉砕のバイアルは小さいサンプル(3 mLまで)のために使用することができる。
- 粉砕バイアルを冷凍工場のチャンバーに置き、所定の位置にロックします。蓋を閉じます。
- 出芽酵母の場合、サンプルを2分/サイクル(サイクル間の冷却のための2分休憩で)14の破砕速度で合計3サイクル粉砕します。サイクルが完了した後に機械が停止したら、冷凍工場の蓋をゆっくりと開け、粉末状の冷凍セルのライゼでバイアルのロックを慎重に解除し、冷凍工場から取り外します。複数の小さなバイアルを使用する場合は、一度に1つのバイアルを取り除き、ドライアイスに入れる作業を迅速に行います。
注:通常、同じタイプのサンプルで異なるサイズのバイアルを使用する場合、粉砕パラメータを調整する必要はありません。しかし、サイクル数や粉砕率などの粉砕パラメータは、ライスの容易さに応じて、異なるサンプルタイプについて経験的に決定する必要があります。ほとんどの哺乳類細胞と軟組織は、10の破砕速度で1〜2サイクルでライジングします。細菌、酵母、フライ幼虫、成体のフルーツハエなどのサンプルをライゼするのが難しく、最大速度で3〜6サイクルを必要とし、骨、歯などの硬い組織は最大10サイクルを必要とするかもしれません。解凍されたサンプルの小さなアリコートは、粉砕の前後に顕微鏡下で見ることができ、リシス効率を決定するために無傷の未分解細胞の数をカウントする。出芽酵母からのタンパク質およびDNAの放出に対する凍結粉砕パラメータの影響の優れた分析が以前に発表された。 - サンプルが解凍しないように迅速に作業し、開口部ツールを使用して、端部の1つを慎重に緩めます(研削中にやや緩くなったように見えるものを選びます)。次に、長い鉗子(ドライアイス付きのバケツで事前に冷やされている)を使用して、インプロクタバーを取り除きます。粉砕されたサンプルを反転して、LN2 で事前に冷やし、ドライアイスに保つポリスチレン計量皿に粉砕バイアルをタップして収集します。
- 粉末状の冷凍細胞のライセートをすべて粉砕バイアルから回収したら、粉末状のライセートを計量皿から50 mLチューブに戻し、最良の結果を得るために次に説明する遅い解凍ステップに直ちに進みます。あるいは、冷凍粉末のライセートを-80°Cで一晩保存しますが、これはいくらか劣化する可能性があります。
- 氷と水の50%のスラリーでアイスバケツを準備し、磁気スターラーと攪拌板の上に置きます。サンプルを保持するために、ワイヤーラックまたは別の適切なラックを氷のスラリーに浸します。
- 次いで、磁気攪拌機を用いてスラリーを一定に撹拌して氷のスラリー浴のサンプルをゆっくりと解凍する。融解氷を置き換えるために氷を追加します(氷のバケツがあふれないように、一部の水を捨てる必要があります)。いくつかの阻害剤は水溶液バッファー内の半分の命が短いので、サンプルが解凍を開始したら、さらにプロテアーゼ阻害剤カクテル( 材料表を参照)と10 μMプロテアソーム阻害剤MG-132をライセートに加えます(約30分後)。
- 5分ごとにチューブの外側に形成された氷を取り除き、解凍プロセスを迅速化します。室温以上でサンプルを急速に解凍すると、著しい劣化を招く可能性があることに注意してください。
- サンプルが完全に解凍された後(サンプルの量に応じて1時間以上かかる場合があります)、4°Cの冷蔵卓上遠心分離機で〜3,220 gでリセートを遠心分離し、細胞デブリの大部分をリセートから取り除きます。
- 上清を氷上で予冷した50 mLポリカーボネート遠心管に移し、サンプルを4°Cで20分間16,000 gで遠心分離する( 材料表を参照)。
- チューブ内の液体カラムの中心から透明な全細胞抽出物からなる透明な上清のみを、白濁した脂質層(図2)を邪魔することなく、冷やした血清学的ピペットを使用して冷蔵15mLチューブに移します。脂質や破片の持ち越しを防ぐために、遠心分離管内のライセートをすべて除去しないでください。遠心分離管の残ったライセートは、複数回遠心分離して、各スピン後に少量の抽出物を回収することができる。
注:脂質と変性タンパク質は曇り/乳白色に見え、液体/空気界面の上部にあり、時には不溶性の細胞破片で構成されるペレット自体の上に位置します(図2)。
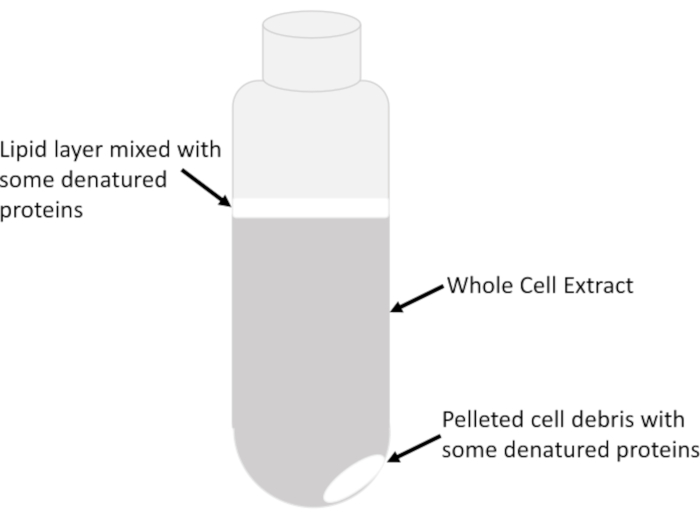
図2:遠心管内の層を抽出します。20分間16,000gでの遠心分離後のチューブ内の全細胞抽出物の主要な目に見える特徴が示されている。各特徴の相対的な存在量は、サンプルの種類、細胞の増殖期(指数対静止)、細胞の再懸濁に使用されるリシスバッファーの量およびリシス効率に依存する。この図の大きなバージョンを表示するには、ここをクリックしてください。
- 遠心分離物 5分間の残りのライセートは、より多くの抽出物を回収する。できるだけ多くの透明な抽出物を回収するために必要な場合は、この5分の遠心分離を複数回繰り返します。ペレット近くのチューブの底に近い脂質を含む濁ったライセートを捨てます。
- これらの抽出物は、タンパク質複合体精製、免疫沈降法、プロテオーム解析などの下流用途に使用します。
結果
酵母細胞のリシスの2つの異なる方法、すなわち4°Cのガラスビーズ粉砕と-196°Cの自動凍結粉砕法を比較し、両方の方法で調製された細胞抽出物中の相対的な回復タンパク質を評価した。この研究のために、私たちは出芽酵母株YAG 1177(MAT a lys2-810 leu2-3,-112 ura3-52 his3-Δ200 trp1-1[am] ubi1-Δ1::TRP1 ubi2-Δ2:ura3 ubi3-Δub-を使用することを選択しました。 2 ubi4-Δ2:::LEU2 [pUB39] [pUB221])
ディスカッション
酵母由来の天然タンパク質の研究の限界は、その硬い細胞壁による酵母細胞の非効率的なリシスである。いくつかの方法が開発されていますが、私たちの手の中で最も一貫した効率的な方法は、ポップコーンとして凍結された酵母細胞の凍結粉砕です。この方法は、他のリシス法と比較して出芽酵母からの高品質の全細胞抽出物の信頼性の高い調製を可能にする。代表的な結果は、クリオグ?...
開示事項
著者らは開示するものは何もない。
謝辞
グンジャン研究所の研究は、国立衛生研究所、国立科学財団、フロリダ州保健省からの資金によって支援されています。学部生のジョン・パーカーに技術支援を感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 50 mL polycarbonate tubes with screw caps | Beckman | 357002 | Centrifuge tubes |
| BD Bacto Peptone | BD Biosiences | 211677 | Yeast YPD media component |
| BD Bacto Yeast Extract | BD Biosiences | 212750 | Yeast YPD media component |
| Beckman Avanti centrifuge | Beckman | B38624 | High speed centrifuge |
| Beckman JLA-9.1000 | Beckman | 366754 | Rotor |
| D-(+)-Dextrose Anhydrous | MP Biomedicals | 901521 | Yeast YPD media component |
| Eppendorf A-4-44 | Eppendorf | 22637461 | Swinging bucket rotor |
| Eppendorf refrigerated centrifuge 5810 R | Eppendorf | 22625101 | Refrigerated centrifuge |
| Glycerol | SIGMA-ALDRICH | G5150-1GA | Volume excluder and cryoprotectant |
| HEPES | FisherBiotech | BP310-100 | Buffer |
| HIS6 antibody | Novagen | 70796 | Antibody for HIS tag |
| KCl | SIGMA-ALDRICH | P9541-1KG | Salt for maintaining ionic strength |
| MG-132 | CALBIOCHEM | 474790 | Proteasome Inhibitor |
| Phosphatase inhibitor cocktail | ThermoFisher Scientific | A32957 | Phosphatase inhibitor cocktail |
| Ponceau S | SIGMA | P7170-1L | Protein Stain |
| Protease inhibitor cocktail | ThermoFisher Scientific | A32963 | Protease inhibitor cocktail |
| Rotor JLA 25.500 | Beckman | JLA 25.500 | Rotor |
| Sodium Butyrate | EM Science | BX2165-1 | Histone Deacetylase Inhibitor |
| Sodium Fluoride | Sigma-Aldrich | S6521 | Phosphatase Inhibitor |
| Sodium Vanadate | MP Biomedicals | 159664 | Phosphatase Inhibitor |
| Sodium β-glycerophosphate | Alfa Aesar | 13408-09-8 | Phosphatase Inhibitor |
| Spex Certiprep 6850 freezer mill | SPEX Sample Prep | 6850 | Freezer Mill |
| TALON Metal Affinity Resin | BD Biosiences | 635502 | For pulling down HIS tagged proteins |
| Tween 20 | VWR International | VW1521-07 | Non-ionic detergent |
| β-Mercaptoethanol | AMRESCO | M131-250ML | Reducing agent |
参考文献
- Botstein, D., Chervitz, S. A., Cherry, J. M. Yeast as a model organism. Science. 277 (5330), 1259-1260 (1997).
- Dunn, B., Wobbe, C. R. Preparation of protein extracts from yeast. Current Protocols in Molecular Biology. , (2001).
- Holm, C., Meeks-Wagner, D. W., Fangman, W. L., Botstein, D. A rapid, efficient method for isolating DNA from yeast. Gene. 42 (2), 169-173 (1986).
- Nandakumar, M. P., Marten, M. R. Comparison of lysis methods and preparation protocols for one- and two-dimensional electrophoresis of Aspergillus oryzae intracellular proteins. Electrophoresis. 23 (14), 2216-2222 (2002).
- Silva, G. A. D., Bernardi, T. L., Schaker, P. D. C., Menegotto, M., Valente, P. Rapid yeast DNA extraction by boiling and freeze-thawing without using chemical reagents and DNA purification. Brazilian Archives of Biology and Technology. 55, 319-327 (2012).
- Baldwin, C., Robinson, C. W. Disruption of Saccharomyces cerevisiae using enzymatic lysis combined with high-pressure homogenization. Biotechnology Techniques. 4 (5), 329-334 (1990).
- Rigaut, G., et al. A generic protein purification method for protein complex characterization and proteome exploration. Nature Biotechnology. 17 (10), 1030-1032 (1999).
- Hudspeth, M. E., Shumard, D. S., Tatti, K. M., Grossman, L. I. Rapid purification of yeast mitochondrial DNA in high yield. Biochimica et Biophysica Acta. 610 (2), 221-228 (1980).
- Szymanski, E. P., Kerscher, O. Budding yeast protein extraction and purification for the study of function, interactions, and post-translational modifications. Journal of Visualized Experiments. (80), e50921 (2013).
- Bzducha-Wróbel, A., et al. Evaluation of the efficiency of different disruption methods on yeast cell wall preparation for β-glucan isolation. Molecules. 19 (12), 20941-20961 (2014).
- Umen, J. G., Guthrie, C. A novel role for a U5 snRNP protein in 3' splice site selection. Genes & Development. 9 (7), 855-868 (1995).
- Smith, B. C., Fisher, D. L., Weedn, V. W., Warnock, G. R., Holland, M. M. A systematic approach to the sampling of dental DNA. Journal of Forensic Sciences. 38 (5), 1194-1209 (1993).
- Sweet, D., Hildebrand, D. Recovery of DNA from human teeth by cryogenic grinding. Journal of Forensic Sciences. 43 (6), 1199-1202 (1998).
- Lorenz, W. W., Yu, Y. S., Dean, J. F. An improved method of RNA isolation from loblolly pine (P. taeda L.) and other conifer species. Journal of Visualized Experiments. (36), e1751 (2010).
- Singh, M. R., Roy, S., Bellare, J. R. Influence of Cryogenic Grinding on Release of Protein and DNA from Saccharomyces cerevisiae. International Journal of Food Engineering. 5 (1), (2009).
- Singh, R. K., Kabbaj, M. H., Paik, J., Gunjan, A. Histone levels are regulated by phosphorylation and ubiquitylation-dependent proteolysis. Nature Cell Biology. 11 (8), 925-933 (2009).
- Liang, D., Burkhart, S. L., Singh, R. K., Kabbaj, M. H., Gunjan, A. Histone dosage regulates DNA damage sensitivity in a checkpoint-independent manner by the homologous recombination pathway. Nucleic Acids Research. 40 (19), 9604-9620 (2012).
- Singh, R. K., Gonzalez, M., Kabbaj, M. H., Gunjan, A. Novel E3 ubiquitin ligases that regulate histone protein levels in the budding yeast Saccharomyces cerevisiae. PLoS One. 7 (5), 36295 (2012).
- Gunjan, A., Verreault, A. A Rad53 kinase-dependent surveillance mechanism that regulates histone protein levels in S. cerevisiae. Cell. 115 (5), 537-549 (2003).
- Gill, P., et al. Identification of the remains of the Romanov family by DNA analysis. Nature Genetics. 6 (2), 130-135 (1994).
- Alain, K., et al. DNA extractions from deep subseafloor sediments: novel cryogenic-mill-based procedure and comparison to existing protocols. Journal of Microbiological Methods. 87 (3), 355-362 (2011).
- Mohammad, F., Buskirk, A. Protocol for Ribosome Profiling in Bacteria. Bio-Protocol. 9 (24), 3468 (2019).
- Liew, Y. J., et al. Identification of microRNAs in the coral Stylophora pistillata. PLoS One. 9 (3), 91101 (2014).
- Lopez de Heredia, M., Jansen, R. P. RNA integrity as a quality indicator during the first steps of RNP purifications: a comparison of yeast lysis methods. BMC Biochem. 5 (14), (2004).
- Grant, L. J., et al. Purified plant cell walls with adsorbed polyphenols alter porcine faecal bacterial communities during in vitro fermentation. Food & Function. 11 (1), 834-845 (2020).
- Lolo, M., et al. Cryogenic grinding pre-treatment improves extraction efficiency of fluoroquinolones for HPLC-MS/MS determination in animal tissue. Analytical and Bioanalytical Chemistry. 387 (5), 1933-1937 (2007).
- Santos, D., et al. Determination of Cd and Pb in food slurries by GFAAS using cryogenic grinding for sample preparation. Analytical and Bioanalytical Chemistry. 373 (3), 183-189 (2002).
- da Silva, E. G. P., et al. Fast method for the determination of copper, manganese and iron in seafood samples. Journal of Food Composition and Analysis. 21 (3), 259-263 (2008).
- Kamogawa, M. Y., Nogueira, A. R. A., Costa, L. M., Garcia, E. E., Nobrega, J. A. A new strategy for preparation of hair slurries using cryogenic grinding and water-soluble tertiary-amines medium. Spectrochimica Acta Part B-Atomic Spectroscopy. 56 (10), 1973-1980 (2001).
- Sillen, A., Hall, G., Richardson, S., Armstrong, R. Sr-87/Sr-86 ratios in modern and fossil food-webs of the Sterkfontein Valley: Implications for early hominid habitat preference. Geochimica Et Cosmochimica Acta. 62 (14), 2463-2473 (1998).
- Nielsen-Marsh, C. M., et al. Sequence preservation of osteocalcin protein and mitochondrial DNA in bison bones older than 55 ka. Geology. 30 (12), 1099-1102 (2002).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved