このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
レーザーフリーヒドロキシルラジカルタンパク質フットプリントによるタンパク質の高次構造解析
要約
このプロトコルは、インラインラジカル線測定とプラズマ光源を使用して、フラッシュ酸化タンパク質のフットプリントを実行する方法を提示する。この方法は、タンパク質研究の高速光化学酸化の再現性を簡素化し、改善するために、危険なUVレーザーを置き換えます。
要約
Hydroxylラジカルプロテインフットプリント(HRPF)は、タンパク質構造の変化、タンパク質とタンパク質の相互作用、またはタンパクリガンド相互作用に関する情報を提供する、新たな有望な高次構造解析技術です。HRPFは、ヒドロキシルラジカル(▪OH)を利用して、タンパク質の溶媒アクセス可能な表面を不可逆的に標識します。HRPFを実行する本質的な複雑さ、コスト、および危険な性質は、バイオ医薬品における広範な採用を実質的に制限しています。これらの要因には、1)実質的な安全対策を必要とする複雑で危険で高価なレーザーの使用が含まれます。2)比較研究を制限する ▪OHのバックグラウンド清掃によって引き起こされるHRPFの再現性。本書は、レーザーフリーHRPFシステムの動作プロトコルを提供します。このレーザーフリーHRPFシステムは、インラインラジカル線量測定を用いた高エネルギー、高圧プラズマ光源フラッシュ酸化技術を利用しています。プラズマ光源は、レーザーベースのHRPFシステムよりも安全で使いやすく、ヒドロキシルラジカルの生成効率が高く、インラインラジカル線量計は研究の再現性を高めます。レーザーフリーHRPFシステムを組み合わせることで、レーザーベースの技術の欠点と限界に対処し、克服します。
概要
タンパク質の立体構造および関連する高次構造(HOS)は、適切な生物学的機能および異常な挙動1の主要な決定因子である。同じことが、その構造と機能活性が生産と環境の様々な側面に依存しているバイオ医薬品にも当てはまります。HOSのバイオ医薬品の変化は、望ましくない薬理学的および患者免疫学的応答2、3に起因する有害薬物反応(ADR)に関連している。ADRの出現は、タンパク質HOSがバイオ医薬品の安全性と有効性において果たす重要な役割をバイオ医薬品業界に警告し、HOS分析4を新しく改良する必要性を確立しました。
ヒドロキシルラジカルプロテインフットプリント(HRPF)は、タンパク質HOSの変化を追跡する有望な技術です。HRPFは、OH▪を伴うタンパク質の外装の不可逆的な標識を伴い、続いて質量分析(MS)分析を行い、タンパク質5、6、7の溶媒アクセス可能表面を同定する。HRPFは、タンパク質HOSとその機能8、9の欠陥の検出に成功し、モノクローナル抗体(mAb)10、11、12、13のHOSを特徴付け、リガンド14の結合Kdを決定し、さらに多くの15、16、17、18、19を検出する。HRPF用の▪OHを生成する一般的な方法は、タンパク質の高速光化学酸化(FPOP)であり、H2O2の光起こしから▪OHを生成するために高エネルギー、高速UVレーザーを採用している。ほとんどの場合、FPOPは、呼吸および眼の損傷を避けるために相当な保護手段を要求する危険ガス(KrF)を採用した高価なエキシマレーザーを使用する20。吸入の危険を避けるために、他の人は、有毒ガスの使用を排除するが、依然として高価であり、重要な運用上の専門知識を必要とし、目の怪我からユーザーを保護するために広範な迷子光制御を要求する周波数四足数ネオジムイットリウムアルミニウムガーネット(Nd:YAG)レーザー21を使用しています。
HRPFを用いて十分な情報を得ることができるが、バイオ医薬品の幅広い採用は満たされていない。限られたHRPFの採用のための2つの障壁は、1)実質的な安全対策20を必要とする危険で高価なレーザーの使用を含む;2)比較研究を制限する ▪OHのバックグラウンド清掃によって引き起こされるHRPFの再現性を22.レーザー使用に取って代わる高速、高エネルギープラズマフラッシュ光度ユニットは、FPOPを安全に簡単に行うために開発されました。HRPF実験の再現性を向上させるために、リアルタイムラジカルドジメトリーが実施される。
HRPFの実践は、▪OH22のバックグラウンド清掃に起因する再現性の不足によって制限されている。▪OHはタンパク質トポグラフィーの優れたプローブですが、調製物に見られる多くの成分と反応するため、標的タンパク質を酸化するために利用可能なラジカルの有効濃度を測定する必要があります。バッファー調製、過酸化水素濃度、リガンド特性、または光分解の変動は、HOS微分試験において曖昧さを生み出す対照群と実験群の間に酸化的な違いをもたらす可能性がある。リアルタイムラジカル・ドジメトリーの追加により、OH負荷▪効果の調整が可能になり、HRPF実験中の信頼性と再現性が向上します。FPOPにおけるラジカル・ドシメトリーの使用は、他の場所で説明されている23,24,25、最近の刊行26で詳しく論じられています。ここでは、新しいフラッシュ光反応系とリアルタイムの量体系を用いて、エキシマレーザーを用いた場合のFPOP実験におけるペプチド酸化のレベルを比較して、馬のアポミオグロビン(aMb)を標識する。
プロトコル
1. キャピラリーチューブの取り付け
- シリカ切断石を使用して、250 μm 内径(ID)シリカ毛管を27インチに切断します。毛細管の端部がきれいな、まっすぐな切り傷を点検しなさい。
- ポリイミドコーティングを焼き払って、長さ約15mmの2つの窓を作成します。「下端」から始めて、最初の光起点窓を「下端」から90mm離れ、「下端」から225mm離れた場所に向けて行います。
注:コーティングが燃え尽きると、毛細管は非常に壊れやすいです。 - ポート 5 でナットとフェルールを外し、フェルールの円錐の端のすぐ向こうにキャピラリーの「下端」を挿入します(図 1A)。
- 光起こしモジュールで、光起細胞キャップをまっすぐ引き出して取り外し、キャピラリーを所定の位置に保持する磁気的に取り付けられた金属マスクを取り外します。
注意:光起こセルキャップの内側は湾曲したミラーであり、ミラーに触れるものは何も許可しないでください。 - ドーシメータモジュールで、左側のタブを押して、ドーシメーターセルを開き、右に開いて、その後、その細胞を振ります。キャピラリーを所定の位置に保持する磁気マウントクリップを取り外します。
- 毛細管を、光起細胞の基部の溝付きチャネルに入れる。毛細血管ウィンドウを、光度セルウィンドウで中央に配置します。片方の手でキャピラリーを所定の位置に持ち、マグネットマスクを加えてキャピラリーを所定の位置に保持します。光のキャップを元の位置に戻します。
- 毛細管を、ドーシメータセルの基部の溝溝に入れる。第2のキャピラリーウィンドウを、線量計細胞の基部の中央にある小さな開口部の上に中央に配置します。片方の手でキャピラリーを所定の位置に持ち、キャピラリーを所定の位置に保持する位置に2つの磁気クリップを置きます。クリックが閉じるまで、ドーシメータ セルを閉じます。
- 製品コレクターのキャピラリーリフトの上に、ひざ付きナットを通して毛細管を挿入します(図1B)。キャピラリーをバイアルの底のすぐ上まで伸ばします。
注: 実験中にキャピラリーが急流溶液に沈み込むには、キャピラリーがバイアルの底に到達することが重要です。
2. インジェクションループの取り付け
- 外径1/16インチ、内径0.015インチ(381μm)のテフロンチューブを使用してください。式 1 に従って、目的の体積に必要なチューブの長さを計算します。
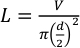
ここでLは管の長さをミリメートルで、Vはマイクロリットルで所望の容積、dは管内径をミリメートル単位で表します。射出体積が25μL、内径が381μmの場合、チューブ長さは約219.3mmである必要があります。
注:20 μL 未満のボリュームの場合は、内径 0.01 インチの PTFE チューブを使用してください。 - チューブカッターを使用して、テフロンチューブを必要な長さにカットします。チューブの端部がきれいなストレートカットを確認します。
- ナットの 1 つを通して新しい射出ループの一端を挿入し、チューブの端に新しいフェルールを配置します。フェルールとナットを所定の位置に保持し、チューブを注入バルブのポート 3 に挿入して、バルブに底を付けます。チューブをしっかりと保持し、ナットフィンガーをしっかりと締めます。レンチを使用して、ナットを1/4ターンさらに締めます。アセンブリを削除して検査します。
メモ:フェルールの位置が固定されていない場合は、再インストールして1/8ターンを締め付けます。ループの反対側で繰り返します。 - 両端にナットと固定フェルールが付いたら、一方の端をポート 3 に緩やかにねじ込み、もう一方の端をポート 6 にねじ込みます (図 1C)。一度位置に両側を引き締めて指を締め、1/4はレンチで指を締めます。
3. 光度システムを初期化する
- 次の順序で光の起動モジュールをオンにする:(1)流体モジュール(2)光起合モジュール(3)ドシメトリーモジュール(4)製品コレクタ、最後に(5)システムコンピュータを起動し、制御ソフトウェアを起動します
メモ:ドーシメータモジュールを最低30分はコールドスタートからウォームアップしてください。 - シリンジポンプの「バルブ」位置(図1D)から10 mLのバッファに完全に沈入管。「廃棄物」位置(図1D)からチューブを配置し、注射器ポートのポート2(図1C)から空の容器にチューブを置いて廃棄物を回収します。
注:タンパク質が中断されるバッファを使用してください。推奨されるバッファは 10 mM NaPO4 です。 - 製品コレクターカルーセルで、Hと6のマークの位置に1.5 mLマイクロ遠心チューブを配置します。
- 250 μm ID キャピラリーを使用する場合は、負荷流量を 500 μL/min、処理流量を 7.5 μL/min に、廃棄物流量を 500 μL/min に設定します。
- 式 2 を使用して半自動モードを使用するフラッシュ遅延、製品時間、および無駄な時間を計算します。
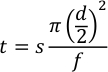
ここで、t は分単位の時間、s は毛管距離をミリメートル単位、d は毛管の内径をミリメートル、f は μL/min の流量です。- フラッシュ遅延の場合、キャピラリーの「下端」から最初のウィンドウまでの距離は、およそ90mmでなければなりません。250 μm ID キャピラリーと処理流量 7.5 μL/m を使用すると、サンプルは約 35 秒で光度のウィンドウに到着します。サンプルが到着する前に 1 Hz で 2 回のフラッシュが発生するので、フラッシュ遅延を 33 秒に設定します。
- 製品時間については、注入液が射出弁から毛細管の端まで流れるのにかかる時間を入力します。27インチ長い250 μm IDキャピラリーの場合、製品時間を4.5分に設定します。
- 無駄な時間については、注入された溶液の総量が収集される時間を入力します。このとき、製品コレクターは製品位置から廃棄物バイアルに移動します。噴射量が25μL、流量が7.5μL/minの場合は、無駄な時間を7.8分に設定します。
- 注入弁を負荷位置に設定して、25 μLのHPLC級水を注入口に注入して、注入ループを5回リンスアウトします。
- システムの残りの部分をフラッシュするために手動で「射出位置」に注入弁を回します。制御ソフトウェアの [プロセス(アウト)]を 選択して、水の流れを開始します。流れている間、製品コレクタの下にある 「上」 を選択して、キャピラリーの終わりを見ることができるように、製品コレクターのキャピラリーリフトを上げます。液滴が形成されるまで毛細血管を通って水を流す。
メモ:液滴が形成されない場合は、インジェクタバルブに漏れが発生していないか確認してください。漏れがある場合は、ナットを緩め、キャピラリーを取り付け直し、締め直します。
4. 実際 の▪OH収量を決定し、バッファーからのラジカル清掃効果をテストします。
- コントロールソフトウェアで、[ プロセス (Out)]を選択してフローを開始します。設定タブで、フラッシュ電圧を0 Vに設定します。[データ量]セクションの[ 手動制御 ]タブで、[ データの開始 + オートゼロ]を選択します。
- 製品バイアル(H)と廃棄物バイアル(6)の位置を選択します。
- [Ready]を選択し、手動でインジェクションバルブを負荷位置まで下げ、100 mM H 2 O 2の25 μLを1mMH2O2で注入ポートに注入します。注入したら、注入弁を注入位置まで手動で回す。
注: これは自動的にシステムが流れを開始し、プラズマ源をオンにして、線量測定データを取得するようにトリガします。 - 設定タブに移動し、フラッシュ電圧を変更して、電圧を上げます。500 V、750 V、1000 V、および 1250 V を使用してステップ 4.2 および 4.3 を繰り返します。
- 計算タブを選択して、各サンプルの平均吸光度を決定します。まず、[ 選択] をクリックし、ピーク吸光度の始点と終点を手動で選択します。使用可能なスペースに、サンプルの説明を書きます。取得したすべてのデータに対して繰り返します。
注意:気泡が形成され、ドーシメータの読み取り値が急上昇する可能性があります。平均吸光度を決定するデータを選択する場合は、気泡のある領域を省略します。 - Excelでデータをコピーして貼り付けて、各電圧のアデニン吸光度の平均変化を計算し、したがって、OH収量 ▪有効を決定する。
- 各条件の有効な ▪OH 歩留まりを正規化するために複数のサンプル条件(異なるバッファ/添加剤)が使用されている場合は、手順 4.1 ~ 4.6 を繰り返します。
5. 高次構造の変化を検出するタンパク質の改変。
- 4 mM アデニンと 20 μM のタンパク質を 1 対 1 の比率で混合し、2 mM アデニンと 10 μM タンパク質を含む溶液を作ります。
- 0.3 mg/mLカタラーゼを使用して余分な過酸化水素を分解し、残りのラジカルをクエンチするために35 mMメチオニンアミドを作ります。アリコート25 μLのクエンチ溶液を200 μLマイクロチューブに入れ、同量のクエンチとラベル付けされた製品が混合されるようにします。
- H2O2~200mMを薄め、氷の上に置いてください。
- 設定タブのコントロールソフトウェアで、フラッシュ電圧を0Vで開始して、バックグラウンドでの酸化を決定します。
- 手動制御タブで、[ データの開始 + オートゼロ] を選択し、[ プロセス (アウト)]、次に [準備完了] を選択し、最後に射出バルブを負荷位置まで下にします。
- 製品のコレクトカルーセルの位置 1 にクエンチ溶液を配置します。システムコントロールソフトウェアで、製品バイアルを1に変更します。
- 注射の直前に、12.5 μLのアデニンとタンパク質混合物を12.5 μLのH2O2と混合して、1 mM アデニン、5 μMタンパク質、および100 mM H 2 O2の最終濃度を作ります。ピペットを上下に軽く混ぜ、素早くスピンダウンし、混合後10秒以内に注入ポートを使用して25μLを注入します。
- 注入弁を注入位置に切り替え、サンプルが処理されている間待ちます。
- 500 V、750 V、1000 V、および1250 Vで取得を繰り返し、各電圧測定を三重で行います。
- ステップ4.5で説明したように平均吸光度を計算する。すべてのデータをコピーして Excel に貼り付けます。
6. システムをシャットダウンする
- すべてのサンプルを採取した後、注入バルブを負荷位置に下げて注射器ポートとサンプルループを洗い流し、25 μLのHPLC水を5回注入します。
- 注入弁を注入位置まで回し、残りのシステムをHPLC水で洗い流します。
- フローを停止し、システム制御ソフトウェアを閉じ、製品コレクタ、ドーシメータモジュール、光起調モジュール、最後に流体モジュールから始まるモジュールをオフにします。
7. 試料調製および液体クロマトグラフィー質量分析
- 50 mMトリスと1 mM CaCl 2の存在下で、サンプルを80°Cで20分間インキュベートしてタンパク質を変性させる。室温にサンプルを冷却し、タンパク質に1:20トリプシンを加えます。サンプル混合で37°Cで一晩タンパク質を消化する。翌朝、サンプルを95°Cに10分間加熱してトリプシン消化を終了する。
- UPLCシステムに接続された高解像度LC-MS/MSシステムを使用してペプチドを検出します。
- サンプルをトラップカラム(300 μm ID X 5 mm 100 Å孔サイズ、5 μm粒径)にロードし、5.0 μL/mLで3分間、2%溶媒B(アセトニトリルと0.1%ギ酸)を含む水で洗浄します。
- C18ナノカラム(0.75mm x 150mm、2μm粒径、100Å孔径)のペプチドを、溶媒A(0.1%のギ酸を含む水)と溶媒Bの間の勾配で300 nL/minの流量で分離します。ペプチド溶出の勾配は、22分間で2〜35%Bに線形増加し、5分間で95%Bに上昇し、3分間保持して3分間保持し、3分間にわたって2%Bに戻し、9分間保持してカラムを再平衡化します。
- 正のイオンモードでデータを取得します。噴霧電圧を2400Vに設定し、イオン転写管の温度を300°Cに設定します。
- 250~2000 m/zの完全MSスキャンを取得し、その後、最も豊富なペプチドイオンの上位8個に対して8回のデータ依存MS/MSスキャンを行います。35%正規化されたエネルギーで衝突誘発解離を使用して、ペプチドをフラグメント化します。
- タンパク質配列と関連するタンパク質分解酵素を含む必要なFASTAファイルに対して利用可能なタンパク質分析ソフトウェアを使用してMS/MS分析から検出されたすべての未修飾ペプチドを同定します。
- HRPFデータ処理ソフトウェアを使用して、修飾ペプチドを検索および定量化します。同定されたペプチドごとに酸化の程度は、修飾ペプチドの合計クロマトグラフィーピーク面積を式3を用いて改変されたペプチドの総クロマトグラフィーピーク面積で割ることによって計算される。
P = [I( 一酸化) X 1 + I (二重酸化 ) X 2 + I(三重酸化 ) X 3 + ../I ( 1酸化性 ) + I (二重酸化) + I( 三重酸化 ) + I(三酸化型 )
ここでPはペプチド分子あたりの平均酸化事象数を示し、Iは非酸化ペプチド(Iunoxキシ化)とn酸化事象を有するペプチドのピーク面積を表す。
8. 差動分析の場合は、2番目の条件でステップ 5 から 7 を繰り返します。
注:再現性を確認するために、各条件の技術的な三重化に加えて、2つの生物学的複製が推奨されます。
結果
リアルタイムのドサイメトリーと組み合わせた高圧血漿源により、高次タンパク質構造の変化をより正確に観察するために、▪OH収量をより適切に制御できます。アデニンの添加は有効なリアルタイムラジカルのドシメータを可能にする。酸化の際、アデニンは265nmでUV吸光度を失う(図2A)。アデニン吸光度の変化は、HRPFに利用可能なラジカルの濃度に直接関係し...
ディスカッション
HRPF実験中にタンパク質の適切な標識を確実に行うためには、いくつかの重要なステップがあります。まず、適切な流量とソースフラッシュレートが、試料の各ボーラスが一度照射されるのを確実にするために選択される。これにより、タンパク質が新たに形成された ▪OHの単一のボーラスにさらされることが保証されます。タンパク質が酸化されると、より高次のタンパク質構造を?...
開示事項
E.E.C.、J.S.S.、S.R.W.は、高次タンパク質構造解析のための技術の製品化を目指す初期段階のGenNext Technologies, Inc.に対する大きな財政的関心を開示しています。提供された代表的なデータは、金銭的な利益相反を持たないS.K..M.によってレビューされています。
謝辞
この研究は、国立一般医学研究所(R43GM125420およびR44GM125420)によって資金提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | any brand is sufficient |
| 50 µL SGE Gastight Syringes | Fisher Scientific | SG-00723 | |
| Acclaim PepMap 100 C18 nanocolumn (0.75 mm X 150 mm, 2 µm) | Thermo Scientific | ||
| Acetonitrile with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS120-500 | |
| Apomyoglobin | Sigma-Aldrich | ||
| Catalase | Sigma-Aldrich | C9322 | |
| Centrifuge | Eppendorf | 022625501 | |
| Delicate Task Wipers | Fisher Scientific | 06-666A | |
| Hydrogen Peroxide | Fisher Scientific | H325-100 | any 30% hydrogen peroxide is sufficient |
| Methionine amide | Chem-Impex | 03109 | |
| Microcentrifuge | Thermo Scientific | 75002436 | |
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | other high resolution instruments (e.g. Q exactive Orbitrap or Orbitrap Fusion) can be used |
| Pierce Trypsin Protease, MS Grade | Thermo Scientific | 90058 | |
| Polymicro Cleaving Stone, 1" x 1" x 1/32” | Molex | 1068680064 | any capillary tubing cutter is sufficient |
| UPLC | Thermo Scientific | ||
| Water with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS118-500 | |
| Water, LC/MS Grade | Fisher Scientific | W6-4 |
参考文献
- Nagarkar, R. P., Murphy, B. M., Yu, X., Manning, M. C., Al-Azzam, W. A. Characterization of protein higher order structure using vibrational circular dichroism spectroscopy. Current Pharmaceutical Biotechnology. 14 (2), 199-208 (2013).
- Giezen, T. J., Schneider, C. K. Safety assessment of biosimilars in Europe: a regulatory perspective. Generics and Biosimilars Initiative Journal. , 1-8 (2014).
- Giezen, T. J., Mantel-Teeuwisse, A. K., Strauss, S. Safety-related regulatory actions for biologicals approved in the United States and the Europena Union. Journal of the American Medical Society. 300 (16), 1887-1896 (2008).
- Gabrielson, J. P., Weiss, W. F. Technical decision-making with higher order structure data: starting a new dialogue. Journal of Pharmaceutical Sciences. 104 (1), 1240-1245 (2015).
- Brenowitz, M., Erie, D. A., Chance, M. R. Catching RNA polymerase in the act of binding: intermediates in transcription illuminated by synchrotron footprinting. Proceedings of the National Academy of Sciences U S A. 102 (13), 4659-4660 (2005).
- Guan, J. Q., Takamoto, K., Almo, S. C., Reisler, E., Chance, M. R. Structure and dynamics of the actin filament. Biochemistry. 44 (9), 3166-3175 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photochemical oxidataion to locate heme binding and conformataional changes in myoglobin. International Journal of Mass Spectrometry. 259 (2007), 124-129 (2007).
- Li, K. S., Shi, L., Gross, M. L. Mass Spectrometry-Based Fast Photochemical Oxidation of Proteins (FPOP) for Higher Order Structure Characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Watson, C., Sharp, J. S. Conformational Analysis of Therapeutic Proteins by Hydroxyl Radical Protein Footprinting. American Association of Pharmaceutical Scientists Journal. 14 (2), 206-217 (2012).
- Deperalta, G., et al. Structural analysis of a therapeutic monoclonal antibody dimer by hydroxyl radical footprinting. mAbs. 5 (1), 86-101 (2013).
- Jones, L. M., et al. Complementary MS methods assist conformational characterization of antibodies with altered S-S bonding networks. Journal of American Society of Mass Spectrometry. 24 (6), 835-845 (2013).
- Storek, K. M., et al. Monoclonal antibody targeting the β-barrel assembly machine of Escherichia coli is bactericidal. Proceedings of the National Academy of Sciences. , (2018).
- Vij, R., et al. A targeted boost-and-sort immunization strategy using Escherichia coli BamA identifies rare growth inhibitory antibodies. Scientific Reports. 8 (1), 7136 (2018).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Lu, Y., et al. Fast Photochemical Oxidation of Proteins Maps the Topology of Intrinsic Membrane Proteins: Light-Harvesting Complex 2 in a Nanodisc. Analytical Chemistry. 88 (17), 8827-8834 (2016).
- Marty, M., Zhang, H., Cui, W., Gross, M., Sligar, S. Interpretation and Deconvolution of Nanodisc Native Mass Spectra. Journal of American Society of Mass Spectrometry. 25, (2013).
- Johnson, D. T., Di Stefano, L. H., Jones, L. M. Fast photochemical oxidation of proteins(FPOP): A powerful mass spectrometry based structural proteomics tool. Journal of Biological Chemistry. , (2019).
- Chea, E. E., Jones, L. M. Analyzing the structure of macromolecules in their native cellular environment using hydroxyl radical footprinting. Analyst. 143 (4), 798-807 (2018).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical chemistry. 90 (12), 7721-7729 (2018).
- Linde. . Linde Specialty Gases of North America. , (2009).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77 (18), 5814-5822 (2005).
- Niu, B., Zhang, H., Giblin, D., Rempel, D. L., Gross, M. L. Dosimetry determines the initial OH radical concentration in fast photochemical oxidation of proteins (FPOP). Journal of American Society of Mass Spectrometry. 26 (5), 843-846 (2015).
- Misra, S. K., Orlando, R., Weinberger, S. R., Sharp, J. S. Compensated Hydroxyl Radical Protein Footprinting Measures Buffer and Excipient Effects on Conformation and Aggregation in an Adalimumab Biosimilar. American Association of Pharmaceutical Scientists Journal. 21 (5), 87 (2019).
- Olson, L. J., Misra, S. K., Ishihara, M., Battaile, K. P., Grant, O. C., Sood, A., Woods, R. J., Kim, J. P., Tiemeyer, M., Ren, G., Sharp, J. S., Dahms, N. M. Allosteric regulation of lysosomal enzyme recognition by the cation-independent mannose 6-phosphate receptor. Communications Biology. 3 (1), 498 (2020).
- Sharp, J. S., Misra, S. K., Persoff, J. J., Egan, R. W., Weinberger, S. R. Real Time Normalization of Fast Photochemical Oxidation of Proteins Experiments by Inline Adenine Radical Dosimetry. Analytical Chemistry. 90 (21), 12625-12630 (2018).
- Misra, S. K., Sharp, J. S. Enabling Real-Time Compensation in Fast Photochemical Oxidations of Proteins for the Determination of Protein Topography Changes. Journal of Visualized Experiments. 163, (2020).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of American Society of Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemical Reviews. 120 (10), 4355 (2020).
- Jones, L. M., Sperry, B. J., Carroll, A. J., Gross, M. L. Fast photochemical oxidation of proteins for epitope mapping. Analytical chemistry. 83 (20), 7657-7661 (2011).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical chemistry. 89 (4), 2250-2258 (2017).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Kiselar, J. G., Janmey, P. A., Almo, S. C., Chance, M. R. Structural analysis of gelsolin using synchrotron protein footprinting. Molecular and Cellular Proteomics. 2 (10), 1120-1132 (2003).
- Chea, E. E., Deredge, D. J., Jones, L. M. Insights on the Conformational Ensemble of Cyt C Reveal a Compact State during Peroxidase Activity. Biophysical Journal. 118 (1), 128-137 (2020).
- Poor, T. A., et al. Probing the paramyxovirus fusion (F) protein-refolding event from pre- to postfusion by oxidative footprinting. Proceedings of the National Academy of Sciences U S A. 111 (25), 2596-2605 (2014).
- Roush, A. E., Riaz, M., Misra, S. K., Weinberger, S. R., Sharp, J. S. Intrinsic Buffer Hydroxyl Radical Dosimetry Using Tris(hydroxymethyl)aminomethane. Journal of American Society of Mass Spectrometry. 31 (2), 169-172 (2020).
- Everett, E. A., Falick, A. M., Reich, N. O. Identification of a critical cysteine in EcoRI DNA methyltransferase by mass spectrometry. Journal of Biological Chemistry. 265 (29), 17713-17719 (1990).
- Sanderson, R. J., Mosbaugh, D. W. Identification of specific carboxyl groups on uracil-DNA glycosylase inhibitor protein that are required for activity. Journal of Biological Chemistry. 271 (46), 29170-29181 (1996).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved