Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Empreinte de protéines de radicaux hydroxyles sans laser pour effectuer une analyse structurelle d’ordre supérieur des protéines
Dans cet article
Résumé
Ce protocole présente une méthode pour utiliser la dosimétrie radicale en ligne et une source de lumière plasma pour effectuer l’empreinte des protéines d’oxydation flash. Cette méthode remplace le laser UV dangereux pour simplifier et améliorer la reproductibilité de l’oxydation photochimique rapide des études de protéines.
Résumé
L’empreinte des protéines du radical hydroxyle (HRPF) est une technique d’analyse structurelle d’ordre supérieur émergente et prometteuse qui fournit des informations sur les changements dans la structure des protéines, les interactions protéine-protéine ou les interactions protéine-ligand. HRPF utilise des radicaux hydroxyles(▪OH) pour étiqueter de manière irréversible la surface accessible au solvant d’une protéine. La complexité inhérente, le coût et la nature dangereuse de l’exécution du FPRH ont considérablement limité l’adoption à grande échelle dans la biopharmaceutique. Ces facteurs comprennent : 1) l’utilisation de lasers compliqués, dangereux et coûteux qui exigent des précautions de sécurité importantes; et 2) l’irréprochabilité du HRPF causée par le nettoyage de fond de ▪l’OH qui limitent les études comparatives. Cette publication fournit un protocole pour le fonctionnement d’un système HRPF sans laser. Ce système HRPF sans laser utilise une technologie d’oxydation flash de source de lumière plasma à haute énergie et à haute pression avec dosimétrie radicale en ligne. La source de lumière plasmatique est plus sûre, plus facile à utiliser et plus efficace pour générer des radicaux hydroxyles que les systèmes HRPF à base de laser, et le dosimètre radicalaire en ligne augmente la reproductibilité des études. Combiné, le système HRPF sans laser résout et surmonte les lacunes et les limites mentionnées des techniques laser.
Introduction
La conformation des protéines et la structure d’ordre supérieur (HOS) associée sont les principaux déterminants de la bonne fonction biologique et du comportement aberrant1. Il en va de même pour les produits biopharmaceutiques, dont la structure et l’activité fonctionnelle dépendent de divers aspects de leur production et de leur environnement. Les changements biopharmaceutiques dans hos ont été liés aux réactions défavorables de drogue (ADR) attribuées à la pharmacologie indésirable et à la réponse immunologique patient2,3. L’apparition des ADR a alerté l’industrie biopharmaceutique sur le rôle essentiel que joue le HOS protéique dans la sécurité et l’efficacité des produits biothérapeutiques, et ils ont établi le besoin d’analyses HOS nouvelles et améliorées4.
L’empreinte des protéines radicales hydroxyles (HRPF) est une technique prometteuse pour suivre l’évolution de la protéine HOS. Hrpf implique le marquage irréversible de l’extérieur d’une protéine avec ▪OH suivi d’une analyse par spectrométrie de masse (MS) pour identifier la surface accessible au solvant de la protéine5,6,7. HRPF a été utilisé avec succès pour détecter des défauts dans la protéine HOS et sa fonction8,9,caractériser le HOS d’anticorps monoclonaux (mAb)10,11,12,13,déterminer la liaison Kd d’un ligand14,et bien plusencore 15,16,17,18,19. Une méthode courante pour générer l’OH ▪pour HRPF est l’oxydation photochimique rapide des protéines (FPOP), qui utilise des lasers UV rapides à haute énergie pour produire de l'▪OH à partir de la photolyse deH2O2. Pour la plupart, FPOP utilise des lasers excimer coûteux utilisant des gaz dangereux (KrF) qui exigent des garanties substantielles pour éviter les blessures respiratoires et oculaires20. Pour éviter les risques d’inhalation, d’autres ont utilisé des lasers grenat en aluminium à yttrium en néodyme quadruple de fréquence (Nd: YAG)21, qui éliminent l’utilisation de gaz toxiques mais sont toujours coûteux, nécessitent une expertise opérationnelle importante et exigent des contrôles de lumière parasite étendus pour protéger les utilisateurs contre les blessures oculaires.
Bien qu’il soit possible d’obtenir de nombreux renseignements à l’aide du HRPF, l’adoption à grande échelle dans le secteur biopharmaceutique n’a pas été respectée. Deux obstacles à l’adoption limitée du HRPF comprennent: 1) l’utilisation de lasers dangereux et coûteux qui exigent des précautions de sécurité substantielles20; et 2) l’irréprochabilité du HRPF causée par le nettoyage de fond des ▪OH qui limitent les études comparatives22. Pour supplanter l’utilisation du laser, une unité de photolyse flash plasma à haute vitesse et à haute énergie a été développée pour effectuer en toute sécurité le FPOP d’une manière facile. Pour améliorer l’irréprochabilité des expériences HRPF, une dosimétrie radicale en temps réel est mise en œuvre.
La pratique du HRPF a été limitée par l’irréprochabilité attribuée au nettoyage de fond de ▪OH22. Bien que les ▪OH soient d’excellentes sondes de la topographie des protéines, elles réagissent également avec de nombreux constituants présents dans les préparations, ce qui rend nécessaire de mesurer la concentration efficace de radical disponible pour oxyder une protéine cible. Les variations dans la préparation tampon, la concentration de peroxyde d’hydrogène, les propriétés de ligand, ou la photolyse peuvent avoir comme conséquence des différences d’oxydation entre le groupe témoin et le groupe expérimental qui créent l’ambiguïté dans des études différentielles hos. L’ajout de la dosimétrie radicale en temps réel permet l’ajustement de l’effet ▪charge OH et augmente donc la confiance et la reproductibilité lors d’une expérience HRPF. L’utilisation de la dosimétrie radicale dans le FPOP a été décrite ailleurs23,24,25,et est discutée plus en détail dans une publication récente26. Ici, nous décrivons l’utilisation d’un nouveau système de photolyse flash et dosimétrie en temps réel pour étiqueter l’apo-myoglobine équine (aMb), en comparant les niveaux d’oxydation peptidique dans une expérience FPOP à celle obtenue lors de l’utilisation d’un laser excimer.
Protocole
1. Installation du tube capillaire
- À l’aide d’une pierre clivante de silice, cliver 250 μm diamètre intérieur (ID) capillaire de silice à 27 pouces. Vérifiez les extrémités capillaires pour une coupe propre et droite.
- Créez deux fenêtres d’environ 15 mm de longueur en brûlant le revêtement en polyimide. À partir de l'«extrémité inférieure », la première fenêtre de photolyse est éloignée de 90 mm de l'«extrémité inférieure » et la deuxième fenêtre de dosimètre à 225 mm de l'«extrémité inférieure ».
REMARQUE: Une fois que le revêtement est brûlé, le capillaire est très fragile. - Dévissez l’écrou et la virole au port 5 et insérez l'«extrémité inférieure » du capillaire juste au-delà de l’extrémité conique de la virole(figure 1A).
- Sur le module de photolyse, retirez le capuchon de la cellule de photolyse en le tirant directement, puis retirez le masque métallique monté magnétiquement qui maintiendra le capillaire en place.
ATTENTION: À l’intérieur du capuchon de la cellule de photolyse se trouve un miroir incurvé, ne laissez rien toucher le miroir. - Sur le module du dosimètre, ouvrez la cellule du dosimètre en appuyant sur la languette à gauche et balancez la cellule du dosimètre ouverte vers la droite. Retirez les clips montés magnétiquement qui maintiendront le capillaire en place.
- Placez le capillaire dans le canal rainuré à la base de la cellule de photolyse. Centrez la fenêtre capillaire avec la fenêtre de la cellule de photolyse. Tout en maintenant le capillaire en position d’une main, ajoutez le masque magnétique pour maintenir le capillaire en place. Replacez le bouchon de photolyse en position.
- Placez le capillaire dans le canal rainuré à la base de la cellule du dosimètre. Centrez la deuxième fenêtre capillaire sur la petite ouverture au centre de la base de la cellule du dosimètre. Tout en maintenant le capillaire en position d’une main, placez les deux clips magnétiques en position pour maintenir le capillaire en place. Fermez la cellule du dosimètre jusqu’à ce qu’elle clique sur fermé.
- Insérez le capillaire à travers l’écrou moleté au sommet de l’ascenseur capillaire du collecteur du produit(Figure 1B). Étendre le capillaire juste au-dessus du fond du flacon.
REMARQUE: Il est important que le capillaire atteigne le fond du flacon pour s’assurer que le capillaire est immergé dans la solution d’extinction lors de l’exécution d’une expérience.
2. Installation d’une boucle d’injection
- Utilisez un tube en téflon d’un diamètre extérieur de 1/16 po et d’un diamètre intérieur de 0,015 po (381 μm). Suivez l’équation 1 pour calculer la longueur des tubes nécessaires pour le volume désiré à l’aide de l’équation 1.
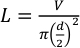
Où L est la longueur du tube en millimètres, V est le volume désiré en microlitres et d est le diamètre intérieur du tube en millimètres. Pour un volume d’injection de 25 μL et un diamètre intérieur de 381 μm, la longueur du tube doit être d’environ 219,3 mm.
NOTA : Pour les volumes inférieurs à 20 μL, utiliser des tubes en PTFE d’un diamètre intérieur de 0,01 ». - Coupez le tube en téflon à la longueur nécessaire à l’aide d’un coupe-tube. Vérifiez les extrémités du tube pour une coupe propre et droite.
- Insérez une extrémité de la nouvelle boucle d’injection à travers l’un des écrous et placez une nouvelle virole sur l’extrémité du tube. Maintenez la virole et l’écrou en place et insérez le tube dans l’orifice 3 de la vanne d’injection jusqu’à ce qu’il fine dans la vanne. Tenez fermement le tube et serrez le doigt de l’écrou serré. À l’aide d’une clé, serrez l’écrou 1/4 tourner plus loin. Retirez et inspectez l’assembly.
REMARQUE: Si la virole n’est pas fixée en position, réinstallez et serrez 1/8 tourner plus loin. Répétez l’opération avec l’autre extrémité de la boucle. - Une fois que les deux extrémités ont un écrou et une virole fixe, visser lâchement une extrémité au port 3 et l’autre extrémité au port 6(Figure 1C). Une fois en position, serrez les deux côtés pour serrer les doigts serrés, puis 1/4 tournez devant le doigt serré avec une clé.
3. Initialiser le système de photolyse
- Allumez les modules de photolyse dans l’ordre suivant : (1) Module fluidique (2) Module photolyse (3) Module de dosimétrie (4) Collecteur de produits, et enfin (5) l’ordinateur système et lancer le logiciel de contrôle
REMARQUE : Laissez le module de dosimètre au moins une demi-heure se réchauffer après un démarrage à froid. - Tube complètement immergé de la position « Valve » sur la pompe de seringue(Figure 1D)dans 10 mL de tampon. Placer le tube de la position « Déchets »(figure 1D)et le tube de l’orifice 2 sur le port de seringue(figure 1C)dans un récipient vide pour collecter les déchets.
REMARQUE: Utilisez le tampon dans lequel votre protéine sera suspendue. Un tampon recommandé est 10 mM NaPO4. - Sur le carrousel Product Collector, placer des tubes microcentrifuges de 1,5 mL à la position marquée H et 6.
- Si vous utilisez un capillaire ID de 250 μm, réglez le débit de charge à 500 μL/min, le débit de traitement à 7,5 μL/min et le débit de déchets à 500 μL/min.
- Calculez le délai d’éclair, le temps de produit et le temps perdu pour utiliser le mode semi-automatique à l’aide de l’équation 2.
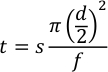
Où t est le temps en minutes, s est la distance capillaire en millimètres, d est le diamètre intérieur du capillaire en millimètres et f est le débit en μL/min.- Pour le délai d’éclair, la distance est d’environ 90 mm entre « l’extrémité inférieure » du capillaire et la première fenêtre. En utilisant un capillaire ID de 250 μm et un débit de traitement de 7,5 μL/min, l’échantillon arrivera à la fenêtre de photolyse en environ 35 secondes. Permettez à deux flashs à 1 Hz de se produire avant l’arrivée des échantillons, donc réglez le délai de flash à 33 secondes.
- Pour le temps du produit, entrez le temps que prendra la solution injectée pour s’écouler de la vanne d’injection à l’extrémité du capillaire. Pour un capillaire ID de 27 po de long de 250 μm, réglez la durée du produit sur 4,5 minutes.
- Pour la perte de temps, entrez le temps que le volume total de solution injecté prendra pour être collecté. À ce stade, le collecteur de produits passera de la position du produit au flacon de déchets. Pour un volume d’injection de 25 μL et un débit de 7,5 μL/min, réglez le temps perdu à 7,8 minutes.
- Rincez la boucle d’injection cinq fois en injectant 25 μL d’eau de qualité HPLC dans l’orifice d’injection avec la vanne d’injection réglée sur la position de charge.
- Tournez manuellement la vanne d’injection jusqu’à la « position d’injection » pour rincer le reste du système. Sélectionnez Traiter (Out) sur le logiciel de contrôle pour commencer à couler de l’eau. Pendant qu’il coule, soulevez l’ascenseur capillaire du collecteur de produit en sélectionnant Vers le haut sous le collecteur de produits afin que vous puissiez voir la fin du capillaire. Faire circuler l’eau à travers le capillaire jusqu’à ce qu’une gouttelette se forme.
REMARQUE : Si une gouttelette ne se forme pas, vérifiez que la vanne de l’injecteur ne contient pas de fuites. En cas de fuite, desserrez l’écrou, réinstallez le capillaire et retentiez.
4. Déterminer le rendement réel en ▪OH pour tester les effets de balayage radicalaire du tampon.
- Dans le logiciel de contrôle, démarrez le flux en sélectionnant Processus (sortie). Dans l’onglet Paramètres, définissez la tension de flash sur 0 V. Sous l’onglet Contrôle manuel dans la section des données du dosimètre, sélectionnez Démarrer les données + AutoZero.
- Sélectionnez la position du flacon de produit (H) et du flacon de déchets (6).
- Sélectionnez Prêt, tournez manuellement la vanne d’injection vers le bas à la position de charge, et injectez 25 μL d’adénine 1 mM avec 100 mMH2O2 dans le port d’injection. Une fois injecté, tournez manuellement la valve d’injection jusqu’à la position d’injection.
Remarque : Cela déclenche automatiquement le système pour commencer le flux, allumer la source de plasma et acquérir les données de dosimétrie. - Augmentez la tension en accédant à l’onglet Paramètres et en modifiant la tension flash. Répétez les étapes 4.2 et 4.3 en utilisant 500 V, 750 V, 1000 V et 1250 V. Effectuez la lecture de l’absorbance de l’adénine à chaque tension en trois exemplaires.
- Sélectionnez l’onglet Calculs pour déterminer l’absorbance moyenne de chaque échantillon. Tout d’abord, cliquez sur Sélectionner, puis sélectionnez manuellement le début et la fin de l’absorbance maximale. Dans l’espace disponible, écrivez une description de l’exemple. Répétez l’opération pour toutes les données acquises.
REMARQUE: Des bulles peuvent se former provoquant un pic dans la lecture du dosimètre. Lorsque vous sélectionnez des données pour déterminer l’absorbance moyenne, omettez les zones avec des bulles. - Copiez et collez des données dans Excel pour calculer la variation moyenne de l’absorbance de l’adénine pour chaque tension, déterminant ainsi le rendement effectif ▪OH.
- Répétez les étapes 4.1 à 4.6 si plusieurs conditions d’échantillonnage (différents tampons/additifs) sont utilisées pour normaliser le rendement efficace en ▪en OH pour chaque condition.
5. Modification de la protéine pour détecter les changements dans la structure d’ordre supérieur.
- Mélanger 4 mM d’adénine avec 20 μM de protéines dans un rapport de un à un pour obtenir une solution contenant 2 mM d’adénine et 10 μM de protéines.
- Faire la solution de trempe en utilisant 0,3 mg/mL de catalase pour décomposer l’excès de peroxyde d’hydrogène et 35 mM d’méthionine amide pour tremper les radicaux restants. Aliquote 25 μL de solution de trempe dans un microtube de 200 μL de sorte qu’un volume égal de trempe et le produit étiqueté soient mélangés.
- DiluerH2O2à 200 mM et garder sur la glace.
- Dans l’onglet Paramètres du logiciel de contrôle, démarrez la tension d’éclair à 0 V pour déterminer toute oxydation en arrière-plan.
- Dans l’onglet de contrôle manuel, sélectionnez Démarrer les données + AutoZero, suivi de Traiter (sortie),puis Prêt, et enfin baisser la vanne d’injection jusqu’à la position de charge.
- Placez une solution de trempe en position 1 sur le carrousel de collecte du produit. Sur le logiciel de contrôle du système, remplacez le flacon du produit par 1.
- Immédiatement avant l’injection, mélanger 12,5 μL du mélange d’adénine et de protéines avec 12,5 μL deH2O2pour obtenir une concentration finale de 1 mM d’adénine, 5 μM de protéines et 100 mMH2O2. Pipeter doucement de haut en bas pour mélanger, tourner rapidement vers le bas et injecter 25 μL à l’aide de l’orifice d’injection dans les 10 secondes suivant le mélange.
- Passez la vanne d’injection à la position d’injection et attendez pendant le traitement de l’échantillon.
- Répétez l’acquisition avec 500 V, 750 V, 1000 V et 1250 V. Effectuez chaque mesure de tension en trois exemplaires.
- Calculer l’absorbance moyenne telle que décrite à l’étape 4.5. Copiez et collez toutes les données dans Excel.
6. Arrêtez le système
- Une fois que tous les échantillons ont été prélevés, rincer l’orifice de la seringue et la boucle de l’échantillon en fixant la valve d’injection à la position de charge et injecter 25 μL d’eau HPLC cinq fois.
- Tournez la vanne d’injection jusqu’à la position d’injection pour rincer le reste du système avec de l’eau HPLC.
- Arrêtez le flux, fermez le logiciel de contrôle du système, et éteignez les modules en commençant par le collecteur de produit, le module de dosimètre, le module de photolyse, puis enfin le module fluidique.
7. Préparation de l’échantillon et chromatographie liquide-spectrométrie de masse
- Dénaturer la protéine en incubant des échantillons à 80 °C pendant 20 min en présence de Tris 50 mM et de CaCl2 1mM. Refroidir les échantillons à température ambiante et ajouter 1:20 trypsine aux protéines. Digérer la protéine pendant la nuit à 37 °C avec mélange d’échantillons. Le lendemain matin, mettez fin à la digestion à la trypsine en chauffant les échantillons à 95 °C pendant 10 minutes.
- Détecter les peptides à l’aide d’un système LC-MS/MS haute résolution connecté à un système UPLC.
- Chargez d’abord l’échantillon sur la colonne du piège (300 μm ID X 5 mm 100 Å taille des pores, granulométrie 5 μm) et lavez à 5,0 μL/mL pendant 3 min avec de l’eau contenant 2 % de solvant B (acétonitrile et 0,1 % d’acide formique).
- Séparer les peptides sur une nanocolonne C18 (0,75 mm x 150 mm, granulométrie de 2 μm, taille des pores de 100 Å) à un débit de 300 nL/min avec un gradient entre le solvant A (eau contenant 0,1 % d’acide formique) et le solvant B. Le gradient pour l’élution peptidique consiste en une augmentation linéaire de 2 à 35% B sur 22 min, rampé à 95% B sur 5 min et maintenu pendant 3 min pour laver la colonne, puis retourné à 2% B sur 3 min et maintenu pendant 9 min pour rééquilibrer la colonne.
- Acquérir les données en mode ionique positif. Réglez la tension de pulvérisation à 2400 V et la température du tube de transfert d’ions à 300 °C.
- Acquérir les scans complets de MS de 250-2000 m/z suivis de huit balayages ms/ms dépendants des données suivants sur les huit ions peptidiques les plus abondants. Utilisez la dissociation induite par collision à 35% d’énergie normalisée pour fragmenter les peptides.
- Identifier tous les peptides non modifiés détectés lors de l’analyse MS/MS à l’aide d’un logiciel d’analyse des protéines disponible par rapport au fichier FASTA nécessaire contenant la séquence protéolytique et l’enzyme protéolytique pertinente.
- Rechercher et quantifier des peptides modifiés à l’aide d’un logiciel de traitement de données HRPF. L’étendue de l’oxydation pour chaque peptide identifié est calculée en divisant la surface de crête chromatographique additionnée d’un peptide modifié par la surface de crête chromatographique totale de ce peptide modifié et non modifié à l’aide de l’équation 3.
P = [I(oxydé proprement) X 1 + I (doublement oxydé) X 2 + I(triplement oxydé) X 3 + .../[Non oxydé + I(oxydé proprement) + I(doublement oxydé) + I(triplement oxydé) ...]
où P désigne le nombre moyen d’événements d’oxydation par molécule peptidique, et I représente la zone de crête du peptide non oxydé(non oxydé)et du peptide avec n événements d’oxydation.
8. Pour une étude différentielle, répétez les étapes 5 à 7 sur la deuxième condition.
NOTA : Pour confirmer la reproductibilité, deux répétitions biologiques en plus des triples techniques pour chaque condition sont recommandées.
Résultats
La source de plasma à haute pression couplée à la dosimétrie en temps réel permet un meilleur contrôle de ▪rendement en OH pour observer plus précisément les changements dans la structure des protéines d’ordre supérieur. L’ajout d’adénine permet un dosimètre radicalaire efficace en temps réel. Lors de l’oxydation, l’adénine perd son absorbance UV à 265 nm(figure 2A). Le changement dans l’absorbance de l’adénine est directement lié à la concentrati...
Discussion
Il y a plusieurs étapes critiques pour assurer le bon étiquetage des protéines pendant toute expérience HRPF. Tout d’abord, un débit et un débit d’éclair à la source appropriés sont choisis pour s’assurer que chaque bolus de l’échantillon est irradié une fois. Cela garantit que la protéine est exposée à un seul bolus de ▪OH nouvellement formé. Une fois qu’une protéine est oxydée, la structure protéique d’ordre supérieur peut être modifiée. Pour être sûr que la structure pro...
Déclarations de divulgation
E.E.C., J.S.S. et S.R.W. divulguent une participation financière importante dans GenNext Technologies, Inc., une société en démarrage qui cherche à commercialiser des technologies pour l’analyse de la structure des protéines d’ordre supérieur. Les données représentatives fournies ont été examinées par S.K.M., qui n’a pas de conflit d’intérêts financier.
Remerciements
Ces travaux ont été financés par l’Institut national des sciences médicales générales (R43GM125420 et R44GM125420).
matériels
| Name | Company | Catalog Number | Comments |
| 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | any brand is sufficient |
| 50 µL SGE Gastight Syringes | Fisher Scientific | SG-00723 | |
| Acclaim PepMap 100 C18 nanocolumn (0.75 mm X 150 mm, 2 µm) | Thermo Scientific | ||
| Acetonitrile with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS120-500 | |
| Apomyoglobin | Sigma-Aldrich | ||
| Catalase | Sigma-Aldrich | C9322 | |
| Centrifuge | Eppendorf | 022625501 | |
| Delicate Task Wipers | Fisher Scientific | 06-666A | |
| Hydrogen Peroxide | Fisher Scientific | H325-100 | any 30% hydrogen peroxide is sufficient |
| Methionine amide | Chem-Impex | 03109 | |
| Microcentrifuge | Thermo Scientific | 75002436 | |
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | other high resolution instruments (e.g. Q exactive Orbitrap or Orbitrap Fusion) can be used |
| Pierce Trypsin Protease, MS Grade | Thermo Scientific | 90058 | |
| Polymicro Cleaving Stone, 1" x 1" x 1/32” | Molex | 1068680064 | any capillary tubing cutter is sufficient |
| UPLC | Thermo Scientific | ||
| Water with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS118-500 | |
| Water, LC/MS Grade | Fisher Scientific | W6-4 |
Références
- Nagarkar, R. P., Murphy, B. M., Yu, X., Manning, M. C., Al-Azzam, W. A. Characterization of protein higher order structure using vibrational circular dichroism spectroscopy. Current Pharmaceutical Biotechnology. 14 (2), 199-208 (2013).
- Giezen, T. J., Schneider, C. K. Safety assessment of biosimilars in Europe: a regulatory perspective. Generics and Biosimilars Initiative Journal. , 1-8 (2014).
- Giezen, T. J., Mantel-Teeuwisse, A. K., Strauss, S. Safety-related regulatory actions for biologicals approved in the United States and the Europena Union. Journal of the American Medical Society. 300 (16), 1887-1896 (2008).
- Gabrielson, J. P., Weiss, W. F. Technical decision-making with higher order structure data: starting a new dialogue. Journal of Pharmaceutical Sciences. 104 (1), 1240-1245 (2015).
- Brenowitz, M., Erie, D. A., Chance, M. R. Catching RNA polymerase in the act of binding: intermediates in transcription illuminated by synchrotron footprinting. Proceedings of the National Academy of Sciences U S A. 102 (13), 4659-4660 (2005).
- Guan, J. Q., Takamoto, K., Almo, S. C., Reisler, E., Chance, M. R. Structure and dynamics of the actin filament. Biochemistry. 44 (9), 3166-3175 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photochemical oxidataion to locate heme binding and conformataional changes in myoglobin. International Journal of Mass Spectrometry. 259 (2007), 124-129 (2007).
- Li, K. S., Shi, L., Gross, M. L. Mass Spectrometry-Based Fast Photochemical Oxidation of Proteins (FPOP) for Higher Order Structure Characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Watson, C., Sharp, J. S. Conformational Analysis of Therapeutic Proteins by Hydroxyl Radical Protein Footprinting. American Association of Pharmaceutical Scientists Journal. 14 (2), 206-217 (2012).
- Deperalta, G., et al. Structural analysis of a therapeutic monoclonal antibody dimer by hydroxyl radical footprinting. mAbs. 5 (1), 86-101 (2013).
- Jones, L. M., et al. Complementary MS methods assist conformational characterization of antibodies with altered S-S bonding networks. Journal of American Society of Mass Spectrometry. 24 (6), 835-845 (2013).
- Storek, K. M., et al. Monoclonal antibody targeting the β-barrel assembly machine of Escherichia coli is bactericidal. Proceedings of the National Academy of Sciences. , (2018).
- Vij, R., et al. A targeted boost-and-sort immunization strategy using Escherichia coli BamA identifies rare growth inhibitory antibodies. Scientific Reports. 8 (1), 7136 (2018).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Lu, Y., et al. Fast Photochemical Oxidation of Proteins Maps the Topology of Intrinsic Membrane Proteins: Light-Harvesting Complex 2 in a Nanodisc. Analytical Chemistry. 88 (17), 8827-8834 (2016).
- Marty, M., Zhang, H., Cui, W., Gross, M., Sligar, S. Interpretation and Deconvolution of Nanodisc Native Mass Spectra. Journal of American Society of Mass Spectrometry. 25, (2013).
- Johnson, D. T., Di Stefano, L. H., Jones, L. M. Fast photochemical oxidation of proteins(FPOP): A powerful mass spectrometry based structural proteomics tool. Journal of Biological Chemistry. , (2019).
- Chea, E. E., Jones, L. M. Analyzing the structure of macromolecules in their native cellular environment using hydroxyl radical footprinting. Analyst. 143 (4), 798-807 (2018).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical chemistry. 90 (12), 7721-7729 (2018).
- Linde. . Linde Specialty Gases of North America. , (2009).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77 (18), 5814-5822 (2005).
- Niu, B., Zhang, H., Giblin, D., Rempel, D. L., Gross, M. L. Dosimetry determines the initial OH radical concentration in fast photochemical oxidation of proteins (FPOP). Journal of American Society of Mass Spectrometry. 26 (5), 843-846 (2015).
- Misra, S. K., Orlando, R., Weinberger, S. R., Sharp, J. S. Compensated Hydroxyl Radical Protein Footprinting Measures Buffer and Excipient Effects on Conformation and Aggregation in an Adalimumab Biosimilar. American Association of Pharmaceutical Scientists Journal. 21 (5), 87 (2019).
- Olson, L. J., Misra, S. K., Ishihara, M., Battaile, K. P., Grant, O. C., Sood, A., Woods, R. J., Kim, J. P., Tiemeyer, M., Ren, G., Sharp, J. S., Dahms, N. M. Allosteric regulation of lysosomal enzyme recognition by the cation-independent mannose 6-phosphate receptor. Communications Biology. 3 (1), 498 (2020).
- Sharp, J. S., Misra, S. K., Persoff, J. J., Egan, R. W., Weinberger, S. R. Real Time Normalization of Fast Photochemical Oxidation of Proteins Experiments by Inline Adenine Radical Dosimetry. Analytical Chemistry. 90 (21), 12625-12630 (2018).
- Misra, S. K., Sharp, J. S. Enabling Real-Time Compensation in Fast Photochemical Oxidations of Proteins for the Determination of Protein Topography Changes. Journal of Visualized Experiments. 163, (2020).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of American Society of Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemical Reviews. 120 (10), 4355 (2020).
- Jones, L. M., Sperry, B. J., Carroll, A. J., Gross, M. L. Fast photochemical oxidation of proteins for epitope mapping. Analytical chemistry. 83 (20), 7657-7661 (2011).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical chemistry. 89 (4), 2250-2258 (2017).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Kiselar, J. G., Janmey, P. A., Almo, S. C., Chance, M. R. Structural analysis of gelsolin using synchrotron protein footprinting. Molecular and Cellular Proteomics. 2 (10), 1120-1132 (2003).
- Chea, E. E., Deredge, D. J., Jones, L. M. Insights on the Conformational Ensemble of Cyt C Reveal a Compact State during Peroxidase Activity. Biophysical Journal. 118 (1), 128-137 (2020).
- Poor, T. A., et al. Probing the paramyxovirus fusion (F) protein-refolding event from pre- to postfusion by oxidative footprinting. Proceedings of the National Academy of Sciences U S A. 111 (25), 2596-2605 (2014).
- Roush, A. E., Riaz, M., Misra, S. K., Weinberger, S. R., Sharp, J. S. Intrinsic Buffer Hydroxyl Radical Dosimetry Using Tris(hydroxymethyl)aminomethane. Journal of American Society of Mass Spectrometry. 31 (2), 169-172 (2020).
- Everett, E. A., Falick, A. M., Reich, N. O. Identification of a critical cysteine in EcoRI DNA methyltransferase by mass spectrometry. Journal of Biological Chemistry. 265 (29), 17713-17719 (1990).
- Sanderson, R. J., Mosbaugh, D. W. Identification of specific carboxyl groups on uracil-DNA glycosylase inhibitor protein that are required for activity. Journal of Biological Chemistry. 271 (46), 29170-29181 (1996).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon