JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
레이저가 없는 하이드록실 라디칼 단백질 발자국은 단백질의 고주 구조 분석을 수행합니다.
요약
이 프로토콜은 플래시 산화 단백질 발자국을 수행하기 위해 인라인 라디칼 도시메트리와 플라즈마 광원을 사용하는 방법을 제시합니다. 이 방법은 단백질 연구의 빠른 광화학 산화의 재현성을 단순화하고 개선하기 위해 위험한 UV 레이저를 대체합니다.
초록
하이드록실 라디칼 단백질 발자국(HRPF)은 단백질 구조, 단백질 단백질 상호 작용 또는 단백질 리간드 상호 작용의 변화에 대한 정보를 제공하는 신흥 및 유망한 고주 구조 분석 기술입니다. HRPF는 하이드록실 라디칼(▪ OH)을 사용하여 단백질의 용매 접근 표면을 돌이킬 수 없이 라벨을 붙입니다. HRPF 수행의 내재된 복잡성, 비용 및 위험성은 바이오 제약분야에서 광범위하게 채택되는 것을 실질적으로 제한했습니다. 이러한 요인은 다음과 같습니다 : 1) 상당한 안전 예방 조치를 요구하는 복잡하고 위험하며 비싼 레이저의 사용; 및 2) 비교 연구를 제한하는 ▪OH의 배경 청소에 의한 HRPF의 불경한. 이 간행물은 레이저가 없는 HRPF 시스템의 작동을 위한 프로토콜을 제공합니다. 이 레이저 없는 HRPF 시스템은 라인 라디칼 도시트라이를 갖춘 고에너지 고압 플라즈마 광원 플래시 산화 기술을 활용합니다. 플라즈마 광원은 레이저 기반 HRPF 시스템보다 하이드록실 라디칼을 생성하는 데 더 안전하고 사용하기 쉬우며 효율적이며, 인라인 라디칼 도지미터는 연구의 재현성을 증가시킵니다. 레이저가 없는 HRPF 시스템은 레이저 기반 기술의 언급된 단점과 한계를 해결하고 극복합니다.
서문
단백질 형태 및 관련 고차 구조(HOS)는 적절한 생물학적 기능 및 비정상적인 동작 1의 주요결정요인이다. 구조와 기능 활동이 생산 및 환경의 다양한 측면에 의존하는 바이오 의약품도 마찬가지입니다. HOS의 바이오 의약품 변화는 바람직하지 않은 약리학 및 환자 면역 학적 반응2,3에기인하는 불리한 약물 반응 (ADR)에 연결되었습니다. ADRs의 출현은 바이오 제약 업계에 단백질 HOS가 생물 치료제의 안전성과 효능에서 중요한 역할을 하고 있으며, 새롭고 향상된 HOS 분석4의필요성을 확립했습니다.
하이드록실 라디칼 단백질 발자국(HRPF)은 단백질 HOS의 변화를 추적하는 유망한 기술입니다. HRPF는 단백질5,6,7의용매 접근 표면을 확인하기 위해 질량 분석법 (MS) 분석을 따르는 ▪OH를 가진 단백질의 외관의 돌이킬 수없는 라벨링을 포함한다. HRPF는 단백질 HOS 및 그 기능8,9에서결함을 검출하는 데 성공적으로 사용되었으며, 단일 클론 항체 (mAb)10,11,12,13의HOS를 특성화하고 리간드14의결합 Kd를 결정하고15,16,17,18,19. HRPF에 대한 ▪OH를 생성하는 일반적인 방법은 H 2O2의사진 에서 ▪OH를 생산하기 위해 고에너지, 빠른 UV 레이저를 사용하는 단백질 (FPOP)의 빠른 광화학 산화입니다. 대부분의 경우, FPOP는 호흡기 및 눈 손상을 피하기 위해 상당한 안전 장치를 요구하는 유해 가스 (KrF)를 사용하는 고가의 엑시머 레이저를사용합니다(20). 흡입 위험을 피하기 위해 다른 사람들은 독성 가스사용을 없애고 상당한 운영 전문 지식을 필요로 하며 눈 손상으로부터 사용자를 보호하기 위해 광범위한 길잃은 광 제어를 요구하는 주파수 네이드뮴 이트리움 알루미늄 가넷(Nd:YAG)레이저(21)를사용했습니다.
충분한 정보는 HRPF를 사용하여 얻을 수 있지만, 바이오 제약에서 광범위한 채택은 충족되지 않았습니다. 제한된 HRPF 채택을위한 두 가지 장벽은 다음과 같습니다 : 1) 상당한 안전 예방 조치를 요구하는 위험하고 비싼 레이저의 사용20; 및 2) 비교연구(22)를제한하는 ▪OH의 배경 청소에 의한 HRPF의 불경성. 레이저 사용을 대체하기 위해 고속, 고에너지 플라즈마 플래시 사진 용해 장치가 개발되어 FPOP를 용이하게 하는 방식으로 안전하게 수행했습니다. HRPF 실험의 불가능성을 개선하기 위해 실시간 라디칼 도시메트리가 구현됩니다.
HRPF의 관행은 ▪OH22의배경 청소에 기인하는 불경한 성에 의해 제한되었습니다. ▪OH는 단백질 지형의 우수한 프로브이지만, 그들은 또한 준비에서 발견 된 많은 성분과 반응하여 표적 단백질을 산화할 수있는 급진적 인 활성농도를 측정할 필요가 있습니다. 완충 제제, 과산화수소 농도, 리간드 특성 또는 광석의 변화는 HOS 차동 연구에서 모호성을 생성하는 대조군과 실험 군 간의 산화 차이를 초래할 수 있다. 실시간 라디칼 도시메트리를 첨가하면 OH 하중▪효과를 조정할 수 있으므로 HRPF 실험 중 신뢰와 재현성을 증가시킵니다. FPOP에서 급진적 인 dosimetry의 사용은 다른 곳에서 설명되었다23,24,25,그리고 최근 간행물 에서 자세히논의된다 26. 여기서, 우리는 새로운 플래시 사진 분석 시스템과 실시간 dosimetry의 사용을 설명하여 말 아포-묘글로빈(aMb)을 라벨링하여, FPOP 실험에서 펩티드 산화 수준을 엑시머 레이저를 사용할 때 얻은 것과 비교합니다.
프로토콜
1. 모세관 튜브 설치
- 실리카 갈라진 돌을 사용하여 250 μm 내경(ID) 실리카 모세관을 27인치로 크리브합니다. 모세관 끝을 확인하여 깨끗하고 직선적인 컷을 확인합니다.
- 폴리이미드 코팅을 태워 길이가 약 15mm인 두 개의 창을 만듭니다. "하부 엔드"에서 시작하여 첫 번째 사진 석기 창을 "하부 끝"에서 90mm 떨어진 곳에 만들고 두 번째 도시계 창은 "하부 끝"에서 225mm 떨어진 곳에 있습니다.
참고 : 코팅이 연소되면 모세관은 매우 취약합니다. - 포트 5에서 너트와 페룰을 풀고 모세관의 "하부 끝"을 발효의 원추형끝(도 1A)을삽입한다.
- 포토 리시스 모듈에서, 바로 당겨서 포토 리시스 셀 캡을 제거한 다음 모세관을 제자리에 고정시키는 자기 장착 금속 마스크를 제거합니다.
주의: 포토리시스 셀 캡 내부에는 곡선 거울이 있으며, 거울을 만지는 것을 허용하지 않습니다. - 도지계 모듈에서, 왼쪽의 탭을 밀어 서 도시계 셀을 열고 오른쪽에 열린 도지계 셀을 스윙. 모세관을 제자리에 고정시키는 자기 장착 클립을 제거합니다.
- 모세관을 사진 의 바닥에 있는 홈 채널에 놓습니다. 모세관 창을 포토리시스 셀 창으로 중앙에 두는 다. 모세관을 한 손으로 제자리에 고정하는 동안, 자기 마스크를 추가하여 모세혈관을 제자리에 고정시합니다. 포토리시스 캡을 다시 배치합니다.
- 도시계 셀의 베이스에 있는 홈 채널에 모세관을 놓습니다. 두 번째 모세관 창을 도시미터 세포 기지의 중앙에 있는 작은 조리개에 집중합니다. 모세관을 한 손으로 제자리에 고정하는 동안 두 개의 자기 클립을 위치에 놓아 모세관을 제자리에 고정시하십시오. 도지계 셀이 닫을 때까지 닫습니다.
- 모세관을 상품 수집기의 모세관 리프트 위에 누른 너트를 통해 삽입한다(도1B). 모세관을 유리병 바닥 바로 위로 확장합니다.
참고: 모세관이 유리병 의 바닥에 도달하여 실험을 실행하는 동안 모세관이 담금질 용액에 침수되도록 하는 것이 중요합니다.
2. 사출 루프 설치
- 외경 1/16"과 0.015"(381 μm)의 내지름이 있는 테플론 튜브를 사용합니다. 방정식 1을 따라 방정식 1을 사용하여 원하는 볼륨에 필요한 튜브 길이를 계산합니다.
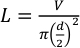
L이 튜브의 길이가 밀리미터인 경우, V는 마이크로리터에서 원하는 부피이며 d는 튜브 내경을 밀리미터 단위로 한다. 25 μL의 주입 부피와 381 μm의 내경의 경우 튜브 길이는 약 219.3 mm여야합니다.
참고: 20 μL 미만의 볼륨의 경우 내지0.01"의 내경을 가진 PTFE 튜브를 사용합니다. - 튜브 커터를 사용하여 테프론 튜브를 필요한 길이로 자릅니다. 튜브 의 끝을 확인하여 깨끗하고 직선컷을 확인하십시오.
- 너트 중 하나를 통해 새 주입 루프의 한쪽 끝을 삽입하고 튜브의 끝에 새로운 ferrule을 배치합니다. 페룰과 너트를 제자리에 고정하고 밸브에서 바닥이 나올 때까지 주입 밸브의 포트 3에 튜브를 삽입합니다. 튜브를 단단히 잡고 너트 손가락을 단단히 조입니다. 렌치를 사용하여 너트 1/4 회전을 더 조입니다. 어셈블리를 제거하고 검사합니다.
참고: ferrule이 위치에 고정되지 않은 경우 1/8 회전을 다시 설치하고 조입니다. 루프의 다른 쪽 끝으로 반복합니다. - 양 끝이 너트와 고정 된 ferrule을 가지고 나면, 느슨하게 포트 3에 한쪽 끝을 나사 와 포트 6(그림 1C)에다른 끝. 일단 자세로 양쪽을 꽉 조이면 1/4는 렌치로 손가락을 단단히 돌립니다.
3. 사진 투하 시스템 초기화
- 다음 순서로 포토리시스 모듈을 켜십시오: (1) 유체 모듈 (2) Photolysis 모듈 (3) Dosimetry 모듈 (4) 제품 수집기, 마지막으로 (5) 시스템 컴퓨터 및 제어 소프트웨어를 실행
참고: Dosimeter 모듈이 추운 시작부터 워밍업을 위해 적어도 반 시간 동안 허용합니다. - 주사기펌프(도 1D)의"밸브" 위치에서 10mL 의 완충액으로 튜브를 완전히 침수합니다. "폐기물"위치(도 1D)에서튜브를 주사기 포트(도1C)에배치하여 빈 용기에 배치하여 폐기물을 수거합니다.
참고: 단백질이 중단될 버퍼를 사용합니다. 권장 버퍼는 10mM NaPO4입니다. - 제품 수집기 회전 목마에 1.5 mL 마이크로 센심 분리기 튜브를 H와 6으로 표시된 위치에 놓습니다.
- 250 μm ID 모세관을 사용하는 경우 하중 유량을 500 μL/min으로 설정하고 유량은 7.5 μL/min으로 설정하고 폐기물 유량은 500 μL/min으로 설정합니다.
- 플래시 지연, 제품 시간 및 낭비 시간을 계산하여 방정식 2를 사용하여 반자동 모드를 사용합니다.
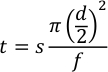
t가 분 단위로 시간인 경우, s는 밀리미터의 모세관 거리이며, d는 모세관의 내경체가 밀리미터이고, f는 μL/min의 유량이다.- 플래시 지연의 경우 거리는 모세관의 "하부 끝"에서 첫 번째 창까지이며, 이는 약 90mm여야 합니다. 250 μm ID 모세관및 7.5 μL/분의 처리 유량을 사용하여 샘플은 약 35초 만에 사진 용해 창에 도착합니다. 샘플이 도착하기 전에 1Hz에서 두 번의 깜박임이 발생하도록 허용하므로 플래시 지연을 33초로 설정합니다.
- 제품 시간 동안 주입 된 용액이 주입 된 시간을 입력하면 사출 밸브에서 모세관의 끝으로 흐르는 데 걸리는 시간이 소요됩니다. 27" 길이 250 μm ID 모세관의 경우 제품 시간을 4.5분으로 설정합니다.
- 폐기물 시간의 경우 주입된 용액의 총 부피를 수집하는 데 걸리는 시간을 입력합니다. 이때 제품 수집기는 제품 위치에서 폐기물 바이알로 이동합니다. 25 μL의 주입 부피와 7.5 μL/min의 유량의 경우 폐기물 시간을 7.8분으로 설정합니다.
- 주입 밸브가 하중 위치로 설정된 주입 포트에 HPLC 급 물 25 μL을 주입하여 주입 루프를 5번 헹구는다.
- 수동으로 사출 밸브를 '주입 위치'로 돌려 시스템의 나머지 부분을 플러시합니다. 제어 소프트웨어에서 프로세스(Out)를 선택하여 물이 흐르기 시작합니다. 흐르는 동안 모세관의 끝을 볼 수 있도록 제품 수집기 에서 위로를 선택하여 제품 수집기 모세관 리프트를 올립니다. 물방울이 형성될 때까지 모세관을 통해 물을 흐르십시오.
참고: 액자가 형성되지 않으면 인젝터 밸브에서 누출 여부를 확인합니다. 누출이 있는 경우 너트를 풀고 모세관을 다시 앉히고 다시 조입니다.
4. 버퍼에서 급진적 인 청소 효과를 테스트하기 위해 실제 ▪OH 수율을 결정합니다.
- 제어 소프트웨어에서 프로세스(Out)를선택하여 흐름을 시작합니다. 설정 탭에서 플래시 전압을 0 V로 설정합니다. 도지계 데이터 섹션의 수동 제어 탭에서 데이터 시작 + AutoZero를선택합니다.
- 제품 바이알(H) 및 폐 바이알(6)의 위치를 선택합니다.
- 준비 를선택하고, 수동으로 사출 밸브를 부하 위치로 돌리고 1mM Adenine의 25 μL을 주입하고 100 mM H 2 O2를주입 포트에 주입하십시오. 주입되면 주입 위치로 주입 밸브를 수동으로 켭니다.
참고: 이렇게 하면 시스템이 자동으로 흐름을 시작하고 플라즈마 소스를 켜고 dosimetry 데이터를 수집합니다. - 설정 탭으로 탐색하고 플래시 전압을 변경하여 전압을 증가시면 전압을 증가시합니다. 500 V, 750 V, 1000 V 및 1250 V. 트리플리케이츠의 각 전압에서 아데닌 흡광도의 판독을 수행하여 4.2 및 4.3 단계를 반복합니다.
- 계산 탭을 선택하여 각 샘플의 평균 흡광도를 결정합니다. 먼저 선택(Select)을클릭한 다음 피크 흡광도의 시작과 끝을 수동으로 선택합니다. 사용 가능한 공간에서 샘플에 대한 설명에 적는다. 획득한 모든 데이터에 대해 반복합니다.
참고: 거품이 형성되어 체계 판독이 급증할 수 있습니다. 평균 흡광도를 결정하기 위해 데이터를 선택할 때 거품이 있는 영역을 생략합니다. - Excel에서 데이터를 복사하고 붙여넣기하여 각 전압에 대한 아데닌 흡광도의 평균 변화를 계산하여 효과적인 ▪OH 수율을 결정합니다.
- 반복 단계 4.1-4.6 여러 샘플 조건 (다른 버퍼 / 첨가제)이 각 조건에 대한 유효 ▪OH 수율을 정상화하는 데 사용되는 경우.
5. 고차 구조의 변화를 감지하기 위해 단백질을 수정합니다.
- 4mM 아데닌과 20 μM의 단백질을 1대1 비율로 혼합하여 2mM 아데닌과 10 μM 단백질을 함유한 용액을 만듭니다.
- 0.3 mg/mL 카탈라아제로 담금질 용액을 만들어 과산화수소와 35m 메티오닌 아마이드를 분해하여 남은 래디칼을 담금질하십시오. Aliquot 25 μL의 담금질 용액이 200 μL 마이크로튜브로 결합되어 동일한 양의 담금질과 라벨이 부착된 제품이 혼합됩니다.
- H2O2 ~ 200mMM을 희석하고 얼음을 유지합니다.
- 설정 탭의 제어 소프트웨어에서 플래시 전압을 0V로 시작하여 배경 산화를 결정합니다.
- 수동 제어 탭에서 데이터 시작 + AutoZero를선택하고 프로세스(Out)다음 준비한다음 마지막으로 사출 밸브를 부하 위치로 바릅니다.
- 제품 수집 회전 목마에 배치 1에 담금질 용액을 배치합니다. 시스템 제어 소프트웨어에서 제품 바이알을 1로 변경합니다.
- 주입 직전에 아데닌과 단백질 혼합물의 12.5 μL을 H2O2의 12.5 μL과 혼합하여 1mM Adenine, 5 μM 단백질 및 100 mM H 2 O2의최종 농도를 만듭니다. 부드럽게 파이펫 위아래로 섞고, 빠르게 회전하고, 혼합 후 10초 이내에 사출 포트를 사용하여 25 μL을 주입합니다.
- 사출 밸브를 주입 위치로 전환하고 샘플이 처리되는 동안 기다립니다.
- 500V, 750V, 1000 V 및 1250 V. 트리플리케이트로 각 전압 측정을 수행하여 반복 합니다.
- 4.5 단계에서 설명된 바와 같이 평균 흡광도를 계산합니다. 모든 데이터를 복사하여 Excel에 붙여넣습니다.
6. 시스템 종료
- 모든 샘플을 수집한 후, 주사 밸브를 하중 위치로 설정하여 주사기 포트 및 샘플 루프를 플러시하고 HPLC 물의 25 μL을 5회 주입한다.
- 주입 밸브를 주입 위치로 돌려 시스템의 나머지 부분을 HPLC 물로 플러시합니다.
- 흐름을 중지하고 시스템 제어 소프트웨어를 닫고 제품 수집기, 체계 모듈, photolysis 모듈로 시작하는 모듈을 끄고 마지막으로 유동성 모듈을 끕니다.
7. 샘플 준비 및 액체 크로마토그래피 질량 분광법
- 50m Tris 및 1mM CaCl2의존재시 20분 동안 80°C에서 샘플을 배양하여 단백질을 분해한다. 실온에 샘플을 냉각하고 단백질에 1:20 트립신을 추가합니다. 샘플 혼합으로 단백질을 37°C에서 밤새 소화합니다. 다음날 아침, 10분 동안 샘플을 95°C로 가열하여 트립신 소화를 종료합니다.
- UPLC 시스템과 연결된 고해상도 LC-MS/MS 시스템을 사용하여 펩티드를 감지합니다.
- 샘플을 먼저 트랩 컬럼(300 μm ID X 5mm 100 Å 모공 크기, 5 μm 입자 크기)에 로드하고 5.0 μL/mL에서 3분 동안 2% 용매 B(아세토니틀및 0.1% 포믹산)를 함유한 물로 세척합니다.
- C18 나노컬럼(0.75mm x 150mm, 2 μm 입자 크기, 100 Å 모공 크기)의 펩티드를 300nL/min의 유량으로 분리하여 용매 A(0.1%의 포믹산을 함유한 물)와 용매 B 사이의 그라데이션을 갖는다. 펩타이드 용출에 대한 그라데이션은 22분 이상 2~35% B에서 선형 증가로 구성되며, 5분 이상 95% B로 진입하여 3분 동안 열을 세척한 다음 3분 동안 2%B로 돌아와 컬럼을 다시 평형화하기 위해 9분 동안 유지하였다.
- 양수 이온 모드에서 데이터를 획득합니다. 스프레이 전압을 2400 V로 설정하고 이온 이송 튜브의 온도를 300°C로 설정합니다.
- 250-2000 m/z에서 전체 MS 스캔을 획득한 다음 8개의 후속 데이터 의존MS/MS 스캔을 상위 8개 펩타이드 이온에 검사합니다. 펩티드를 단편화하기 위해 35% 정규화된 에너지에서 충돌 유도 된 해리를 사용합니다.
- 단백질 서열 및 관련 단백질 서열을 포함하는 필요한 FASTA 파일에 대해 사용 가능한 단백질 분석 소프트웨어를 사용하여 MS/MS 분석에서 검출된 모든 수정되지 않은 펩티드를 식별한다.
- HRPF 데이터 처리 소프트웨어를 사용하여 수정된 펩티드를 검색하고 정량화합니다. 확인된 각 펩타이드에 대한 산화의 정도는 수정된 펩타이드의 총 크로마토그래피 피크 영역에 의해 수정된 펩타이드의 합산 크로마토그래픽 피크 영역을 방정식 3을 사용하여 수정및 수정되지 않은 것으로 계산한다.
P = [I(singly 산화) X 1 + I (이중 산화) X 2 + I(트립 산화) X 3 + .../[Iunoxidized + I(노래 산화+ I) + I(이중 산화)+ I(삼중 산화) + I(삼중 산화)
여기서 P는 펩티드 분자당 평균 산화 이벤트 수를 나타내고, 나는 산화되지 않은 펩티드(Iunoxidized)와 n 산화 이벤트를 가진 펩티드의 피크 영역을 나타낸다.
8. 차동 연구의 경우 두 번째 조건에서 5-7 단계를 반복하십시오.
참고: 재현성을 확인하려면 각 조건에 대한 기술적 삼중 생물 외에도 두 개의 생물학적 복제가 권장됩니다.
결과
실시간 dosimetry와 결합된 고압 플라즈마 소스는 ▪OH 항복을 더 잘 제어하여 고차 단백질 구조의 변화를 보다 정확하게 관찰할 수 있게 합니다. 아데닌을 첨가하면 효과적인 실시간 라디칼 도지계가 가능합니다. 산화시, 아데닌은 265 nm(그림 2A)에서UV 흡광도를 잃는다. 아데닌 흡광도의 변화는 따라서 효과적으로 버퍼, 부형제, 리간드(그림 2B)와?...
토론
HRPF 실험 중에 단백질의 적절한 라벨링을 보장하기 위한 몇 가지 중요한 단계가 있습니다. 첫째, 적절한 유량 및 소스 플래시 레이트가 선택되어 시료의 각 볼러스가 한 번 조사되도록 합니다. 이것은 단백질이 새로 형성된 ▪OH의 단 하나 bolus에 드러나는 것을 보장합니다. 단백질이 산화되면, 더 높은 차순 단백질 구조를 변경할 수 있습니다. 네이티브 단백질 구조가 조사되고 있다고 확신...
공개
E.E..C. J.S.S.와 S.R.W.는 고차 단백질 구조 분석을 위한 기술을 상용화하고자 하는 초기 단계 회사인 GenNext Technologies, Inc.에 상당한 재정적 관심을 공개합니다. 제공된 대표 데이터는 재정적 이해 상충이 없는 S.K.M에 의해 검토되었습니다.
감사의 말
이 작품은 일반 의학의 국립 연구소에 의해 투자되었다 (R43GM125420 및 R44GM125420).
자료
| Name | Company | Catalog Number | Comments |
| 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | any brand is sufficient |
| 50 µL SGE Gastight Syringes | Fisher Scientific | SG-00723 | |
| Acclaim PepMap 100 C18 nanocolumn (0.75 mm X 150 mm, 2 µm) | Thermo Scientific | ||
| Acetonitrile with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS120-500 | |
| Apomyoglobin | Sigma-Aldrich | ||
| Catalase | Sigma-Aldrich | C9322 | |
| Centrifuge | Eppendorf | 022625501 | |
| Delicate Task Wipers | Fisher Scientific | 06-666A | |
| Hydrogen Peroxide | Fisher Scientific | H325-100 | any 30% hydrogen peroxide is sufficient |
| Methionine amide | Chem-Impex | 03109 | |
| Microcentrifuge | Thermo Scientific | 75002436 | |
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | other high resolution instruments (e.g. Q exactive Orbitrap or Orbitrap Fusion) can be used |
| Pierce Trypsin Protease, MS Grade | Thermo Scientific | 90058 | |
| Polymicro Cleaving Stone, 1" x 1" x 1/32” | Molex | 1068680064 | any capillary tubing cutter is sufficient |
| UPLC | Thermo Scientific | ||
| Water with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS118-500 | |
| Water, LC/MS Grade | Fisher Scientific | W6-4 |
참고문헌
- Nagarkar, R. P., Murphy, B. M., Yu, X., Manning, M. C., Al-Azzam, W. A. Characterization of protein higher order structure using vibrational circular dichroism spectroscopy. Current Pharmaceutical Biotechnology. 14 (2), 199-208 (2013).
- Giezen, T. J., Schneider, C. K. Safety assessment of biosimilars in Europe: a regulatory perspective. Generics and Biosimilars Initiative Journal. , 1-8 (2014).
- Giezen, T. J., Mantel-Teeuwisse, A. K., Strauss, S. Safety-related regulatory actions for biologicals approved in the United States and the Europena Union. Journal of the American Medical Society. 300 (16), 1887-1896 (2008).
- Gabrielson, J. P., Weiss, W. F. Technical decision-making with higher order structure data: starting a new dialogue. Journal of Pharmaceutical Sciences. 104 (1), 1240-1245 (2015).
- Brenowitz, M., Erie, D. A., Chance, M. R. Catching RNA polymerase in the act of binding: intermediates in transcription illuminated by synchrotron footprinting. Proceedings of the National Academy of Sciences U S A. 102 (13), 4659-4660 (2005).
- Guan, J. Q., Takamoto, K., Almo, S. C., Reisler, E., Chance, M. R. Structure and dynamics of the actin filament. Biochemistry. 44 (9), 3166-3175 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photochemical oxidataion to locate heme binding and conformataional changes in myoglobin. International Journal of Mass Spectrometry. 259 (2007), 124-129 (2007).
- Li, K. S., Shi, L., Gross, M. L. Mass Spectrometry-Based Fast Photochemical Oxidation of Proteins (FPOP) for Higher Order Structure Characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Watson, C., Sharp, J. S. Conformational Analysis of Therapeutic Proteins by Hydroxyl Radical Protein Footprinting. American Association of Pharmaceutical Scientists Journal. 14 (2), 206-217 (2012).
- Deperalta, G., et al. Structural analysis of a therapeutic monoclonal antibody dimer by hydroxyl radical footprinting. mAbs. 5 (1), 86-101 (2013).
- Jones, L. M., et al. Complementary MS methods assist conformational characterization of antibodies with altered S-S bonding networks. Journal of American Society of Mass Spectrometry. 24 (6), 835-845 (2013).
- Storek, K. M., et al. Monoclonal antibody targeting the β-barrel assembly machine of Escherichia coli is bactericidal. Proceedings of the National Academy of Sciences. , (2018).
- Vij, R., et al. A targeted boost-and-sort immunization strategy using Escherichia coli BamA identifies rare growth inhibitory antibodies. Scientific Reports. 8 (1), 7136 (2018).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Lu, Y., et al. Fast Photochemical Oxidation of Proteins Maps the Topology of Intrinsic Membrane Proteins: Light-Harvesting Complex 2 in a Nanodisc. Analytical Chemistry. 88 (17), 8827-8834 (2016).
- Marty, M., Zhang, H., Cui, W., Gross, M., Sligar, S. Interpretation and Deconvolution of Nanodisc Native Mass Spectra. Journal of American Society of Mass Spectrometry. 25, (2013).
- Johnson, D. T., Di Stefano, L. H., Jones, L. M. Fast photochemical oxidation of proteins(FPOP): A powerful mass spectrometry based structural proteomics tool. Journal of Biological Chemistry. , (2019).
- Chea, E. E., Jones, L. M. Analyzing the structure of macromolecules in their native cellular environment using hydroxyl radical footprinting. Analyst. 143 (4), 798-807 (2018).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical chemistry. 90 (12), 7721-7729 (2018).
- Linde. . Linde Specialty Gases of North America. , (2009).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77 (18), 5814-5822 (2005).
- Niu, B., Zhang, H., Giblin, D., Rempel, D. L., Gross, M. L. Dosimetry determines the initial OH radical concentration in fast photochemical oxidation of proteins (FPOP). Journal of American Society of Mass Spectrometry. 26 (5), 843-846 (2015).
- Misra, S. K., Orlando, R., Weinberger, S. R., Sharp, J. S. Compensated Hydroxyl Radical Protein Footprinting Measures Buffer and Excipient Effects on Conformation and Aggregation in an Adalimumab Biosimilar. American Association of Pharmaceutical Scientists Journal. 21 (5), 87 (2019).
- Olson, L. J., Misra, S. K., Ishihara, M., Battaile, K. P., Grant, O. C., Sood, A., Woods, R. J., Kim, J. P., Tiemeyer, M., Ren, G., Sharp, J. S., Dahms, N. M. Allosteric regulation of lysosomal enzyme recognition by the cation-independent mannose 6-phosphate receptor. Communications Biology. 3 (1), 498 (2020).
- Sharp, J. S., Misra, S. K., Persoff, J. J., Egan, R. W., Weinberger, S. R. Real Time Normalization of Fast Photochemical Oxidation of Proteins Experiments by Inline Adenine Radical Dosimetry. Analytical Chemistry. 90 (21), 12625-12630 (2018).
- Misra, S. K., Sharp, J. S. Enabling Real-Time Compensation in Fast Photochemical Oxidations of Proteins for the Determination of Protein Topography Changes. Journal of Visualized Experiments. 163, (2020).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of American Society of Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemical Reviews. 120 (10), 4355 (2020).
- Jones, L. M., Sperry, B. J., Carroll, A. J., Gross, M. L. Fast photochemical oxidation of proteins for epitope mapping. Analytical chemistry. 83 (20), 7657-7661 (2011).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical chemistry. 89 (4), 2250-2258 (2017).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Kiselar, J. G., Janmey, P. A., Almo, S. C., Chance, M. R. Structural analysis of gelsolin using synchrotron protein footprinting. Molecular and Cellular Proteomics. 2 (10), 1120-1132 (2003).
- Chea, E. E., Deredge, D. J., Jones, L. M. Insights on the Conformational Ensemble of Cyt C Reveal a Compact State during Peroxidase Activity. Biophysical Journal. 118 (1), 128-137 (2020).
- Poor, T. A., et al. Probing the paramyxovirus fusion (F) protein-refolding event from pre- to postfusion by oxidative footprinting. Proceedings of the National Academy of Sciences U S A. 111 (25), 2596-2605 (2014).
- Roush, A. E., Riaz, M., Misra, S. K., Weinberger, S. R., Sharp, J. S. Intrinsic Buffer Hydroxyl Radical Dosimetry Using Tris(hydroxymethyl)aminomethane. Journal of American Society of Mass Spectrometry. 31 (2), 169-172 (2020).
- Everett, E. A., Falick, A. M., Reich, N. O. Identification of a critical cysteine in EcoRI DNA methyltransferase by mass spectrometry. Journal of Biological Chemistry. 265 (29), 17713-17719 (1990).
- Sanderson, R. J., Mosbaugh, D. W. Identification of specific carboxyl groups on uracil-DNA glycosylase inhibitor protein that are required for activity. Journal of Biological Chemistry. 271 (46), 29170-29181 (1996).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유