Method Article
大腸菌における天然接合プラスミドを介した遺伝子水平伝播の検出
要約
コンジュゲーションは、プラスミドDNAを2つの異なる細胞に動員することにより、遺伝子の水平伝播を媒介し、有益な遺伝子の拡散を促進します。この研究では、接合プラスミド、ドナー、およびレシピエントの鑑別マーカーを使用してトランスコンジュゲーションを検出することに基づいて、接合プラスミド転移を効率的に検出するために広く使用されている方法について説明しています。
要約
抱合は、グラム陰性菌の水平遺伝子導入を促進する主要なメカニズムの1つです。この研究では、2つの天然に存在するプラスミドを例として使用して、天然に存在する接合プラスミドの動員を研究する方法について説明します。これらのプロトコルは、ドナー、レシピエント、および接合プラスミドにおける選択可能なマーカーの異なる存在に依存しています。具体的には、記載される方法には、1)天然の接合プラスミドの同定、2)固体培養における抱合速度の定量、および3)ポリメラーゼ連鎖反応(PCR)によるトランスコンジュガントレシピエントにおける抗生物質耐性遺伝子およびプラスミドレプリコンタイプの診断的検出が含まれる。ここで説明するプロトコルは、環境中に見られる細菌における抗生物質耐性遺伝子を運ぶ接合プラスミドの存在をスクリーニングするために、水平遺伝子導入の進化生態学を研究するという文脈で開発されました。培養におけるこれらの実験で観察された接合プラスミドの効率的な転写は、一般的な遺伝子水平伝播、特に抗生物質耐性の拡大を促進するメカニズムとしての接合の生物学的関連性を強調しています。
概要
1946年、レーダーバーグとテイタム1は、現在抱合として知られている大腸菌K-12の性的プロセスについて説明しました。細菌結合は、細菌細胞(ドナー)が細胞間直接接触によって一方向に遺伝物質を別の細胞(レシピエント)に移すプロセスです。抱合は細菌2,3に広く分布しているが、接合機構を発現するドナー細胞の画分は通常非常に小さい4。
プラスミドは、自律複製する染色体外DNAエレメントです。プラスミドの複製と維持に関与する遺伝子に加えて、プラスミドは重金属や抗生物質への曝露などの環境問題への適応に関与する遺伝子の貨物を頻繁に運びます5。接合プラスミドは、レシピエント細胞への導入を可能にし、導入後の持続性をサポートする特殊な遺伝子のセットを持つプラスミドのクラスです6。接合プラスミドのサイズは、シュードモナドータ門(プロテオバクテリアと同義)の細菌で21.8 kbから1.35 Mbまで変化し、中央値は約100 kb 5,7です。それらはまた、おそらく宿主への代謝負荷を低く保つために、一般的に低いコピー数を有する8,9。
典型的な接合装置は、転移起点(oriT)、リラクターゼ、IV型結合タンパク質、IV型分泌系(ドナーがレシピエントと接触できるピルスと呼ばれる管状の構造)の4つの要素で構成されています6。接合プラスミドを保有する細胞のごく一部のみが接合機構を発現しますが4、プラスミドが適応度の利点を提供する場合、トランスコンジュガントは集団内で急速に拡大する可能性があります。異なる生息地から収集された大腸菌分離株の35%から80%以上は、少なくとも1つの抗生物質に対する耐性を付与する遺伝子を有する接合プラスミドを有する10,11;したがって、接合プラスミドによって媒介される水平遺伝子導入は、抗生物質耐性遺伝子の世界的な普及を推進する主要なメカニズムである12。
実験室培養で行われた交配実験は、接合頻度が、レシピエント細胞の性質、成長段階、細胞密度、ドナーとレシピエントの比率、コンジュゲーションが液体培地または固体培地のどちらで行われるか、炭素、酸素、胆汁酸塩、金属濃度、哺乳類細胞の存在、温度、pH、および交配時間を含む複数の要因によって影響を受けることを示しています13,14、15.
この研究では、特定の宿主株における接合プラスミドの存在を検出し、固体培養における接合速度を定量化し、レシピエント細胞へのそれらの移動を再確認するためのプロトコルについて説明します。これらのプロトコルは、研究に適した天然の接合プラスミドを同定するための最初のステップとして使用できます。それらは、複数の供給源(環境、共生、および病原性)から得られた細菌中の接合プラスミドの存在を大規模(数十から数百のドナー)でスクリーニングするように設計されているため、最小限の簡略化されたステップを使用します。
さらに、所与の接合プラスミドの動員が検出に使用された抗生物質とは無関係であるかどうか(すなわち、選択中の抗生物質耐性遺伝子の関連性)を検出し、異なる環境分離株で見出される2つの接合プラスミドの抱合率を比較するための試験が示されている。
関連する接合プラスミドの遺伝的構成(プラスミドレプリコンおよび抗生物質耐性遺伝子構成)に基づいて、プロトコルの各ステップを変更して、接合速度に影響を与える可能性のあるさまざまな要因の影響を研究することができます。
一般実験計画:
交配実験のセットアップに必要な必須成分は、ドナー細胞、レシピエント株、およびドナー(抗生物質A)、レシピエント(抗生物質B)、およびトランスコンジュガント(抗生物質AおよびB)を選択するための固体培地です。トランスコンジュガントは、ドナーの接合プラスミドを安定に維持するレシピエント細胞です。
ドナー細胞は抗生物質(抗生物質A)に耐性があり、レシピエント細胞(抗生物質B)を選択するために使用されるマーカーまたはマーカーに耐性があります。抗生物質耐性遺伝子のゲノム位置(すなわち、それが染色体に位置するか、ドナー細胞のプラスミドに位置するか)は、レシピエントへの抗生物質耐性マーカーの動員(ドナー−レシピエント間の直接接触後)がドナー提供マーカーがプラスミド中にあったことを意味するので、先験的に知る必要はない。
レシピエント株(接合プラスミドを取ることが知られている)は、ドナーに存在しない安定した選択マーカーを持っている必要があります。この選択可能なマーカーは、一般に、染色体に位置する抗生物質または殺生物剤に耐性がある。交配実験に使用するレシピエントの選択は、一部の 大腸菌 株が接合プラスミドを摂取する能力が異なるため、非常に重要です16。
これらの成分が確立されると、ドナーとレシピエントのペアが接触した後に抗生物質(AとB)の両方を含む培地上で増殖するコロニーは、推定トランスコンジュガントです(図1)。これは、ドナーが抗生物質Aを含む培地では増殖できるが抗生物質Bを含む培地では増殖できないこと、およびレシピエントが抗生物質Bを含む培地では増殖できるが、抗生物質Aでは増殖できないことを前提としています。最初のテストは、トランスコンジュガントコロニーの接合プラスミドに見られる遺伝子の検出(ポリメラーゼ連鎖反応[PCR]遺伝子の増幅、またはその他の方法による)で構成されます。2番目のテストでは、ラクトース代謝に基づく差動コロニーカラーマーカーを使用します。異なるコロニーの色は、マッコンキー寒天の使用によって明らかになります。寒天中の乳糖は、乳糖発酵(lac+)微生物による発酵源として使用できます。これらの微生物は、pHを下げる有機酸、特に乳酸を生成します。ニュートラルレッドは、pHが6.8を下回るとオフホワイトから明るい赤/ピンクに変わる培地に含まれるpHインジケーターです17。したがって、 大腸菌 ラクトース陽性株はマッコンキー寒天培地でより大きなピンク色のコロニーを生成しますが、ラクトース陰性株はマッコンキー寒天培地で淡黄色で小さなコロニーを生成します。
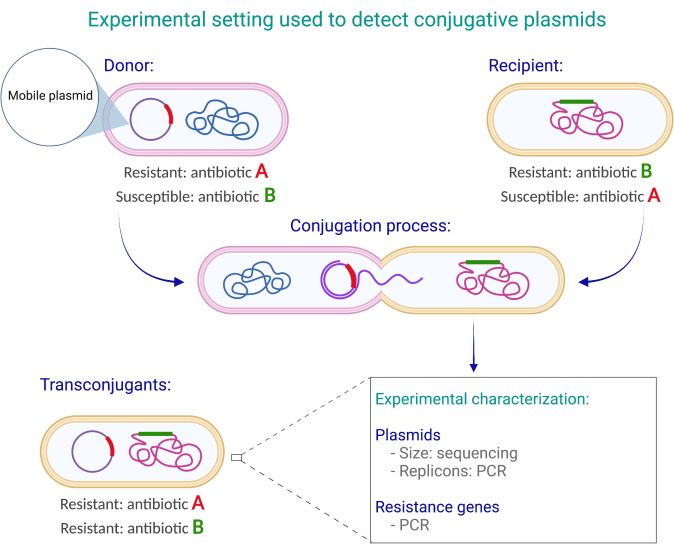
図1:ドナー株における接合プラスミドの存在を検出するために使用される実験デザイン。 この例では、ドナーは抗生物質Aに対する耐性を付与する抗生物質耐性遺伝子を有する接合プラスミドを有するが、抗生物質Bに感受性である。 逆に、レシピエントは抗生物質Bからの保護を与える染色体耐性決定基を有するが、抗生物質Aに感受性である。 トランスコンジュガントは抗生物質(AおよびB)の両方に耐性がある。 抗生物質Aに対する耐性を付与するドナーの接合プラスミドと、抗生物質Bからの保護を付与するレシピエントの染色体を持っているためです。 この 図の拡大版を表示するには、ここをクリックしてください。
ドナーとレシピエントのペアの共役率は(所与の実験条件下で)トランスコンジュガントの数をドナーの数またはレシピエントの数で割ることによって計算できます。第1の速度は、ドナー16、18による接合機構の機能的発現を示すドナー細胞の画分を示し、一方、第2の速度は、接合プラスミド19、20を取るレシピエントの能力を示す。この研究では、特に明記しない限り、コンジュゲート速度は、トランスコンジュガントになるレシピエント細胞の割合(すなわち、レシピエント当たりの割合)を表す。
ここでは、1人の大腸菌レシピエントと2人の大腸菌ドナーを含む2つの独立した交配実験が報告されています。さらに、異なる抗生物質を使用してドナーの1人のトランスコンジュガントを選択し、接合プラスミドに見られる抗生物質耐性遺伝子のいずれかで単一の多剤耐性プラスミドを選択できることを確認しました。
この研究で使用されたドナー株とレシピエント株は、この実験システムのすべてのコンポーネントを理解するために完全に配列決定されています。ただし、これらのプロトコルは、未知の配列の宿主における接合プラスミドの存在をスクリーニングするように設計されており、この実験状況でも使用できます。ただし、この場合、関連する遺伝子が最初に配列決定されます。
プロトコルで使用されるドナー株とレシピエント株は次のとおりです。
ドナー1。 大腸菌 SW4955は、バトンルージュ(米国LA)の湖で収集されました。134,797 bp の接合プラスミド (p134797) と IncFIC(FII) および IncFIB (AP001918) のレプリコンがあります。この接合プラスミドは、第3世代セファロスポリン(blaCTX-M-55)、アミノグリコシド(aac(3)-IIa および aadA1)、フェニコール(catA2)、テトラサイクリン(tet(A))、トリメトプリム(dfrA14)、およびスルホンアミド(sul3)に対する耐性を付与する遺伝子を有する。p134797 の完全なマップについては、 図 2A を参照してください。 大腸菌 SW4955はラクトース陽性で、マッコンキー寒天培地にピンク色のコロニーを生成します。
ドナー2。 大腸菌 SW7037はエリー湖(米国オハイオ州オタワ郡)で採集された。101,718 bp の接合プラスミド (p101718) と IncI1-I(Alpha) レプリコンを担持しています。この接合プラスミドは、β-ラクタム(blaCMY-2)に対する耐性を付与する遺伝子を有する。p101718 の完全なマップについては、 図 2B を参照してください。 大腸菌 SW7037もラクトース陽性であり、マッコンキー寒天培地上にピンク色のコロニーを生成します。
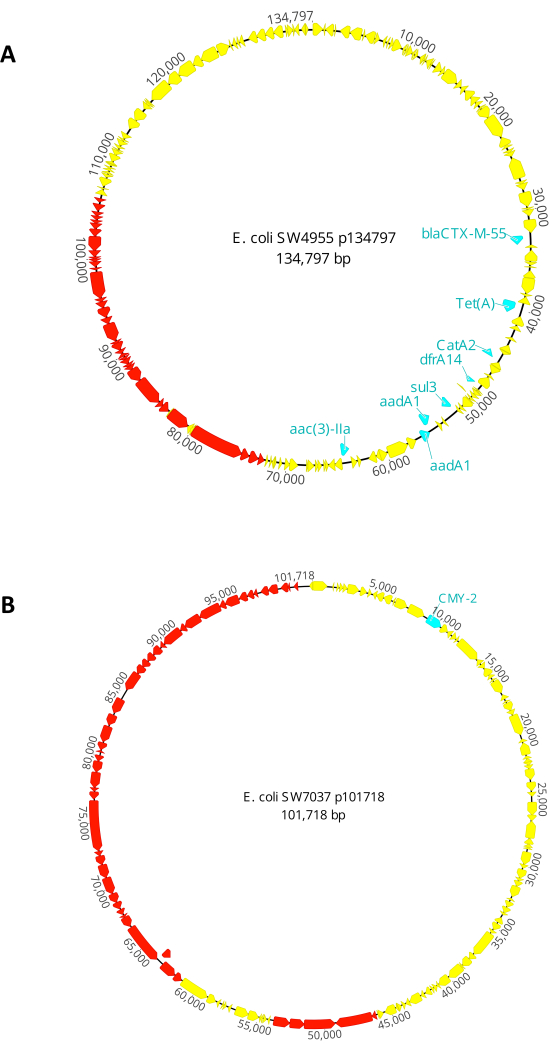
図2:本研究で用いた接合プラスミドの遺伝子マップ 。 (a)プラスミドp134797、 大腸菌 株SW4955に見られる接合プラスミド。(b)プラスミドp101718、 大腸菌 SW7037株に見られる接合プラスミド。抗生物質耐性遺伝子は青色で強調表示され、接合装置に属する遺伝子は赤色で強調表示されます。 この図の拡大版を表示するには、ここをクリックしてください。
受信者。 大腸菌 LMB100 が受信者として使用されます。これは、リファンピン(100 mg / L)およびストレプトマイシン(100 mg / L)に耐性のあるプラスミドレス株です。2つの抗生物質に耐性があると、結果の解釈を妨げる耐性変異がドナーに発生する可能性が低くなります。さらに、 大腸菌 LMB100は乳糖陰性であり、マッコンキー寒天培地上で淡黄色で小さなコロニー(大きなピンク色のコロニーとは対照的に)を生成するため、2つのドナー株と区別できます。
ドナーが乳糖陰性の場合、乳糖陽性レシピエント(例:大腸菌J53)の使用をお勧めします 。 LMB100およびJ53株は、他のラボで使用することができます。ヘラルド・コルテス・コルテス博士に住所とフェデックス番号を添えてリクエストを送信してください。
ドナー、レシピエント、およびトランスコンジュガントを選択してカウントするために必要な固体培地は、直径100 mmのペトリ皿に入ったマッコンキー寒天培地です。次の抗生物質の追加が必要です:(i)培地A:ドナーを数え、レシピエントがこの抗生物質で成長できないようにするためのカルベニシリン(100 mg / L)。(ii)培地B:リファンピン(100 mg / L)+ストレプトマイシン(100 mg / L)レシピエントを数え、ドナーがこれら2つの抗生物質で成長できないようにします。(iii)培地AB:カルベニシリン(100 mg / L)+リファンピン(100 mg / L)+ストレプトマイシン(100 mg / L)トランスコンジュガントを取得してカウントします。(iv)培地C:研究したすべての分離株にストリークする抗生物質はありません。
大腸菌SW4955および大腸菌SW7037から大腸菌LMB100への接合プラスミドの接合率を比較した。さらに、p134797 接合プラスミド (SW4955 株) の場合、抗生物質カルベニシリン (100 mg/L) をゲンタマイシン (2 mg/L)、クロラムフェニコール (25 mg/L)、テトラサイクリン (10 mg/L)、トリメトプリム (20 mg/L)、またはスルファメトキサゾール (100 mg/L) に置き換え、選択に使用した抗生物質耐性マーカーが結果に影響を与えるかどうかを確立します。
プロトコル
1.方法1:活用
- 1日目:ドナーとレシピエントをストリークします。培地C(抗生物質を含まないマッコンキー寒天培地)にグリセロールストックとは別にストリークし、37°Cで一晩インキュベートします。
注:このステップは、実験が純粋な分離株で行われていることを確認し、コロニーの色によってラクトースの表現型を確認するために必要です。 - 2日目:ドナーとレシピエントごとに14.0 mLの培養チューブにラベルを付けます。各ドナーとレシピエントのコロニーを1つ選択し、ミューラーヒントンブロス2 mLを入れた別々の14.0 mL培養チューブで37°C、200 rpmで振とうしながら一晩(18時間)増殖させます。
注:使用した菌株では、18時間の一晩培養後に固定期に達します。 - 3日目:生理食塩水900 μLと一晩培養液100 μLの1:10希釈を使用して、各ドナーとレシピエントの一晩培養(ボルテックスなし)の600 nm(OD600)での光学密度を測定します。
- 滅菌生理食塩水(水中0.85%NaCl)を使用して、各ドナーとレシピエントの光学密度を2.0(OD600 = 2.0)に調整します。
注:OD600 = 2.0の大腸菌の一晩培養では、1.6 x 109 CFU / mLになります。 - 1.5 mLマイクロ遠心チューブに「相手チューブ」というラベルを付け、ドナー株とレシピエント株を示します。各ドナーとレシピエントの調整済み(OD600 = 2.0)懸濁液500 μLを移し、それらを嵌合チューブに入れます(この嵌合チューブには、各ドナーとレシピエントの0.8 x 109 CFU / mLがあります)。相手チューブを反転させて静かに混ぜます。
- 室温で500 x g で10分間、相手チューブを遠心分離します。
- ペレットを乱すことなく、800 μLのコンジュゲーションチューブをピペットで取り出します。コンジュゲーションチューブには200μLが残っているはずです。
注意: 上清を10%漂白剤容器に廃棄して、懸濁液中の細菌を不活性化します。 - インキュベーター内で37°Cで18時間(一晩)インキュベートします。交配は潜伏期間中に起こります。
注:このステップは、共役線を壊さないように振らずに実行する必要があります。 - ネガティブコントロールとして、培地B上のドナーと培地A上のレシピエントの一晩培養物をストリークします。 プレートを37°Cで一晩インキュベートします
- 4日目:800 μLの生理食塩水を嵌合チューブとボルテックスに加えて再懸濁します(これにより、嵌合ミックスが1 mLに再構成されます)。
注:ボルテックスは交配細菌を分解し、培養物を均質化してCFU / mLを定量化します。 - 交配ミックスの1:10希釈液(1 x 100 から1 x 10-7)を準備します。100 μLの希釈液10-5 から10-7 を培地Aと培地Bにプレートし、すべての希釈液を培地ABにプレートします。
注:Dlution 100はきちんとしたチューブを指します。 - プレートを逆さまにしてインキュベーターに入れる前に、プレートを乾かしてください。プレートを37°Cで18時間(一晩)インキュベートします。
- 5日目:ネガティブコントロールを調べて、ドナーの純粋な一晩培養の縞が培地Bで成長せず、レシピエントの純粋な一晩培養が培地Aで成長しなかったことを確認します。
- 数えることができるコロニーと希釈の数を記録します。
- 培地Aのコロニーを数えます。これらはドナーです。コロニーはピンク色でなければなりません(図3A、B)。
- メディアBのコロニーを数えます。これらはレシピエントとトランスコンジュガントです。コロニーは淡黄色でなければなりません(図3C)。
- 培地AB内のコロニーを数えます。これらはトランスコンジュガントです。コロニーは淡黄色になるはずです。
注: カウント可能なプレートを選択します。カウント可能なプレートには30〜300個のコロニーがあります(300を超えるコロニーはカウントが困難であり、30個未満のコロニーはサンプルサイズが小さすぎて元のサンプルを正確に表現できないと見なされます)。
- ドナーごとまたはレシピエントごとの活用の頻度を計算する
ドナーあたりの抱合頻度=(トランスコンジュガント[培地AB]のCFU/mL)/(ドナー[培地A]のCFU/mL)x 100
レシピエントあたりのコンジュゲーションの頻度 = (トランスコンジュガント[培地AB]のCFU/mL)/(レシピエントおよびトランスコンジュガント[メディアB]のCFU/mL)x 100
注:このプロトコルの共役頻度は、トランスコンジュガントをレシピエントの数で割って100を掛けて計算しました。文献によれば、結果として生じる接合の量は、エクスジュゲート頻度、遺伝子導入頻度、コンジュゲーション頻度、組換え収率、プラスミド導入効率、総細菌数に基づくコンジュゲーション頻度、トランスコンジュガントの割合、レシピエント集団におけるトランスコンジュガントの割合、トランスコンジュガント頻度、コンジュゲーション頻度、コンジュゲーション速度の対数、トランスジュゲーション速度定数、交配ペアあたりのコンジュゲーション率、 共役係数、または共役効率20。このプロトコルは、共役効率という用語を指します。 - トランスコンジュガントをグリセロールストックに-80°Cで保存します。 グリセロールストックを準備するためのサブステップに従ってください。
- トランスコンジュガントの単一コロニーを選択し、カルベニシリン(100 mg/L)を添加したミューラーヒントンブロス2 mLを含む5.0 mL培養チューブで、37°C、200 rpmで振とうしながら一晩(18時間)増殖させます。
- トランスコンジュガントの名前を示す極低温バイアルを次のようにラベル化する:Tc +ドナーの名前+培地Aでの選択の抗生物質(トランスコンジュガントであるため、そのバックグラウンドはレシピエント株であることが知られており、これはすでにストレプトマイシンおよびリファンピンに耐性があるが、さらにβ-ラクタム耐性遺伝子を有するプラスミドを有する)。たとえば、TcU1Carb などです。同じドナーからの複数のトランスコンジュガントを保存する必要がある場合(例:TcU1Carb1、TcU1Carb2など)に連続した番号を追加します。
- 一晩培養液1 mLを1 mLの50%グリセロール(v / v、最終グリセロール濃度は25%)に移し、転倒して穏やかに混合します。極低温バイアルをドライアイスに15分間置き、将来の実験のためにクライオバイアルを-80°Cで保管します。
- DNA抽出のために、カルベニシリン(100 mg/L)を添加したミューラーヒントン寒天培地上のグリセロールストックからトラスコンジュゲートをストリークし、37°Cで一晩インキュベートします。次に、手順 2.1 の推奨事項に従います。
2.方法2:大腸 菌 トランスコンジュガントにおける抗生物質耐性遺伝子とプラスミドレプリコンを増幅するためのポリメラーゼ連鎖反応(PCR)
注:ポリメラーゼ連鎖反応(PCR)は、1983年にKary Mullis博士によって開発されました。PCRは、特定のプライマー(複製が進行できるポイントとして機能する特定のDNA配列に相補的なDNAの短い断片、一般に20〜40ヌクレオチドの長さ、理想的にはグアニン-シトシン含有量が40%〜60%)に依存し、変性した二本鎖DNAテンプレートの反対側の鎖にアニールされ、耐熱性DNAポリメラーゼを介して伸長されます。 したがって、反応の次のサイクルのための追加のテンプレートを生成し、元のテンプレート21の指数関数的増幅をもたらす。このプロトコルでは、接合プラスミドに存在するレプリコンおよび耐性遺伝子を増幅し、トランスコンジュガントレシピエント細胞における導入を確認した。
- DNA抽出
- 接合プラスミドによって運ばれる耐性遺伝子の転写を検出するには、トランスコンジュガントDNAを鋳型として使用します。単純なボイルプレップ抽出を使用して、細菌の細胞壁を溶解します。
注意: 単純なボイルプレップ抽出は、細菌の細胞壁を溶解するための簡単なアプローチです。この抽出には、ゲノムDNAと混合したプラスミドDNAが含まれていますが、プライマーは目的の特定のDNA配列に従って設計されているため、このテンプレートはPCRにも有用です。プラスミドを特異的に精製することもできますが、プラスミドサイズが大きくなると収率は低下する傾向があります22。これは便利な停留所です。DNAは-20°Cで数ヶ月間保存できます。
- 接合プラスミドによって運ばれる耐性遺伝子の転写を検出するには、トランスコンジュガントDNAを鋳型として使用します。単純なボイルプレップ抽出を使用して、細菌の細胞壁を溶解します。
- PCR法
- 実験にネガティブコントロールとポジティブコントロールがあることを考えると、1.5 mLチューブ内のすべての反応のためのプールを設定することは有益です。1.5 mLチューブに「プール」とラベルを付け、必要なPCRチューブに遺伝子とサンプルの名前(OXA-U1など)をラベル付けします。
- PCR試薬を1.5 mLチューブにピペットで入れます:滅菌水、2xマスターミックス、プライマー、およびポリメラーゼ(テンプレートDNAを除く)( 表1を参照)。少なくとも10回上下にピペッティングして穏やかに混ぜます。ずっと氷の上に保管してください。
注意: 反応混合物または試薬の汚染を避けるために手袋を着用してください。試薬の汚染を防ぐために、試薬の開封と混合、および同じ領域でのサンプルの取り扱いは避けてください。試薬をアイスバケットに入れて、ヌクレアーゼ活性と非特異的プライミングを抑制します。反応を設定する前に、それらを完全に解凍させてください。実験中は試薬を氷上に保管してください。TaqポリメラーゼはpH変化に敏感であるため、最後に追加されます。劣化やミスフォールディングを避けるためにバッファリングする必要があります。プライマーの配列を表2−抗生物質耐性遺伝子−および表3−レプリコン23、24、25、26、27、28に列挙する。 - テンプレートDNAを対応するチューブに追加します。PCRチューブに気泡を入れたり、キャップを固定したりしないでください。
- PCRチューブをサーマルサイクラーに入れます。
- プログラムを起動します。表 2 (耐性遺伝子)および 表3 (レプリコン)の各遺伝子について記載されているプログラム設定を参照してください。
注:実行後、サンプルを4°Cに保持するようにPCRプログラムを設定します。- レプリコンのPCR条件:94°Cで5分間の変性を1サイクル、続いて94°Cで1分間の変性を30サイクル、60°Cで30秒間アニーリング、72°Cで1分間伸長、72°Cで5分間の1サイクルの最終延長。
注:これらはCarattoliらによって標準化された条件です29。
- レプリコンのPCR条件:94°Cで5分間の変性を1サイクル、続いて94°Cで1分間の変性を30サイクル、60°Cで30秒間アニーリング、72°Cで1分間伸長、72°Cで5分間の1サイクルの最終延長。
- プログラムが終了したら、PCRチューブを4°Cで保管します。
注:これは便利な停止ポイントです。PCR産物は-20°Cで数ヶ月間保存できます - 250 mLフラスコに0.6 gのアガロースを量り、60 mLの1xトリスアセテート-EDTA(TAE)バッファーを加えて、1%アガロースゲルを調製します(別のゲルサイズが必要な場合は試薬を調整します)。アガロースがフラスコにこぼれないように、アガロースを電子レンジで注意深くゆっくりと溶かし、約55°C(10〜15分)まで冷却します。
- 超高純度の脱イオン水と分析グレードの試薬を使用して、すべての試薬を調製します。
- TAE 50xバッファー(トリス塩基、酢酸、およびEDTA)(1 L):トリス塩基121.1 g+ EDTA372.24 gを混合し、500 mLの蒸留水を加えます。マグネチックスターラーで溶かします。酢酸57.1mLを加え、蒸留水を総量1,000mLまで加える。
- オートクレーブを15psi、121°Cで15分間行い、準備ができるまで室温で最大6か月間保管します。
- TAE 1xバッファー(1 L):20 mLのTAE 50xバッファーと980 mLの蒸留水を混ぜ合わせて混合します。
- ヒュームフードの下で、溶かしたアガロースに6 μLのSYBR Greenを加え、混合して、こぼれないように粘着テープで密封したトレイ(およびコーム)に注ぎます。濁りが現れるまでゲルを15〜25分間セットします。
注:他の代替染料も入手可能です30,31,32,33。 - 粘着テープをはがし、ゲルを電気泳動チャンバーに入れ、ゲルを覆うのに十分な1x TAEバッファーを追加します。
- 5 μLの6x DNAゲルローディング色素を各反応の25 μLと混合します。少なくとも10回上下にピペッティングして穏やかに混ぜます。
注:予想される産物を視覚化するために、すべてのPCR産物をゲルにロードする必要はありません(対応する色素の量を調整します)。残りのPCRサンプルは保存し、-20°Cで数ヶ月間保存することができます。 - 混合物をウェルに入れ(気泡の導入を避け)、チャンバーの蓋を下ろします。
注:ネガティブコントロールから始めて、最初のサンプルを2番目のウェルにロードします(最初のサンプルをはしごに予約します)。 - 4 μLの1 kb DNAラダーを最初のウェルにロードします。
- 電気泳動を120 V、400 mA、および60分で実行します。
- UV光(UVイルミネーターを使用)の下でゲルを視覚化し、画像を記録します。
注:PCR産物が存在する場合、染色されたDNAバンドは、標準のUVトランスイルミネーター、可視光トランスイルミネーター、またはレーザーベースのスキャナーを使用して検出できます。
| 試薬 | ストックソリューション | 50 μL反応液に添加量(1回) | 最終的な | 例: ボリューム | 例: ボリューム | 例:各チューブに添加した容量 ファイナル Vol. 50 μL | ポジティブコントロールに追加された音量 | ネガティブコントロールに追加された音量 |
| 濃度 | 1x に追加されました | 3x (プール) に追加 | ||||||
| 滅菌水 | - | 21.5 μL (50 μLまで) | - | 21.5 μL | 64.5 μL | 49 μL/チューブ | 49 μL/チューブ | 49 μL/チューブ |
| マスターミックス | 2倍速 | 25 μL | 1倍速 | 25 μL | 75 μL | |||
| フォワードプライマー | 25 μM | 1 μL | 0.5 μM | 1 μL | 3 μL | |||
| リバースプライマー | 25 μM | 1 μL | 0.5 μM | 1 μL | 3 μL | |||
| テンプレートDNA | 可変 (100–200 ng/μL) | 可変 (1 μL) | 変数 | 0.5 μL | 1.5 μL | |||
| ポリメラーゼ | 5単位/μL | 0.5 μL | 2.5 単位 | - | - | 1 μL(トランスコンジュガント) | 1 μL (ドナー) | 1 μL (レシピエント) |
| 注:q.s.は、必要な量を意味する量子サティスのラテン語の略語です。 | ||||||||
表1:PCR試薬および3反応用プール(一例)。
| プライマー(5'-3'方向にリストされている配列) | PCRプログラム | 予想されるサイズ |
| (BP)参照 | ||
| ブラCTX-M グループ 1 | 94 °C 7分 | (864)23名 |
| CTX-M: GGTTAAAAATCACTGCGYC | 94 oC 50 s (35 サイクル) | |
| CTX-M: TTGGTGACGATTTTAGCCGC | 50 oC 40 s | |
| 68 °C 1分 | ||
| 68 °C 5分 | ||
| ブラCMY-2 | 95 °C 3分 | (1855)24名 |
| CMY-2-F: GATTCCTTGGACTCTTCAG | 95 oC 30 s (30 サイクル) | |
| CMY-2-R: TAAAACCAGGTTCCCAGATAGC | 53 oC 30 s | |
| 72 oC 30 s | ||
| 72 °C 3分 | ||
| aac(3')-II | 94 °C 5分 | (237)25名 |
| aac(3')-II-F: ACTGTGATGGGATACGCGTC | 94 oC 30 s (32 サイクル) | |
| aac(3')-II-R: CTCCGTCAGCGTTTCAGCTA | 60 oC 45 s | |
| 72 °C 2分 | ||
| 72 °C 8分 | ||
| アーダ | 94 °C 5分 | (283)26名 |
| aadA1: GCAGCGCAATGACATTCTTG | 94 °C 1分(35サイクル) | |
| aadA2: ATCCTTCGGCGCGATTTTG | 60 °C 1分 | |
| 72 °C 1分 | ||
| 72 °C 8分 | ||
| スル-3 | 94 °C 5分 | (799)27名 |
| sul-3-F: GAGCAAGATTTTGGAATCG | 94 °C 1分(30サイクル) | |
| sul-3-R: CATCTGCAGCTAACCTAGGGCTTTGGA | 51 °C 1分 | |
| 72 °C 1分 | ||
| 72 °C 5分 | ||
| テト(A) | 95 °C 5分 | (957)28名 |
| TETA-1: GTAATTCTGAGCACTGTCGC | 95 oC 30 s (23 サイクル) | |
| TETA-2: CTGCCTGGACAACATTGCTT | 62 oC 30 s | |
| 72 oC 45 s | ||
| 72 °C 7分 | ||
| ティッカーある | 95 °C 5分 | (302) この作品 |
| dfrA-F: CATACCCTGGTCCGCGAAAG | 95 °C 1分(30サイクル) | |
| 55 °C 1分 | ||
| dfrA-R: CGATGTCGATCGTCAAGTG | 72 °C 1分 | |
| 72 °C 7分 | ||
| catA2 | 95 °C 5分 | |
| catA2-F: GACCCGGTCTTTACTGTCTTTTC | 95 °C 1分(25サイクル) | (225)この作品 |
| catA2-R: TCCGGTGGTGATATCAGATTAATAAT | 60 °C 1分 | |
| 72 °C 1分 | ||
| 72 °C 7分 |
表2:診断耐性遺伝子を増幅するために使用されるプライマーおよびPCRプログラム。
| レプリコン | プライマー(5'-3'配向でリストされた配列) | ターゲット | PCRプログラム | 予想されるサイズ (bp) | |||
| 株式会社フィブ | F: TCTGTTTATTCTTTTTACTGTCCAC | レパ | 94 °C 5分 | 683 | |||
| R: CTCCCGTCGCTTCAGGGCATT | 94 °C 1分(30サイクル) | ||||||
| 60 oC 30 s | |||||||
| 株式会社フィック | F: GTGAACTGGCAGATGAGGAGG | repA2 | 72 °C 1分 | 262 | |||
| R: TTCTCCTCGTCGCCAAACTAGAT | 72 °C 5分 | ||||||
| IncI1 | F: CGAAAGCCGGACGGCAGAA | ティッカー | 139 | ||||
| R: TCGTCGTTCCGCCAAGTTCGT | |||||||
表3:PCRベースのレプリコンタイピング29によってプラスミドp134797およびp101718を分類するために使用されたプライマーおよびPCRプログラム。
結果
ドナーSW4955およびSW7037が配列決定されていることを考えると、これら2つのドナー株は、それらのゲノム配列で同定された耐性遺伝子プロファイルに対応する耐性表現型を有すると予想される。プラスミドp134797(SW4955由来)は、第3世代セファロスポリン(blaCTX-M-55)に対する耐性、およびアミノグリコシド(aac(3)-IIaおよびaadA1)、フェニコール(catA2)、テトラサイクリン(tet(A))、トリメトプリム(dfrA14)、およびスルホンアミド(sul3)に対する耐性を付与する遺伝子を有する。SW4955染色体には抗生物質耐性遺伝子は見つからなかった。プラスミドp101718(SW7037由来)は、第3世代セファロスポリン(blaCTX-M-55)に対する耐性を付与すると予想される抗生物質耐性遺伝子(blaCMY-2)を1つだけ持っています。ここでも、SW7037染色体に抗生物質耐性遺伝子は見つかりませんでした。
交配後、上記のすべての耐性遺伝子を持つ2つの接合プラスミドの転写の検出が期待されました。ポジティブコンジュゲーション検出は、トランスコンジュガントとして同定されたレシピエントがコンジュゲーションプラスミドを獲得しているはずであることを意味します。トランスコンジュガント中の接合プラスミドの診断耐性遺伝子の存在(PCRによって検出される)も予想された。
本明細書に記載の方法を例示するために、2つの 大腸菌 環境単離物がコンジュゲーションによってプラスミドDNAを転写する能力を試験した。実験設定の概略図を 図1に示します。2人のドナー(大腸菌 SW4955およびSW7037)と1人のレシピエント(大腸菌 LMB100)を独立して交配させた。 大腸菌 SW4955 (p134797) および SW7037 (p101718) で見つかった接合プラスミドの遺伝子マップを 図 2 に示します。
接合プラスミドをカルベニシリンで選択し、リファンピンとストレプトマイシンを使用してカウンター選択し、その耐性決定基はレシピエントの染色体にあります。MacConkey寒天培地を使用して、乳糖陽性ドナー(大きなピンク色のコロニーを生成する)をレシピエントおよびトランスコンジュガント(乳糖陰性で、より小さな淡黄色のコロニーを生成する)と区別しました(図3)。
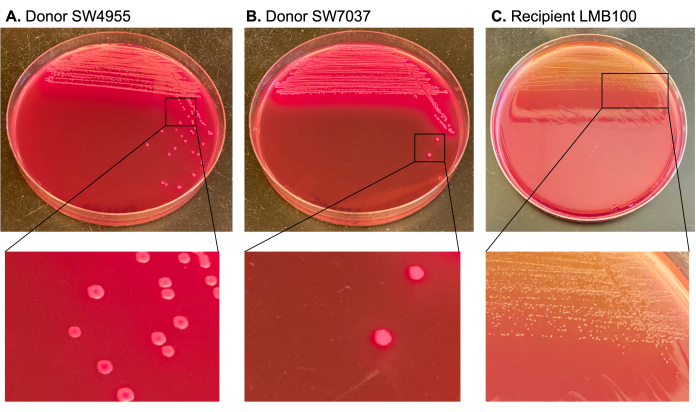
図3:MacConkey寒天培地上のドナーコロニーとレシピエントコロニーの異なるカラーマーカーの図。 ドナーに対応するコロニーは、ラクトース陽性であるため、マッコンキー寒天培地ではピンク色です。これは、ドナーを、淡黄色で小さい(乳糖陰性)レシピエントコロニーと区別します。 この図の拡大版を表示するには、ここをクリックしてください。
接合型プラスミドp134797の動員が検出され、5つの抗生物質のそれぞれがプラスミドに見られる5つの異なるクラスの耐性遺伝子に対応しました。
株SW4955の接合効率(CE)を計算する方法の例が提示されます。株SW4955を用いたアッセイから、172CFUのトランスコンジュガントを希釈10-2からプレート内で計数した。対応する希釈液(10-2)からのレシピエントの数は10,300,000 CFU/mLでした(表4)。
CEは次のように計算されます。
CE = (174 CFU/mL)/(10,300,000 CFU/mL)
CE= 1.67 x 10-5トランスコンジュガント/レシピエント
| LMB100 | カルベリシリンで選択されたSW4955株からのトランスコンジュガント | 共役効率 | ||
| 希釈 | CFU(カウントプレート) | 約CFU/mL | CFU(カウントプレート) | |
| 100 | 合流 | 1.03E+09 | ||
| 10-1. | 合流 | 1.03E+08 | ||
| 10-2. | 合流 | 10300000 | 172 | 1.67 x 10-5 トランスコンジュガント/レシピエント |
| 10-3. | 合流 | 1030000 | ||
| 10-4. | 合流 | 103000 | ||
| 10-5. | 合流 | 10300 | ||
| 10-6. | 合流 | 1030 | ||
| 10-7. | 103 | 103 | ||
| CE の計算に使用される値は太字で強調表示されています。 | ||||
表4:株SW4955の接合効率(CE)を計算する方法の例。
結果を表5に示し、レシピエント当たりの抱合効率として 表した。SW4955株をドナーとして得られた5つの接合効率は、すべて同じ桁以内でした。これらの結果は、接合プラスミドの動員がトランスコンジュガント同定に用いた選択に関係なく検出できることを示している。
株SW7037をドナーとして使用すると、得られた接合効率は3桁低かった。これらの結果は、異なるドナーおよびプラスミドタイプの結合効率を同じレシピエントと比較することを可能にする。
| 共役効率 | |||||
| 濾す | カルベニシリン(100 mg / L) | ゲンタマイシン(2 mg / L) | クロラムフェニコール(25 mg / L) | テトラサイクリン(10 mg / L) | トリメトプリム(20 mg / L) |
| SW4955 | 1.67×10-5 | 5.67×10-5 | 2.17×10-5 | 7.62×10-5 | 1.36×10-5 |
| SW7037 | 2.14×10-6 | ||||
表5:選択に使用した抗生物質に応じた大腸菌ドナー株SW4955およびSW7037の結合効率。
トランスコンジュガントにおいて、レプリコンおよび試験した2つの接合プラスミドにコードされるすべての耐性遺伝子、ならびにそれらに対応するレプリコンの存在をPCRによって確認した。これらの診断PCR反応に用いた条件を 表3に示す。
診断用ゲルを図4に示す。これらのゲルは、トランスコンジュガントにおける予想されるレプリコンの存在を確認します (p1347975 の IncFIC および IncFIB、および p101718 の IncI1)。また、予想される抗生物質耐性遺伝子、すなわちp1347975についてはbla CTX-M-55群(β-ラクタム)、aac(3)-IIおよびaadA(アミノグリコシド)、catA2(フェニコール)、tet(A)(テトラサイクリン)、dfrA14(トリメトプリム)、およびsul3(スルホンアミド)はp1347975、p101718についてはblaCMY-2の存在も確認された(図4)。
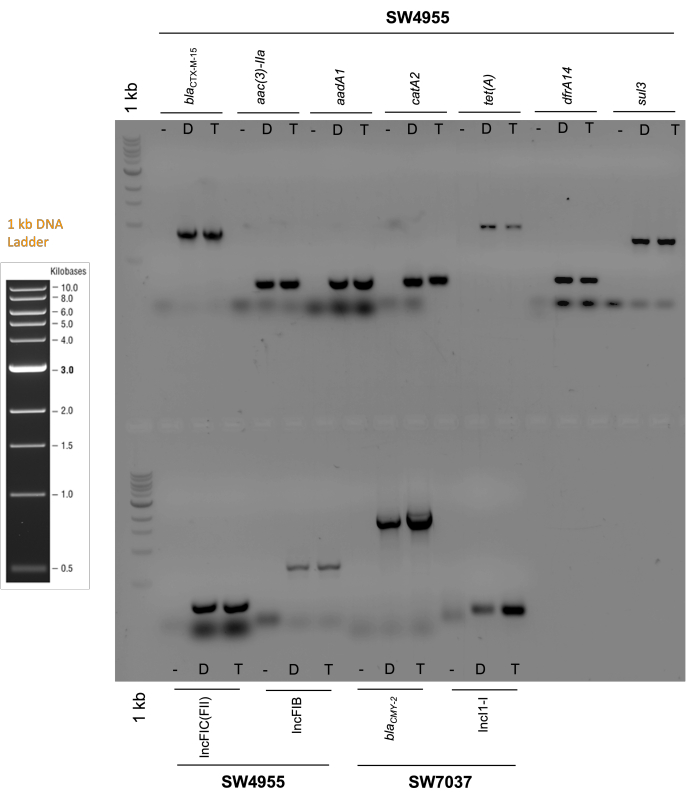
図4:診断用電気泳動ゲル。 ドナー細胞(それぞれ株SW4955およびSW7037)および対応するLMB100トランスコンジュガントにおける接合プラスミドp134797およびp101718に見られる抗生物質耐性遺伝子およびプラスミドレプリコンのPCR産物のゲル電気泳動。カルベニシリンをプラスミド選択に用いた。略語。-:陰性対照(陰性対照としてLMB100株のDNAを用いた);D:ドナー;T:トランスコンジュガント。予想されるアンプリコンサイズ:blaCTX-M-55:864 pb; aac(3)-IIa: 237 bp; aadA1: 283 bp; カテゴリA2: 225 bp; tet(A): 957 bp; dfrA14: 302 bp; スル3: 799 BP;IncFIC(FII): 262 bp;IncFIB: 683 bp; ブラCMY-2: 1,855 bp;IncI1-I: 139 bp.ゲルは1%のアガロースを有する。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
接合プラスミドは、組換えと水平遺伝子導入を通じて、特定の環境環境内の遺伝子の共同プールへのアクセスを提供します34。したがって、接合プラスミドは、細菌が時間的スケールで複数の条件(抗生物質に対する耐性、金属に対する耐性、金属の獲得、バイオフィルム形成、病原性遺伝子などを含む)に適応することを可能にする機能を獲得および付与できる進化的実体です。
この研究は、細菌中の接合プラスミドを同定するためのプロトコルを提示します。プロトコルが機能するためには、培地AおよびBのコントロールによって示されるように、使用されるマーカーがドナー株とレシピエント株を区別する必要があります。また、プラスミドを保有する細胞を効果的に選択する必要があります。嵌合反応は重要です。この反応では、ドナーとレシピエントは、抱合が起こるために(ピリを介して)長時間接触する必要があります。不十分なインキュベーション時間や培養物の振とうや攪拌など、細胞間の接触を妨げる可能性のあるものはすべて、結合の効率を低下させます。一部のレシピエント株は接合に難治性であるため、レシピエントの選択も重要です35,36。LMB100は、複数の非適合性タイプのプラスミドを採取できるため、最適なレシピエントとして提案されています。
結合率は、各プラスミド-ドナー染色体ペア、レシピエント、および環境条件に特異的です。特定のプラスミドのコンジュゲーションは、成長期、細胞密度、ドナーとレシピエントの比率、コンジュゲーションが液体培地で行われるか固体培地で行われるか、炭素、酸素、胆汁酸塩、金属濃度、哺乳類細胞の存在、温度、pH、交配時間など、多数の変数に敏感であることがわかっています13,14,15.これらの相互作用の研究は、詳細に研究される接合プラスミドの初期検出に依存します。したがって、ここで説明するプロトコルを変更して、さまざまな実験変数の影響を調べることもできますが、これにはスクリーニングされたドナーの数を制限するという犠牲が伴います。また、ヘルパープラスミド(すなわち、トランス6の動員可能なプラスミドに欠けている機能を提供することによって結合を可能にするプラスミド)を有する宿主中の動員可能なプラスミドを同定することもできる。
接合プラスミドの配列を知ることが推奨される(ただし、上記のように結合を検出するために必要ではない)。ドナーがラクトース陰性(MacConkey寒天培地上で黄色のコロニーを産生する)の場合、ラクトース陽性レシピエント(MacConkey寒天培地上でピンク色のコロニーを産生する)を使用して、真のトランスコンジュガントと、トランスコンジュガントを選択するために培地上で増殖できるドナー細胞を区別できます。ここで説明するプロトコルは、腸内細菌科の環境、共生、および病原性のメンバーからの接合プラスミドを検出するように設計されています。ただし、適切なドナー、レシピエント、抗生物質、およびカラーマーカーを持つ任意の細菌種で使用できます。他の細菌におけるこれらの重要な成分の同定には、複数のドナー、レシピエント、およびマーカー(抗生物質および色)を使用した系統的研究が必要です。このプロトコルは、グラム陽性菌ではテストされていません。.
ここに提示された方法のいくつかの変形が文献で報告されており、最近レビューされた20。定性的結果はさまざまな方法(例えば、エクスコンジュガント頻度および遺伝子導入頻度)で参照することもでき、無次元単位を使用してコンジュゲーション効率(mL/CFU x h)を報告することができる20。
ここで説明するプロトコルは、既存の方法16、18、19、20ではこれまで対処されていなかったいくつかの制限を解決する。まず、適切なレシピエントが特定されました。第二に、真のトランスコンジュガントを選択するために2つの抗生物質(リファンピンとストレプトマイシン)を使用することで、ドナーが自然突然変異誘発を通じてトランスコンジュガントを選択するために使用される抗生物質の1つに対する耐性を進化させる可能性が最小限に抑えられます。偽のトランスコンジュガントは、ドナーによって大量に産生される酵素(例えば、β-ラクタマーゼ)によるトランスコンジュガントを選択するために使用される抗生物質の加水分解による傍観者保護からも生じ得る。第三に、交配の産物が真のトランスコンジュガント(すなわち、ドナーの接合プラスミドを安定に取り込んだレシピエント細胞)であることを保証するために、いくつかの実験的対照が含まれる。ここでは、トランスコンジュガントの真正性をテストするための2つの独立した方法、すなわちMacConkey寒天培地の比色マーカーと、レシピエントの接合プラスミドのレプリコンと抗生物質耐性遺伝子のPCR検出を紹介しました。また、ここで説明するプロトコルは、腸内細菌科(エシェリヒア、クレブシエラ、エンテロバクター、シトロバクター、サルモネラ、赤痢菌、およびその他の種)の環境、共生、および病原性のメンバーから接合プラスミドを単離および特徴付けるように設計されています。
共役のメカニズムを理解するために、実験的観測はコンピュータシミュレーションを使用してモデル化されています。これらの予測モデルは、ドナー、レシピエント、およびトランスコンジュガントの特定の増殖密度に対するプラスミド導入の頻度を推定します。エンドポイントモデルとして知られるモデルは、液体培養におけるプラスミド転写速度がそれを超える閾値を発見し、転写速度は細胞密度、ドナー:レシピエント比、および交配時間の影響を受けないと結論付けました19。蛍光 in situ ハイブリダイゼーション(FISH)は、トランスコンジュガントプラスミド検出の代替方法として使用されています。FISHは、DNAプローブとターゲットDNAのハイブリダイゼーションにより、蛍光顕微鏡を使用したプラスミドの可視化を可能にします。したがって、FISHは、異なる細胞集団37にわたるプラスミドフローの視覚的検出を可能にするが、トランスコンジュガントが選択とは対照的に視覚的スクリーニングによって検出される場合、ここに提示された方法と同じレベルの感度を有しない。
生態系のさまざまな構成要素(診療所、農業、下水、野生生物、家畜、土壌、河川、湖)を介して抗生物質耐性遺伝子を分散させている接合プラスミドの生物学を理解することが非常に必要です。要約すると、ここに記載されているプロトコルに示されている簡略化された実験条件は、大規模なドナーのスクリーニングを容易にし、したがって、さまざまなソースからの接合プラスミドに由来する水平遺伝子導入の研究のための重要なツールを表しています。これらは、複数の供給源および細菌からの接合プラスミドにおける抗生物質耐性遺伝子または他の臨床的に関連する遺伝子の有病率を調査するために使用できます。それらはまた、 in vivo (例えば、脊椎動物の腸内)での抱合の研究および接合の効率を調節する条件の研究に適応させることができる。これらの研究はすべて、多剤耐性接合プラスミドの動員が多剤耐性の拡大にどのように寄与するかを理解するのに大いに役立ちます。
開示事項
著者は開示するものは何もありません。
謝辞
LM-B、IMG、AT、およびISは、LM-Bに授与されたNIH助成金GM055246によってサポートされました。GC-Cは、2017-18年度と2018-19年度の2年連続でUCメクサス-コナシットポスドクフェローシップを授与されました。ジェネンテック財団が後援するアカデミックインスピレーションネットワーク(GAIN)メンタリングプログラムの学生のセージチャベスとペッパーセントクレアに、実験の技術サポートに心から感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 1.5 mL tube racks | Thermo Fisher Scientific | 22-313630 | It can be replaced for another brand |
| 2.0 mL Cryogenic vials | Corning | 430659 | It can be replaced for another brand |
| 14 mL conical tubes | FALCON | 149597 | It can be replaced for another brand |
| 14 mL tube racks | Thermo Fisher Scientific | 8850 | It can be replaced for another brand |
| 1 kb DNA ladder | New England Biolabs | N0552G | |
| 250 mL sterile flasks | PYREX | 5320 | It can be replaced for another permanent marker brand |
| Acetic acid | Fisher Scientific | A38SI-212 | It can be replaced for another brand |
| Agarose (molecular biology grade, multipurpose) | Fisher Scientific | BP160-500 | It can be replaced for another brand |
| Carbenicillin | GOLD BIOTECHNOLOGY | C-103-50 | It can be replaced for another brand |
| Disposable inoculating loops: 1 µL | Fisherbrand | 22-363-595 | It can be replaced for another brand |
| DNA gel loading dye 6x | New England Biolabs | B7024S | It can be replaced for another brand |
| EDTA | Fisher Scientific | BP119-500 | It can be replaced for another brand |
| Electronic digital scale | Denver Instrument | APX-2001 | It can be replaced for another brand |
| Eppendorf conical tubes: 1.5 mL | Eppendorf | 14-282-302 | It can be replaced for another brand |
| Equipment for agarose gel electrophoresis | Fisher Scientific | FP300 | It can be replaced for another brand |
| Ethanol-resistant markers | Sharpie | 37001; 37002; 37003 | It can be replaced for another brand |
| Gel documentation systems (UVP GelSolo) | Analytakjena | SP-1108; 849-97-0936-01; 95-0612-01 | It can be replaced for another brand |
| Gloves | X-GEN | 44-100M | Any nitrile gloves brand works |
| Ice bucket | Thermo Fisher Scientific | 432128 | It can be replaced for another brand |
| MacConkey agar | BD DIFCO | 212123 | |
| Master Mix | BioLabs | M0496S | It can be replaced for another brand |
| Methanol | Fisher Scientific | A452-4 | It can be replaced for another brand |
| Microcentrifuge tubes: 2.0 mL | Fisherbrand | 05-408-138 | It can be replaced for another brand |
| Mueller Hinton agar | BD DIFCO | DF0479-17-3 | |
| Mueller Hinton broth | BD DIFCO | 275730 | |
| NucleoSpin Plasmid, Mini kit for plasmid DNA | Takara Bio USA, Inc., MACHEREY-NAGEL | 740588. 250 | |
| PCR tube racks | AXYGEN | R96PCRFSP | It can be replaced for another brand |
| PCR tubes (0.2 mL) | AXYGEN | PCR-02-C | It can be replaced for another brand |
| Rifampicin | Fisher Scientific | 50-164-7517 | |
| Set of micropipettes that dispense between 1 - 10 μL (P10), 2–20 μL (P20), 20–200 μL (P200) and 200–1000 μL (P1000) | RAININ | 17008648; 17008650; 17008652; 17008653 | Micropipettes should be calibrated; can be replaced for another brand |
| Sodium chloride | Fisher Scientific | S671-3 | It can be replaced for another brand |
| Spectrophotometer | Thermo Scientific | 335905P | It can be replaced for another brand |
| Sterilized tips for 10 μL, 200 μL and 1000 μL | Eclipse | 1011-260-000-9; 1018-260-000; 1019-260-000-9 | It can be replaced for another brand |
| Streptomycin | Fisher Scientific | BP910-50 | |
| SYBR Safe DNA gel stain | Thermo Fisher Scientific | S33102 | |
| Taq DNA Polymerase | BioLabs | M0273S | |
| Thermal cycler C1000 Touch | Bio-Rad | 1851196 | It can be replaced for another brand |
| Tris | Fisher Scientific | BP153-500 | It can be replaced for another brand |
参考文献
- Lederberg, J., Tatum, E. Gene recombination in Escherichia coli. Nature. 158 (4016), 558 (1946).
- de la Cruz, F., Frost, L. S., Meyer, R. J., Zechner, E. L. Conjugative DNA metabolism in Gram-negative bacteria. FEMS Microbiology Reviews. 34 (1), 18-40 (2010).
- Grohmann, E., Muth, G., Espinosa, M. Conjugative plasmid transfer in gram-positive bacteria. Microbiology and Molecular Biology Reviews. 67 (2), 277-301 (2003).
- Koraimann, G., Wagner, M. A. Social behavior and decision making in bacterial conjugation. Frontiers in Cellular and Infection Microbiology. 4, 54 (2014).
- Dziewit, L., et al. Diversity and role of plasmids in adaptation of bacteria inhabiting the Lubin copper mine in Poland, an environment rich in heavy metals. Frontiers in Microbiology. 6, 152 (2015).
- Smillie, C., Garcillán-Barcia, M. P., Francia, M. V., Rocha, E. P., de la Cruz, F. Mobility of plasmids. Microbiology and Molecular Biology Reviews. 74 (3), 434-452 (2010).
- Balbuena-Alonso, M. G., et al. Genomic analysis of plasmid content in food isolates of E. coli strongly supports its role as a reservoir for the horizontal transfer of virulence and antibiotic resistance genes. Plasmid. 123-124, 102650 (2022).
- San Millan, A., MacLean, R. C. Fitness costs of plasmids: A limit to plasmid transmission. Microbiol Spectrum. 5 (5), (2017).
- Coluzzi, C., Garcillán-Barcia, M. P., de la Cruz, F., Rocha, E. P. C. Evolution of plasmid mobility: Origin and fate of conjugative and nonconjugative plasmids. Molecular Biology and Evolution. 39 (6), 1-23 (2022).
- Bevan, E. R., et al. Molecular characterization of plasmids encoding blaCTX-M from faecal Escherichia coli in travellers returning to the UK from South Asia. The Journal of Hospital Infection. 114, 134-143 (2021).
- Minja, C. A., Shirima, G., Mshana, S. E. Conjugative plasmids disseminating CTX-M-15 among human, animals and the environment in Mwanza Tanzania: a need to intensify one health approach. Antibiotics. 10 (7), 836 (2021).
- Carattoli, A. Plasmids and the spread of resistance. International Journal of Medical Microbiology. 303 (6-7), 298-304 (2013).
- Sengupta, M., Austin, S. Prevalence and significance of plasmid maintenance functions in the virulence plasmids of pathogenic bacteria. Infection and Immunity. 79 (7), 2502-2509 (2011).
- Alderliesten, J. B., et al. Effect of donor-recipient relatedness on the plasmid conjugation frequency: a meta-analysis. BMC Microbiology. 20 (1), 135 (2020).
- Neil, K., Allard, N., Rodrigue, S. Molecular mechanisms influencing bacterial conjugation in the intestinal microbiota. Frontiers in Microbiology. 12, 673260 (2021).
- Liu, G., Bogaj, K., Bortolaia, V., Olsen, J. E., Thomsen, L. E. Antibiotic-induced, increased conjugative transfer is common to diverse naturally occurring ESBL plasmids in Escherichia coli. Frontiers in Microbiology. 10, 2119 (2019).
- Suchman, E. Polymerase chain reaction protocol. American Society for Microbiology. , 1-14 (2011).
- Watanabe, T. Infective heredity of multiple drug resistance in bacteria. Bacteriological Reviews. 27 (1), 87-115 (1963).
- Simonsen, L., Gordon, D. M., Stewart, F. M., Levin, B. R. Estimating the rate of plasmid transfer: an end-point method. Journal of General Microbiology. 136 (11), 2319-2325 (1990).
- Huisman, J. S., et al. Estimating plasmid conjugation rates: A new computational tool and a critical comparison of methods. Plasmid. 121, 102627 (2022).
- Jung, B., Hoilat, G. J. MacConkey medium. StatPearls. , (2022).
- . NucleoSpin Plasmid DNA purification Available from: https://www.mn-net.com/media/pdf/45/51/02/Instruction-NucleoSpin-Plasmid.pdf (2022)
- Jiang, X., et al. Detection of extended-spectrum beta-lactamases in clinical isolates of Pseudomonas aeruginosa. Antimicrobial Agents and Chemotherapy. 50 (9), 2990-2995 (2006).
- Stapleton, P. D., Shannon, K. P., French, G. L. Carbapenem resistance in Escherichia coli associated with plasmid-determined CMY-4 beta-lactamase production and loss of an outer membrane protein. Antimicrobial Agents and Chemotherapy. 43 (5), 1206-1210 (1999).
- Ben Yahia, H., et al. Detection of CTX-M-15 harboring Escherichia coli isolated from wild birds in Tunisia. BMC Microbiology. 18 (1), 26 (2018).
- Madsen, L., Aarestrup, F. M., Olsen, J. E. Characterisation of streptomycin resistance determinants in Danish isolates of Salmonella Typhimurium. Veterinary Microbiology. 75 (1), 73-82 (2000).
- Perreten, V., Boerlin, P. A new sulfonamide resistance gene (sul3) in Escherichia coli is widespread in the pig population of Switzerland. Antimicrobial Agents and Chemotherapy. 47 (3), 1169-1172 (2003).
- Guardabassi, L., Dijkshoorn, L., Collard, J. M., Olsen, J. E., Dalsgaard, A. Distribution and in-vitro transfer of tetracycline resistance determinants in clinical and aquatic Acinetobacter strains. Journal of Medical Microbiology. 49 (10), 929-936 (2000).
- Carattoli, A., et al. Identification of plasmids by PCR-based replicon typing. Journal of Microbiological Methods. 63 (3), 219-228 (2005).
- Green, M. R., Sambrook, J. Agarose gel electrophoresis. Cold Spring Harbor protocols. 2019 (1), (2019).
- Singer, V. L., Lawlor, T. E., Yue, S. Comparison of SYBR Green I nucleic acid gel stain mutagenicity and ethidium bromide mutagenicity in the Salmonella/mammalian microsome reverse mutation assay (Ames test). Mutation Research. 439 (1), 37-47 (1999).
- Tuma, R. S., et al. Characterization of SYBR Gold nucleic acid gel stain: a dye optimized for use with 300-nm ultraviolet transilluminators. Analytical Biochemistry. 268 (2), 278-288 (1999).
- Oatey, P. Imaging fluorescently stained DNA with CCD technology How to increase sensitivity and reduce integration times. Biotechniques. 42 (3), 376-377 (2007).
- Norman, A., Hansen, L. H., Sørensen, S. J. Conjugative plasmids: vessels of the communal gene pool. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 364 (1527), 2275-2289 (2009).
- Du, L., Liu, R. H., Ying, L., Zhao, G. R. An efficient intergeneric conjugation of DNA from Escherichia coli to mycelia of the lincomycin-producer Streptomyces lincolnensis. International Journal of Molecular Sciences. 13 (4), 4797-4806 (2012).
- Kirk, J. A., Fagan, R. P. Heat shock increases conjugation efficiency in Clostridium difficile. Anaerobe. 42, 1-5 (2016).
- Esteves, G. M., Pereira, J. A., Azevedo, N. F., Azevedo, A. S., Mendes, L. Friends with benefits: An inside look of periodontal microbes' interactions using fluorescence in situ hybridization-Scoping review. Microorganisms. 9 (7), 1504 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved