このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
AMEBaS:分極単一細胞のレシオメトリック蛍光タイムラプスの自動正中線抽出とバックグラウンドサブトラクション
要約
分極した単一細胞の細胞内動態を解析する現在の方法は、多くの場合、手作業であり、標準化されていません。この原稿では、単一偏光細胞の正中線抽出を自動化し、ユーザーフレンドリーなオンラインインターフェースでタイムラプスからの時空間挙動を定量化するための新しい画像解析パイプラインを紹介します。
要約
細胞極性は、空間的に集中した分子と構造の集合体によって確立される巨視的な現象であり、細胞内レベルでの特殊なドメインの出現で最高潮に達します。これは、細胞分裂、成長、遊走などの主要な生物学的機能の根底にある非対称な形態学的構造の発達に関連しています。さらに、細胞極性の破壊は、癌や胃異形成などの組織関連疾患に関連しています。
個々の偏光細胞における蛍光レポーターの時空間動態を評価する現在の方法では、細胞の長軸に沿って正中線をトレースする手作業が必要になることが多く、時間がかかり、強いバイアスが発生しがちです。さらに、レシオメトリック分析では、2つの蛍光チャンネルを使用してレポーター分子の不均一な分布を補正できますが、バックグラウンド減算手法は恣意的であることが多く、統計的な裏付けがありません。
この論文では、細胞極性(花粉管/根毛の成長と細胞質イオン動態)のモデルを使用して、単一細胞の時空間的挙動を自動化および定量化するための新しい計算パイプラインを紹介します。レシオメトリック画像を処理し、細胞内の動態と増殖の定量的表現を抽出するために、3段階のアルゴリズムが開発されました。最初のステップでは、背景からセルをセグメント化し、ピクセル強度空間の閾値化技術によってバイナリマスクを生成します。2 番目のステップでは、スケルトン化操作によってセルの正中線を通るパスをトレースします。最後に、3番目のステップでは、処理されたデータをレシオメトリックタイムラプスとして提供し、レシオメトリックキモグラフ(すなわち、経時的な1D空間プロファイル)を生成します。成長する花粉管から遺伝子コードされた蛍光レポーターで取得したレシオメトリック画像からのデータを使用して、分析法のベンチマークを行いました。このパイプラインにより、分極された細胞の正中線に沿った時空間動態をより速く、より偏りが少なく、より正確に表現できるため、細胞極性を調べるために利用できる定量的ツールキットが進歩します。AMEBaS Pythonのソースコードは、https://github.com/badain/amebas.git
概要
細胞極性は、空間的に集中した分子と構造の集合体の協調的な作用が、特殊な形態学的細胞内ドメインの確立で最高潮に達する基本的な生物学的プロセスです1。細胞分裂、増殖、遊走はこのような極性部位に依存していますが、その喪失は上皮組織関連疾患の癌と関連しています2。
頂端に増殖する細胞は極性の劇的な例であり、先端の極性部位は通常、細胞外の手がかりに再配向する3。これらには、神経突起、真菌菌糸、根毛、花粉管の発達が含まれ、複数の細胞プロセスが細胞の先端からすねに向かって顕著な違いを示します。特に花粉管では、アクチン重合、小胞輸送、イオン濃度が著しく分極し、先端に焦点を絞った勾配を示す4。花粉管は顕花植物の雄性配偶体であり、単一細胞で知られている最速の成長率の1つで細胞の頂点でのみ成長することにより、精子細胞を胚珠に送達する役割を担っています。カルシウム5(Ca2+)やプロトン6(H+)などのイオンの先端に焦点を絞った勾配は、花粉管の成長を維持する上で主要な役割を果たし、二重受精で最高潮に達する主要な生物学的機能を達成するために不可欠です5,6。したがって、頂端増殖細胞の正中線に沿った時空間動態を定量的に解析する手法は、分極増殖の根底にある細胞および分子メカニズムを調査するために不可欠です7,8,9。研究者は、細胞の正中線(列など)のピクセル強度を経時的(行など)で表すマトリックスであるキモグラフを使用することが多く、対角線上の細胞の成長と移動を視覚化できます(図1)。キモグラフは、その有用性にもかかわらず、正中線を手作業でトレースして抽出されることが多く、バイアスや人為的ミスが発生しやすく、かなり手間がかかります。これは、AMEBaSと名付けられた本明細書で紹介するパイプラインの最初の特徴である正中線抽出の自動化された方法を必要とします:偏光された単一細胞のレシオメトリック蛍光タイムラプスのユートマティックMidline EエクストラクションおよびBackground Sウブトラクション。
実験手順に関しては、単一細胞における目的のイオン/分子/種の定量的イメージングは、遺伝的にコードされた蛍光プローブ10を用いて達成することができる。拡大し続ける選択肢の中で、レシオメトリックプローブは、目的の分子に結合/非結合するときに異なる蛍光波長を発するため、最も正確なプローブの1つです11。これにより、2つのチャンネルの比からチャンネル特異的なバックグラウンドを差し引いた値を使用することで、プローブの細胞内濃度の空間的不均一性を補正することができます。しかし、各チャンネルや時点の背景閾値を推定することは、画像の隅が中心に対して光度が変動するシェーディングなどの効果や、蛍光色素の退色(光退色)による時間的に変化することが多いため、複雑な作業になる可能性があります12。複数の方法が考えられるが、本稿では、Isodataアルゴリズム13で得られたセグメンテーション閾値を用いてバックグラウンド強度を自動的に決定し、これを標準として多項式回帰によってフレーム間で平滑化することを提案している。しかし、12で除去した標的細胞とは無関係な蛍光の不均一性に由来する空間成分は、この方法では無視された。自動閾値はいくつかの方法で実行できますが、Isodataアルゴリズムは経験的に最良の結果を生み出しました。したがって、自動バックグラウンド値減算とレシオメトリック計算は、AMEBaS(図1)の2番目の主な機能であり、これらをまとめると、デュアルチャンネル蛍光顕微鏡画像のスタックを入力として受け取り、細胞の正中線とチャンネル固有のバックグラウンドを推定し、バックグラウンド減算、平滑化、および外れ値除去後に両方のチャンネルとその比率(メイン出力#1)のキモグラフを出力します。 レシオメトリック画像のスタック(メイン出力#2)と一緒に。
AMEBaSは、花粉特異的LAT52プロモーター下で発現したCa2+ (CaMeleon)8 またはpH(pH)6 レシオメトリックセンサーを用いて、顕微鏡下で得られた成長中のシロイヌナズナ花粉管の蛍光タイムラプスで試験されました。倒立顕微鏡、表面照射型カメラ(2560画素×2160画素、画素サイズ6.45μm)、蛍光イルミネーター、水浸対物レンズ63倍、1.2NAを組み合わせて、各チャンネルの画像を4秒ごとに撮影した。CaMeleonのフィルター設定は、励起426-450 nm(CFP)および505-515 nm(YFP)、発光458-487 nm(CFP)および520-550 nm(YFP)であり、pHluorinでは励起318-390 nm(DAPI)および428-475 nm(FITC)、発光435-448 nm(DAPI)および523-536 nm(FITC)であった。Zenodoでテストするための完全なデータセットが追加されました(DOI:10.5281/zenodo.7975350)14。
さらに、パイプラインは、UBQ10プロモーター17の制御下で遺伝的にコードされたCa2+レポーターNES-YC3.6を発現するシロイヌナズナの根毛を用いて、前述のようにライトシート顕微鏡(SPIM)でイメージングを行った15,16。ライトシート顕微鏡のカメラ取得、サンプル変換、シャッターを制御する自作のLabViewソフトウェアにより、2つのcpVenusチャンネルとCFPチャンネルの観察だけでなく、それらの比率をリアルタイムで視覚化することもできました。タイムラプスの各比率画像は、3 μm間隔のサンプルの15スライスから得られたcpVenus蛍光チャンネル画像とCFP蛍光チャンネル画像の間の最大強度投影(MIP)を表しています。MIPのタイムラプスcpVenus/CFP比を保存し、AMEBaS解析に直接使用しました。
このパイプラインは、複数の種類の増殖細胞と遊走細胞を扱うことができますが、花粉管、根毛、真菌菌糸など、フレーム間に成長しない細胞質領域が対応している先端のみで成長する成長細胞を分析するために特別に設計されました。このような対応が存在しない場合、ユーザーはステップ 1.3.1.1 で complete_skeletonization オプションを選択する必要があります (詳細については、「ディスカッション」セクションを参照してください)。
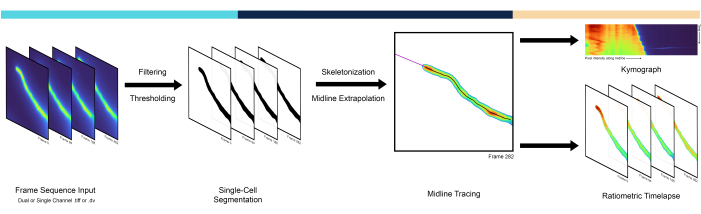
図 1: パイプライン ワークフローの概要。 AMEBaSパイプラインは、シングルセルセグメンテーション、ミッドライントレーシング、キモグラフ生成の3つの主要なステップで、顕微鏡のタイムラプスを解析および処理します。 この図の拡大版をご覧になるには、ここをクリックしてください。
プロトコル
1.インタラクティブノートブックプロトコル
Jupyter Notebook は、以下の手順の基になっている Google Colab at https://colab.research.google.com/github/badain/amebas/blob/main/AMEBAS_Colab.ipynb を使用して、Web 上で直接使用できます。または、Jupyter Notebook は https://github.com/badain/amebas で入手でき、ダウンロードして Jupyter でローカルに実行するように構成できます (Anaconda は簡単でクロスプラットフォームのインストールプロセスを提供できます)。完全な試験データはZenodo(https://doi.org/10.5281/zenodo.7975350)にあり、pHまたはCa2+ レポーターのいずれかを発現するシロイヌナズナ花粉管のシングルチャンネルおよびデュアルチャンネルデータが含まれています14。パイプラインはいくつかの部分に分かれており、ユーザー固有のオプションを設定した後、再生ボタンをクリックすることですべてのステップを実行できます。本研究に必要なファイルは、AMEBaS-のメインzipフォルダ(Supplementary Coding File 1)にあります。
- Jupyter Notebookを開き、タイムラプスファイルを読み取ります。
- 上記の Google Colab のインタラクティブ ノートブックのホームページに移動するか、GitHub から AMEBaS_Local.ipynb ノートブックをダウンロードして開きます。
- 入力データと出力データのディレクトリ設定を準備します。
- ローカルバージョンを使用する場合は、蛍光タイムラプスをTIFFファイルまたはDVファイルとして、プログラムのルートフォルダーにある dataという名前のフォルダー 内に配置します。生成されたデータを受け取るには、 out という名前のフォルダーを作成する必要があります。次に、セットアップ コード ブロックを実行します。
- Google Colabでノートブックを使用している場合は、 セットアップ コードブロックを実行して、 データ と 出力 フォルダーを自動的に生成します。
- [ファイル入力] コード ブロックを実行し、再生ボタンをクリックしてタイム ラプス データを読み取ります。ノートブックのGoogle Colabバージョンを使用している場合は、[ファイルの選択]ボタンをクリックして、タイムラプスファイルをデータフォルダーに直接アップロードします。
注:チャンネル数は、画像のサイズに基づいて自動的に検出されます。 - 'verbose' パラメーターを True または False に設定して、各ステップの追加出力を生成するかどうかを選択します。
- メインセルとセグメントをバックグラウンドから検出します(図2)。
- [Single Cell Segmentation] コード ブロックを実行し、再生ボタンをクリックして、対象のセルを背景から自動的に分離します。
注: メディアン フィルターとガウス フィルターは、不要なノイズを除去するための前処理ステップとして適用され、Isodata しきい値処理によって前景を背景からセグメント化し、最大領域の領域を分離して不要なアーティファクトを除去します。- 'sigma' 変数のガウスによって使用されるシグマ値を調整して、セグメンテーション マスクの滑らかさを微調整します。デフォルト値は 2.0 です。
- 変数 estimate を False に設定して Isodata から推定されたしきい値を直接 保存するか、 True に設定して局所多項式回帰 (LOESS) を使用して隣接するフレーム間で平滑化します。 n_points 変数を変更して、その機能を微調整します。デフォルト値は 40 です。
- [Single Cell Segmentation] コード ブロックを実行し、再生ボタンをクリックして、対象のセルを背景から自動的に分離します。
- セルの延長に沿って正中線をトレースします(図3)。
- 再生ボタンをクリックして [Cell Midline Tracing] コード ブロックを実行し、Lee の方法18 を使用してセルを自動的にスケルトン化し、線形外挿によって最後のスケルトンの先端を拡張します。
- 最後のフレームでのみ正中線をトレースするか、 complete_skeletonization 引数を調整してフレームごとに 1 回トレースするかを選択します。
注: すべてのフレームがスケルトン化されると、外挿はスキップされます。 - interpolation_fraction変数を調整して、外挿中に補間されるスケルトンのポイントの割合を設定します。デフォルト値は 0.25 です。
- 変数 extrapolation_length を変更して、正中線外挿の長さを選択します。既定値は -1 で、スケルトンが最も近いエッジまで延長されます。
- 最後のフレームでのみ正中線をトレースするか、 complete_skeletonization 引数を調整してフレームごとに 1 回トレースするかを選択します。
- 再生ボタンをクリックして [Cell Midline Tracing] コード ブロックを実行し、Lee の方法18 を使用してセルを自動的にスケルトン化し、線形外挿によって最後のスケルトンの先端を拡張します。
- 各チャンネルのキモグラフを生成します( 図4)。
- 再生ボタンをクリックして最初の Data Visualization コードブロックを実行し、両方のチャンネルのキモグラフを自動的に生成します。
- 変数 kymograph_kernel を調整して、平滑化に使用するガウス カーネルのサイズを選択します。
注: これは、ピクセル強度が平均化される近傍のサイズ (ピクセル単位) に対応します。既定値は 3 ピクセル x 3 ピクセルです。 - 非拡張スケルトンは、強度を適切に表示するためにカスタムカラーマップを使用する必要があるキャップ付きキモグラフを生成します。背景色である黒に割り当てる強度の割合を選択し、 shift_fraction 変数を調整します。デフォルト値は 0.7 です。
- 変数 kymograph_kernel を調整して、平滑化に使用するガウス カーネルのサイズを選択します。
- 再生ボタンをクリックして最初の Data Visualization コードブロックを実行し、両方のチャンネルのキモグラフを自動的に生成します。
- チャネル間の比率を計算します(図5)。
- 再生ボタンをクリックして 2 つ目の Data Visualization コード ブロックを実行すると、レシオメトリック キモグラフとレシオメトリック タイムラプスが自動的に生成されます(図 6)。
注意: この手順は、デュアルチャンネルタイムラプスを使用している場合にのみ使用できます。ステップ1.2.1.2で保存したバックグラウンド強度の閾値は、各チャンネルから差し引かれます。- switch_ratio変数を調整して、比率計算時に分子と分母として使用されるチャネルの順序を切り替えます。既定値は False です。
- smooth_ratio変数を調整して、中央値フィルターパスでタイムラプス比をさらに平滑化する必要があるかどうかを選択します。既定値は False です。
- 分母チャネルの低信号によって生成される外れ値を、 reject_outliers 変数を操作して削除するかどうかを選択します。既定値は True で、外れ値を第 3 四分位数 (値の 75% がある) より上の四分位範囲の 1.5 倍の値として定義します。
- レシオメトリック出力の背景を変数 background_ratioを調整してエクスポートする必要があるかどうかを選択します。既定値は False で、0 に置き換えられます。
- 再生ボタンをクリックして 2 つ目の Data Visualization コード ブロックを実行すると、レシオメトリック キモグラフとレシオメトリック タイムラプスが自動的に生成されます(図 6)。

図2:シングルセルセグメンテーションステップ。 フィルタリング、閾値処理、エリアラベリングなどの画像処理技術を使用して、目的の信号を分離します(ステップ1.2)。この特定のデータには、最低強度:2556、中央値:3441、および最高強度:32125の値がありました。 この図の拡大版をご覧になるには、ここをクリックしてください。

図3:正中線追跡の概要-単一細胞の正中線は、その骨格(白)を計算することによって得られます。先端(マゼンタ)は、スケルトンの終端にある最後の点から線形に外挿されます(ステップ1.3)。この構図では、正中線とその先端の両方が元のセルに重ね合わされています。この図の拡大版をご覧になるには、ここをクリックしてください。
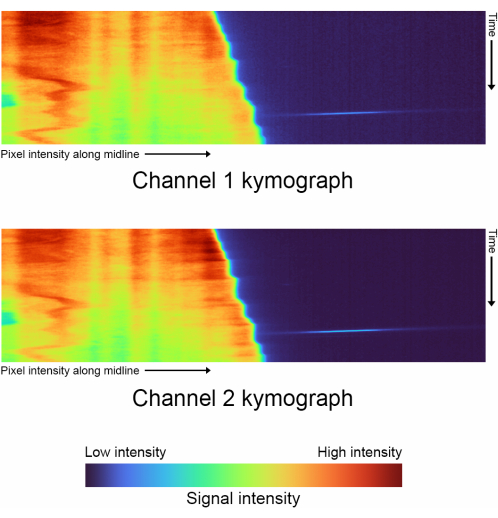
図4:タイムラプスのキモグラフ - 「complete_skeletonization」をオフにした状態で生成された各チャンネルのキモグラフの比較(ステップ1.4)。縦軸は時間の進行を表し、横軸は外挿された正中線経路とそれに続く単一のセルの平均強度をプロットします。この特定のデータでは、カラーマップはチャネル 1 の最低強度: 2886、中央値: 3167、最高強度: 21021 の値を表します。チャンネル2の最低輝度:3030、中央値:3400、最高輝度:29688。 この図の拡大版をご覧になるには、ここをクリックしてください。

図5:バックグラウンド閾値平滑化 - バックグラウンドセグメンテーション閾値は、アイソデータアルゴリズムによって推定され、局所多項式回帰によって平滑化されます(ステップ1.5)。 この図の拡大版をご覧になるには、ここをクリックしてください。
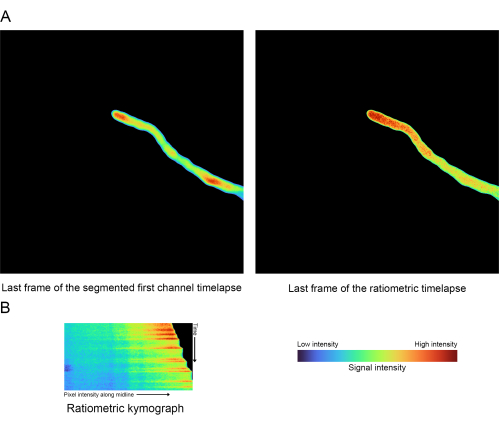
図6:レシオメトリックの結果-(A)レシオメトリックタイムラプスの最後のフレームとセグメント化された元の最初のチャネルとの比較。(B)レシオメトリックタイムラプスから生成されたキモグラフ(ステップ1.5)。この図の拡大版をご覧になるには、ここをクリックしてください。
2.バッチモードプロトコル
- ファイル pipeline.py をAMEBaS GitHubリポジトリーからダウンロードし、データと同じディレクトリに配置します。
- コマンドラインでプログラムファイルの後にファイルの場所を入力します。
- 必要に応じて、パイプラインの内部ステップを示す位置引数として - -v を含めます。
- セル セグメンテーションの準備としてガウス フィルターの前処理手順で使用されるシグマ値に - -s を含めます。既定値は 2 です。
- タイムラプスの各フレームの正中線をトレースするには、- -a を含めます。既定では、パイプラインは最後のフレームのみを使用します。
- - -f を含めると、補間に使用するスケルトンの分数 [0,1] が選択されます。デフォルト値は 0.25 です。
- -e を含めると、外挿されたスケルトンの長さをピクセル単位で選択できます。既定値は -1 で、スケルトンが最も近いエッジまで延長されます。
- - -sf を含めると、外挿されていないキモグラフで背景にシフトされる色範囲の割合を選択できます。デフォルト値は 0.7 です。
- --k を含めて、キモグラフのガウス フィルタリングで使用されるカーネルのサイズを決定します。既定値は 3 です。
- フレーム固有のバックグラウンド閾値強度の LOESS 多項式回帰を介してグローバル背景閾値強度を推定するには、- -eb を含めます。
- パラメータ - -n を変更して、背景しきい値の LOESS スムージングで使用されるポイント数をカスタマイズします。デフォルト値は 40 です。
- タイムラプスにチャンネルが2つある場合は、 -r または- -switch_ratioなど、比率計算時に分子と分母として使用するチャンネルを切り替えます。デフォルトでは、2番目のチャネルが分子で、1番目のチャネルが分母です。
- - -sm 引数を指定したメディアンフィルターパスでタイムラプス比をさらに平滑化する必要があるかどうかを選択します。既定値は False です。
- レシオメトリックタイムラプス生成中に異常な強度を持つピクセルを拒否するには、 -o を含めます。
- レシオメトリック出力の背景を引数 - -b を使用してエクスポートする必要があるかどうかを選択します。既定値は False で、0 に置き換えられます。
- Enter キーを押して実行します。出力は、プログラムファイルと同じディレクトリに生成されます。
結果
AMEBaSパイプラインは、蛍光顕微鏡の画像スタックから偏光した単一細胞の正中線ダイナミクスの抽出を自動化するため、時間がかからず、人為的ミスも発生しにくくなります。この方法では、増殖中の単一細胞でキモグラフとレシオメトリック画像スタック(図1)を生成することにより、これらの時間経過を定量化します。単一細胞の遊走に作用するように調整すること?...
ディスカッション
ここで紹介する新しい方法は、偏光細胞の蛍光顕微鏡画像スタックの解析を合理化および自動化するための強力なツールです。ImageJ Kymographプラグインなど、文献に記載されている現在の方法では、対象の偏光細胞の正中線を手動でトレースする必要がありますが、この作業は時間がかかるだけでなく、人為的ミスも発生しやすい作業です。このパイプラインにおける正中線の定義は、スケル...
開示事項
この原稿の著者は、競合する金銭的利益やその他の利益相反がないことを宣言します。
謝辞
著者らは、FAPESP助成金2015/22308-2、2019/23343-7、2019/26129-6、2020/06744-5、2021/05363-0、CNPq、NIH R01助成金GM131043、およびNSF助成金MCB1714993、MCB1930165の財政的支援に感謝します。根毛データは、Andrea Bassi教授とAlex Costa教授の指導の下、インフラストラクチャを使用して作成されました。
資料
| Name | Company | Catalog Number | Comments |
| Github | Github | https://github.com/badain/amebas | |
| Google Colab | https://colab.research.google.com/github/badain/amebas/blob/main/AMEBAS_Colab.ipynb |
参考文献
- Drubin, D. G., Nelson, W. J. Origins of cell polarity. Cell. 84 (3), 335-344 (1996).
- Wodarz, A., Näthke, I. Cell polarity in development and cancer. Nature Cell Biology. 9 (9), 1016-1024 (2007).
- Palanivelu, R., Preuss, D. Pollen tube targeting and axon guidance: parallels in tip growth mechanisms. Trends in Cell Biology. 10 (12), 517-524 (2000).
- Portes, M. T., et al. . The Pollen Tube Oscillator: Integrating Biophysics and Biochemistry into Cellular Growth and Morphogenesis. Rhythms in Plants: Dynamic Responses in a Dynamic Environment. , (2015).
- Wudick, M. M., et al. CORNICHON sorting and regulation of GLR channels underlie pollen tube Ca2+ homeostasis. Science. 360 (6388), 533-536 (2018).
- Hoffmann, R. D., et al. Plasma membrane H+-ATPases sustain pollen tube growth and fertilization. Nature Communications. 11 (1), 1-15 (2020).
- Michard, E., et al. Glutamate receptor-like genes form Ca2+ channels in pollen tubes and are regulated by pistil D-serine. Science. 332 (6028), 434-437 (2011).
- Damineli, D. S., Portes, M. T., Feijó, J. A. Oscillatory signatures underlie growth regimes in Arabidopsis pollen tubes: computational methods to estimate tip location, periodicity, and synchronization in growing cells. Journal of Experimental Botany. 68 (12), 3267-3281 (2017).
- Li, K., et al. An optimized genetically encoded dual reporter for simultaneous ratio imaging of Ca2+ and H+ reveals new insights into ion signaling in plants. New Phytologist. 230 (6), 2292-2310 (2021).
- Sadoine, M., et al. Designs, applications, and limitations of genetically encoded fluorescent sensors to explore plant biology. Plant Physiology. 187 (2), 485-503 (2021).
- Grenzi, M., et al. Illuminating the hidden world of calcium ions in plants with a universe of indicators. Plant Physiology. 187 (2), 550-571 (2021).
- Munglani, G., Vogler, H., Grossniklaus, U. Fast and flexible processing of large FRET image stacks using the FRET-IBRA toolkit. PLoS Computational Biology. 18 (4), 1009242 (2022).
- Ridler, T., Calvard, S. Picture thresholding using an iterative selection method. IEEE Transactions on Systems, Man, and Cybernetics. 8 (8), 630-632 (1978).
- Portes, M. T., Feijó, J. . Growing Arabidopsis pollen tubes expressing genetically encoded reporters for calcium and pH. , (2023).
- Candeo, A., Doccula, F. G., Valentini, G., Bassi, A., Costa, A. Light sheet fluorescence microscopy quantifies calcium oscillations in root hairs of Arabidopsis thaliana. Plant & Cell Physiology. 58 (7), 1161-1172 (2017).
- Romano Armada, N., et al. In vivo light sheet fluorescence microscopy of calcium oscillations in Arabidopsis thaliana. Methods in Molecular Biology. 1925, 87-101 (2019).
- Krebs, M., et al. FRET-based genetically encoded sensors allow high-resolution live cell imaging of Ca2+ dynamics. The Plant Journal. 69 (1), 181-192 (2012).
- Lee, T. -. C., Kashyap, R. L., Chu, C. -. N. Building skeleton models via 3-D medial surface axis thinning algorithms. CVGIP: Graphical Models and Image Processing. 56 (6), 462-478 (1994).
- Nunez-Iglesias, J., Blanch, A. J., Looker, O., Dixon, M. W., Tilley, L. A new Python library to analyse skeleton images confirms malaria parasite remodelling of the red blood cell membrane skeleton. PeerJ. 6, 4312 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved