Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
AMEBaS : Extraction automatique de la ligne médiane et soustraction de l’arrière-plan des time-lapses de fluorescence ratiométrique de cellules uniques polarisées
Dans cet article
Résumé
Les méthodes actuelles d’analyse de la dynamique intracellulaire des cellules uniques polarisées sont souvent manuelles et manquent de standardisation. Ce manuscrit présente un nouveau pipeline d’analyse d’images pour automatiser l’extraction médiane de cellules polarisées uniques et quantifier le comportement spatio-temporel à partir de timelapses dans une interface en ligne conviviale.
Résumé
La polarité cellulaire est un phénomène macroscopique établi par un ensemble de molécules et de structures concentrées dans l’espace qui aboutissent à l’émergence de domaines spécialisés au niveau subcellulaire. Il est associé au développement de structures morphologiques asymétriques qui sous-tendent des fonctions biologiques clés telles que la division, la croissance et la migration cellulaires. De plus, la perturbation de la polarité cellulaire a été liée à des troubles tissulaires tels que le cancer et la dysplasie gastrique.
Les méthodes actuelles d’évaluation de la dynamique spatio-temporelle des rapporteurs fluorescents dans les cellules polarisées individuelles impliquent souvent des étapes manuelles pour tracer une ligne médiane le long de l’axe principal des cellules, ce qui prend du temps et est sujet à de forts biais. De plus, bien que l’analyse ratiométrique puisse corriger la distribution inégale des molécules rapporteures à l’aide de deux canaux de fluorescence, les techniques de soustraction de fond sont souvent arbitraires et manquent de support statistique.
Ce manuscrit présente un nouveau pipeline de calcul pour automatiser et quantifier le comportement spatio-temporel de cellules individuelles à l’aide d’un modèle de polarité cellulaire : la croissance du tube pollinique et des poils racinaires et la dynamique des ions cytosoliques. Un algorithme en trois étapes a été développé pour traiter des images ratiométriques et extraire une représentation quantitative de la dynamique et de la croissance intracellulaires. La première étape segmente la cellule à partir de l’arrière-plan, produisant un masque binaire grâce à une technique de seuillage dans l’espace d’intensité des pixels. La deuxième étape trace un chemin à travers la ligne médiane de la cellule grâce à une opération de squelettisation. Enfin, la troisième étape fournit les données traitées sous forme de timelapse ratiométrique et produit un kymographe ratiométrique (c’est-à-dire un profil spatial 1D dans le temps). Des données provenant d’images ratiométriques acquises avec des rapporteurs fluorescents génétiquement codés à partir de tubes polliniques en croissance ont été utilisées pour comparer la méthode. Ce pipeline permet une représentation plus rapide, moins biaisée et plus précise de la dynamique spatio-temporelle le long de la ligne médiane des cellules polarisées, faisant ainsi progresser la boîte à outils quantitative disponible pour étudier la polarité cellulaire. Le code source Python d’AMEBaS est disponible à l’adresse suivante : https://github.com/badain/amebas.git
Introduction
La polarité cellulaire est un processus biologique fondamental dans lequel l’action concertée d’un ensemble de molécules et de structures concentrées dans l’espace aboutit à l’établissement de domaines morphologiques subcellulaires spécialisés1. La division, la croissance et la migration cellulaires dépendent de ces sites de polarité, tandis que sa perte a été associée au cancer dans les troubles liés aux tissus épithéliaux2.
Les cellules à croissance apicale sont un exemple dramatique de polarité, où le site de polarité à l’extrémité se réoriente généralement vers des signaux extracellulaires3. Il s’agit notamment des neurites en développement, des hyphes fongiques, des poils racinaires et des tubes polliniques, où de multiples processus cellulaires présentent des différences prononcées de l’extrémité de la cellule vers la tige. Dans les tubes polliniques, en particulier, la polymérisation de l’actine, le trafic des vésicules et les concentrations ioniques sont nettement polarisés, montrant des gradients focalisés sur la pointe4. Les tubes polliniques sont les gamétophytes mâles des plantes à fleurs et sont responsables de l’acheminement des spermatozoïdes vers l’ovule en se développant exclusivement à l’apex de la cellule à l’un des taux de croissance les plus rapides connus pour une seule cellule. Les gradients focalisés sur la pointe des ions tels que le calcium 5 (Ca2+) et les protons 6 (H+) jouent un rôle majeur dans le maintien de la croissance du tube pollinique, ce qui est essentiel pour accomplir sa fonction biologique principale qui aboutit à une double fécondation 5,6. Ainsi, les méthodes quantitatives d’analyse de la dynamique spatio-temporelle le long de la ligne médiane des cellules à croissance apicalle sont essentielles pour étudier les mécanismes cellulaires et moléculaires sous-jacents à la croissance polarisée 7,8,9. Les chercheurs utilisent souvent des kymographes, c’est-à-dire une matrice qui représente l’intensité des pixels de la ligne médiane de la cellule (p. ex., les colonnes) dans le temps (p. ex., les lignes), ce qui permet de visualiser la croissance et la migration des cellules en diagonale (figure 1). Malgré leur utilité, les kymographes sont souvent extraits en traçant manuellement la ligne médiane, étant sujets à des biais et à des erreurs humaines tout en étant plutôt laborieux. Cela nécessite une méthode automatisée d’extraction de la ligne médiane qui est la première caractéristique du pipeline présenté ici et qui est appelée AMEBaS : unextraction utomatique Midline Eet Background Subtraction des timelapses de fluorescence ratiométriques de cellules uniques polarisées.
En termes de procédures expérimentales, l’imagerie quantitative d’ions/molécules/espèces d’intérêt dans des cellules individuelles peut être réalisée avec des sondes fluorescentes génétiquement codées10. Parmi les choix en constante expansion, les sondes ratiométriques sont l’une des plus précises car elles émettent différentes longueurs d’onde de fluorescence lorsqu’elles sont liées/non liées aux molécules d’intérêt11. Cela permet de corriger l’hétérogénéité spatiale de la concentration intracellulaire de la sonde en utilisant le rapport de deux canaux avec leur bruit de fond spécifique au canal soustrait. Cependant, l’estimation du seuil d’arrière-plan pour chaque canal et point temporel peut être une tâche complexe car il varie souvent dans l’espace en raison d’effets tels que l’ombrage, où les coins de l’image ont une variation de luminosité par rapport au centre, et dans le temps en raison de la décoloration du fluorophore (photoblanchiment)12. Bien qu’il existe plusieurs méthodes possibles, ce manuscrit propose de déterminer automatiquement l’intensité du bruit de fond à l’aide du seuil de segmentation obtenu avec l’algorithme Isodata13, qui est ensuite lissé à travers les trames par régression polynomiale comme norme. Cependant, les composantes spatiales issues de l’hétérogénéité de fluorescence non liées à la cellule cible éliminée dans12 ont été ignorées par cette méthode. Le seuillage automatique peut être effectué par plusieurs méthodes, mais l’algorithme Isodata a produit les meilleurs résultats empiriques. Ainsi, la soustraction automatique de la valeur de fond et le calcul ratiométrique sont la deuxième caractéristique principale d’AMEBaS (Figure 1), qui, pris ensemble, reçoit en entrée une pile d’images de microscopie à fluorescence à double canal, estime la ligne médiane de la cellule et le fond spécifique au canal, et produit des kymographes des deux canaux et de leur rapport (sortie principale #1) après soustraction de l’arrière-plan, lissage et suppression des valeurs aberrantes, ainsi qu’une pile d’images ratiométriques (sortie principale #2).
AMEBaS a été testé avec des time-lapses de fluorescence de tubes polliniques d’Arabidopsis en croissance obtenus au microscope, soit avec des capteurs ratiométriques Ca2+ (CaMeleon)8 ou pH (pHluorin)6 exprimés sous le promoteur LAT52 spécifique au pollen. Les images de chaque canal ont été prises toutes les 4 s couplées à un microscope inversé, une caméra frontale éclairée (2560 pixels × 2160 pixels, taille de pixel 6,45 μm), un illuminateur à fluorescence et un objectif à immersion dans l’eau 63x, 1,2NA. Les réglages des filtres utilisés pour CaMeleon étaient : excitation 426-450 nm (CFP) et 505-515 nm (YFP), émission 458-487 nm (CFP) et 520-550 nm (YFP), tandis que pour pHluorin, excitation 318-390 nm (DAPI) et 428-475 nm (FITC), émission 435-448 nm (DAPI) et 523-536 nm (FITC). Un ensemble complet de données a été ajouté pour les tests chez Zenodo (DOI : 10.5281/zenodo.7975350)14.
De plus, le pipeline a été testé avec des données sur les poils racinaires, où l’imagerie a été réalisée avec un microscope à feuille de lumière (SPIM) comme décrit précédemment 15,16 avec des poils racinaires d’Arabidopsis exprimant le rapporteur Ca2+ génétiquement codé NES-YC3.6 sous le contrôle du promoteur UBQ1017. Le logiciel maison LabView qui contrôlait l’acquisition de la caméra, la translation de l’échantillon et l’obturateur du microscope à nappe de lumière a permis l’observation des deux canaux cpVenus et CFP, mais aussi la visualisation de leur rapport en temps réel. Chaque image de rapport du time-lapse représentait une projection d’intensité maximale (MIP) entre les images des canaux fluorescents cpVenus et CFP obtenues à partir de 15 tranches de l’échantillon espacées de 3 μm. Le rapport cpVenus/CFP en time-lapse des MIP a été enregistré et directement utilisé pour l’analyse AMEBaS.
Bien que ce pipeline puisse fonctionner avec plusieurs types de cellules en croissance et en migration, il a été spécialement conçu pour analyser les cellules en croissance qui se développent exclusivement à l’extrémité, telles que les tubes polliniques, les poils racinaires et les hyphes fongiques, où il existe une correspondance des régions cytoplasmiques non croissantes entre les cadres. En l’absence d’une telle correspondance, l’utilisateur doit choisir l’option complete_skeletonization à l’étape 1.3.1.1 (voir la section Discussion pour plus de détails).
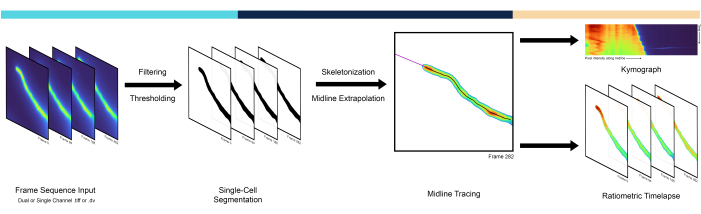
Figure 1 : Vue d’ensemble du flux de travail du pipeline. Le pipeline AMEBaS analyse et traite les timelapses microscopiques en trois étapes principales : la segmentation d’une cellule unique, le traçage de la ligne médiane et la génération de kymographes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Protocole de bloc-notes interactif
Le bloc-notes Jupyter peut être utilisé directement sur le Web à l’aide de Google Colab à https://colab.research.google.com/github/badain/amebas/blob/main/AMEBAS_Colab.ipynb, où les instructions ci-dessous ont été basées. Le notebook Jupyter est également disponible à l’adresse https://github.com/badain/amebas, où il peut être téléchargé et configuré pour s’exécuter localement dans Jupyter (Anaconda peut fournir un processus d’installation simple et multiplateforme). Des données d’essai complètes peuvent être trouvées sur Zenodo (https://doi.org/10.5281/zenodo.7975350), contenant des données à canal unique et double de tubes polliniques d’Arabidopsis exprimant soit le pH, soit les rapporteurs de Ca2+ 14. Le pipeline a été divisé en parties, où chaque étape peut être exécutée en cliquant sur le bouton de lecture après avoir défini les options spécifiques à l’utilisateur. Les fichiers nécessaires à cette étude sont disponibles dans le dossier zip principal d’AMEBaS (Supplementary Coding File 1).
- Ouvrez le bloc-notes Jupyter et lisez les fichiers time-lapse.
- Accédez à la page d’accueil du bloc-notes interactif dans Google Colab mentionné ci-dessus ou téléchargez et ouvrez le bloc-notes AMEBaS_Local.ipynb à partir de GitHub.
- Préparez la configuration du répertoire pour les données d’entrée et de sortie :
- Si vous utilisez la version locale, placez le time-lapse de fluorescence sous forme de fichier TIFF ou de fichier DV dans un dossier nommé data qui doit se trouver dans le dossier racine du programme. Un dossier nommé out doit être créé pour recevoir les données générées. Exécutez ensuite le bloc de code d’installation.
- Si vous utilisez le bloc-notes sur Google Colab, exécutez le bloc de code Setup pour générer automatiquement les dossiers de données et de sortie .
- Exécutez le bloc de code d’entrée de fichier pour lire les données de time-lapse en cliquant sur le bouton de lecture . Si vous utilisez la version Google Colab du bloc-notes, cliquez sur le bouton Choisir un fichier pour télécharger directement le fichier time-lapse dans le dossier de données .
REMARQUE : Le nombre de canaux sera automatiquement détecté en fonction de la dimension de l’image. - Choisissez si des sorties supplémentaires de chaque étape seront générées en définissant le paramètre 'verbose' sur True ou False.
- Détectez la cellule principale et le segment à partir de l’arrière-plan (Figure 2).
- Exécutez le bloc de code Segmentation de cellule unique pour séparer automatiquement la cellule d’intérêt de l’arrière-plan en cliquant sur le bouton de lecture .
REMARQUE : Les filtres médians et gaussiens seront appliqués en tant qu’étape de prétraitement pour supprimer le bruit indésirable avant de segmenter le premier plan de l’arrière-plan par seuillage Isodata et d’isoler la région de la plus grande zone pour supprimer les artefacts indésirables.- Ajustez la valeur sigma utilisée par la gaussienne dans la variable 'sigma' pour affiner le lissage du masque de segmentation. La valeur par défaut est 2.0.
- Définissez l’estimation de la variable sur False pour stocker directement le seuil estimé à partir d’Isodata ou sur True pour le lisser sur les trames voisines à l’aide de la régression polynomiale locale (LOESS). Affinez sa fonction en changeant la variable n_points . La valeur par défaut est 40.
- Exécutez le bloc de code Segmentation de cellule unique pour séparer automatiquement la cellule d’intérêt de l’arrière-plan en cliquant sur le bouton de lecture .
- Tracez la ligne médiane le long de l’extension de la cellule (Figure 3).
- Exécutez le bloc de code Cell Midline Tracing en cliquant sur le bouton de lecture pour squelettiser automatiquement la cellule à l’aide de la méthode18 de Lee et étendre la pointe du dernier squelette par extrapolation linéaire.
- Choisissez de tracer la ligne médiane uniquement sur la dernière image ou une fois par image en ajustant l’argument complete_skeletonization .
REMARQUE : Lorsque toutes les images sont squelettées, l’extrapolation est ignorée. - Définissez la fraction de points du squelette à interpoler lors de l’extrapolation en ajustant la variable interpolation_fraction . La valeur par défaut est 0,25.
- Choisissez la longueur de l’extrapolation médiane en modifiant la variable extrapolation_length. La valeur par défaut est -1, ce qui étend le squelette jusqu’à l’arête la plus proche.
- Choisissez de tracer la ligne médiane uniquement sur la dernière image ou une fois par image en ajustant l’argument complete_skeletonization .
- Exécutez le bloc de code Cell Midline Tracing en cliquant sur le bouton de lecture pour squelettiser automatiquement la cellule à l’aide de la méthode18 de Lee et étendre la pointe du dernier squelette par extrapolation linéaire.
- Générez des kymographes pour chaque canal ( Figure 4).
- Exécutez le premier bloc de code de visualisation de données en cliquant sur le bouton de lecture pour générer automatiquement des kymographes pour les deux canaux.
- Choisissez la taille du noyau gaussien utilisé pour le lissage en ajustant la variable kymograph_kernel.
REMARQUE : Il correspond à la taille du voisinage (en pixels) sur lequel les intensités de pixels sont calculées. La valeur par défaut est de 3 pixels x 3 pixels. - Les squelettes non étendus génèrent des Kymographes plafonnés qui doivent utiliser une palette de couleurs personnalisée pour afficher correctement leurs intensités. Choisissez le pourcentage de fraction des intensités qui sera attribué à la couleur de fond, le noir, en ajustant la variable shift_fraction . La valeur par défaut est 0,7.
- Choisissez la taille du noyau gaussien utilisé pour le lissage en ajustant la variable kymograph_kernel.
- Exécutez le premier bloc de code de visualisation de données en cliquant sur le bouton de lecture pour générer automatiquement des kymographes pour les deux canaux.
- Calculez le rapport entre les canaux (Figure 5).
- Exécutez le deuxième bloc de code de visualisation de données en cliquant sur le bouton de lecture pour générer automatiquement un kymographe ratiométrique et un timelapse ratiométrique (Figure 6).
REMARQUE : Cette étape n’est disponible que lors de l’utilisation de timelapses à double canal. Le seuil d’intensité de fond enregistré à l’étape 1.2.1.2 est soustrait de chaque canal.- Ajustez la variable switch_ratio pour changer l’ordre des canaux utilisés comme numérateur et dénominateur lors des calculs de ratio. La valeur par défaut est False.
- Choisissez si le timelapse de ratio doit être lissé davantage avec une passe de filtre médiane en ajustant la variable smooth_ratio . La valeur par défaut est False.
- Choisissez de supprimer ou non les valeurs aberrantes produites par le signal faible du canal du dénominateur en manipulant la variable reject_outliers . La valeur par défaut est True et définit les valeurs aberrantes comme des valeurs 1,5 fois l’intervalle interquartile au-dessus du troisième quartile (où se trouvent 75 % des valeurs).
- Indiquez si l’arrière-plan de la sortie ratiométrique doit être exporté en ajustant la variable background_ratio. La valeur par défaut est False, ce qui la remplace par des zéros.
- Exécutez le deuxième bloc de code de visualisation de données en cliquant sur le bouton de lecture pour générer automatiquement un kymographe ratiométrique et un timelapse ratiométrique (Figure 6).

Figure 2 : Étape de segmentation d’une seule cellule. Des techniques de traitement d’image telles que le filtrage, le seuillage et l’étiquetage de zone sont utilisées pour isoler le signal d’intérêt (étape 1.2). Ces données particulières avaient les valeurs suivantes pour l’intensité la plus faible : 2556, la médiane : 3441 et l’intensité la plus élevée : 32125. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Vue d’ensemble du tracé de la ligne médiane - La ligne médiane de la cellule unique est obtenue en calculant son squelette (blanc). La pointe (magenta) est extrapolée linéairement à partir des derniers points à l’extrémité du squelette (étape 1.3). Dans cette composition, la ligne médiane et sa pointe sont superposées à la cellule d’origine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
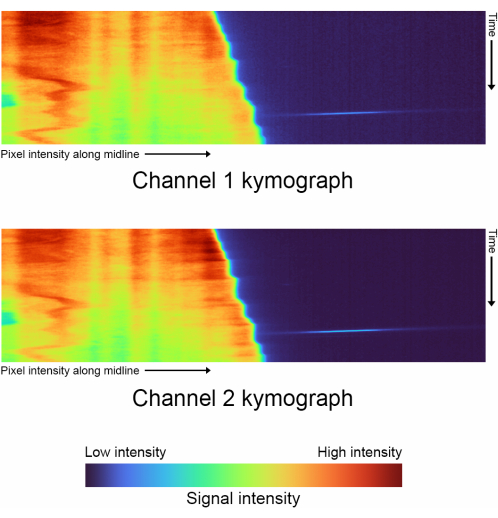
Figure 4 : Kymographes de Timelapse - Comparaison des kymographes pour chaque canal généré avec 'complete_skeletonization' désactivé (étape 1.4). L’axe vertical décrit la progression du temps, et l’axe horizontal représente l’intensité moyenne de la trajectoire extrapolée de la ligne médiane suivie d’une seule cellule. Pour ces données particulières, la palette de couleurs représente les valeurs suivantes pour Canal 1 Intensité la plus basse : 2886, Médiane : 3167, Intensité la plus élevée : 21021. Canal 2 Intensité la plus basse : 3030, Médiane : 3400, Intensité la plus élevée : 29688. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Lissage du seuil d’arrière-plan : le seuil de segmentation de l’arrière-plan est estimé à l’aide de l’algorithme isodata, puis lissé par régression polynomiale locale (étape 1.5). Veuillez cliquer ici pour voir une version agrandie de cette figure.
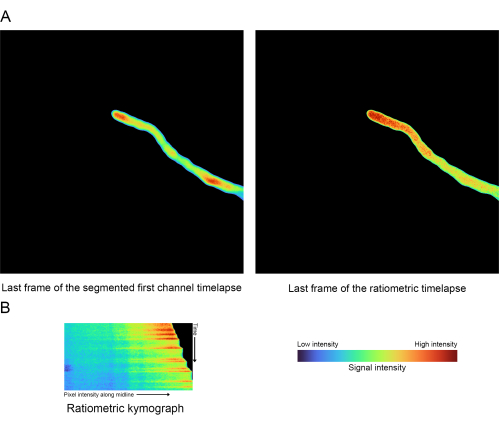
Figure 6 : Résultats ratiométriques - (A) Comparaison entre la dernière image du timelapse ratiométrique et le premier canal d’origine segmenté. (B) Kymographe généré à partir du timelapse ratiométrique (étape 1.5). Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Protocole en mode batch
- Téléchargez et placez le fichier pipeline.py à partir du référentiel GitHub AMEBaS dans le même répertoire que les données.
- Tapez l’emplacement du fichier sur la ligne de commande après le fichier programme.
- Incluez - -v comme argument positionnel pour afficher les étapes internes du pipeline, si vous le souhaitez.
- Incluez l’option --s à choisir dans la valeur sigma utilisée dans l’étape de prétraitement du filtre gaussien en vue de la segmentation des cellules. La valeur par défaut est 2.
- Incluez - -a pour tracer la ligne médiane de chaque image du laps de temps. Par défaut, le pipeline n’utilise que la dernière image.
- Incluez - -f pour choisir la fraction [0,1] du squelette à utiliser dans l’interpolation. La valeur par défaut est 0,25.
- Incluez -e pour choisir la longueur en pixels du squelette extrapolé. La valeur par défaut est -1, ce qui étend le squelette jusqu’à l’arête la plus proche.
- Incluez -- sf pour choisir la fraction de la gamme de couleurs qui sera décalée vers l’arrière-plan dans les kymographes non extrapolés. La valeur par défaut est 0,7.
- Incluez - -k pour déterminer la taille du noyau utilisé dans le filtrage gaussien du kymographe. La valeur par défaut est 3.
- Incluez - -eb pour estimer l’intensité du seuil de bruit de fond global via la régression polynomiale LOESS des intensités de seuil de fond spécifiques à la trame.
- Personnalisez le nombre de points utilisés dans le lissage LOESS des valeurs de seuil d’arrière-plan en modifiant le paramètre - -n. La valeur par défaut est 40.
- Permutez les canaux utilisés comme numérateur et dénominateur lors des calculs de ratio, y compris - r ou - -switch_ratio, si le timelapse comporte deux canaux. Par défaut, le deuxième canal est le numérateur et le premier est le dénominateur.
- Choisissez si le timelapse du rapport doit être lissé davantage avec une passe de filtre médian avec l’argument - -sm . La valeur par défaut est False.
- Incluez -o pour rejeter les pixels avec des intensités anormales lors de la génération ratiométrique en time-lapse .
- Indiquez si l’arrière-plan de la sortie ratiométrique doit être exporté à l’aide de l’argument - -b. La valeur par défaut est False, ce qui la remplace par des zéros.
- Appuyez sur Entrée pour exécuter. La sortie sera générée dans le même répertoire que le fichier programme.
Résultats
Le pipeline AMEBaS automatise l’extraction de la dynamique médiane des cellules uniques polarisées à partir d’empilements d’images de microscopie à fluorescence, ce qui le rend moins chronophage et moins sujet aux erreurs humaines. La méthode quantifie ces laps de temps en générant des kymographes et des empilements d’images ratiométriques (Figure 1) dans des cellules individuelles en croissance. Il peut être ajusté pour travailler sur la migration de cellules individuelles...
Discussion
La nouvelle méthode présentée ici est un outil puissant pour rationaliser et automatiser l’analyse des piles d’images de microscopie à fluorescence de cellules polarisées. Les méthodes actuelles décrites dans la littérature, telles que les plugins ImageJ Kymograph, nécessitent un traçage manuel de la ligne médiane de la cellule polarisée d’intérêt, une tâche qui prend non seulement du temps, mais qui est également sujette à des erreurs humaines. Étant donné que la définition de la ligne médiane...
Déclarations de divulgation
Les auteurs de ce manuscrit ne déclarent aucun intérêt financier concurrent ou autre conflit d’intérêts.
Remerciements
Les auteurs remercient les subventions 2015/22308-2, 2019/23343-7, 2019/26129-6, 2020/06744-5, 2021/05363-0, le CNPq, la subvention NIH R01 GM131043 et les subventions NSF MCB1714993, MCB1930165 pour le soutien financier. Les données sur les poils racinaires ont été produites avec l’infrastructure et sous la supervision du professeur Andrea Bassi et du professeur Alex Costa.
matériels
| Name | Company | Catalog Number | Comments |
| Github | Github | https://github.com/badain/amebas | |
| Google Colab | https://colab.research.google.com/github/badain/amebas/blob/main/AMEBAS_Colab.ipynb |
Références
- Drubin, D. G., Nelson, W. J. Origins of cell polarity. Cell. 84 (3), 335-344 (1996).
- Wodarz, A., Näthke, I. Cell polarity in development and cancer. Nature Cell Biology. 9 (9), 1016-1024 (2007).
- Palanivelu, R., Preuss, D. Pollen tube targeting and axon guidance: parallels in tip growth mechanisms. Trends in Cell Biology. 10 (12), 517-524 (2000).
- Portes, M. T., et al. . The Pollen Tube Oscillator: Integrating Biophysics and Biochemistry into Cellular Growth and Morphogenesis. Rhythms in Plants: Dynamic Responses in a Dynamic Environment. , (2015).
- Wudick, M. M., et al. CORNICHON sorting and regulation of GLR channels underlie pollen tube Ca2+ homeostasis. Science. 360 (6388), 533-536 (2018).
- Hoffmann, R. D., et al. Plasma membrane H+-ATPases sustain pollen tube growth and fertilization. Nature Communications. 11 (1), 1-15 (2020).
- Michard, E., et al. Glutamate receptor-like genes form Ca2+ channels in pollen tubes and are regulated by pistil D-serine. Science. 332 (6028), 434-437 (2011).
- Damineli, D. S., Portes, M. T., Feijó, J. A. Oscillatory signatures underlie growth regimes in Arabidopsis pollen tubes: computational methods to estimate tip location, periodicity, and synchronization in growing cells. Journal of Experimental Botany. 68 (12), 3267-3281 (2017).
- Li, K., et al. An optimized genetically encoded dual reporter for simultaneous ratio imaging of Ca2+ and H+ reveals new insights into ion signaling in plants. New Phytologist. 230 (6), 2292-2310 (2021).
- Sadoine, M., et al. Designs, applications, and limitations of genetically encoded fluorescent sensors to explore plant biology. Plant Physiology. 187 (2), 485-503 (2021).
- Grenzi, M., et al. Illuminating the hidden world of calcium ions in plants with a universe of indicators. Plant Physiology. 187 (2), 550-571 (2021).
- Munglani, G., Vogler, H., Grossniklaus, U. Fast and flexible processing of large FRET image stacks using the FRET-IBRA toolkit. PLoS Computational Biology. 18 (4), 1009242 (2022).
- Ridler, T., Calvard, S. Picture thresholding using an iterative selection method. IEEE Transactions on Systems, Man, and Cybernetics. 8 (8), 630-632 (1978).
- Portes, M. T., Feijó, J. . Growing Arabidopsis pollen tubes expressing genetically encoded reporters for calcium and pH. , (2023).
- Candeo, A., Doccula, F. G., Valentini, G., Bassi, A., Costa, A. Light sheet fluorescence microscopy quantifies calcium oscillations in root hairs of Arabidopsis thaliana. Plant & Cell Physiology. 58 (7), 1161-1172 (2017).
- Romano Armada, N., et al. In vivo light sheet fluorescence microscopy of calcium oscillations in Arabidopsis thaliana. Methods in Molecular Biology. 1925, 87-101 (2019).
- Krebs, M., et al. FRET-based genetically encoded sensors allow high-resolution live cell imaging of Ca2+ dynamics. The Plant Journal. 69 (1), 181-192 (2012).
- Lee, T. -. C., Kashyap, R. L., Chu, C. -. N. Building skeleton models via 3-D medial surface axis thinning algorithms. CVGIP: Graphical Models and Image Processing. 56 (6), 462-478 (1994).
- Nunez-Iglesias, J., Blanch, A. J., Looker, O., Dixon, M. W., Tilley, L. A new Python library to analyse skeleton images confirms malaria parasite remodelling of the red blood cell membrane skeleton. PeerJ. 6, 4312 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon