Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
AMEBaS: Автоматическое выделение срединной линии и вычитание фона ратиометрических флуоресцентных таймлапсов поляризованных одиночных ячеек
В этой статье
Резюме
Современные методы анализа внутриклеточной динамики поляризованных одиночных клеток часто являются ручными и не стандартизированы. В этой рукописи представлен новый конвейер анализа изображений для автоматизации срединного извлечения одиночных поляризованных ячеек и количественной оценки пространственно-временного поведения по временным интервалам в удобном онлайн-интерфейсе.
Аннотация
Полярность клеток — это макроскопическое явление, устанавливаемое совокупностью пространственно концентрированных молекул и структур, кульминацией которых является появление специализированных доменов на субклеточном уровне. Он связан с развитием асимметричных морфологических структур, которые лежат в основе ключевых биологических функций, таких как деление клеток, рост и миграция. Кроме того, нарушение полярности клеток связано с тканевыми расстройствами, такими как рак и дисплазия желудка.
Современные методы оценки пространственно-временной динамики флуоресцентных репортеров в отдельных поляризованных клетках часто включают ручные шаги по трассировке средней линии вдоль главной оси клеток, что отнимает много времени и подвержено сильным смещениям. Кроме того, несмотря на то, что ратиометрический анализ может исправить неравномерное распределение репортерных молекул с помощью двух флуоресцентных каналов, методы вычитания фона часто произвольны и не имеют статистической поддержки.
В данной статье представлен новый вычислительный конвейер для автоматизации и количественной оценки пространственно-временного поведения отдельных клеток с использованием модели полярности клеток: рост пыльцевой трубки/корневых волосков и цитозольная ионная динамика. Был разработан трехступенчатый алгоритм для обработки ратиометрических изображений и извлечения количественного представления о внутриклеточной динамике и росте. На первом шаге ячейка отделяется от фона, создавая двоичную маску с помощью метода порогового значения в пространстве интенсивности пикселей. На втором шаге прослеживается путь по средней линии клетки с помощью операции скелетирования. Наконец, на третьем шаге обработанные данные представлены в виде ратиометрического таймлапса и получен ратиометрический кимограф (т.е. 1D-пространственный профиль во времени). Для тестирования метода были использованы данные ратиометрических изображений, полученных с помощью генетически закодированных флуоресцентных репортеров из растущих пыльцевых трубок. Этот конвейер позволяет быстрее, менее смещенно и более точно представлять пространственно-временную динамику вдоль средней линии поляризованных клеток, тем самым расширяя количественный инструментарий, доступный для исследования полярности клеток. Исходный код AMEBaS Python доступен по адресу: https://github.com/badain/amebas.git
Введение
Полярность клеток является фундаментальным биологическим процессом, в котором согласованное действие набора пространственно концентрированных молекул и структур достигает кульминации в создании специализированных морфологических субклеточныхдоменов. Деление, рост и миграция клеток зависят от таких участков полярности, в то время как их потеря связана с раком при заболеваниях, связанных с эпителиальной тканью2.
Апикально растущие клетки являются ярким примером полярности, когда участок полярности на кончике обычно переориентируется на внеклеточные сигналы3. К ним относятся развивающиеся невриты, грибковые гифы, корневые волоски и пыльцевые трубки, где множественные клеточные процессы демонстрируют выраженные различия от кончика клетки к ножке. В пыльцевых трубках, в частности, полимеризация актина, транспорт везикул и концентрация ионов заметно поляризуются, демонстрируя градиенты, сфокусированные на кончиках4. Пыльцевые трубки являются мужскими гаметофитами цветковых растений и отвечают за доставку сперматозоидов к яйцеклетке, растя исключительно на верхушке клетки с одной из самых быстрых скоростей роста, известных для одной клетки. Градиенты ионов, таких как кальций 5 (Ca2+) и протоны 6 (H+), играют важную роль в поддержании роста пыльцевой трубки, что необходимо для выполнения ее основной биологической функции, кульминацией которой является двойное оплодотворение 5,6. Таким образом, количественные методы анализа пространственно-временной динамики вдоль срединной линии апикально растущих клеток имеют важное значение для изучения клеточных и молекулярных механизмов, лежащих в основе поляризованного роста 7,8,9. Исследователи часто используют кимографы, т.е. матрицы, представляющие интенсивность пикселей средней линии клетки (например, столбцов) во времени (например, строк), что позволяет визуализировать рост и миграцию клеток по диагонали (рис. 1). Несмотря на свою полезность, кимографы часто извлекаются путем ручного отслеживания средней линии, что подвержено предвзятости и человеческим ошибкам, а также довольно трудоемко. Это требует автоматизированного метода выделения срединной линии, который является первой особенностью представленного здесь конвейера под названием AMEBaS: Automatic Midline Extraction and Background Sутракции ратиометрической флуоресценции поляризованных одиночных клеток.
С точки зрения экспериментальных процедур, количественная визуализация интересующих ионов/молекул/видов в отдельных клетках может быть достигнута с помощью генетически кодируемых флуоресцентных зондов10. Среди постоянно расширяющегося выбора ратиометрические зонды являются одними из самых точных, поскольку они излучают различные длины волн флуоресценции при связывании/развязываниис интересующими молекулами. Это позволяет корректировать пространственную неоднородность внутриклеточной концентрации зонда за счет соотношения двух каналов с вычитанием их канального специфического фона. Однако оценка фонового порога для каждого канала и точки времени может быть сложной задачей, поскольку он часто изменяется в пространстве из-за таких эффектов, как затенение, когда углы изображения имеют изменение яркости относительно центра, и во времени из-за затухания флуорофора (фотообесцвечивание)12. Несмотря на то, что существует множество возможных методов, в данной работе предлагается определять интенсивность фона автоматически, используя порог сегментации, полученный с помощью алгоритма Isodata13, который затем сглаживается по кадрам с помощью полиномиальной регрессии в качестве стандарта. Однако пространственные компоненты, проистекающие из флуоресцентной гетерогенности, не связанной с клеткой-мишенью, удаленной в12, были проигнорированы этим методом. Автоматическое пороговое определение может быть выполнено несколькими методами, но алгоритм Isodata дал наилучшие результаты эмпирически. Таким образом, автоматическое вычитание значения фона и ратиометрический расчет являются второй основной особенностью AMEBaS (рис. 1), которая в совокупности получает на вход стопку изображений двухканальной флуоресцентной микроскопии, оценивает срединную линию ячейки и канальный фон, а также выдает кимографы обоих каналов и их соотношение (основной выход #1) после вычитания фона, сглаживания и удаления выбросов. вместе со стопкой ратиометрических изображений (основной выход #2).
AMEBaS был протестирован с помощью флуоресцентных таймлапсов растущих пыльцевых трубок Arabidopsis, полученных под микроскопом, либо с помощью ратиометрических сенсоров Ca2+ (CaMeleon)8 или pH (pHluorin)6 , экспрессированных под пыльцевым промотором LAT52. Изображения с каждого канала делались каждые 4 с в сочетании с инвертированным микроскопом, камерой с фронтальной подсветкой (2560 пикселей × 2160 пикселей, размер пикселя 6,45 мкм), флуоресцентным осветителем и объективом с погружением в воду 63x, 1,2NA. Для CaMeleon использовались следующие настройки фильтров: возбуждение 426-450 нм (CFP) и 505-515 нм (YFP), излучение 458-487 нм (CFP) и 520-550 нм (YFP), в то время как для pHluorin возбуждение 318-390 нм (DAPI) и 428-475 нм (FITC), излучение 435-448 нм (DAPI) и 523-536 нм (FITC). Для тестирования в Zenodo был добавлен полный набор данных (DOI: 10.5281/zenodo.7975350)14.
Кроме того, трубопровод был протестирован с данными корневых волосков, где визуализация была выполнена с помощью светового листового микроскопа (SPIM), как описано ранее15,16, с корневыми волосками арабидопсиса, экспрессирующими генетически кодируемыйCa2+ репортер NES-YC3.6 под контролем промотора UBQ10 17. Самодельное программное обеспечение LabView, которое управляло захватом камеры, перемещением образца и затвором светового микроскопа, позволяло наблюдать два канала cpVenus и CFP, а также визуализировать их соотношение в режиме реального времени. Каждое изображение интервальной съемки представляло собой проекцию максимальной интенсивности (MIP) между изображениями флуоресцентных каналов cpVenus и CFP, полученными из 15 срезов образца, расположенных на расстоянии 3 мкм друг от друга. Интервальная съемка соотношения cpVenus/CFP MIP была сохранена и непосредственно использована для анализа AMEBaS.
Хотя этот конвейер может работать с несколькими типами растущих и мигрирующих клеток, он был специально разработан для анализа растущих клеток, которые растут исключительно на кончике, таких как пыльцевые трубки, корневые волоски и гифы грибов, где существует соответствие нерастущих цитоплазматических областей между рамками. Если такого соответствия нет, пользователь должен выбрать вариант complete_skeletonization на шаге 1.3.1.1 (подробнее см. раздел Обсуждение).
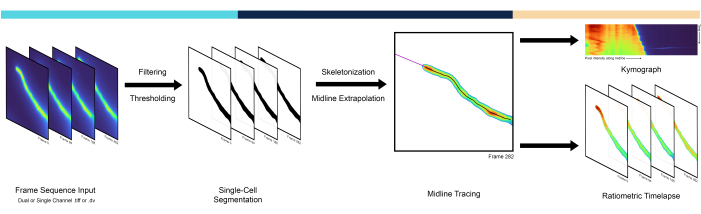
Рисунок 1: Обзор рабочего процесса конвейера. Конвейер AMEBaS анализирует и обрабатывает микроскопические таймлапсы в три основных этапа: сегментация отдельных клеток, трассировка срединных линий и генерация кимографа. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
протокол
1. Протокол интерактивной записной книжки
Записную книжку Jupyter можно использовать непосредственно в Интернете с помощью Google Colab в https://colab.research.google.com/github/badain/amebas/blob/main/AMEBAS_Colab.ipynb, где были основаны приведенные ниже инструкции. Кроме того, записная книжка Jupyter доступна на https://github.com/badain/amebas, где ее можно загрузить и настроить для локального запуска в Jupyter (Anaconda может обеспечить простой и кроссплатформенный процесс установки). Полные данные испытаний можно найти в Zenodo (https://doi.org/10.5281/zenodo.7975350), содержащие одно- и двухканальные данные пыльцевых трубок арабидопсиса, экспрессирующих рН или Ca2+ 14. Конвейер разделен на части, где каждый шаг может быть выполнен нажатием кнопки воспроизведения после установки пользовательских параметров. Необходимые файлы для этого исследования доступны в основной zip-папке AMEBaS (Supplementary Coding File 1).
- Откройте записную книжку Jupyter и прочтите файлы интервальной съемки.
- Перейдите на домашнюю страницу интерактивной записной книжки в Google Colab, упомянутой выше, или загрузите и откройте записную книжку AMEBaS_Local.ipynb с GitHub.
- Подготовьте настройку каталога для входных и выходных данных:
- Если вы используете локальную версию, поместите интервальную съемку флуоресценции в виде файла TIFF или DV в папку с именем data , которая должна находиться в корневой папке программы. Для получения сгенерированных данных необходимо создать папку с именем . Затем запустите блок кода установки.
- Если вы используете блокнот в Google Colab, запустите блок кода установки , чтобы автоматически создать папки данных и исходящих данных.
- Запустите блок кода File Input , чтобы прочитать данные интервальной съемки, нажав кнопку воспроизведения . Если вы используете версию блокнота Google Colab, нажмите кнопку «Выбрать файл», чтобы напрямую загрузить файл замедленной съемки в папку данных .
ПРИМЕЧАНИЕ: Количество каналов будет автоматически определено в зависимости от размера изображения. - Выберите, будут ли создаваться дополнительные выходные данные каждого шага, установив для параметра verbose значение True или False.
- Определите основную ячейку и сегмент на фоне (рисунок 2).
- Запустите блок кода Сегментация по одной ячейке, чтобы автоматически отделить интересующую ячейку от фона, нажав кнопку воспроизведения .
ПРИМЕЧАНИЕ: Медианный и Гауссовы фильтры будут применены в качестве шага предварительной обработки для удаления нежелательного шума перед сегментированием переднего плана от фона с помощью порогового значения Isodata и изоляции области наибольшей области для удаления нежелательных артефактов.- Отрегулируйте значение сигмы, используемое Гауссом, в переменной 'sigma', чтобы точно настроить сглаживание маски сегментации. Значение по умолчанию — 2.0.
- Установите переменную estimate в False , чтобы сохранить пороговое значение, оцененное непосредственно из Isodata, или в True , чтобы сгладить его по соседним фреймам с помощью локальной полиномиальной регрессии (LOESS). Отрегулируйте его функцию, изменив переменную n_points . Значение по умолчанию — 40.
- Запустите блок кода Сегментация по одной ячейке, чтобы автоматически отделить интересующую ячейку от фона, нажав кнопку воспроизведения .
- Проведите срединную линию вдоль расширения ячейки (рис. 3).
- Запустите блок кода Трассировка средней линии ячейки, нажав кнопку воспроизведения, чтобы автоматически скелетизировать ячейку с помощью метода18 Ли и удлинить кончик последнего скелета с помощью линейной экстраполяции.
- Выберите трассировку средней линии только в последнем кадре или один раз за кадр, настроив аргумент complete_skeletonization .
ПРИМЕЧАНИЕ: Когда все кадры скелетизированы, экстраполяция пропускается. - Задайте долю точек в скелете, которые будут интерполированы во время экстраполяции, настроив переменную interpolation_fraction . Значение по умолчанию — 0,25.
- Выберите длину экстраполяции средней линии, изменив переменную extrapolation_length. Значение по умолчанию равно -1, что расширяет скелет до ближайшего края.
- Выберите трассировку средней линии только в последнем кадре или один раз за кадр, настроив аргумент complete_skeletonization .
- Запустите блок кода Трассировка средней линии ячейки, нажав кнопку воспроизведения, чтобы автоматически скелетизировать ячейку с помощью метода18 Ли и удлинить кончик последнего скелета с помощью линейной экстраполяции.
- Сгенерируйте кимограммы для каждого канала ( рисунок 4).
- Запустите первый блок кода визуализации данных , нажав кнопку воспроизведения , чтобы автоматически создать кимограммы для обоих каналов.
- Выберите размер гауссова ядра, используемого для сглаживания, настроив переменную kymograph_kernel.
ПРИМЕЧАНИЕ: Соответствует размеру окрестности (в пикселях), по которой усредняются интенсивности пикселов. Значение по умолчанию — 3 x 3 пикселя. - Непротяженные скелеты генерируют кимографы с ограничениями, которые должны использовать пользовательскую цветовую карту для правильного отображения их интенсивности. Выберите долю интенсивности, которая будет присвоена цвету фона, черному, настроив переменную shift_fraction . Значение по умолчанию — 0,7.
- Выберите размер гауссова ядра, используемого для сглаживания, настроив переменную kymograph_kernel.
- Запустите первый блок кода визуализации данных , нажав кнопку воспроизведения , чтобы автоматически создать кимограммы для обоих каналов.
- Рассчитайте соотношение между каналами (рисунок 5).
- Запустите второй блок кода Data Visualization , нажав кнопку воспроизведения , чтобы автоматически создать ратиометрический кимограф и ратиометрический таймлапс (рис. 6) .
ПРИМЕЧАНИЕ: Этот шаг доступен только при использовании двухканальной таймлапс-съемки. Порог интенсивности фона, сохраненный на шаге 1.2.1.2, вычитается из каждого канала.- Настройте переменную switch_ratio , чтобы переключить порядок каналов, используемых в качестве числителя и знаменателя во время вычисления соотношения. Значение по умолчанию — False.
- Выберите, нужно ли дополнительно сглаживать таймлапс соотношения с помощью прохода фильтра медианы, настроив переменную smooth_ratio . Значение по умолчанию — False.
- Выберите, следует ли удалять выбросы, вызванные низким сигналом канала знаменателя, манипулируя переменной reject_outliers . По умолчанию имеет значение True и определяет выбросы как значения, в 1,5 раза превышающие межквартильный диапазон выше третьего квартиля (где находится 75 % значений).
- Выберите, нужно ли экспортировать фон в выводе ратиометрии, настроив переменную background_ratio. Значение по умолчанию — False, которое заменяет его нулями.
- Запустите второй блок кода Data Visualization , нажав кнопку воспроизведения , чтобы автоматически создать ратиометрический кимограф и ратиометрический таймлапс (рис. 6) .

Рисунок 2: Шаг сегментации одиночных ячеек. Методы обработки изображений, такие как фильтрация, пороговые значения и маркировка областей, используются для изоляции интересующего сигнала (шаг 1.2). Эти данные имели следующие значения: Самая низкая интенсивность: 2556, Медиана: 3441 и Самая высокая интенсивность: 32125. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.

Рисунок 3: Обзор трассировки срединной линии - Средняя линия одиночной ячейки получается путем вычисления ее скелета (белый). Кончик (пурпурный) экстраполируется линейно от последних точек в конце скелета (шаг 1.3). В этой композиции и срединная линия, и ее кончик накладываются на исходную клетку. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
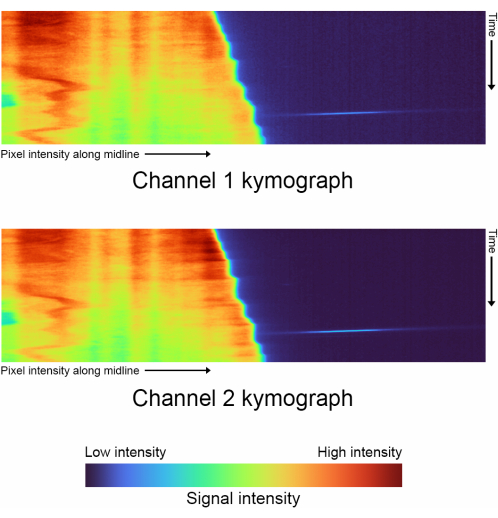
Рисунок 4: Кимографы Timelapse - Сравнение кимографов для каждого канала, сгенерированного с выключенным 'complete_skeletonization' (шаг 1.4). Вертикальная ось описывает ход времени, а горизонтальная ось отображает среднюю интенсивность экстраполированной средней линии, по которой следует одна ячейка. Для этих конкретных данных цветовая карта представляет следующие значения для канала 1: Самая низкая интенсивность: 2886, Медиана: 3167, Максимальная интенсивность: 21021. Канал 2 Самая низкая интенсивность: 3030, Медиана: 3400, Максимальная интенсивность: 29688. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.

Рисунок 5: Сглаживание фонового порога - Порог фоновой сегментации оценивается с помощью алгоритма изоданных, а затем сглаживается с помощью локальной полиномиальной регрессии (шаг 1.5). Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
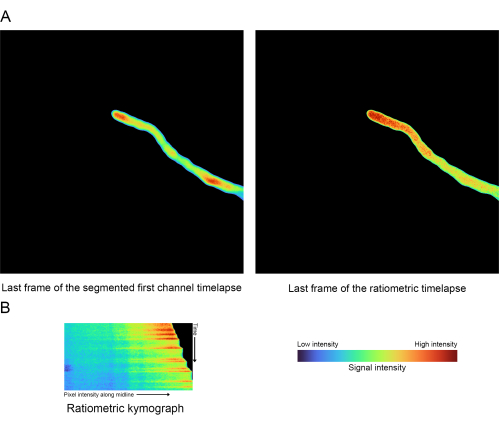
Рисунок 6: Ратиометрические результаты - (A) Сравнение между последним кадром ратиометрического таймлапса и сегментированным исходным первым каналом. (B) Кимограф, сгенерированный на основе ратиометрического таймлапса (шаг 1.5). Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
2. Протокол пакетного режима
- Скачайте и поместите файл pipeline.py из репозитория AMEBaS GitHub в тот же каталог, что и данные.
- Введите местоположение файла в командной строке после файла программы.
- При необходимости включите - -v в качестве позиционного аргумента, чтобы показать внутренние шаги конвейера.
- Включите - -s для выбора значения сигмы, используемого на этапе предварительной обработки фильтра Гаусса при подготовке к сегментации ячеек. Значение по умолчанию — 2.
- Включите - -a для трассировки средней линии для каждого кадра таймлапса. По умолчанию конвейер использует только последний кадр.
- Включите - -f , чтобы выбрать дробь [0,1] скелета, которая будет использоваться в интерполяции. Значение по умолчанию — 0,25.
- Включите -e , чтобы выбрать длину экстраполированного скелета в пикселях. Значение по умолчанию равно -1, что расширяет скелет до ближайшего края.
- Включите - -sf для выбора доли цветового диапазона, которая будет смещена на задний план в неэкстраполированных кимографах. Значение по умолчанию — 0,7.
- Включите - -k для определения размера ядра, используемого в кимографе Гауссова фильтрация. Значение по умолчанию — 3.
- Включите - -eb для оценки глобальной пороговой интенсивности фона с помощью полиномиальной регрессии LOESS для пороговых интенсивностей фона для конкретного кадра.
- Настройте количество точек, используемых при сглаживании LOESS фоновых пороговых значений, изменив параметр - -n. Значение по умолчанию — 40.
- Поменяйте местами каналы, используемые в качестве числителя и знаменателя во время вычисления соотношения, включая - r или - -switch_ratio, если таймлапс имеет два канала. По умолчанию второй канал является числителем, а первый — знаменателем.
- Выберите, нужно ли дополнительно сглаживать таймлапс соотношения с помощью прохода медианного фильтра с аргументом - -sm . Значение по умолчанию — False.
- Включите -o , чтобы отклонять пиксели с аномальной интенсивностью во время создания ратиометрической интервальной съемки.
- Выберите, должен ли фон в выводе ratiometric экспортироваться, используя аргумент - -b. Значение по умолчанию — False, которое заменяет его нулями.
- Нажмите enter , чтобы запустить. Вывод будет сгенерирован в том же каталоге, что и файл программы.
Результаты
Конвейер AMEBaS автоматизирует извлечение динамики срединной линии поляризованных одиночных клеток из стеков изображений флуоресцентной микроскопии, что делает его менее трудоемким и менее подверженным человеческим ошибкам. Метод количественно оценивает эти временные интервалы путе?...
Обсуждение
Новый метод, представленный здесь, является мощным инструментом для оптимизации и автоматизации анализа стеков изображений поляризованных клеток флуоресцентной микроскопии. Современные методы, описанные в литературе, такие как плагины ImageJ Kymograph, требуют ручной трассировки средней л?...
Раскрытие информации
Авторы данной рукописи заявляют об отсутствии конкурирующих финансовых интересов или иных конфликтов интересов.
Благодарности
Авторы выражают благодарность грантам FAPESP 2015/22308-2, 2019/23343-7, 2019/26129-6, 2020/06744-5, 2021/05363-0, CNPq, гранту NIH R01 GM131043 и грантам NSF MCB1714993, MCB1930165 за финансовую поддержку. Данные о корневых волосах были получены с помощью инфраструктуры и под руководством профессора Андреа Басси и профессора Алекса Коста.
Материалы
| Name | Company | Catalog Number | Comments |
| Github | Github | https://github.com/badain/amebas | |
| Google Colab | https://colab.research.google.com/github/badain/amebas/blob/main/AMEBAS_Colab.ipynb |
Ссылки
- Drubin, D. G., Nelson, W. J. Origins of cell polarity. Cell. 84 (3), 335-344 (1996).
- Wodarz, A., Näthke, I. Cell polarity in development and cancer. Nature Cell Biology. 9 (9), 1016-1024 (2007).
- Palanivelu, R., Preuss, D. Pollen tube targeting and axon guidance: parallels in tip growth mechanisms. Trends in Cell Biology. 10 (12), 517-524 (2000).
- Portes, M. T., et al. . The Pollen Tube Oscillator: Integrating Biophysics and Biochemistry into Cellular Growth and Morphogenesis. Rhythms in Plants: Dynamic Responses in a Dynamic Environment. , (2015).
- Wudick, M. M., et al. CORNICHON sorting and regulation of GLR channels underlie pollen tube Ca2+ homeostasis. Science. 360 (6388), 533-536 (2018).
- Hoffmann, R. D., et al. Plasma membrane H+-ATPases sustain pollen tube growth and fertilization. Nature Communications. 11 (1), 1-15 (2020).
- Michard, E., et al. Glutamate receptor-like genes form Ca2+ channels in pollen tubes and are regulated by pistil D-serine. Science. 332 (6028), 434-437 (2011).
- Damineli, D. S., Portes, M. T., Feijó, J. A. Oscillatory signatures underlie growth regimes in Arabidopsis pollen tubes: computational methods to estimate tip location, periodicity, and synchronization in growing cells. Journal of Experimental Botany. 68 (12), 3267-3281 (2017).
- Li, K., et al. An optimized genetically encoded dual reporter for simultaneous ratio imaging of Ca2+ and H+ reveals new insights into ion signaling in plants. New Phytologist. 230 (6), 2292-2310 (2021).
- Sadoine, M., et al. Designs, applications, and limitations of genetically encoded fluorescent sensors to explore plant biology. Plant Physiology. 187 (2), 485-503 (2021).
- Grenzi, M., et al. Illuminating the hidden world of calcium ions in plants with a universe of indicators. Plant Physiology. 187 (2), 550-571 (2021).
- Munglani, G., Vogler, H., Grossniklaus, U. Fast and flexible processing of large FRET image stacks using the FRET-IBRA toolkit. PLoS Computational Biology. 18 (4), 1009242 (2022).
- Ridler, T., Calvard, S. Picture thresholding using an iterative selection method. IEEE Transactions on Systems, Man, and Cybernetics. 8 (8), 630-632 (1978).
- Portes, M. T., Feijó, J. . Growing Arabidopsis pollen tubes expressing genetically encoded reporters for calcium and pH. , (2023).
- Candeo, A., Doccula, F. G., Valentini, G., Bassi, A., Costa, A. Light sheet fluorescence microscopy quantifies calcium oscillations in root hairs of Arabidopsis thaliana. Plant & Cell Physiology. 58 (7), 1161-1172 (2017).
- Romano Armada, N., et al. In vivo light sheet fluorescence microscopy of calcium oscillations in Arabidopsis thaliana. Methods in Molecular Biology. 1925, 87-101 (2019).
- Krebs, M., et al. FRET-based genetically encoded sensors allow high-resolution live cell imaging of Ca2+ dynamics. The Plant Journal. 69 (1), 181-192 (2012).
- Lee, T. -. C., Kashyap, R. L., Chu, C. -. N. Building skeleton models via 3-D medial surface axis thinning algorithms. CVGIP: Graphical Models and Image Processing. 56 (6), 462-478 (1994).
- Nunez-Iglesias, J., Blanch, A. J., Looker, O., Dixon, M. W., Tilley, L. A new Python library to analyse skeleton images confirms malaria parasite remodelling of the red blood cell membrane skeleton. PeerJ. 6, 4312 (2018).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены