Method Article
초저입력 캐리어-CAGE를 사용한 전사 시작 사이트 매핑
요약
유전자 발현(CAGE)의 캡 분석은 단일 뉴클레오티드 분해능에서 RNA 폴리머라제 II 전사 시작 부위를 포착하기 위해 mRNA 5'ends의 게놈 전반의 정량적 매핑을 위한 방법이다. 이 작품은 나노그램 양의 총 RNA를 사용하여 고품질 라이브러리의 생성을 위한 낮은 입력(SLIC-CAGE) 프로토콜을 설명합니다.
초록
유전자 발현(CAGE)의 캡 분석은 RNA 폴리머라제 II 전사 개시 부위(TSSs)의 단일 뉴클레오티드 분해능 검출에 사용되는 방법이다. TSS의 정확한 검출은 핵심 프로모터의 식별 및 발견을 향상시킵니다. 또한, 활성 인핸서는 양방향 전사 개시의 서명을 통해 검출 될 수있다. 여기서 설명된 초저 입력 반송파-CAGE(SLIC-CAGE)를 수행하기 위한 프로토콜이 있다. 이러한 CAGE 프로토콜의 SLIC 적응은 관심 있는 샘플에 추가되는 시험관 내 전사 RNA 담체 혼합물을 사용하여 RNA 양을 인위적으로 증가시킴으로써 RNA 손실을 최소화하여 총 나노그램 양에서 라이브러리 준비를 가능하게 합니다. RNA (즉, 수천 개의 세포). 캐리어는 예상된 DNA 라이브러리 단편 길이 분포를 모방하여 균질한 담체의 풍부에 의해 야기될 수 있는 편견을 제거합니다. 프로토콜의 마지막 단계에서, 반송파는 호밍 엔도누클리스를 통해 분해를 통해 제거되고 표적 라이브러리는 증폭된다. 표적 샘플 라이브러리는 호밍 엔도뉴클로스 인식 사이트가 길기 때문에(18~27bp 사이) 진핵 게놈에서 존재할 확률이 매우 낮기 때문에 분해로부터 보호됩니다. 최종 결과는 차세대 염기서열 분석이 준비된 DNA 라이브러리입니다. 시퀀싱까지 프로토콜의 모든 단계는 6일 이내에 완료할 수 있습니다. 캐리어 준비는 하루 종일 작동해야합니다. 그러나, 대량으로 제조될 수 있고 -80°C에서 냉동 보관할 수 있다. 일단 서열화되면, 판독은 게놈 전체 의 단일 뉴클레오티드 분해능 TSSs를 얻기 위하여 가공될 수 있습니다. TSSs는 유전자 규칙에 대한 통찰력을 제공하는 코어 프로모터 또는 증강인자 발견을 위해 사용될 수 있습니다. 프로모터에 집계되면 데이터는 5'중심 식 프로파일링에도 사용할 수 있습니다.
서문
유전자 발현(CAGE)의 캡 분석은 RNA 폴리머라제 II 전사 개시 부위(TSSs)의 단일 뉴클레오티드 분해능 게놈 전체 매핑에 사용되는 방법1. 정량적 특성으로 인해 5'엔드 중심 식 프로파일링도 가능합니다. TSSs를 둘러싼 영역(대략 40 bp 상류 및 다운스트림)은 핵심 프로모터이며 RNA 폴리머라제 II및 일반 전사 인자 결합이 결합되는 물리적 위치를 나타낸다(이전에 검토된 2,3). TSS의 정확한 위치에 대한 정보는 코어 프로모터 발견 및 프로모터 역학 모니터링에 사용될 수 있습니다. 또한, 액티브 인핸서가 양방향 전사의 서명을 나타내기 때문에, CAGE 데이터는 또한 인핸서 역학4의 증강 발견 및 모니터링에 사용될 수 있다. CAGE 방법론은 최근 인코드 5, modENCODE6및 FANTOM 프로젝트7과 같은 중요한 연구 프로젝트에서광범위한 응용 프로그램 및 사용으로 인해 인기가 높아졌습니다. 또한, TSS 정보는 또한 질병 특이적 TSS가 진단 목적으로 사용될 수 있기 때문에 건강하고 병이 있는 조직을 구별하는 데 중요한 것으로 입증되고있다8.
TSS 매핑을 위한 여러 가지 방법(CAGE, RAMPAGE, STRT, nanoCAGE, nanoCAGE-XL, 올리고 캡핑)을 사용할 수 있지만, 우리와 다른 사람들은 최근 CAGE가 거짓 긍정 수가 가장 적은 진정한 TSS를 캡처하는 가장 편견없는 방법임을 보여주었습니다9 , 10. 최근 CAGE 프로토콜, nAnT-iCAGE11은제한 효소를 사용하여 짧은 태그로 조각을 절단하는 것을 피하고 PCR 증폭을 사용하지 않기 때문에 TSS 프로파일링을위한 가장 편견없는 프로토콜입니다. nAnT-iCAGE 프로토콜의 한계는 다량의 시작 물질(예를 들어, 각 샘플에 대해 총 RNA의 5 μg)에 대한 요구사항이다. 구체적인 생물학적으로 관련된 질문에 대답하기 위해, 그러한 다량의 시작 물질(예를 들어, FACS 분류 세포 또는 초기 배아 단계)을 얻는 것은 종종 불가능하다. 마지막으로, nAnT-iCAGE가 성공하면 각 샘플에서 DNA 라이브러리 물질의 1-2 ng만 사용할 수 있어 달성 가능한 시퀀싱 깊이를 제한합니다.
총 RNA의 나노그램만을 사용하여 TSS 프로파일링을 가능하게 하기 위해, 우리는 최근에 초저 입력 캐리어-CAGE 10(SLIC-CAGE, 그림1)을 개발했습니다. SLIC-CAGE는 높은 복잡성 라이브러리를 얻기 위해 총 RNA의 10ng만 필요로 합니다. 당사의 프로토콜은 총 5 μg의 RNA 물질을 달성하기 위해 관심 있는 RNA에 추가된 신중하게 설계된 합성 RNA 담체에 의존합니다. 합성 담체는 초과균질 분자에 의해 야기될 수 있는 잠재적인 편견을 피하기 위하여 길이 분포에 있는 표적 DNA 라이브러리를 모방합니다. 담체의 서열은 대장균 류실-tRNA 합성 유전자의 서열에 기초한다(표 1)는 두 가지 이유로. 첫째, 최종 라이브러리에 있는 운반체의 남은 것은, 순서가 있더라도, 진핵 게놈에 지도되지 않을 것입니다. 둘째, 대장균은 중증 종이므로, 하우스키핑 유전자는 SLIC-CAGE에 적합한 온도 범위에 최적화되어 있습니다. 담체 서열은 또한 운반체 RNA 분자로부터 유래된 DNA의 특이적 분해를 허용하기 위해 호밍 엔도뉴클로스 인식 부위와 함께 내장된다. 호밍 엔도뉴클레아제 인식 사이트가 길기 때문에 대상, 샘플 유래 라이브러리는 그대로 유지됩니다(I-CeuI = 27 bp; I-SceI = 18 bp) 및 진핵 게놈에서 통계적으로 발견될 가능성이 낮다. 캐리어의 특정 분해 및 크기 배제에 의한 단편의 제거 후, 대상 라이브러리는 PCR 증폭 및 차세대 시퀀싱에 대한 준비된다. 시작 RNA 양 (1-100 ng)에 따라, 13-18 PCR 증폭 주기 사이 요구 될 것으로 예상 된다. 각 샘플당 DNA의 최종 양은 5-50 ng 사이, 매우 깊은 시퀀싱을 위한 충분한 물자를 산출합니다. 총 RNA의 단지 1-2 ng를 사용하는 경우에, 진정한 TSS는 검출될 수 있습니다; 그러나 라이브러리의 복잡성은 낮을 것으로 예상됩니다. 마지막으로 SLIC-CAGE는 nAnT-iCAGE 프로토콜11을기반으로 하므로 시퀀싱 전에 최대 8개의 샘플을 다중화할 수 있습니다.
프로토콜
1. 캐리어의 준비
-
체외 전사를 위한 DNA 템플릿의 준비
- 41 μL의 물, 5x HF 버퍼의 20 μL, 2.5 mM dNTP의 8 μL, 10 μL의 10 μL의 독특한 전진 프라이머 (PCR_GN5_f1, 표2; 프라이머를 물에 용해하고 희석하여 각 PCR 템플릿에 대한 PCR 믹스를 준비) , 합성 담체 유전자 및 1 μL Phusion 폴리머라아제를 함유하는 2 ng/μL 템플릿 플라스미드의 10 μL. 파이펫팅하여 PCR 믹스를 혼합합니다. 모든 10 템플릿에 대한 마스터 믹스는 한 번에 준비 될 수있다 (11 반응을 준비).
- PCR 혼합물의 90 μL을 각 10 μM 리버스 프라이머의 10 μL에 첨가합니다(PCR_N6_r1-r10, 표2). 파이펫팅으로 섞으세요.
- PCR은 다음 프로그램을 사용하여 템플릿을 증폭시다: 60초의 경우 98°C, (10초의 경우 98°C, 30초의 경우 50°C, 30초의 경우 72°C) 35사이클, 72°C는 10분 동안, 4°C에서 유지한다.
-
PCR 증폭 DNA 템플릿의 젤 정제
- 1% 아가로즈 젤을 준비하십시오 (저용융 아가로스권장).
- 부피를 줄이려면, 저온(30-40°C)에서 진공 농축기를 사용하여 PCR 반응 혼합물을 100 μL에서 20 μL 총 부피로 농축합니다.
- 6x 로딩 염료 의 6 μL을 추가하고 잘 섞고 젤에 적재하십시오. 사용되는 전기 동공 탱크 (5-10 V / cm)에 적합한 전압에서 1x TAE 버퍼에서 30 분 동안 전기 동극을 실행하십시오. 병렬로 100 bp 또는 1,000 bp DNA 사다리를 실행합니다.
- 깨끗한 메스를 사용하여 대상 PCR 생성물이 함유된 젤 슬라이스를 절제한다. 과도한 아가로즈 젤을 피하십시오. 젤 추출 키트를 사용하여 PCR 제품을 정화하십시오 (제조업체의 지침에 따라).
참고: 아가로즈 겔로부터 분리된 DNA의 A260/A230 비율은 전형적으로 낮다 (0.1-0.3). 예상 대상 제품 및 부가 제품은 그림 2A에나와 있습니다. 100 μL PCR 반응의 예상 수율은 1.2-3 μg입니다. 반응은 더 높은 수율을 얻기 위해 확대될 수 있습니다.
-
담체 분자의 생체 외 내 전사
- 제조자의 지시에 따라 T7 RNA 폴리머라제를 사용하여 시험관내 담체 RNA를 전사한다. 10-20 μL 반응을 설정하십시오 (권장 키트는 재료표에 있습니다).
- RNA 정제 키트를 사용하여 시험관 내 전사 RNA를 정제한다. 제조업체의 표준 지침에 따라 DNase I을 사용하여 용액에 DNA 소화를 설정하고 RNA를 50 μL의 물로 용해시다. 용출 수율을 높이려면 원심 분리 전에 5 분 동안 열에 물을 둡니다.
참고: 열의 최대 결합 용량을 초과하지 않도록주의하십시오 (재료 표에언급 된 키트에서 용량은 최대 100 μg입니다). PCR 템플릿 1-10(길이 1 kbp 내지 200 bp)으로부터의 예상 수율은 10 μL 에서 25-50 μg이다. 반응은 담체 분자의 더 큰 주식을 얻기 위하여 확장될 수 있습니다.
-
생체 외에서 전사 된 캐리어 RNA 분자의 캡핑
- 캡핑 혼합을 10x 캡핑 버퍼 의 2 μL, 1 0 mM GTP의 1 μL, 1 μL 의 2 mM SAM (갓 희석) 및 담체 RNA 당 백시니아 캡핑 효소 1 μL을 결합하여 준비한다.
- 65°C에서 10분 동안 총 부피와 변성 15 μL로 각 담체 분자의 최대 10 μg를 혼합합니다. 이차 구조물 형성을 방지하기 위해 즉시 얼음 위에 놓습니다.
- 변성담체 RNA를 캡핑 믹스의 5 μL과 혼합하고 37°C에서 1시간 동안 배양한다.
- RNA 정제 키트를 사용하여 덮인 RNA 분자를 정화하십시오 - 제조업체의 클린업 프로토콜을 따르십시오. 30 μL의 물에 RNA를 용출하십시오. 용출 수율을 높이려면 원심 분리 전에 5 분 동안 열에 물을 둡니다.
참고: 마이크로볼륨 분광광도계를 사용하여 농도를 측정합니다. 예상 A260/A280 비율은 >2이고 A260/A230은 >2입니다. 일부 RNA 샘플의 경우 A260/A230은 1.3-2 사이일 수 있습니다. 뚜껑이 없는 RNA의 10 μg를 사용할 때 예상되는 수율은 뚜껑이 있는 RNA의 9-10 μg이다.
- 표3에 기재된 양을 조합하여 캡식 및 언캡되지 않은 담체의 혼합을 준비한다. 튜브를 가볍게 두어 잘 섞고 마이크로볼륨 분광광도계를 사용하여 농도를 측정합니다.
참고: 상반신 전사 반응에 맞추기 위해 더 높은 농도의 담체가 요구되는 경우(아래 참조), 반송파 혼합물은 원하는 최종 농도에 도달할 때까지 저온(30-35°C)에서 진공 농축기를 사용하여 농축될 수 있다. 단계 2-14는 무라타 외11에 의해보고 된 표준 nAnT-iCAGE 프로토콜에서 수정됩니다
2. 역 전사
- RT 프라이머의 1 μL(물에 용해된 2.5 mM TCT-N6, 서열은 보충표1참조), 총 RNA 의 10 ng 및 캐리어 믹스의 4,990 ng(표 3)를 저결합 PCR 플레이트에서 총 부피의 10 μL에 결합한다. 튜브를 가볍게 두핑하여 섞습니다.
참고: 샘플 RNA가 역전사를 위해 너무 희석된 경우(아래 참조), 적절한 양의 담체와 결합하고, 진공 농축기를 사용하여 총 부피 9 μL에 농축하고 RT 프라이머의 1 μL을 첨가한다. 담체 얼을 첨가하여, 총 5 μg의 RNA에 도달하여 샘플 손실을 방지한다. - 65 °C에서 2.1 단계에서 혼합물을 5 분 동안 가열하고 즉시 얼음위에 두어 비틀기를 방지하십시오.
-
역전사(RT) 혼합을 준비한다.
- 각 샘플에 대해 6.1 μL의 물(RNase- 및 DNase-free), 5x 첫 번째 가닥 완충액의 7.6 μL, 0.1 M DTT의 1.9 μL, 10 mM dNTP의 1 μL, 7.6 μL의 트레할로스/소르비톨 믹스를 결합합니다(무라타 외11의레시피 참조) 및 3.8 μl의 6>재료표). 튜브를 가볍게 두켜 잘 섞으세요.
- 28 μL의 RT 혼합물을 RNA, 담체 및 RT 프라이머(총 부피 38 μL)로 PCR 튜브에 추가합니다. 파이펫팅으로 잘 섞으세요.
참고: 이 혼합물은 트레할로오스/소르비톨로 인해 점성이 높습니다. 눈에 띄게 균일할 때까지 섞으세요. - 다음 프로그램을 사용하여 열 사이클러에서 배양: 30s에 25°C, 50°C에서 60분 동안, 4°C에서 유지한다.
-
CDNA의 정화:SPRI 자기 구슬을 이용한 RNA 하이브리드
- 권장 RNAE- 및 DNase-free SPRI 비드의 68.4 μL을 RT 혼합물의 38 μL에 추가합니다(비드 대 샘플 비 1.8:1). 파이펫팅으로 잘 섞고 실온(RT)에서 5분 동안 배양합니다.
- 5 분 동안 자기 스탠드에 구슬을 분리. 상급을 버리고 70 % 에탄올 (갓 제조)의 200 μL로 두 번 구슬을 세척합니다.
참고: 에탄올은 혼합하지 않고 튜브가 자기 스탠드에있는 동안 구슬에 추가됩니다. 첨가된 에탄올은 즉시 제거된다. 시료 손실로 이어질 수 있으므로 세차 하는 동안 구슬을 잃지 않도록 주의 해야 합니다. - 튜브가 마그네틱 스탠드에 있는 동안 에탄올의 모든 흔적을 제거합니다. P10 파이펫을 사용하여 튜브 밖으로 에탄올 방울을 제거하고 밀어 낼 수 있습니다. 구슬을 말리지 마십시오.
- 37°C에서 예열된 물 42 μL을 구슬에 넣고 60배 위아래로 파이펫팅하여 시료를 다루합니다.
참고: 발포에 비드 (즉, 바운드 샘플)가 손실 될 수 있으므로 파이펫팅에 의해 발포가 발생하지 않도록주의하십시오. - 뚜껑 없이 37°C에서 5분 동안 배양하여 미량의 에탄올 증발을 허용한다.
- 5 분 동안 자기 스탠드에 구슬을 분리하고 새 접시에 상판을 전송합니다.
참고: 비드 이월을 피하면서 샘플 손실을 방지하기 위해 모든 상급을 검색하려고합니다. P10 파이펫을 사용하여 마지막 샘플 물방울을 가져옵니다.
3. 산화
- 정제된 RT 반응에 1 M NaOAc(pH 4.5)의 2 μL을 첨가합니다. 파이펫팅으로 혼합하고 250 mM NaIO4의 2 μL을 추가하고 다시 섞습니다.
- 45 분 동안 얼음에 배양. 빛을 피하기 위해 알루미늄 호일로 접시를 덮습니다.
- 산화 혼합물에 Tris-HCl (pH 8.5)의 16 μL을 첨가하여 pH를 중화시다.
- 산화 된 cDNA를 정화 : SPRI 자기 구슬을 사용하여 RNA 하이브리드. 산화 혼합의 60 μL에 SPRI 비드 108 μL을 추가합니다(샘플 비에 대한 1.8:1 비드). 단계 2.6.1-2.6.6에 설명된 대로 정제를 반복합니다. 37 °C로 예열 된 물 42 μL을 사용하여 용루.
참고: NaIO4 의 1 mg 당 18.7 μL의 물을 추가하여 250 mM NaIO4를신선하게 준비하십시오. NaIO 4는 빛에 민감합니다. 따라서 용액을 알루미늄 호일로 덮인 튜브 또는 내광성 튜브에 보관하십시오.
4. 생물항제
- 정제된 산화 시료를 함유하는 튜브에 1 M NaOAc(pH 6.0)의 4 μL을 넣고 파이펫팅하여 혼합합니다.
- 10 mM 비오틴 용액 4 μL을 추가하고 파이펫팅하여 혼합하고 빛을 피하기 위해 열 사이클러에서 23 °C에서 2 시간 동안 배양하십시오.
참고: 50 mg의 비오틴을 DMSO 13.5 mL와 혼합하여 비오틴 용액을 준비합니다. 일회용 알리쿼트(aliquots)를 만들고 -80°C에서 얼어 붙습니다. - SPRI 자기 구슬을 사용하여 생체 생물학적 cDNA:RNA 하이브리드를 정화합니다. 12 μL의 2-프로판올을 넣고 파이펫팅하여 섞습니다. SPRI 비드 108 μL(샘플 비에 1.8:1 비드)을 추가하고 단계 2.6.1-2.6.6에 설명된 대로 정제를 반복합니다. 37 °C에서 예열된 42 μL의 물을 사용하여 용루.
참고: 프로토콜은 여기에서 일시 중지될 수 있으며, 샘플은 -80°C에서 동결됩니다.
5화 RNase I 소화불량
- 각 샘플당 RNase I(10 U/μL)의 0.5 μL로 완충된 RNase I 의 4.5 μL을 혼합하여 RNase I 혼합물을 준비한다. 파이펫팅으로 섞으세요.
- 각 정제 된 샘플 (총 45 μL)에 혼합물 5 μL을 추가합니다. 파이펫팅하여 혼합하고 37 °C에서 30 분 동안 배양합니다.
6. 스트렙타비딘 구슬의 준비
- 각 샘플에 대해 스트렙타비딘 비드 슬러리의 30 μL을 0.38 μL의 20 mg/mL tRNA와 혼합합니다. 30 분 동안 얼음에 배양하고 튜브를 가볍게 하여 5 분마다 섞습니다.
참고: 스트렙타비딘 비드 슬러리를 병을 가볍게 두드리면 파이펫팅을 할 수 있습니다. tRNA 용액은 무라타 외11에 따라 제조되어야 한다. - 마그네틱 스탠드의 구슬을 2-3분 동안 분리합니다.
- 완충A의 15 μL로 재중단하여 구슬을 세척하고 자석 스탠드에 있는 구슬을 2-3분 동안 분리하고 상한체를 제거한다. 세척을 반복하고 상급제를 제거합니다.
- 완충A의 105 μL에서 구슬을 다시 중단하고 tRNA의 20 mg/mL의 0.19 μL을 추가합니다. 파이펫팅으로 잘 섞으세요.
참고: 구슬은 사용하기 전에 신선하게 준비해야합니다. RNase I 소화 동안 구슬의 준비를 시작합니다. 여러 샘플의 경우 단일 튜브에 함께 구슬을 준비합니다.
7. 캡 트랩핑
-
샘플 바인딩
- 준비된 스트렙타비딘 비드 105 μL을 RNase I 처리 샘플의 45 μL에 첨가합니다. 파이펫팅을 통해 잘 섞고 37°C에서 30분 동안 배양합니다.
- 마그네틱 스탠드의 구슬을 2-3분 동안 분리합니다.
-
세탁 구슬
- 세척 버퍼 A의 150 μL을 추가하고 파이펫팅하여 구슬을 다시 일시 중단합니다. 마그네틱 스탠드의 구슬을 2-3분 동안 분리하고 상한을 제거합니다.
- 세척 버퍼 B의 150 μL을 추가하고 파이펫팅으로 구슬을 다시 일시 중단합니다. 마그네틱 스탠드의 구슬을 2-3분 동안 분리하고 상한을 제거합니다.
- 세척 버퍼 C의 150 μL을 추가하고 파이펫팅으로 비드를 다시 일시 중단합니다. 마그네틱 스탠드의 구슬을 2-3분 동안 분리하고 상한을 제거합니다.
참고: 버퍼 B와 C는 37°C로 예열되어야 한다. 세척 버퍼 A, B 및 C에 대한 조리법은 Murata 외11에 설명되어 있습니다.
-
cDNA 방출
- 58.5 μL의 물을 10x RNase I 버퍼의 6.5 μL과 혼합하여 1x RNase I 완충물을 준비합니다.
- 1x RNase I 버퍼의 35 μL에서 비드를 다시 일시 중단합니다. 95°C에서 5분 동안 배양하고 cDNA의 재결합을 방지하기 위해 2분 동안 얼음에 직접 전달합니다. 얼음으로 옮기는 동안 뚜껑을 잡고 압력이 쌓일 수 있습니다.
- 마그네틱 스탠드에서 2-3분 동안 구슬을 분리하고 상판을 새 플레이트로 옮김.
- 1x RNase I 버퍼의 30 μL에서 비드를 다시 일시 중단합니다. 자기 스탠드에 구슬을 2-3 분 동안 분리하고 이전에 수집 된 상판으로 상구체를 전송합니다 (용출 된 cDNA의 총 부피는 약 65 μL여야합니다).
8. RNase H와 RNase I 소화에 의한 RNA 제거
- 시료당 2.4 μL의 물, 0.5 μL의 RNase I 버퍼, 0.1 μL의 RNase H 및 2 μL의 RNase I를 결합합니다.
- 방출된 cDNA 샘플의 65 μL에 혼합물의 5 μL을 첨가하고 파이펫팅하여 혼합한다. 37°C에서 15분 동안 배양하고 4°C에서 유지합니다.
- SPRI 자기 구슬을 사용하여 RNase 소화 믹스에서 cDNA를 정화합니다. 126 μL의 SPRI 비드를 70 μL의 분해 반응을 추가하고 파이펫팅으로 혼합합니다. 2.6.1-2.6.6에서 SPRI 비드 정제에 대해 설명한 대로 정제 단계를 따르십시오. 설명된 바와 같이 37°C에서 예열된 42 μL의 물을 사용하여 용루.
- RNase I 의 4.5 μL의 10x RNase I 버퍼 및 RNase I의 0.5 μL을 결합하여 혼합하여 준비한다.
- 정제된 cDNA 샘플의 40 μL에 RNase 혼합물 의 5 μL을 첨가합니다. 파이펫팅하여 37°C에서 30분 동안 배양합니다.
- SPRI 자기 구슬을 사용하여 샘플을 정화합니다. 81 μL의 SPRI 비드를 45 μL의 분해 반응을 추가하고 파이펫팅으로 혼합합니다. 2.6.1-2.6.6에서 SPRI 비드 정제에 대해 설명한 대로 정제 단계를 따르십시오. 기재된 바와 같이 42 μL의 물을 사용하여 용루.
9. 5' 링커 의 결찰
- 진공 농축기를 사용하여 정제된 cDNA 샘플을 4 μL에 농축합니다. 온도를 30-35 °C로 유지하십시오. 파이펫을 사용하여 체적을 테스트합니다. 시료가 완전하게 건조된 경우, 4 μL의 물을 첨가하여 용해하십시오.
참고: 시료 손실을 방지하기 위해 완전성을 위해 건조를 피하는 것이 좋습니다. - 농축 된 샘플을 95 °C에서 5 분 동안 배양하고 즉시 2 분 동안 얼음에 두어 발란을 방지하십시오. 압력 축적으로 인해 뚜껑이 튀어 나오게 될 수 있기 때문에 튜브를 옮기는 동안 뚜껑을 잡습니다.
- 55°C에서 2.5 μM 5' 링커의 4 μL을 5분 동안 인큐베이팅하고 즉시 얼음 위에 2분 동안 두어 발란을 방지합니다.
- 2.5 μM 5' 링커의 4 μL을 시료의 4 μL과 혼합합니다.
참고: 5' 링커는 보충표2, 보충표3, 보충표4, 보충표5에 따라 준비되어야 한다. 사용 전에 10μM NaCl을 사용하여 10 μM 5' 링커를 2.5 μM 농도로 희석합니다. - 혼합 5' 링커에 결찰 프리믹스 16 μL(재료 표참조)을 넣고 시료를 파이펫팅하여 잘 섞습니다. 16°C에서 16시간 동안 배양한다.
- SPRI 자기 비드를 사용하여 결찰 혼합물을 정화합니다. SPRI 비드 43.2 μL을 추가하고 2.6.1-2.6.6 단계를 따릅니다. 37°C에서 예열된 42 μL의 물을 사용하여 설명한 바와 같이 용루.
- 9.6단계에서 수행된 정제를 72 μL의 SPRI 비드를 전달된 상급물질(샘플 비에 1.8:1 비드)에 첨가하여 반복한다.
참고: 5' 링커에는 시퀀싱 전에 최대 8개의 샘플을 풀링할 수 있는 바코드가 포함되어 있습니다(Murata 등11 및 보충 표1에 설명된 대로 8개의 삼중뉴클레오티드 바코드를 사용할 수 있음).
10. 3' 링커 의 결찰
- 9.1단계에서 설명한 바와 같이 진공 농축기를 사용하여 정제된 샘플을 4 μL로 농축한다.
- 농축 된 샘플을 95 °C에서 5 분 동안 배양하고 즉시 2 분 동안 얼음에 두어 발란을 방지하십시오. 압력 축적으로 인해 뚜껑이 튀어 나오기 때문에 튜브를 옮기는 동안 뚜껑을 잡습니다.
- 65°C에서 2.5 μM 3' 링커의 4 μL을 5분 동안 인큐베이팅하고 즉시 얼음 위에 2분 동안 두어 발란을 방지합니다.
- 농축 된 샘플의 4 μL에 2.5 μM 3 '링커의 4 μL을 추가합니다.
- 결찰 프리믹스 16 μL을 추가하고 파이펫팅으로 잘 섞습니다. 16°C에서 16시간 동안 배양한다.
- SPRI 자기 비드를 사용하여 결찰 혼합물을 정화합니다. SPRI 비드 43.2 μL을 추가하고 2.6.1-2.6.6 단계를 따릅니다. 37°C로 예열된 42 μL의 물을 사용하여 기술된 바와 같이 용루.
참고: 3' 링커는 보충 표 6 및 보충 표7에 따라 준비되어야 합니다. 100 μM NaCl을 사용하여 10 μM 3' 링커를 2.5 μM 농도로 희석한다.
11화 탈인
- 4 μL의 물, 5 μL의 10x SAP 완충제 및 1 μL의 SAP 효소를 결합하여 SAP 혼합물을 준비한다.
- 10 μL의 SAP 혼합물을 정제된 결개 시료(총 부피 50 μL)에 넣고 열순환기에 인큐베이트하여 30분 동안 37°C, 65°C에서 15분, 4°C에서 유지한다.
12. 우라실 특정 절제 효소를 사용하여 3' 링커 상부 가닥의 분해
- 우라실 특이적 절제 효소 2μL을 dephosphorylated 샘플에 추가하고, 피펫팅하여 혼합하고 열사이클러에서 배양하여 37°C를 30분, 95°C에서 5분 동안, 얼음에 즉시 2분 간 두세요. 조각난 상부 가닥의 재내막을 방지합니다.
- 반응 혼합물을 52 μl 혼합물에 93.6 μL의 SPRI 자기 비드를 첨가하고 파이펫팅하여 잘 혼합하여 반응 혼합물을 정제한다. 정화 단계 2.6.1-2.6.6을 반복합니다. 설명된 바와 같이 37°C에서 예열된 42 μL의 물로 용루.
13. 두 번째 가닥 합성
- 10x DNA 폴리머라제 반응 완충액 5 μL, 물 2 μL, 1 μL 의 1 μL, 50 μM nAnT-iCAGE 제2 가닥 프라이머(서열은 보충표1) 및 D의 1 μL을 결합하여 제2 가닥 합성 믹스(시료당 부피가 표현된다)를 준비한다. NA 외핵구아제 결핍 폴리머라제(재료 표에서권장 폴리머라제 참조).
- 정제된 샘플에 10 μL의 혼합물을 추가하고 파이펫팅을 통해 잘 혼합합니다(총 부피는 50 μL). 다음 프로그램을 사용하여 열 사이클러에서 배양: 5분 동안 95°C, 55°C에서 5분, 72°C를 30분 동안, 4°C에서 유지한다.
14. 엑소뉴클레아제 I을 사용하여 두 번째 가닥 합성 프라이머의 분해
- 엑소뉴클레아제 I의 1 μL을 제2 가닥 합성 혼합물에 첨가한다. 파이펫팅을 통해 잘 혼합하고 37°C에서 30분 동안 배양한 다음 4°C에서 유지하였다.
- 엑소뉴클레아제 I-처리 시료의 51 μL에 SPRI 자기 비드 91.8 μL을 첨가하여 이중 가닥 DNA를 정제하였다. 2.6.1-2.6.6에 설명된 반복 정제 단계. 기재된 바와 같이 37°C로 예열된 42 μL의 물로 용해한다.
- 9.1단계에서 설명한 바와 같이 진공 농축기를 사용하여 15 μL까지 샘플을 농축한다.
15. 질 및 수량 통제
- 농축 된 샘플의 1 μL을 사용하고 DNA 품질 분석기에서 고감도 DNA 칩을 실행하십시오. 예상 프로파일/수량은 그림3에 표시됩니다.
16. 캐리어 저하의 첫 번째 라운드
- 2 μL의 물, 2 μL의 10x 제한 효소 완충제, 1 μL의 I-SceI 및 1 μL의 I-CeuI를 결합하여 분해 혼합을 준비한다.
- 분해 혼합물의 6 μL을 농축 된 샘플의 14 μL에 넣고 파이펫팅하여 혼합합니다. 37°C에서 3시간 동안 인큐베이션한 다음 65°C에서 20분 비활성화하고 4°C에서 유지한다.
- SPRI 자기 비드를 사용하여 분해 혼합을 정화합니다. 5 μL의 물을 추가하여 분해 혼합물의 부피를 늘리고 SPRI 비드 45 μL(시료 비에 1.8:1 비드)을 추가합니다. 단계 2.6.1-2.6.6에 설명된 대로 반복 정제. 37 °C로 예열 된 물 42 μL로 용해하십시오.
- 9.1단계에서 설명한 바와 같이 총 부피의 42 μL ~ 20 μL의 용출 샘플을 농축한다.
17. 저하 레벨 제어 및 PCR 증폭 주기 수 결정
- 전체 라이브러리(어댑터 믹스)를 증폭하기 위한 qPCR 믹스를 준비합니다. 3.8 μL의 물을 결합하고, 5 μL의 qPCR 프리믹스(2x), 0.1 μL 의 10 μM 어댑터_f1 프라이머(5'-AATGATACGGCGACCACCACCGA-3'), 및 0.1 μL 의 10 μM 어댑터_r1 프라이머(5'-CAAGCAGAAGACACACACACGA-3')는 각 샘플에 대해 (권장되는 q.c.의 재료 표 참조).
- 9 μL의 qPCR 어댑터 믹스를 16.4단계에서 1μL의 시료와 결합하고 파이펫팅을 통해 잘 섞습니다.
- 담체(carrier mix)로부터 유래된 DNA를 증폭하기 위한 qPCR 혼합물을 준비한다. 3.8 μL의 물, 5 μL의 qPCR 프리믹스(2x), 0.1 μl 의 10 μM 캐리어_f1 프라이머(5'-GCGGGCGCGCGTTTCACACAC-3'), 각 시료에 대해 0.1 μL의 10μl의 어댑터_r1 프라이머를 결합합니다.
- 9 μL의 qPCR 캐리어 믹스를 16.4 단계에서 샘플의 1 μL과 결합하고 파이펫팅으로 잘 혼합하십시오.
- qPCR 프로그램 설정: 3분 동안 95°C(20초의 경우 95°C, 20초의 경우 60°C, 20초동안 72°C)를 40회 반복한 다음 계측기별 변성 곡선(65-95°C)을 반복하고 4°C에서 유지합니다.
참고: 샘플을 물로 대체하여 음의 제어를 준비합니다.
18. 대상 라이브러리의 PCR 증폭
- 6 μL의 물, 0.5 μL의 10 μM 어댑터_f1 프라이머, 0.5 μL의 10 μM 어댑터_r1 프라이머 및 25 μL의 PCR 프리믹스(2x)를 결합하여 PCR 증폭 믹스를 준비합니다. 파이펫팅으로 혼합합니다(권장PCR 프리믹스는 재료 표 참조).
- 16.4단계에서 시료의 18 μL에 PCR 혼합물의 32 μL을 첨가한다. 파이펫팅으로 철저히 섞으세요.
- PCR 증폭 설정: 3분 동안 95°C, (20초의 경우 98°C, 15s의 경우 60°C, 2분동안 72°C) 12-18사이클, 72°C에서 2분 간, 4°C에서 유지한다.
참고: 정확한 PCR 사이클 수는 qPCR 결과에 의해 결정되며 어댑터 프라이머 믹스로 얻은 Ct 값에 해당합니다(PCR 사이클의 수는 Ct 값과 동일합니다). - 증폭된 시료의 90 μL에 SPRI 자기 비드를 50 μL에 첨가하여 증폭된 시료를 정화하고 파이펫팅을 통해 철저히 혼합합니다. 2.6.1-2.6.6단계에 설명된 정제 단계를 반복합니다. 기재된 바와 같이 42 μL의 물을 사용하여 샘플을 용출한다.
19. 캐리어 저하 의 두 번째 라운드
- 16.1-16.3단계를 반복합니다.
- SPRI 자기 비드를 사용하여 분해 혼합을 정화합니다. 시료에 10 μL의 물을 추가하여 부피를 늘리고 SPRI 비드 30 μL(시료 비1:1 비드)과 혼합합니다. 단계 2.6.1-2.6.6에 설명된 대로 반복 정제. 기재된 바와 같이 37°C에서 예열된 42 μL의 물로 용해한다.
- 용출 된 샘플을 총 부피의 42 μL에서 30 μL까지 농축하십시오.
20. 라이브러리 크기 선택
- 24 μL의 SPRI 자기 비드를 19.3 단계에서 샘플의 30 μL과 혼합합니다. (0.8:1 비드 대 샘플 비율) 단계 2.6.1-2.6.6에 설명된 대로 정제 단계를 반복합니다. 설명된 대로 42 μL의 물로 샘플을 용해시낸다.
- 9.1단계에서 기재된 바와 같이 샘플을 약 14 μL로 농축한다.
21. 품질 관리
-
크기 분포 평가
- 고감도 DNA 칩상에서 시료의 1 μL을 실행합니다. 예상 결과는 그림4에 표시됩니다.
참고: 200 bp보다 짧은 조각이 표시되는 경우(그림 4A,C의 예 참조), 크기 선택(단계 20.1-20.2)은 짧은 조각이 제거될 때까지 반복되어야 합니다(그림4B,D). 일반적으로 하나의 추가 크기 선택으로 충분합니다. 짧은 조각의 양이 심한 경우(그림 4C에서와 같이), 비드 대 샘플 비율은 0.6:1로 감소시켜야 합니다.
- 고감도 DNA 칩상에서 시료의 1 μL을 실행합니다. 예상 결과는 그림4에 표시됩니다.
-
캐리어 열화 품질 관리
- 17.1-17.5단계를 반복합니다.
참고: HS DNA 칩 런(region analysis)에서 추정된 라이브러리의 농도에 따라, 샘플은 qPCR 이전에 희석될 필요가 있다. 시료의 0.5 μL을 사용하여 시료 손실을 방지하고 물에서 100-500x를 희석하십시오 (1-20 pg/μL 최종 농도로 희석). 어댑터와 캐리어 믹스로 얻은 Ct 값 간의 예상 차이는 5-10입니다.
- 17.1-17.5단계를 반복합니다.
-
라이브러리 정량화
- 100 mg/mL 람다 DNA 표준의 20 μL과 1x TE의 980 μL을 혼합하여 람다 DNA 표준의 작동 희석을 준비합니다(DNA 정량화 키트에 제공된 20x TE를 희석하여 준비). 람다 DNA의 희석은 -20°C에서 저장될 수 있다.
- 보충표8에 따라 희석된 람다 표준 및 1x TE를 혼합하여 람다 DNA 표준 직렬 희석을 준비한다.
참고: 정확도를 높이려면 모든 튜브에 100 μL의 1x TE 버퍼를 추가하고 희석된 람다의 부피당 필요에 따라 1x TE 부피를 제거하는 것이 좋습니다. 라이브러리의 1 μL 이상을 사용하지 마십시오. 이 측정을 위해 384 개의 웰 플레이트를 사용하는 것이 좋습니다.
결과
본 보고서는 시작 총 RNA 물질의 나노그램으로부터 시퀀싱 준비 라이브러리를 얻기 위한 전체SLIC-CAGE 프로토콜을 설명한다(도 1). 합성 RNA 담체 혼합물을 얻기 위해, 먼저, PCR 담체 템플릿을 제조하고 정제하여 PCR 측 제품을 제거해야 한다(도2A). 각 PCR 템플릿(총 10개)은 공통 정방향이지만 상이한역프라이머(표 2)를 사용하여 생성되지만, 합성 RNA 담체의 크기 가변성을 가능하게 하는 PCR 템플릿의 상이한 길이로 이어진다. 일단 정제되면, PCR 템플릿은 담체 분자의 시험관 내 전사에 사용된다. 템플릿이 겔 정제되는 경우 단일 RNA 담체 생성물이 예상된다(도 2B의대표적인 겔 분석 참조). 캐리어의 제조는 필요에 따라 업스케일링될 수 있고, 제조될 때, 향후 사용을 위해 -80°C에서 혼합 및 냉동될 수 있다.
PCR 증폭의 16-18 사이클과 결합된 권장 최소량의 샘플 총 RNA(10ng)를 사용하여 높은 복잡성SLIC-CAGE 라이브러리를 달성할 수 있습니다. 최종 라이브러리를 증폭하는 데 필요한 PCR 사이클의 수는 사용된 총 입력 RNA의 양에 따라 크게 좌우됩니다(예상 사이클 수는 표4에 제시되어 있음).
첫 번째 분해 후 qPCR 결과(17단계)에서 adaptor_f1 또는 carrier_f1 프라이머를 사용하여 얻은 Ct 값 간의 예상 차이는 1-2이며, adaptor_f1로 얻은 Ct 값은 carrier_f1보다 낮습니다.
최종 라이브러리에서 단편 길이의 분포는 평균 단편 크기가 700-900 bp인 200-2,000 bp 사이입니다(바이오분석기 소프트웨어를 사용한 영역 분석에 기초, 도 4B,D). 그림 4A,C에제시된 바와 같이 짧은 조각은 크기 배제의 추가 라운드(단계 20-21)에 의해 제거되어야 합니다. 이러한 짧은 조각은 대상 라이브러리가 아닌 PCR 증폭 아티팩트입니다. 짧은 조각은 시퀀싱 흐름 셀에서 더 나은 클러스터를 가지며 시퀀싱 문제가 발생할 수 있습니다.
샘플당 얻어진 라이브러리 재료의 예상 량은 5-50 ng 사이입니다. 현저히 낮은 양은 프로토콜 동안의 샘플 손실을 나타낸다. 얻어진 낮은 수량이 시퀀싱에 충분하면(풀링된 라이브러리의 2-3ng가 필요함) 라이브러리는 더 낮은 복잡성일 수 있습니다(아래 참조).
시퀀싱 머신에 따라 유량 셀에 로드된 라이브러리의 양을 최적화해야 할 수 있습니다. Illumina HiSeq 2500을 사용하면 8-12pM SLIC-CAGE 라이브러리를 로드하면 평균 150-2억 개의 읽기가 제공되며 읽기의 80%는 품질 점수 Q30을 임계값으로 통과합니다.
얻어진 판독값은 기준 게놈에 매핑된 후 [50 bp 읽기의 경우, Bowtie212는 시드 서열당 0개의 불일치를 허용하는 기본 파라미터와 함께 사용될 수 있다(22 bp)]. 예상 매핑 효율성은 총 RNA 입력 량에 따라 달라지며 표5에 나와 있습니다. 고유하게 매핑된 판독값은 R 그래픽 및 통계 컴퓨팅환경(13)에 로드되고 CAGEr(생체 전도체 패키지14)를사용하여 처리될 수 있다. 패키지 비네팅은 따라하기 쉽고 매핑된 데이터의 워크플로우 및 처리를 자세히 설명합니다. 라이브러리 복잡성의 쉬운 시각적 제어는 프로모터 폭의 분포이며, 저복잡성 라이브러리는 인위적으로 좁은 프로모터를 가질 것이다(그림5A,총 RNA의 1ng에서 파생된 SLIC-CAGE 라이브러리, 자세한 내용은 이전 참조) 10). 그러나, 심지어 낮은 복잡성 SLIC-CAGE 라이브러리는 낮은 / 중간 입력 TSS 매핑에 대한 대체 방법보다 더 큰 정밀도, 진정한 CTSS의 식별을 할 수 있습니다 (그림5B,C).
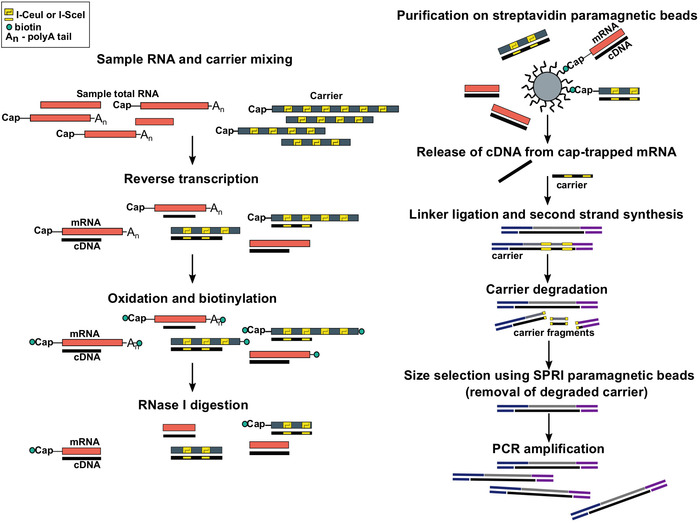
그림 1: SLIC-CAGE 프로토콜의 단계입니다. 샘플 RNA는 총 RNA 물질의 5 μg를 달성하기 위해 RNA 담체 혼합물과 혼합된다. cDNA는 역전사를 통해 합성되고 캡은 나트륨 주위를 사용하여 산화됩니다. 산화는 비오틴 히드라지드를 사용하여 캡에 비오틴을 부착할 수 있게 합니다. 비오틴은 mRNA의 3′ 끝에 부착되다, 그것은 또한 나트륨 주기도 사용하여 산화로. mRNA에서 비오틴을 제거하기 위해:cDNA 하이브리드 불완전 합성 cDNA와 3′ mRNA의 끝에서, 샘플은 RNase I. mRNA의 끝에 도달 cDNA로 처리됩니다 다음 스트렙 타비딘 자기 구슬에 친화도 정제에 의해 선택된다 ( 캡 트래핑)을 참조하십시오. cDNA의 출시 후, 5′- 그리고 3′링커는 결찰. 담체로부터 유래된 라이브러리 분자는 I-SceI 및 I-CeuI 호밍 엔도뉴클레스를 사용하여 분해되고 단편은 SPRI 자기 비드를 사용하여 제거된다. 그런 다음 라이브러리가 PCR 증폭됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
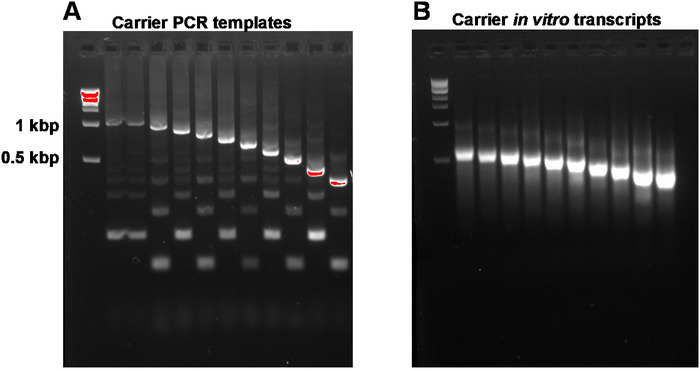
도 2: 대표적인 겔-체외 전사체 내 전사체 및 담체 PCR 템플릿의 겔 분석. (a) 겔 정제 전에 캐리어 PCR 템플릿: 제 1 웰은 1 kbp 마커를 포함하고, 그 다음에 캐리어 PCR 템플릿 1, 1-10. (B) 담체 체외 전사체: 제 1 웰은 1 kbp 마커를 포함하고, 그 다음에 담체 전사체 1-10을 함유한다. 담체 전사체는 로딩 전에 95°C에서 5분 동안 가열하여 변성하였다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
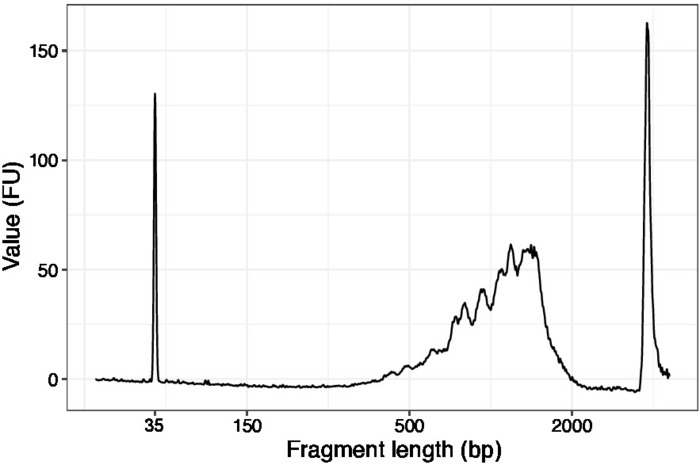
그림 3: 1차 담체 분해 전에 SLIC-CAGE의 대표적인 DNA 품질(고감도 DNA 칩) 트레이스. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
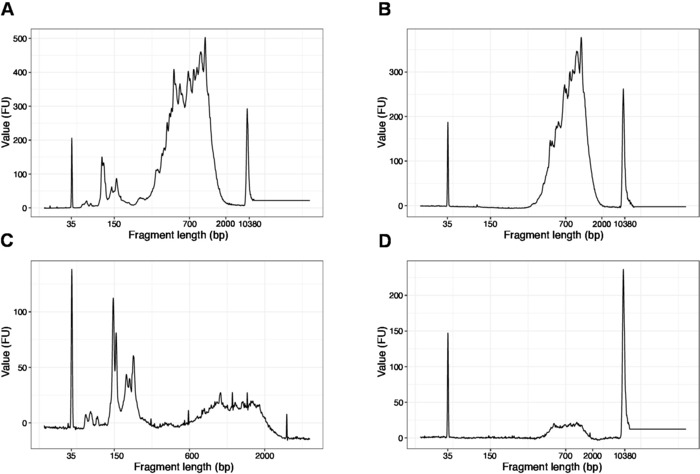
그림 4: PCR 증폭 후 SLIC-CAGE 라이브러리의 대표적인 DNA 품질(고감도 DNA 칩) 트레이스. (A) 짧은 조각을 제거하기 위해 추가 크기 선택이 필요한 SLIC-CAGE 라이브러리입니다. (B) SLIC-CAGE 라이브러리는 0.6x SPRI 비드를 사용하여 크기 선택 후 샘플 비율을 사용합니다. (C) 짧은 조각의 제거를 위한 크기 선택이 필요한 낮은 출력 량의 SLIC-CAGE 라이브러리. (D) SLIC-CAGE 라이브러리는 0.6:1 SPRI 비드를 사용하여 크기 선택 후 낮은 출력량을 시료 비율로 사용한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
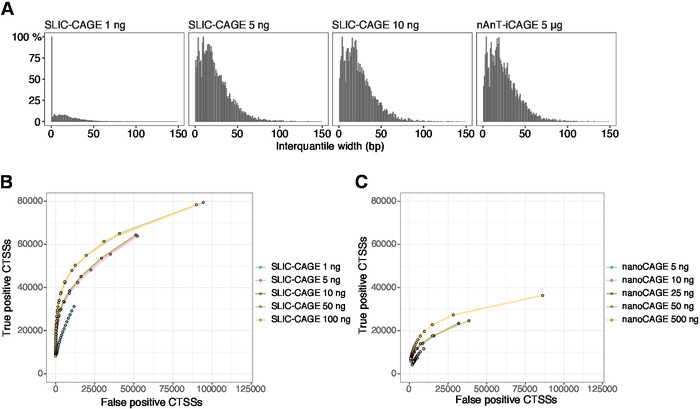
그림 5: SLIC-CAGE 라이브러리의 유효성 검사. (a) SLIC-CAGE 라이브러리에서 태그 클러스터 인터퀀티들 폭의 분포는 S. 세레비시아 총 RNA의 1, 5, 또는 10 ng로부터 제조되고, S. 세레비시아 총 RNA의 5 μg로부터 제조된 nAnT-iCAGE 라이브러리에서 제조하였다. 1ng SLIC-CAGE 라이브러리에 있는 많은 양의 좁은 태그 클러스터는 낮은 복잡성을 나타냅니다. (B) S. 세레비시아 SLIC-CAGE 라이브러리의 CTSS 식별을 위한 ROC 곡선. 모든 S. 세레비시아 nAnT-iCAGE CTSS는 트루 세트로 사용되었습니다. (C) S. 세레비시아 나노케이지 라이브러리에서 CTSS 식별을 위한 ROC 곡선. 모든 S. 세레비시아 nAnT-iCAGE CTSS는 트루 세트로 사용되었습니다. ROC 곡선의 비교는 SLIC-CAGE가 CTSS 식별에서 나노케이지를 능가한다는 것을 보여줍니다. 어레이익스프레스 E-MTAB-6519의 데이터가 사용되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 1: 담체 합성 유전자의 서열. I-SceI 사이트는 보라색으로 대담하고 기울임꼴로 표시되며 I-CeuI 인식 사이트는 녹색입니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
| 캐리어 | 역프라이머 5'-3' | PCR 제품 길이 / bp | |
| 1개 | PCR_N6_r1: NNNNNNCTACGTGTCGCAGACGAATT | 1034년 | |
| 2개 | PCR_N6_r2: NNNNNNTATATCCAGATCGTTGAGCTGC | 966 | |
| 3개 | PCR_N6_r3: NNNNNCACTGGGCTTTACG | 889 | |
| 4개 | PCR_N6_r4: NNNNNNGCCGTCGATAACTTTTCGTTCGTTTCGT | 821 | |
| 5개 | PCR_N6_r5: NNNNNagTTGACCGCAGTTTC | 744 | |
| 6개 | PCR_N6_r6: NNNNNgTGAAGAATTTTTCCCCCCCCCC | 676 | |
| 7명 | PCR_N6_r7: NNNNNNCTCGCGCGTT카타AC | 599 | |
| 8개 | PCR_N6_r8: NNNNN타타CGCGGTGTCGTAC | 531 | |
| 9개 | PCR_N6_r9: NNNNNACCCCCGCCTTGCAGG | 454 | |
| 10개 | PCR_N6_r10: NNNNNNCAGGACGTTTTTGCCCAGCA | 386 | |
| * 전달 프라이머는 모든 캐리어 템플릿에 대해 동일합니다. 밑줄은 T7 프로모터 서열이다. PCR_GN5_f1: 타타그액카타타GNNNNNCAGCGCGCTCTCTCtTA | |||
표 2: 캐리어 템플릿 증폭을 위한 프라이머. 전달 프라이머는 모든 캐리어 템플릿에 대해 동일합니다. 밑줄은 T7 프로모터 서열이다. PCR_GN5_f1: 타타치아그락카타타GNNNNNCAGCGCGCTCTCTCtA. 상이한 역프라이머를 사용하여, PCR 템플릿 및 따라서 다른 길이의 캐리어 RNA가 생성된다.
| 캐리어 | 길이 | 제한되지 않은/μg | 캡 /μg |
| 1개 | 1034년 | 3.96 | 0.45 |
| 2개 | 966 | 8.36 | 0.95 |
| 3개 | 889 | 4.4 | 0.5 |
| 4개 | 821 | 6.6 | 0.75 |
| 5개 | 744 | 4.4 | 0.5 |
| 6개 | 676 | 3.08 | 0.35 |
| 7명 | 599 | 4.4 | 0.5 |
| 8개 | 531 | 3.96 | 0.45 |
| 9개 | 454 | 2.64 | 0.3 |
| 10개 | 386 | 2.2 | 0.25 |
표 3: RNA 담체 혼합. 캐리어 믹스 0.3-1 kbp의 총 49 μg: 언캡 = 44 μg, 캡핑 = 5 μg.
| 총 RNA 입력 /ng | PCR 주기 |
| 1 ng | 18세 |
| 2 ng | 17세 |
| 5 ng | 16세 |
| 10 ng | 15-16 |
| 25 ng | 14-15 |
| 50 ng | 13-15 |
| 100 ng | 12-14 |
표 4: 샘플 총 RNA 입력에 대한 의존성에서 예상되는 PCR 사이클 수입니다. 대략적인 사이클 수는 사카로미세세스 세레비시아, 드로소필라 멜라노가스터 및 근막 총 RNA를 사용하여 수행된 실험을 기반으로 합니다.
| 총 RNA 입력/ng | 전체 매핑 % | % 고유 매핑 | % 캐리어 |
| 1 ng | 30개 | 20-30 | 30개 |
| 2 ng | 60 | 20-50 | 10개 |
| 5 ng | 60-70 | 40-60 | 5-10 |
| 10 ng | 60-70 | 40-60 | 5-10 |
| 25 ng | 65-80 | 40-70 | 0-5 |
| 50 ng | 65-80 | 40-70 | 0-3 |
| 100 ng | 70-85 | 40-70 | 0-2 |
표 5: 예상 매핑 효율 및 총 RNA 입력 량의 의존성. 대략적인 숫자는 사카로마이세스 세레비시아및 무스 근육 총 RNA를 사용하여 수행된 실험에 기초하여 제시되고 기초한다.
토론
성공적인 SLIC-CAGE 라이브러리 준비의 경우, 시료 흡착으로 인한 시료 손실을 방지하기 위해 낮은 결합 팁과 튜브를 사용하는 것이 중요합니다. 상급물의 검색과 관련된 모든 단계에서 전체 샘플 볼륨을 복구하는 것이 좋습니다. 프로토콜에 여러 단계가 있기 때문에 연속 샘플 손실은 실패한 라이브러리로 이어질 수 있습니다.
CAGE(nAnT-iCAGE)가 일상적으로 수행되지 않은 경우, 동일한 총 RNA 샘플의 다른 입력 량(10 ng, 20 ng, 50 ng, 100 ng, 200 ng)으로 SLIC-CAGE를 테스트하고 총 RNA 5 μg를 사용하여 제조된 nAnT-iCAGE 라이브러리와 비교하는 것이 가장 좋습니다. nAnT-iCAGE 라이브러리가 실패하는 경우(샘플당 얻은 DNA 라이브러리의 0.5-1 ng 미만), SLIC-CAGE는 작동하지 않을 수 있으며 샘플 손실을 최소화해야 합니다.
상한 되지 않은 RNA 또는 rRNA가 없는 고품질 라이브러리를 보장하는 중요한 단계는 섹션 7에 설명된 캡 트래핑입니다. 스트렙타비딘 비드는 세척 버퍼에 완전히 다시 매달려 있고 세척 버퍼가 다음 세척 단계 또는 cDNA 용출을 계속하기 전에 제거되는 것이 매우 중요합니다.
캐리어 성능 저하의 첫 번째 라운드 후 qPCR의 결과가 adaptor_f1 및 carrier_f1 프라이머의 사용 간에 차이가 없는 경우 프로토콜을 계속 사용하는 것이 좋습니다. 캐리어 저하의 두 번째 라운드 후, Ct 값의 차이가 5 보다 작은 경우, 캐리어 저하의 세 번째 라운드를 권장 합니다. 우리는 필요한 저하의 세 번째 라운드를 발견 한 적이 없다, 그리고 발생하는 경우, 호밍 endonuclease 주식을 교체하는 것이 좋습니다.
얻어진 라이브러리의 최종 양이 시퀀싱을 위해 충분하지 않은 경우 PCR 증폭의 추가 라운드가 프로토콜에 추가될 수 있다. PCR 증폭은 크기 선택에서 피할 수 없는 샘플 손실을 고려하여 시퀀싱에 충분한 재료를 산출하는 데 필요한 최소한의 증폭 주기로 설정할 수 있습니다. SPRI 자기 비드를 사용하여 정화 또는 크기 선택은 모든 작은 (&200 bp) 조각이 제거 될 때까지 수행되어야합니다 (필요한 경우, 샘플 비율에 0.6 :1 구슬을 사용), 라이브러리는 Picogreen을 사용하여 정량화되어야한다.
라이브러리는 단일 엔드 또는 페어링 된 엔드 모드에서 시퀀스할 수 있습니다. 페어링된 말단 시퀀싱을 사용하여 전사체 이소폼에 대한 정보를 얻을 수 있습니다. 또한, 역전사가 랜덤 프라이머(TCT-N 6,N6 임의 의 hexamer인)를 사용하여 수행됨에 따라, 서열화된 3'-말단으로부터의 정보는 PCR 중복을 붕괴시키는 고유 분자 식별자(UMI)로서 사용될 수 있다. 적당한 수의 PCR 증폭 주기가 사용됨에 따라(최대 18), UMI의 사용은 이전에 불필요한 것으로 밝혀졌다.
프로토콜의 핵심은 nAnT-iCAGE11에의존하기 때문에 SLIC-CAGE는 8개의 바코드를 사용합니다. 따라서 8개 이상의 샘플을 다중화하는 것은 현재 지원되지 않습니다. 또한 SLIC-CAGE와 nAnT-iCAGE는 AMPure XP 비드를 사용하여 크기 제외를 통해 링커와 PCR 아티팩트를 제거하도록 설계되었기 때문에 200bp 미만의 RNA를 캡처하는 데 적합하지 않습니다.
SLIC-CAGE는 전체 RNA 물질의 나노그램을 사용하여 전사 개시 부위를 매핑하는 유일한 편견없는 저입력 단일 뉴클레오티드 분해 방법이다. 대안적인 방법은 캡 트래핑 대신에 역전사의 템플릿 스위칭 활성을 바코드 캡핑 RNA(예를 들어, NanoCAGE15 및 NanoPARE16)에의존한다. 템플릿 스위칭으로 인해 이러한 메서드는 TSS 검출에서 시퀀스 별 편향을 나타내며, 거짓 양성 TSS의 수가 증가하고 실제 TSS9,10의수가 감소합니다.
공개
분해성 담체 RNA/DNA에 대한 특허가 작성되었습니다.
감사의 말
이 작품은 웰컴 트러스트 보조금에 의해 지원되었다 (106954) B. L. 및 의료 연구위원회에 수여 (MRC) 핵심 자금 (MC-A652-5QA10). N. C.는 EMBO 장기 펠로우십(EMBO ALTF 1279-2016)의 지원을 받았습니다. E. P. 의학 연구 위원회 영국에 의해 지원 되었다; B. L. 의료 연구 위원회 영국에 의해 지원 되었다 (MC UP 1102/1).
자료
| Name | Company | Catalog Number | Comments |
| 2-propanol, Bioultra, for molecular biology, ≥99.5% | Sigma-Aldrich | 59304-100ML-F | Used in RNAclean XP purification. |
| 3' linkers | Sequences are described in Murata et al. 2014 and Supplementary Table 1 of this manuscript. Annealing of strands to produce 3'linkers is described in the supplementary of this protocol. | ||
| 5' linkers | Sequences are described in Murata et al. 2014 and Supplementary Table 1 of this manuscript. Annealing of strands to produce 5'linkers is described in the supplementary of this protocol. | ||
| Agencourt AMPure XP, 60 mL | Beckman Coulter | A63881 | Purification of DNA |
| Agencourt RNAClean XP Kit | Beckman Coulter | A63987 | Purification of RNA and RNA:cDNA hybrids in CAGE steps. |
| Axygen 0.2 mL Polypropylene PCR Tube Strips and Domed Cap Strips | Axygen (available through Corning) | PCR-0208-CP-C | Or any 8-tube PCR strips (used only for water and mixes). |
| Axygen 1 x 8 strip domed PCR caps | Axygen (available through Corning) | PCR-02CP-C | Caps for PCR plates. |
| Axygen 1.5 mL Maxymum Recovery Snaplock Microcentrifuge Tube | Axygen (available through Corning) | MCT-150-L-C | Low-binding 1.5 mL tubes, used for enzyme mixes or sample concentration. |
| Axygen 96 well no skirt PCR microplate | Axygen (available through Corning) | PCR-96-C | Low-binding PCR plates - have to be used for all steps in the protocol. Note that plates should be cut to contain 2 x 8 wells for easier visibility of the samples |
| Bioanalyzer (or Tapestation): RNA nano and HS DNA kits | Agilent | To determine quality of RNA, efficient size selection and final quality of the library (Tapestation can also be used) | |
| Biotin (Long Arm) Hydrazide | Vector laboratories | SP-1100 | Biotinylation/tagging |
| Cutsmart buffer | NEB | Restriction enzyme buffer | |
| Deep Vent (exo-) DNA Polymerase | NEB | M0259S | Second strand synthesis |
| DNA Ligation Kit, Mighty Mix | Takara | 6023 | Used for 5' and 3'-linker ligation |
| dNTP mix (10 mM each) | ThermoFisher Scientific | 18427013 | dNTP mix for production of carrier templates (or any dNTPs suitable for PCR) |
| Dynabeads M-270 Streptavidin | Invitrogen | 65305 | Cap-trapping. Do not use other beads as these are optimised with the buffers used. |
| DynaMag-2 Magnet | ThermoFisher Scientific | 12321D | Magnetic stand for 1.5 mL tubes - used to prepare Streptavidin beads. |
| DynaMag-96 Side Skirted Magnet | ThermoFisher Scientific | 12027 | Magnetic stand for PCR plates (96 well-plates) - used with cut plates to contain 2 x 8 wells. |
| Ethanol, BioUltra, for molecular biology, ≥99.8% | Sigma-Aldrich | 51976-500ML-F | Used in AMPure washes. Any molecular biology suitable ethanol can be used. |
| Exonuclease I (E. coli) | NEB | M0293S | Leftover primer degradation |
| Gel Loading Dye, Purple (6x), no SDS | NEB | B7025S | agarose gel loading dye |
| HiScribe T7 High Yield RNA Synthesis Kit | New England Biolabs | E2040S | Kit for carrier in vitro transcription |
| Horizontal electrophoresis apparatus | purification of carrier DNA templates from agarose gels | ||
| I-Ceu | NEB | R0699S | Homing endonuclease used for carrier degradation. |
| I-SceI | NEB | R0694S | Homing endonuclease used for carrier degradation. |
| KAPA HiFi HS ReadyMix (2x) | Kapa Biosystems (Supplied by Roche) | KK2601 | PCR mix for target library amplification |
| KAPA SYBR FAST qPCR kit (Universal) 2x | Kapa Biosystems (Supplied by Roche) | KK4600 | qPCR mix to assess degradation efficiency and requiered number of PCR amplification cycles |
| Micropipettes and multichannel micropipettes (0.1-10 µL, 1-20 µL, 20-200 µL) | Gilson | Use of Gilson with the low-binding Sorenson tips is recommended. Other micropippetes might not be compatible. Different brand low-binding tips may not be of equal quality and may increase sample loss. | |
| Microplate reader | For Picogreen concentration measurement of the final library. Microplates are used to allow small volume measurement and reduce sample waste. | ||
| nuclease free water | ThermoFisher Scientific | AM9937 | Or any nuclease (DNase and RNase) free water |
| PCR thermal cycler | incubation steps and PCR amplficication | ||
| Phusion High-Fidelity DNA Polymerase | ThermoFisher Scientific | F530S | DNA polymerase for amplification of carrier templates (or any high fidelity polymerase) |
| QIAquick Gel Extraction Kit (50) | Qiagen | 28704 | Purification of carrier PCR templates from agarose gels. |
| qPCR machine | determining PCR amplification cyle number and degree of carrier degradation | ||
| Quant-iT PicoGreen dsDNA Reagent | ThermoFisher Scientific | P11495 | Used to measure final library concentration - recommended as, in our hands, it is more accurate and reproducible than Qubit. |
| Quick-Load Purple 100 bp DNA Ladder | NEB | N0551S | DNA ladder |
| Quick-Load Purple 1 kb Plus DNA Ladder | NEB | N0550S | DNA ladder |
| Ribonuclease H | Takara | 2150A | Digestion of RNA after cap-trapping. |
| RNase ONE Ribonuclease | Promega | M4261 | Degradation of single stranded RNA not protected by cDNA. |
| RNase-Free DNase Set | Qiagen | 79254 | Removal of carrier DNA templates after in vitro transcription. |
| RNeasy Mini Kit | Qiagen | 74104 | For cleanup of carrier RNA from in vitro transcription or capping |
| Sodium acetate, 1 M, aq.soln, pH 4.5 RNAse free | VWR | AAJ63669-AK | Or any nuclease (DNase and RNase) free solution |
| Sodium acetate, 1 M, aq.soln, pH 6.0 RNAse free | Or any nuclease (DNase and RNase) free solution | ||
| Sodium periodate | Sigma-Aldrich | 311448-100G | Oxidation of vicinal diols |
| Sorenson low binding aerosol barrier tips, MicroReach Guard, volume range 10 μL, Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719390-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 1,000 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719463-1000EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 20 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719412-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| Sorenson low binding aerosol barrier tips, MultiGuard, volume range 200 μL , Graduated | Sorenson (available through SIGMA-ALDRICH) | Z719447-960EA | Low-binding tips - recommended use throughout the protocol to minimise sample loss. |
| SpeedVac Vacuum Concentrator | concentrating samples in various steps to lower volume | ||
| SuperScript III Reverse Transcriptase | ThermoFisher Scientific | 18080044 | Used for reverse transcription (1st CAGE step) |
| Trehalose/sorbitol solution | Preparation is described in Murata et al. 2014. | ||
| Tris-HCl, 1 M aq.soln, pH 8.5 | 1 M solution, DNase and RNase free | ||
| tRNA (20 mg/mL) | tRNA solution. Preparation is described in Murata et al. 2014. | ||
| UltraPure Low Melting Point Agarose | ThermoFisher Scientific | 16520050 | Or any suitable pure low-melt agarose. |
| USB Shrimp Alkaline Phosphatase (SAP) | Applied Biosystems (Provided by ThermoFisher Scientific) | 78390500UN | |
| USER Enzyme | NEB | M5505S | Degradation of 3'linker's upper strand, Uracil Specific Excision Reagent/Enzyme |
| Vaccinia Capping System | NEB | M2080S | Enzymatic kit for in vitro capping of carrier molecules |
| Wash buffer A | Cap trapping washes. Preparation is described in Murata et al. 2014. | ||
| Wash buffer B | Cap trapping washes. Preparation is described in Murata et al. 2014. | ||
| Wash buffer C | Cap trapping washes. Preparation is described in Murata et al. 2014. |
참고문헌
- Shiraki, T., et al. Cap analysis gene expression for high-throughput analysis of transcriptional starting point and identification of promoter usage. Proceedings of the National Academy of Sciences of the United States of America. 100 (26), 15776-15781 (2003).
- Haberle, V., Lenhard, B. Promoter architectures and developmental gene regulation. Seminars in Cell and Developmental Biology. 57, 11-23 (2016).
- Haberle, V., Stark, A. Eukaryotic core promoters and the functional basis of transcription initiation. Nature Reviews Molecular Cell Biology. 19 (10), 621-637 (2018).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Celniker, S. E., et al. Unlocking the secrets of the genome. Nature. 459 (7249), 927-930 (2009).
- Consortium, F., et al. A promoter-level mammalian expression atlas. Nature. 507 (7493), 462-470 (2014).
- Boyd, M., et al. Characterization of the enhancer and promoter landscape of inflammatory bowel disease from human colon biopsies. Nature Communications. 9 (1), 1661(2018).
- Adiconis, X., et al. Comprehensive comparative analysis of 5'-end RNA-sequencing methods. Nature Methods. , (2018).
- Cvetesic, N., et al. SLIC-CAGE: high-resolution transcription start site mapping using nanogram-levels of total RNA. Genome Research. 28 (12), 1943-1956 (2018).
- Murata, M., et al. Detecting expressed genes using CAGE. Methods in Molecular Biology. 1164, 67-85 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9, 357(2012).
- R Core Team. A language and environment for statistical computing. , Available from: https://www.R-project.org/ (2017).
- Haberle, V., Forrest, A. R., Hayashizaki, Y., Carninci, P., Lenhard, B. CAGEr: precise TSS data retrieval and high-resolution promoterome mining for integrative analyses. Nucleic Acids Research. 43 (8), e51(2015).
- Poulain, S., et al. NanoCAGE: A Method for the Analysis of Coding and Noncoding 5'-Capped Transcriptomes. Methods in Molecular Biology. 1543, 57-109 (2017).
- Schon, M. A., Kellner, M. J., Plotnikova, A., Hofmann, F., Nodine, M. D. NanoPARE: parallel analysis of RNA 5' ends from low-input RNA. Genome Research. 28 (12), 1931-1942 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유