Method Article
세포 주기의 G1 단계에서 단일 가닥 DNA 병소 시각화
요약
다음 프로토콜은 세포주기 동기화 후 RPA2 면역형광 염색을 사용하여 세포주기의 G1 단계에서 단일 가닥 DNA 병소를 검출하는 방법을 제시합니다.
초록
DNA는 내인성 및/또는 외인성 원인 모두에서 발생할 수 있는 병변에 대처할 수 있는 전용 세포 복구 경로를 가지고 있습니다. DNA 복구는 DNA 병변의 존재를 인식하고 신호를 보내는 것부터 물리적으로 복구하는 것까지 광범위한 작업을 담당하는 수많은 단백질 간의 협력을 필요로 합니다. 이 과정에서 단일 가닥 DNA(ssDNA)의 흔적이 종종 생성되며, 이는 결국 DNA 중합효소로 채워집니다. 이러한 ssDNA 트랙의 특성(길이와 수 모두)은 이러한 간격을 메우기 위해 모집된 중합효소와 함께 복구 경로 특이적입니다. 이러한 ssDNA 트랙의 시각화는 DNA 복구 메커니즘의 복잡한 역학을 이해하는 데 도움이 될 수 있습니다.
이 프로토콜은 유전독성 스트레스에 따른 ssDNA 병소 형성을 측정하기 위해 G1 동기화 세포를 준비하기 위한 자세한 방법을 제공합니다. 활용하기 쉬운 면역형광 접근법을 사용하여 이질삼합체 복제 단백질 A 복합체(RPA)의 구성 요소인 RPA2를 염색하여 ssDNA를 시각화합니다. RPA2는 유전독성 스트레스 또는 복제 시 발생하는 ssDNA 중간체에 결합하고 안정화하여 DNA 복구 및 DNA 손상 체크포인트 활성화를 제어합니다. 5-에티닐-2'-데옥시우리딘(EdU) 염색은 S상 세포를 배제하기 위해 DNA 복제를 시각화하는 데 사용됩니다. 이 프로토콜은 기존의 비변성 5-bromo-2'-deoxyuridine(BrdU) 기반 분석에 대한 대안적 접근법을 제공하며 S상 외부의 ssDNA 병소 검출에 더 적합합니다.
서문
생명을 유지하기 위해 세포는 게놈 무결성을 유지하기 위해 DNA를 지속적으로 조사하고 복구합니다. 세포는 DNA 스트레스 요인의 내인성(예: 산화, 알킬화, 탈아미노화, 복제 오류) 및 외인성(예: UV, 이온화 조사) 소스로 인해 다양한 유형의 DNA 손상을 축적할 수 있습니다. 이러한 병변을 복구하지 못하면 세포사멸, 세포주기 정지 또는 노화가 발생하여 질병으로 이어질 수 있다1. DNA 병변은 다음과 같은 주요 DNA 복구 경로 중 하나로 해결할 수 있습니다: DR(직접 역전 복구), 주로 알킬화된 염기를 복구합니다2; 부피가 크지 않은 DNA 염기 오류 및 단일 가닥 DNA 절단(SSB)을 표적으로 하는 BER(염기 절제 복구)3; 부피가 크고 나선으로 왜곡된 DNA 병변을 교정하는 NER(뉴클레오티드 절제 복구)4; MMR(mismatch repair)은 주로 DNA 불일치, 삽입/삭제 루프(IDL) 및 특정 염기 손상을 표적으로 합니다5; 이중 가닥 DNA 절단(DSB)에서 모두 활성화되는 NHEJ(비상동 말단 결합) 및 HRR(상동 재조합 복구)6; 및 TLS (translesion synthesis), 이는 DNA 병변 우회 메커니즘7입니다. 이러한 경로는 뚜렷한 기질 특이성을 가지고 있지만, 효율적인 수리를 위한 중복성을 보장하기 위해 이들 사이에 특정 겹침이 있습니다. 이러한 DNA 복구 인자는 암, 노화 및 신경 장애를 치료하기 위한 치료 접근법의 필수 표적이 될 수 있기 때문에 다양한 세포 주기 단계에서 다양한 DNA 복구 경로의 작용을 이해하는 것이 중요합니다 8,9.
단일 가닥 DNA(ssDNA)는 내인성 및 외인성 DNA 손상 물질에 의해 생성된 DNA 병변의 복구로 인해 세포 주기 전반에 걸쳐 생성됩니다. 유전독성 스트레스에 시달리면, ssDNA는 HRR과 MMR이 가장 높은 활성을 갖는 S 및 G2 단계에서 풍부하게 생성되며, DNA 병변을 만났을 때 복제 기계가 멈추거나 붕괴될 때 생성된다 6,10,11. 다른 DNA 복구 경로(예: NHEJ/microhomology-mediated end joining(MMEJ)/single-strand annealing [SSA])도 DSB 복구 동안 ssDNA를 생성한다12. 이러한 ssDNA 흔적은 일반적으로 HR 및 MMR 동안 EXO1, DNA2 및 CtIP와 같은 엑소뉴클레아제, NER 중 XPF 및 XPG와 같은 엔도뉴클레아제, 또는 BER 4,13,14,15,16,17,18,19 동안 POLB와 FEN1의 결합 작용을 통해 수행되는 DNA 절제에서 발생합니다 . 복제 기계의 일로 인해, DNA 헬리카제가 PCNA-결합 복제 중합효소(20) 앞에서 DNA를 풀 때 ssDNA 트랙도 생성된다. 대조적으로, G1 단계에서는 HRR 및 DNA 복제의 부족과 MMR의 제한된 활성으로 인해 생성된 ssDNA 추적의 범위가 줄어들어 검출하기가 더 어렵습니다10,11,21.
세포 ssDNA 추적은 DSB의 형성을 방지하기 위해 보호해야 하는 매우 민감한 구조입니다. 이는 ssDNA 트랙을 RPA로 코팅하여 달성됩니다. RPA는 세포 주기전반에 걸쳐 유비쿼터스로 발현되는 여러 소단위체(RPA1, RPA2 및 RPA3, 각각 RPA70, RPA32 및 RPA14라고도 함)로 구성된 풍부한 이종 삼합체 단백질 복합체입니다. 각 RPA 소단위체는 4-6개의 뉴클레오티드와 상호 작용할 수 있는 DNA 결합 도메인(DBD)을 포함하며, 결합된 소단위체는 안정적인 삼량화 코어를 형성합니다. 전체적으로, RPA는 서브 나노몰 친화도23,24를 가진 대략 20-30의 뉴클레오티드에 결합한다.
종래의 방법은 면역형광(IF) 현미경을 사용하여 BrdU 항체25를 사용하여 게놈 DNA에 통합된 5-bromo-2'-deoxyuridine(BrdU)을 표지하여 ssDNA 병소를 시각화합니다. 이 접근법은 BrdU 항체가 노출된 ssDNA25에서만 BrdU를 검출할 수 있다는 사실에 의존합니다. 이 방법은 간단하지만 몇 가지 제한 사항도 있습니다. 예를 들어, 세포는 실험을 시작하기 전에 BrdU를 통합하도록 전처리되며, 이는 시간이 많이 걸리고 다운스트림 이펙터를 방해할 수 있습니다. 따라서 BrdU 기반 ssDNA 검출은 복제 세포에 국한되며 정지 세포에는 사용할 수 없습니다. 이것은 암과 neurodegeneration과 같은 몇몇 질병에 있는 그것의 중요성에도 불구하고 non-replicating 세포에 있는 DNA 수선을 공부하기 위하여 이 방법의 신청을 제외한다 5,26. 또한, BrdU와 EdU의 구조가 매우 유사하기 때문에, 대부분의 BrdU 항체는 EdU에 대해 교차반응을 나타내는데, 이는 이중 표지 실험을 목표로 할 때 반드시 고려되어야 한다27. RPA 염색은 이전에 주로 S상 세포에서 ssDNA 병소를 보여주기 위해 사용되었습니다. 그러나 일부 논문은 S 상 28,29,30,31,32,33,34,35 밖에서도 성공적으로 사용했습니다. 다음 프로토콜은 RPA의 속성을 효율적으로 활용하여 세포 주기의 G1 단계에서 DNA 손상에 따른 ssDNA 병소를 시각화할 수 있습니다(모든 세포 주기 단계에서 사용할 수 있음).
프로토콜
1. hTERT에 의한 불멸화된 망막 색소 상피세포(RPE1)의 유지
- 37°C에서 5%CO2 가 있는 가습 인큐베이터에서 10% 열 비활성화 소 태아 혈청(Hi-FBS) 및 100μg/mL 페니실린-스트렙토마이신(지금부터 배양 배지라고 함)이 보충된 Dulbecco의 DMEM(Modified Eagle Medium)에서 RPE1 세포주를 유지합니다. 일상적인 배양의 경우 15cm 조직 배양 처리된 접시에서 RPE1 세포를 성장시키고 80-90% 밀도(15cm 접시당 ~16-18 × 106 세포)에 도달하면 분할합니다.
- 분할할 때 배지를 제거하고 10mL의 1x 인산염 완충 식염수(PBS)로 세포를 헹굽니다.
- 3mL의 0.05% 트립신-EDTA를 추가하여 접시 표면 전체를 덮습니다. 세포가 분리될 때까지 트립신을 사용하여 세포를 37°C로 유지합니다.
- 트립신화 후 세포를 배양 배지로 재현탁시키고 실온(RT, 22-25°C)에서 5분 동안 150× g 에서 스핀다운합니다. 상층액을 제거하고 10mL의 배양 배지에 세포를 부드럽게 재현탁시킵니다.
- 1.6-1.8 × 106 세포를 새로운 15cm 접시에 파종합니다(세포 현탁액의 ~1mL).
알림: 모든 조직 배양 작업은 BSL-2 안전 수준에서 수행해야 합니다. 트립신화를 위한 배양 시간은 세포 밀도에 따라 다릅니다. 일반적으로 이 프로세스는 2% 합류 플레이트에 대해 완료하는 데 3-90분이 걸립니다. 세포는 상업적으로 이용 가능한 키트를 사용하여 정기적으로 마이코플라스마 오염을 검사해야 합니다( 재료 표의 예 참조).
2. siRNA knocking of the interest gene (GOI)
- 형질주입 전날 10mL의 배양 배지가 있는 10cm 조직 배양 처리 플레이트에 1.0 × 106 RPE1 세포를 파종합니다.
- transfection 당일, siRNA를 복합체화합니다. 10cm 플레이트의 경우 500μL의 저혈청 transfection 배지에 20nM siRPA2 및 12μL의 지질 기반 transfection 시약의 최종 농도를 사용합니다. 튜브를 튕겨 모든 성분을 부드럽게 혼합하고 RT(22-25°C)에서 5분 동안 배양합니다.
- 복합화된 siRNA 혼합물을 세포에 적가하고 siRNA로 세포를 48시간 동안 배양합니다.
3. RPE1 셀을 G0 단계로 동기화
- 섹션 1에 설명된 대로 2.3단계에서 RPE1 세포를 트립시화합니다(~2 × 106 개 세포).
- 셀 현탁액을 15mL 원심분리 튜브로 옮기고 150× g, RT(22-25°C)에서 5분 동안 원심분리합니다.
- 상층액을 제거하고 세포를 12mL의 PBS에 재현탁시킵니다. RT(22-25°C)에서 150× g 에서 5분 동안 세포를 원심분리합니다. 상층액 제거와 원심분리를 두 번 반복합니다.
- 100μg/mL 페니실린-스트렙토마이신, 1mM 피루브산 나트륨, 15mM HEPES가 보충된 무혈청 DMEM 10mL에 세포를 재현탁시키고 10cm 조직 배양 접시에 담습니다.
참고: 세포가 뭉치는 경향이 있는 경우 1mL의 무혈청 DMEM에 재현탁시키고 P1000 팁을 사용하여 위아래로 5배 피펫팅하여 덩어리를 제거한 후 현탁액을 최종 부피 10mL까지 희석합니다. - 혈청 결핍 24시간 후, 혈청 결핍 세포에 복합 siRNA를 추가하여 섹션 2에 설명된 것과 동일한 절차를 사용하여 두 번째 침묵을 도입합니다.
- G1 방출을 진행하기 전에 RPE1 세포를 혈청이 없는 DMEM에 72시간 동안 유지합니다.
4. 커버슬립 코팅 및 G1 단계로 세포 방출
- 70% 에탄올로 핀셋을 멸균하고 단일 유리 커버슬립(직경 12mm, #1.5 두께[0.17mm])을 24웰 플레이트의 웰에 넣습니다.
- 비트로넥틴 코팅 매트릭스를 PBS로 희석하여 최종 농도 10μg/mL를 얻습니다. 500μL의 비트로넥틴 용액을 커버슬립이 들어 있는 각 웰에 넣고 RT에서 1시간 동안 배양합니다.
- 코팅 용액을 제거하고 PBS 1mL로 커버슬립을 세척합니다.
- 37°C에서 1분 동안 PBS 세척 후 0.05% 트립신 1mL를 사용하여 10cm 조직 배양 처리 플레이트에서 혈청 결핍 RPE1 세포를 분리합니다.
참고: 혈청 결핍 후 세포가 훨씬 더 빨리 분리됩니다. PBS로 세포를 세척할 때 주의하고 짧은 트립신화 시간을 사용하십시오. - 트립신을 비활성화하려면 총 6mL의 배양 배지에 RPE1 세포를 재현탁시킵니다. RT(22-25°C)에서 5분 동안 150× g 을 사용하여 세포를 회전시켜 비활성화된 트립신을 제거합니다.
- 배양 배지 1mL에 세포를 재현탁시키고 세포 수를 측정합니다.
- 총 500μL의 배양 배지에서 코팅된 커버슬립에 4 ×10 개의 RPE1 세포를 파종합니다.
알림: 다운스트림 단계를 진행하기 전에 세포 생존율이 90% 이상인지 확인하십시오. 세포 생존율은 세포 계수 단계에서 트리판 블루 염색을 통해 신속하게 평가할 수 있습니다. - 세포를 배양 배지에 도금한 지 6시간이 지나면 G0 방출 세포는 초기 G1 단계가 됩니다. 세포가 S 단계로 들어가기 시작하기 전에 이 6-12시간 동안 G1에서 실험을 수행합니다.
- DNA 손상을 도입하기 전에 배양 배지에 희석하여 37°C에서 30분 동안 10μM 5-ethynyl-2'-deoxyuridine(EdU)으로 세포를 펄스합니다.
- EdU가 포함된 배지를 제거하고 DNA 손상 유도 중 남아 있는 EdU 혼입을 방지하기 위해 37°C에서 10분 동안 10μM 티미딘으로 세포를 추적합니다.
- 티미딘으로 배지를 제거하고 배양 배지에 희석하여 1시간 동안 250μM H2O2로 세포를 처리합니다.
5. ssDNA의 면역형광 염색
- 1mL의 RT(22-25°C) PBS로 세포를 한 번 세척하여 배지 및 혈청 성분을 제거합니다.
알림: 셀이 분리되거나 건조되지 않도록 셀을 세척할 때 부드럽게 하십시오. 동시에 많은 웰을 처리하지 마십시오. - 추출 전: 세척된 세포를 1mL의 CSK 추출 완충액(표 1)에 넣고 RT(22-25°C)에서 5분 동안 배양합니다.
참고: CSK 사전 추출은 가용성 RPA2를 포함하여 염색질에 결합되지 않은 모든 단백질을 제거합니다.
주의 : Triton X-100은 삼키면 유해하며 피부 자극 및 눈 손상을 유발할 수 있습니다. - 세포에서 CSK 완충액을 제거하고 RT(22-25°C)에서 10분 동안 0.05% Triton X-100을 함유한 3.6% 파라포름알데히드 용액(PBS) 0.5mL를 첨가하여 직접 고정합니다.
주의 : 32 % PFA 스톡에서 3.6 % PFA를 신선하게 준비하는 것이 중요합니다. 파라포름알데히드는 심각한 눈 손상, 피부 자극 및 호흡기 자극을 유발할 수 있습니다. - PFA를 제거하기 위해 0.05% Triton X-100이 함유된 PBS 1mL로 세포를 한 번 세척합니다.
- RT (22-25 °C)에서 15 분 동안 0.5 % Triton X-100을 함유 한 PBS 1mL를 사용하여 세포를 추가로 투과시킵니다.
- 복제 세포 시각화를 위한 EdU click-IT 반응(S상)
- 투과화 용액을 제거하고 1mL의 차단 완충액을 사용하여 세포를 2회 세척합니다(표 1).
주의: 소 혈청 알부민(BSA)은 호흡기 자극을 유발할 수 있습니다. - 1mL의 차단 완충액(표 1)을 추가하고 뚜껑 슬립 함유 플레이트를 RT(22-25°C)에서 10분 동안 부드럽게 흔듭니다.
- 차단 완충액을 제거하고 피콜릴 아지드 647을 함유하는 500μL의 클릭 반응 칵테일을 추가합니다(표 1). 부드러운 흔들기를 사용하여 RT(22-25°C)에서 30분 동안 커버슬립을 배양하고 어둠 속에서 다운스트림 배양을 수행합니다.
참고: BrdU 항체를 사용할 때는 제조업체에서 권장하는 대로 클릭 반응에 두 배의 양(1mL)과 시간(60분)을 사용하여 반응이 포화 상태이고 통합된 EdU가 라벨링되도록 합니다. 이는 BrdU 항체(27)의 교차-반응성을 제한한다.
- 투과화 용액을 제거하고 1mL의 차단 완충액을 사용하여 세포를 2회 세척합니다(표 1).
- 클릭 반응 혼합물을 제거하고 RT(22-25°C)에서 10분 동안 0.05% Triton X-100을 사용하여 PBS로 세포를 2배 세척합니다(그림 1 및 그림 2).
- 1mL의 차단 완충액을 추가하고 실온(22-25°C)에서 30분 동안 배양합니다. 또는 세포를 4°C의 차단 완충액에 밤새 보관합니다.
- 1차 항체(anti-RPA2 rat, 1:1,000 희석)를 RT(22-25°C)에서 250-500μL의 차단 완충액에 2시간 동안 부드럽게 흔들어 적용합니다.
- 0.05% Triton X-100을 함유한 PBS로 세포를 2회 세척하여 대부분의 항체 용액을 빠르게 제거합니다.
- RT(22-25°C)에서 차단 완충액을 사용하여 3 x 10분 동안 셀을 계속 세척합니다.
- 2차 항체(anti-rat Alexa-488, 1:1,000 희석)를 RT(22-25°C)에서 2시간 동안 부드럽게 흔들면서 250-500μL의 차단 완충액에 적용합니다.
- 차단 완충액 2배로 세포를 세척하여 대부분의 2차 항체를 빠르게 제거합니다. RT(22-25°C)에서 0.05% Triton X-100을 함유한 PBS로 3 x 10분 동안 셀을 계속 세척합니다.
- 핵을 대조염색하려면 0.05% Triton X-100 및 1μg/mL 4',6-diamidino-2-phenylindole(DAPI)을 함유한 PBS로 RT(22-25°C)에서 10분 동안 세포를 한 번 세척합니다. RT (22-25 ° C)에서 5 분 동안 PBS로 세포를 한 번 세척하십시오.
- 10μL의 장착 매체/커버슬립을 사용하여 커버 유리를 현미경 슬라이드에 장착합니다. 소금 결정을 제거하기 위해 장착하기 전에 커버슬립을 증류수에 담그십시오. 다음 날 슬라이드를 이미지화하고 4°C에서 몇 주 동안 보관합니다(그림 3).
6. 이미지 획득 및 정량화
- 이미지를 캡처하려면 일상적인 필터 세트가 장착된 사용 가능한 모든 형광 현미경을 사용하여 최소 60-63x 배율, 높은 숫자 조리개 및 오일 대물렌즈로 DAPI, FITC 및 Cy5 채널을 이미지화하여 핵 초점을 시각화하십시오.
참고: 최적의 DAPI 여기(excitation)는 ~359nm입니다. Alexa 488 여기(excitation)는 ~488nm입니다. Alexa 647 여기가 ~ 647 nm입니다. - 이미지 분석을 위해 Fiji/ImageJ에서 이미지 파일을 엽니다.
- DAPI 염색을 사용하여 핵 마스크를 만듭니다(그림 4A-F 및 보충 비디오 S1).
- DAPI 이미지를 엽니다.
- 프로세스 | 대비를 향상시키고 채도가 높은 픽셀을 0.35로 설정합니다.
- 프로세스 | 바이너리 | 마스크로 변환. 바이너리 선택 | 구멍을 채우고 분석(Analyze) | 입자를 분석합니다. 크기를 10-무한대로 설정합니다.
- ROI 관리자에서 모두 표시를 클릭합니다.
- 핵에서 RPA2 병소 찾기(그림 4G,H 및 보충 비디오 S1)
- RPA2 이미지를 엽니다.
- 프로세스 | 최대값을 구합니다. 돌출부를 RPA2 초점(500에서 750 사이)을 강조 표시하는 값으로 설정하여 배경과 분리합니다.
- 마지막으로 ROI Manager에서 측정 버튼을 클릭합니다.
- RawinDen 열의 값을 255(각 초점의 픽셀 강도 최대값)로 나누어 핵 ssDNA 병소의 총 개수를 계산합니다.
- 선호하는 통계 소프트웨어 도구를 사용하여 통계 분석을 수행합니다.
참고: 모든 EdU 양성 세포와 부적절하게 분할된 DAPI 마스크는 분석에서 제외합니다.
- DAPI 염색을 사용하여 핵 마스크를 만듭니다(그림 4A-F 및 보충 비디오 S1).
결과
G1에서 ssDNA 검출의 한계를 극복하기 위해 RPA2를 활용했는데, 이는 ssDNA foci 검출의 특이성과 강도를 모두 향상시킨다35. 정확한 세포 동기화를 달성하기 위해 혈청이 결핍되어 G0 단계로 동기화될 수 있는 RPE1 세포를 사용했습니다. 그런 다음 혈청 박탈 후 혈청을 추가하여 세포주기에 다시 들어가도록 유도 할 수 있습니다. 동기화 효율을 확인하기 위해 세포에 EdU를 표시하고 DNA 함량을 프로피듐-요오드화물로 표지했습니다. 유세포 분석을 통해 정성적 및 정량적 결과를 추가로 수집했습니다(보충 그림 S1A). 점도표는 혈청 결핍 72시간 후 세포의 ~98%가 G0 단계에 있음을 보여줍니다. 6시간 동안 혈청 함유 배지를 첨가한 후, 세포는 세포주기에 재진입하며(그림 1A의 p27 수준 증가에서 볼 수 있듯이) G1에서 ~97%의 세포를 갖는 반면, S기에는 <1%, G2기에는 <2%의 세포만 있습니다(보충 그림 S1A). 세포에 혈청을 첨가한 후 20-28시간 동안 유세포 분석 플롯에 나타낸 바와 같이 세포는 점차적으로 S상을 통과합니다(보충 그림 S1A). 이 세포 동기화 프로토콜은 ~97%의 순수 G1 집단을 제공합니다(혈청 결핍 후 6시간 후 혈청 추가, 72시간). 동기화 효율을 추가로 검증하기 위해 웨스턴 블로팅(Western blotting)을 사용하여 혈청 방출 후 세포주기 마커의 발현을 비교하고(그림 1A 및 보충 그림 S1B) 동시에 EdU 통합 분석을 수행하여 DNA 복제를 시각화했습니다. EdU 염색은 또한 동기화 효율성과 G1 상에서의 DNA 복제 부족을 강조합니다(그림 1B,C).
포유류 세포에서 ssDNA를 검출하는 기존의 방법은 ssDNA에서 BrdU를 검출하는 데 의존합니다. 그림 2A는H2O2및 네오카르지노스타틴(NCS) 처리 시 BrdU 병소는 S상 세포에서만 검출할 수 있는 반면, S상이 아닌 세포에서는 ssDNA 병소가 검출되지 않았음을 보여줍니다. BrdU 항체 염색은 또한 세포 주기 단계 또는 적용된 처리와 무관하게 모든 핵에서 검출될 수 있는 눈에 띄는 핵 배경 염색을 보여주었습니다. 여기에 설명된 EdU 클릭 프로토콜을 사용하여 EdU 및 BrdU 초점의 공동 국소화를 검출할 수 없었으며, 이는 그림 2A의 처리되지 않은 샘플에서 분명합니다. 교차 반응에서 발생하는 BrdU 신호를 완전히 배제하기 위해 EdU 표지를 피하고 cyclin A2를 S-G2 마커로 사용했습니다. 그러나 cyclin A2 염색은 CSK 사전 추출을 허용하지 않았으며, 이 조건에서는 유전독성 스트레스 후에도 BrdU 병소가 관찰되지 않았습니다(보충 그림 S2A). 이는 항-BrdU 기반 ssDNA 염색에 CSK 사전 추출이 필요하다는 사실을 강조합니다. 대조군으로서 변성 조건에서 BrdU 항체 염색을 테스트했습니다. 이것은 통합된 BrdU를 노출시키기 위해 DNA를 열고, 이는 BrdU가 균일하게 통합되었음을 나타낸다(보충 그림 S2B).
대조적으로, RPA2 염색은 S상뿐만 아니라 다른 세포주기 상에서도 NCS 및H2O2의존성 병소 형성을 보여줍니다(그림 2B). 대조군으로서, 우리는 또한 복제가 진행되는 세포에서 ssDNA 축적을 유발하는 HU로 세포를 처리했습니다. 예상했던 대로, EdU 양성 세포에서 RPA2 항체로 HU를 처리했을 때만 신호 증가를 감지하여 이 접근법의 특이성을 강조했습니다. RPA2 항체는 또한 외인성 유전독성 스트레스가 없는 상태에서 복제 중에 자연적으로 발생하는 ssDNA 형성을 검출할 수 있습니다(그림 2B). RPA2 항체의 고감도 특성으로 인해 기존 BrdU 염색이 유전독성 스트레스에 대한 신호를 감지하지 못하는 G1 단계에서 이를 활용하려고 시도했습니다(보충 그림 S2C). 그림 3A는 G1에서도 항-RPA2 항체를 사용할 때H2O2처리 시 ssDNA 병소의 형성이 검출되었음을 보여줍니다. H2O2처리 시 이러한 핵에서 RPA2 병소의 수가 크게 증가했습니다(그림 3B). RPA2의 침묵으로 IF 신호가 사라졌기 때문에 이러한 초점은 RPA2에만 국한되었습니다(그림 3A,B). 그림 3C와 보충 그림 S1C는 이러한 셀에서 RPA2 침묵의 효율성을 보여줍니다. 기존 방법에 비해 ssDNA의 RPA2 기반 검출은 감도가 높기 때문에 G1 상 세포로 응용 분야를 확장할 수 있습니다.
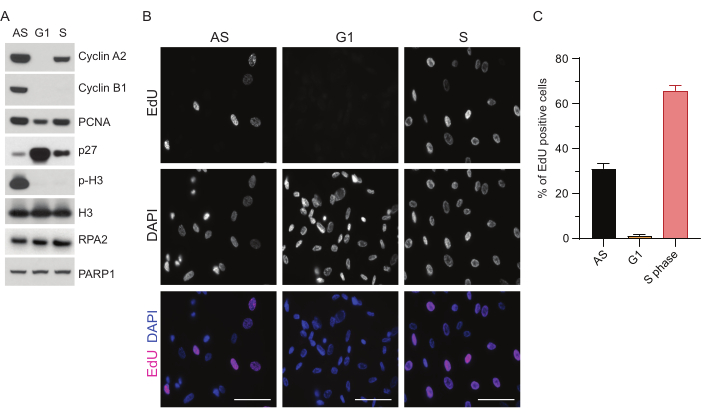
그림 1: 혈청 결핍 후 RPE1 세포의 동기화 효율. (A) 면역블롯은 비동기, G1 및 S상 동기화 RPE1 세포에서 표시된 단백질 수준을 보여줍니다. (B) 대표 이미지는 고정 전 30분 동안 10μM EdU에 노출되고 Click-IT 반응으로 시각화된 비동기, G1 및 S상 동기화 RPE1 세포를 보여줍니다. DAPI는 핵 DNA를 대조염색하는 데 사용되었습니다. 축척 막대 = 50 μm. (C) 그래프는 DAPI로 평가한 전체 세포 집단에서 EdU 양성 세포의 백분율을 보여줍니다. 오차 막대는 평균의 표준 오차를 나타내며 분석된 핵 수는 AS n = 219, G1 n = 630, S n = 437입니다. 약어: RPE1 = hTERT-불멸화된 망막 색소 상피 세포; AS = 비동기; EdU = 5-에티닐-2'-데옥시우리딘; DAPI = 4',6-디아미디노-2-페닐린돌. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
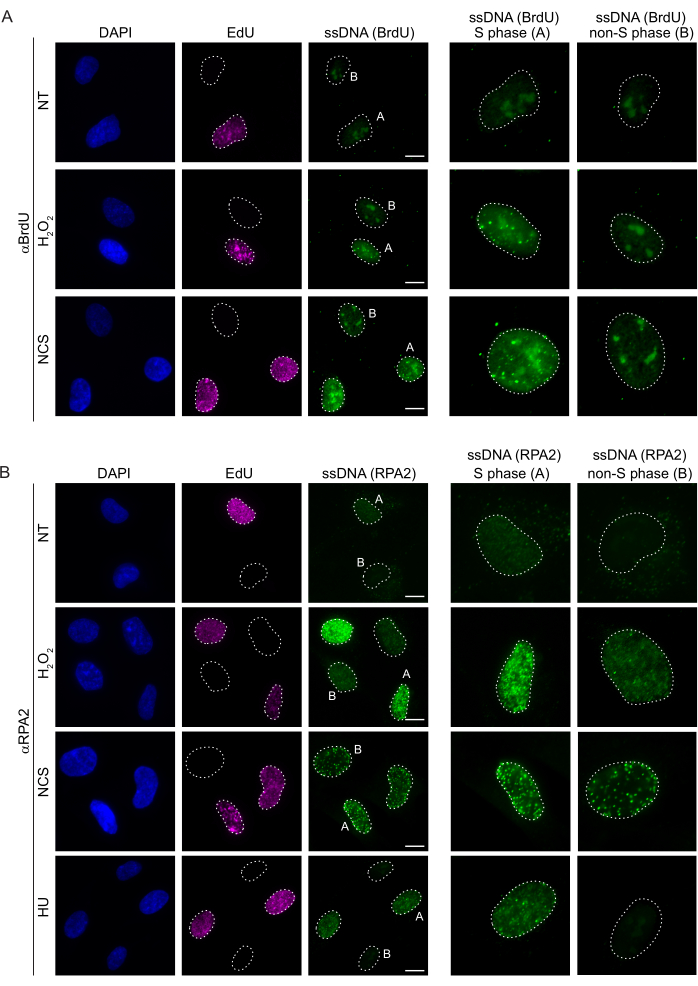
그림 2: DNA 손상 시 BrdU 항체 또는 RPA2 항체를 사용한 ssDNA 검출. (A) 대표 이미지는 αBrdU(녹색)를 사용하여 ssDNA 병소를 보여주고, S상 세포는 EdU(보라색)로 강조 표시되며, DAPI는 핵 DNA(파란색)를 대조염색하는 데 사용되었습니다. RPE1 세포는 추가 처리 전에 48시간 동안 10μM BrdU에 보관했습니다. 48시간 후, 세포를 30분 동안 10μM EdU로 펄스한 후 1시간 동안H2O2(250μM) 또는 4시간 동안 네오카르지노스타틴(0.5μg/mL)을 처리했습니다. 세포는 CSK 사전 추출 후에 고정되었습니다. 흰색 점선은 각 핵의 경계를 나타냅니다. 눈금 막대 = 5μm. 오른쪽의 패널은 표시된 S상 또는 비S상 핵의 확대 이미지입니다. (B) 대표 이미지는 αRPA2 항체(녹색)를 이용한 ssDNA 병소를 보여줍니다. S상 세포는 EdU(보라색)로 강조 표시되며, DAPI는 핵 DNA(파란색)를 대조염색하는 데 사용되었습니다. RPE1 세포를 30분 동안 10μM EdU로 펄스한 후 1시간 H2O2(250μM), 4시간 하이드록시우레아(2mM) 또는 4시간 NCS(0.5μg/mL)를 펄스했습니다. 세포는 CSK 사전 추출 후에 고정되었습니다. 흰색 점선은 각 핵의 경계를 나타냅니다. 눈금 막대 = 10μm. 오른쪽의 패널은 표시된 S상 또는 비S상 핵의 확대 이미지입니다. 약어: ssDNA = 단일 가닥 DNA; BrdU = 5-브로모-2'-데옥시우리딘; DAPI = 4', 6- 디아 미디노 -2- 페닐 린돌; RPE1 = hTERT-불멸화된 망막 색소 상피 세포; EdU = 5-에티닐-2'-데옥시우리딘; NCS = 네오카르지노스타틴; HU = 하이드록시우레아. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
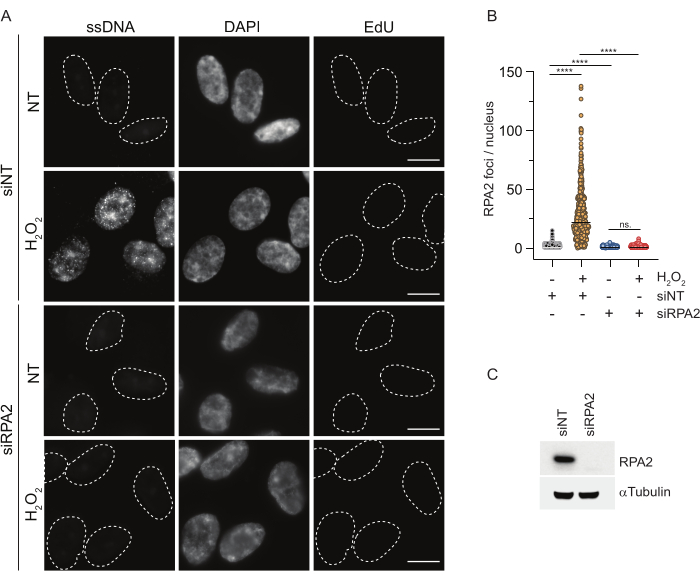
그림 3: RPA2 항체를 사용한 G1 상의 ssDNA 병소 검출. (A) RPE1 세포를 RPA2를 표적으로 하는 siRNA 또는 비표적 siRNA 대조군으로 transfection한 후, G1에서 동기화하고 지시된 곳에서 1시간 동안H2O2(250μM)로 처리하기 전에 30분 동안 10μM EdU로 펄스 라벨링했습니다. DAPI는 핵 DNA를 대조염색하는 데 사용되었습니다. 세포는 CSK 사전 추출 후에 고정되었습니다. 흰색 점선은 각 핵의 경계를 나타냅니다. 스케일 바 = 5 μm. (B) RPA2 병소/핵 수에 대한 측정은 두 개의 독립적인 실험에서 수행되었습니다. 분석 중에는 EdU 음성 세포만 고려되었습니다. 선은 그림의 평균값을 나타냅니다. 통계 분석을 위해 비모수 분산 분석(Kruskal-Wallis)을 수행했습니다. 별은 P < 0.0001을 나타냅니다. 분석된 핵의 수는 다음과 같았다: siNT no H2O2 n = 513, siNT H2O2 n = 603, siRPA2 no H2O2 n = 266, siRPA2 H2O2 n = 536. (C) siRNA knockdown의 효율은 immunoblotting에서 확인할 수 있습니다. 약어: siNT = non-targeting siRNA control; BrdU = 5-브로모-2'-데옥시우리딘; DAPI = 4', 6- 디아 미디노 -2- 페닐 린돌; RPE1 = hTERT-불멸화된 망막 색소 상피 세포; EdU = 5-에티닐-2'-데옥시우리딘. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: 피지를 사용한 ssDNA 병소의 정량화. 피지에서 핵의 RPA2 초점 수치를 평가하는 방법을 보여주는 자세한 단계. (A-E) DAPI 채널을 사용하여 핵 마스크를 만듭니다. (FH) 배경 신호에서 개별 핵 ssDNA 병소를 식별하기 위한 임계값 설정. 약어: ssDNA = 단일 가닥 DNA; DAPI = 4',6-디아미디노-2-페닐린돌. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 세포골격(CSK) 완충액 | |
| 파이프 pH 7.0 | 10 mM |
| 염화나트륨 | 100 밀리미터 |
| EDTA pH 8 | 1 mM |
| 마그네슘화합물 2 | 3 밀리미터 |
| D-자당 | 300 밀리미터 |
| 트리톤 X-100 | 0.20% |
| 인산가수분해효소 억제제 칵테일 | 10mL당 1정 |
| 프로테아제 억제제 칵테일 | 10mL당 1정 |
| ddH2O에 희석 | 해당 사항 없음 |
| 세척 버퍼 | |
| 트리톤 X-100 | 0.05% |
| PBS에 희석된 | 해당 사항 없음 |
| 투과화 완충액 | |
| 트리톤 X-100 | 0.50% |
| PBS에 희석된 | 해당 사항 없음 |
| 고정 솔루션 | |
| 파라포름알데히드 | 3.60% |
| 트리톤 X-100 | 0.05% |
| PBS에 희석된 | 해당 사항 없음 |
| 버퍼 차단 | |
| 소 혈청 알부민(BSA) | 5% |
| 트리톤 X-100 | 0.10% |
| PBS에 희석된 | 해당 사항 없음 |
| Click-iT Plus 반응 칵테일 | |
| Click-iT 반응 완충액 1개 | 435 mL |
| Alexa Fluor PCA 솔루션 | 5 mL |
| CuSO4-구리 보호제 프리믹스 | 10mL |
| 1x Click-iT 완충액 첨가제 | 50 mL |
| 총 부피 | 500 mL |
표 1: 이 프로토콜에 사용되는 버퍼의 구성.
보충 그림 S1. (A) RPE1 세포는 72시간 동안 혈청 결핍을 사용하여 G0 상으로 동기화된 후 혈청을 재도입하여 다른 세포 주기 상으로 방출했습니다. 점도표는 G0/G1, S 또는 G2/M 단계의 세포를 보여주며, 여기서 시간은 혈청 결핍 후 혈청을 다시 첨가한 후의 시간을 나타냅니다. 오른쪽 그래프는 각 조건에서 G0/G1, S 및 G2/M 세포의 백분율을 보여줍니다. FACS 분석은 제조업체의 권장 사항에 따라 EdU 및 프로피듐 요오드화물을 사용하는 시판되는 세포 증식 키트를 사용하여 수행되었습니다. (B) 그림 1에 대한 잘리지 않은 웨스턴 블롯 스캔. 숫자는 분자량 마커를 kDa 단위로 보여줍니다. PARP1을 로딩 컨트롤로 사용하고, 멤브레인을 절단하여 CCNA2, p27(PCNA에 대해 추가로 벗겨짐) 및 pH3(S10)(H3에 대해 추가로 벗겨짐)에 대해 개발된 겔 상에 로딩하였다. CCNB1 및 RPA2는 비교 가능성을 보장하기 위해 동일한 양의 단백질 용해물을 사용하여 별도의 겔에 로드되었습니다. (C) 그림 3에 대한 잘리지 않은 웨스턴 블롯 스캔. 숫자는 분자량 마커를 kDa 단위로 보여줍니다. 약어: EdU = 5-ethynyl-2'-deoxyuridine. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 S2: (A) 대표적인 이미지는 BrdU 항체(녹색)를 사용한 ssDNA 병소를 보여줍니다. S 상 셀은 사이클린 A2 (빨간색)로 강조 표시됩니다. DAPI는 핵 DNA(파란색)를 대조염색하는 데 사용되었습니다. RPE1 세포는 추가 처리 전에 48시간 동안 10μM BrdU에 보관했습니다. 48시간 후, 세포를 고정하기 전에 1시간 동안H2O2(250μM) 또는 4시간 동안 네오카르지노스타틴(0.5μg/mL)으로 처리하였다. 흰색 점선은 각 핵의 경계를 나타냅니다. 척도 막대 = 5 μm. (B) 변성 조건이 있거나 없는 RPE1 세포의 BrdU 염색. 비동기 RPE1 세포를 10μM BrdU로 48시간 동안 전처리했습니다. 스케일 바 = 10 μm. (C) BrdU 병소/핵의 수에 대한 측정은 G1 동기화된 RPE1 세포에서 두 개의 독립적인 실험에서 수행되었습니다. 분석 중에는 EdU 음성 세포만 고려되었습니다. 선은 그림의 평균값을 나타냅니다. 통계 분석을 위해 비모수 분산 분석(Kruskal-Wallis)을 수행했습니다. 'ns'는 유의하지 않은 차이를 나타냅니다. 분석된 핵의 수는 NT n=52, NCS n=105,H2O2n=82였다. 약어: siNT = non-targeting siRNA control; BrdU = 5-브로모-2'-데옥시우리딘; DAPI = 4', 6- 디아 미디노 -2- 페닐 린돌; RPE1 = hTERT-불멸화된 망막 색소 상피 세포; NCS = 네오카르지노스타틴. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 비디오 S1: 피지 기반 RPA2 초점 분석 화면 녹화. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
마이코플라스마가 없는 건강한 세포 배양을 유지하는 것은 위에서 설명한 모든 실험에서 매우 중요합니다. RPE1 세포는 정상적인 배양 배지에서 조직 배양 처리된 플라스틱 용기에 강한 부착을 가지고 있습니다. 그러나 이들의 결합 특성은 혈청이 없는 상태로 보관할 때 현저히 감소합니다. 또한 현미경으로 ssDNA 병소의 고해상도 이미지를 캡처하려면 세포를 0.17mm 두께의 커버 유리에 도금해야 하며, 이는 RPE1 세포의 적절한 부착을 지원할 만큼 친수성이 아닙니다. 적절하게 평평하고 고르게 분포된 세포가 없으면 개별 ssDNA 병소를 시각화하는 것이 매우 어렵습니다. 따라서 적절한 코팅 재료(예: 비트로넥틴)를 선택하고 세포를 G1 단계로 방출한 후 세포가 퍼지고 부착될 수 있도록 적절한 시간(6-12시간)을 두는 것이 중요합니다.
프로토콜의 어려운 부분은 동종 G1 동기화 RPE1 셀을 얻는 것입니다. 이를 위해서는 두 가지 중요한 단계가 필요합니다. 첫째, 효율적인 혈청 결핍을 위해 세포를 트립신화하고, PBS로 철저히 세척하고, 무혈청 배지를 사용하여 새로운 조직 배양 접시에 직접 파종해야 합니다. 혈청을 제거하기 위해 조직 배양 접시에서 세포를 직접 세척하면 효율적인 G0 동기화가 이루어지지 않습니다. 둘째, 세포를 G1 단계로 방출할 때 세포를 다시 트립신화하고 신선한 조직 배양 플레이트에 파종해야 합니다. 유사하게, 배지를 변경하고 혈청 함유 배양 배지를 세포에 추가하는 것만으로는 동기 G1 진입이 발생하지 않습니다. 또한 적절한 G1 진입을 위해서는 코팅된 커버 유리에 있는 세포의 파종 밀도가 특정 밀도 수준이어야 합니다. 완벽한 세포 동기화는 일반적으로 달성할 수 없지만, 여기에 설명된 이 동기화 프로토콜은 ~97%의 순수 G1 집단을 제공합니다. 직경 12mm의 커버슬립에서 RPE1의 권장 시딩 밀도는 약 70%의 밀도로 이미징을 위한 균일한 시야를 획득하기 위해 ~4 × 104 입니다. 시드 밀도가 높을수록 CSK 추출 후 세포가 분리되고 "벗겨지게" 되며 이미지 획득 중에 더 높은 배경 신호가 발생합니다.
배경 신호를 줄이고 양호한 신호 대 잡음비를 달성하려면 1차 및 2차 항체 배양 후 철저한 세척이 필수적입니다. 수많은 세척 단계를 거쳐야 하기 때문에 각 세척 단계에서 우물이 마르지 않도록 하는 것도 중요합니다. 당사는 모든 세척 및 배양 단계에서 최소 0.05%의 Triton X-100을 도포하여 이 아티팩트를 최소화합니다. 우물이 마르면 세포는 변경된 신호 대 잡음비를 나타냈습니다. 이로 인해 현미경에서 모자이크와 같은 패턴이 나타나고 평가를 방해할 수 있습니다. 디콘볼루션과 결합된 Z-stack 이미지 획득은 분석을 개선하기 위해 다양한 초점면에서 초점을 캡처하는 데 도움이 될 수 있습니다.
종래의 방법은 비변성 조건 하에서 혼입된 BrdU의 검출에 의존한다. 그러나 이러한 방법은 균일한 게놈 통합을 보장하기 위해 최소 1-2일(또는 사용된 세포주에서 전체 세포 주기에 해당하는 시간) 동안 고용량의 BrdU를 사용한 세포의 전처리에 의존합니다. 바람직하지 않게도, 광범위한 BrdU 혼입은 세포주기 간섭을 일으킬 수 있다36. 이러한 한계를 해결하기 위해 이 방법은 내인성 RPA2를 사용하여 ssDNA 병소를 검출합니다. 이 접근은 복제 몬 BrdU 합동을 요구하지 않습니다, 또한 포스트 유사분열 세포에서 사용될 수 있습니다. 광범위한 BrdU 통합이 필요하지 않기 때문에 시간을 절약하고 실험 복잡성을 줄입니다. RPA2 염색을 사용하여 ssDNA를 시각화함으로써 2′-deoxy-5-ethynyluridine(EdU) 및 클릭 화학을 사용하여 EdU 27,37,38에 대한 BrdU 항체의 교차 반응 가능성을 피하면서 DNA 복제를 표시할 수 있습니다. BrdU 항체가 EdU27,39와 교차 반응하지 않도록 클릭 반응 중에 통합된 EdU를 적절하게 마스킹하기 위해 특별한 주의를 기울여야 합니다.
마지막으로, BrdU 대신 RPA2를 사용하는 것의 중요한 이점은 S상 외부의 BrdU 염색과 비교할 때 신호 대 잡음비가 우수하다는 것입니다. 비변성 BrdU 염색과 ssDNA를 시각화하는 능력은 복제 세포에서도 S상으로 제한된다는 것을 발견했습니다(그림 2). BrdU 항체는 ssDNA 스트레치에서 충분히 노출된 BrdU에만 결합합니다. RPA2를 포함한 복구 단백질이 ssDNA 스트레치에 결합하면 ssDNA에서 BrdU의 충분한 노출을 억제하거나 방해할 수 있습니다. 또한 BrdU 항체를 사용한 ssDNA 시각화를 위해 CSK 사전 추출이 필요하다는 것을 발견했습니다. 이는 ssDNA 트랙이 항체에서 가볍게 결합된 단백질 성분을 제거하지 않고는 항체에 접근할 수 없기 때문에 가능합니다.
그럼에도 불구하고 이 프로토콜과 관련된 몇 가지 제한 사항이 있습니다. ssDNA 검출에 RPA2를 사용할 때의 한계는 CSK 사전 추출 단계를 최적화해야 한다는 것입니다. 결합되지 않은 과도한 RPA2는 세포를 고정하기 전에 DNA에서 씻어내야 합니다. 한편, 언더추출은 ssDNA에 결합되지 않은 RPA2 단백질 분획으로 인해 높은 백그라운드를 유발합니다. 반면에 과잉 추출은 신호 손실로 이어집니다. BrdU 검출의 경우, BrdU는 DNA에 안정적으로 통합되고 사전 추출에 의해 씻겨 나갈 수 없기 때문에 이는 변수가 아닙니다. 따라서 CSK 사전 추출 시간, 완충액 내 Triton X-100의 양, 부피 및 사전 추출이 수행되는 온도를 신중하게 고려해야 합니다. CSK 사전 추출은 또한 G0/G1 세포와 S/G2 세포를 구별하기 위해 핵 크기의 사용을 제한합니다.
또한 RPA2에서 오는 신호 중 일부가 다른 염색질 결합 단백질 상호 작용기에 결합되어 발생할 가능성을 배제할 수 없습니다. RPA2 항체의 종 특이성도 고려해야 합니다. 이 프로토콜에 사용된 항체는 인간, 생쥐, 쥐, 햄스터, 원숭이 RPA2를 인식할 수 있습니다. 이 접근법의 또 다른 한계는 모든 세포주가 G0 동기화를 위해 혈청에 굶주릴 수 있는 것은 아니라는 것입니다. 대부분의 암세포주는 세포주기 체크포인트를 우회할 수 있으며 혈청이 부족한 배지에서도 증식할 수 있습니다. 혈청 결핍은 DNA 손상을 일으키지 않기 때문에 유익하지만, 적절한 세포 주기 단계 농축이 달성되도록 세포 동기화 효율을 주의 깊게 모니터링해야 합니다. 혈청 박탈에 반응하지 않는 세포의 경우 다른 세포 동기화 방법(예: 유사분열 쉐이크 오프, G2 정지를 위한 CDK1 억제 또는 원심 용출과 같은 비침습적 기술)을 고려해야 합니다. 또 다른 가능한 방법은 비동기 세포(31)의 세포주기 프로파일링을 위한 EdU 및 핵 DNA 함량을 측정하기 위해 고-함량 이미징을 사용하는 것이다. 다운스트림 분석과의 간섭을 방지하기 위해 대체 동기화 방법을 사용하는 것의 의미를 고려해야 합니다. 예를 들어, 문헌에서 자주 사용되는 이중 티미딘 블록 또는 아피디콜린의 사용은 복제 스트레스와 DNA 손상을 초래할 것이다40.
DNA 복구 메커니즘에 대한 연구는 암 및 세포 생물학 분야에서 계속해서 논의의 초점이 되고 있습니다. 여기에 제시된 프로토콜은 세포 준비를 위한 귀중한 접근 방식을 제공하여 DNA 손상 물질에 노출된 시 ssDNA의 시각화 및 정량 분석을 가능하게 합니다. 특히, 이 프로토콜은 ssDNA 결합 단백질인 RPA2의 활용을 강조하여 모든 세포 주기 단계에서 원치 않는 교차 반응을 피하면서 소량의 ssDNA 병소를 시각화하는 높은 특이성을 보여줍니다. RPA2를 사용하면 특히 세포 주기의 G1 단계에서 세포를 분석하려는 연구자에게 많은 이점을 제공합니다. 이 프로토콜은 몇 가지 제한 사항을 고려하고 RPA2 또는 BrdU 염색을 사용하여 ssDNA를 검출할 때 신호 간섭, 원치 않는 배경 잡음 및 교차 반응과 관련된 문제를 해결합니다.
공개
저자는 선언할 경쟁 이익이 없습니다.
감사의 말
저자는 미셸 파가노(Michele Pagano)의 지원과 유용한 통찰력, 원고를 비판적으로 읽어준 애슐리 추이(Ashley Chui)와 샤론 카이사리(Sharon Kaisari), 지속적인 지원을 해준 제프리 에스트라다(Jeffrey Estrada)와 빌마 디아즈(Vilma Diaz)에게 감사를 표한다. 이 연구는 미국 국립보건원(National Institutes of Health)의 보조금 GM136250에 대한 다양성 보충 자료의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| Alpha-tubulin antibody | Sigma-Aldrich | T6074 | primary antibody (1:5,000) |
| Axio Observer Inverted Microscope | Zeiss | na | microscope |
| Bis-Tris Plus Mini Protein Gels, 4-12% | Invitrogen | NW04127BOX | Western Blot |
| Bovine Serum Albumin | Jackson ImmunoResearch | 001-000-162 | blocking |
| BrdU (5-Bromo-2'-deoxyuridine) | Sigma-Aldrich | B5002-100MG | nucleotide analogue |
| BrdU antibody BU1/75 | Abcam | ab6326 | primary antibody (1:500) |
| CellAdhere Dilution Buffer | Stemcell Technologies | 07183 | coating reagent |
| Click-iT Plus EdU Flow Cytometry Assay Kits | Invitrogen | C10632 | flow cytomery |
| Click-iT Plus EdU Cell Proliferation Kit for Imaging, Alexa Fluor 647 dye | Thermo Fisher Scientific | C10640 | click-reaction kit |
| cOmplete ULTRA Protease inhibitor tablets | Sigma-Aldrich | 5892791001 | reagent |
| Countess 3 Automated cell counter | Thermo Scientific | AMQAX2000 | cell counter |
| Coverslip | neuVitro | GG12PRE | tissue culture |
| Cyclin A2 antibody | Santa Cruz Biotechnology | sc-271682 | primary antibody (1:1,000) for IF and WB |
| Cyclin B1 antibody | Santa Cruz Biotechnology | sc-245 | primary antibody (1:5,000) |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D2650-100ML | vehicle control |
| DMEM, high glucose, with HEPES | Gibco | 12430051 | cell culture medium for RPE cells |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | the PBS used throughout the protocol |
| D-Sucrose | Thermo Fisher Scientific | bp220-1 | reagent |
| Eclipse Ti2 Series Epifluorescent Microscope | Nikon | na | microscope |
| EdU (5-Ethynyl-2'-deoxyuridine) | Thermo Fisher Scientific | C10637 | nucleotide analogue |
| Falcon 24-well plate | Corning | 351147 | tissue culture |
| Falcon Cell Culture Dishes 100 mm | Corning | 353003 | tissue culture |
| Fetal Bovine Serum, heat inactivated | Gibco | 16140071 | media supplement |
| Fiji (ImageJ) | NIH | version 1.54f | software and algorithms |
| FxCycle PI/RNase Staining Solution | Invitrogen | F10797 | PI staining |
| Goat anti-mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 555 | Thermo Fisher Scientific | A21422 | secondary antibody (1:1,000) |
| Goat anti-rat IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 488 | Thermo Fisher Scientific | A48262 | secondary antibody (1:1,000) |
| Histone H3 antibody | Abcam | ab1791 | primary antibody (1:10,000) |
| hTERT RPE1 | ATCC | CRL-3216 | cell line |
| Hydrochloric acid | Sigma-Aldrich | H1758-100ML | reagent |
| Hydrogen peroxide 30% soultion | Sigma-Aldrich | H1009-100ML | reagent |
| Hydroxyurea,98% powder | Sigma-Aldrich | H8627-5G | reagent |
| Invitrogen Ultra Pure 0.5 M EDTA pH 8.0 | Thermo Fisher Scientific | 15-575-020 | reagent |
| Lipfectamine RNAiMAX Transfection Reagent | Invitrogen | 13778150 | transfection reagent |
| Magnesium chloride solution 1 M | Sigma-Aldrich | M1028-100ML | reagent |
| MycoFluor | Thermo Fisher | M7006 | Mycoplasma Detection Kit |
| Neocarzinostatin from Streptomyces carzinostaticus | Sigma-Aldrich | N9162-100UG | reagent |
| NuPage MES SDS Running Buffer (20x) | Invitrogen | NP0002 | Western Blot |
| onTARGETplus Human RPA2 siRNA | Dharmacon | L-017058-01-0005 | siRNA |
| p27 antibody | BD Biosciences | 610241 | primary antibody (1:1,000) |
| Paraformaldehyde aqueous solution (32%) | Electron Microscopy Sciences | 50-980-494 | fixative |
| PARP1 antibody | Cell Signaling Technology | 9542S | primary antibody (1:1,000) |
| PCNA antibody | Cell Signaling Technology | 13110S | primary antibody (1:2,000) |
| Penicillin-Streptomycin | Gibco | 15140163 | media supplement |
| pH3 antibody | Cell Signaling Technology | 3377S | primary antibody (1:2,000) |
| PhosSTOP phosphatase inhibitor tablets | Sigma-Aldrich | 4906837001 | reagent |
| PIPES Buffer 0.5 M solution, pH 7.0 | Bioworld | 41620034-1 | reagent |
| Precision Plus Protein Kaleidoscope Prestained Protein Standards | Bio-Rad | 1610395 | Western Blot |
| Prism | GraphPad | version 10 | statistical analysis and graph |
| ProLong Diamond Antifade Mountant | Thermo Scientific | P36961 | mounting media |
| Reduced serum media (Opti-MEM) | Gibco | 31985070 | used for transfection |
| Rpa32/rpa2 antibody (mouse) | EMD Millipore | NA19L | primary antibody (1:1,000) for WB |
| Rpa32/rpa2 antibody (rat) | Cell Signaling Technology | 2208S | primary antibody (1:1,000) for IF |
| Sodium Chloride solution (5 M) | Sigma-Aldrich | S5150 | reagent |
| Sodium Pyruvate (100 mM) | Gibco | 11360070 | media supplement |
| Sodium tetraborate decahydrate | Sigma-Aldrich | B3535-500G | reagent |
| Thermo Scientific Pierce DAPI Nuclear Counterstain | Thermo Scientific | 62248 | nucleic acid stain |
| Thymidine,powder | Sigma-Aldrich | T1985-1G | reagent |
| Triton X-100 aqueous solution (10%) | Sigma-Aldrich | 11332481001 | detergent |
| Trypsin-EDTA (0.5%), no phenol red | Gibco | 1540054 | cell dissociation agent |
| Vitronectin XF | Stemcell Technologies | 07180 | coating reagent |
| ZE5 Cell Analyzer | Bio-Rad | na | flow cytomery |
참고문헌
- Hakem, R. DNA-damage repair; the good, the bad, and ugly. EMBO J. 27 (4), 589-605 (2008).
- Gutierrez, R., O'Connor, T. R. DNA direct reversal repair and alkylating agent drug resistance. Cancer Drug Resist. 4 (2), 414-423 (2021).
- Krokan, H. E., Bjoras, M. Base excision repair. Cold Spring Harb Perspect Biol. 5 (4), a012583 (2013).
- Marteijn, J. A., Lans, H., Vermeulen, W., Hoeijmakers, J. H. Understanding nucleotide excision repair and its roles in cancer and ageing. Nat Rev Mol Cell Biol. 15 (7), 465-481 (2014).
- Li, G. M. Mechanisms and functions of DNA mismatch repair. Cell Res. 18 (1), 85-98 (2008).
- Hustedt, N., Durocher, D. The control of DNA repair by the cell cycle. Nat Cell Biol. 19 (1), 1-9 (2016).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bhat, D. S., et al. Therapeutic disruption of RAD52-ssDNA complexation via novel drug-like inhibitors. NAR Cancer. 5 (2), zcad018 (2023).
- Gupta, P., Saha, B., Chattopadhyay, S., Patro, B. S. Pharmacological targeting of differential DNA repair, radio-sensitizes WRN-deficient cancer cells in vitro and in vivo. Biochem Pharmacol. 186, 114450 (2021).
- Pena-Diaz, J., et al. Noncanonical mismatch repair as a source of genomic instability in human cells. Mol Cell. 47 (5), 669-680 (2012).
- Schroering, A. G., Edelbrock, M. A., Richards, T. J., Williams, K. J. The cell cycle and DNA mismatch repair. Exp Cell Res. 313 (2), 292-304 (2007).
- Scully, R., Panday, A., Elango, R., Willis, N. A. DNA double-strand break repair-pathway choice in somatic mammalian cells. Nat Rev Mol Cell Biol. 20 (11), 698-714 (2019).
- Escribano-Diaz, C., et al. A cell cycle-dependent regulatory circuit composed of 53BP1-RIF1 and BRCA1-CtIP controls DNA repair pathway choice. Mol Cell. 49 (5), 872-883 (2013).
- Genschel, J., Modrich, P. Mechanism of 5'-directed excision in human mismatch repair. Mol Cell. 12 (5), 1077-1086 (2003).
- Hu, J., et al. Nucleotide excision repair in human cells: fate of the excised oligonucleotide carrying DNA damage in vivo. J Biol Chem. 288 (29), 20918-20926 (2013).
- Huertas, P., Jackson, S. P. Human CtIP mediates cell cycle control of DNA end resection and double strand break repair. J Biol Chem. 284 (14), 9558-9565 (2009).
- Keijzers, G., et al. Human exonuclease 1 (EXO1) regulatory functions in DNA replication with putative roles in cancer. Int J Mol Sci. 20 (1), 74 (2018).
- Symington, L. S. End resection at double-strand breaks: mechanism and regulation. Cold Spring Harb Perspect Biol. 6 (8), a016436 (2014).
- Liu, Y., et al. DNA polymerase beta and flap endonuclease 1 enzymatic specificities sustain DNA synthesis for long patch base excision repair. J Biol Chem. 280 (5), 3665-3674 (2005).
- Wold, M. S., Kelly, T. Purification and characterization of replication protein A, a cellular protein required for in vitro replication of simian virus 40 DNA. Proc Natl Acad Sci U S A. 85 (8), 2523-2527 (1988).
- Wienholz, F., Vermeulen, W., Marteijn, J. A. Amplification of unscheduled DNA synthesis signal enables fluorescence-based single cell quantification of transcription-coupled nucleotide excision repair. Nucleic Acids Res. 45 (9), e68 (2017).
- Wold, M. S. Replication protein A: a heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annu Rev Biochem. 66, 61-92 (1997).
- Chen, R., Wold, M. S. Replication protein A: single-stranded DNA's first responder: dynamic DNA-interactions allow replication protein A to direct single-strand DNA intermediates into different pathways for synthesis or repair. Bioessays. 36 (12), 1156-1161 (2014).
- Kang, Y., et al. Alteration of replication protein A binding mode on single-stranded DNA by NSMF potentiates RPA phosphorylation by ATR kinase. Nucleic Acids Res. 51 (15), 7936-7950 (2023).
- Kilgas, S., Kiltie, A. E., Ramadan, K. Immunofluorescence microscopy-based detection of ssDNA foci by BrdU in mammalian cells. STAR Protoc. 2 (4), 100978 (2021).
- Madabhushi, R., Pan, L., Tsai, L. H. DNA damage and its links to neurodegeneration. Neuron. 83 (2), 266-282 (2014).
- Liboska, R., Ligasova, A., Strunin, D., Rosenberg, I., Koberna, K. Most anti-BrdU antibodies react with 2'-deoxy-5-ethynyluridine -- the method for the effective suppression of this cross-reactivity. PLoS One. 7 (12), e51679 (2012).
- Biehs, R., et al. DNA double-strand break resection occurs during non-homologous end joining in G1 but is distinct from resection during homologous recombination. Mol Cell. 65 (4), 671-684.e5 (2017).
- Cruz-Garcia, A., Lopez-Saavedra, A., Huertas, P. BRCA1 accelerates CtIP-mediated DNA-end resection. Cell Rep. 9 (2), 451-459 (2014).
- Ercilla, A., et al. Physiological tolerance to ssDNA enables strand uncoupling during DNA replication. Cell Rep. 30 (7), 2416-2429.e7 (2020).
- Lezaja, A., et al. RPA shields inherited DNA lesions for post-mitotic DNA synthesis. Nat Commun. 12 (1), 3827 (2021).
- Mukherjee, B., Tomimatsu, N., Burma, S. Immunofluorescence-based methods to monitor DNA end resection. Methods Mol Biol. 1292, 67-75 (2015).
- Ochs, F., et al. 53BP1 fosters fidelity of homology-directed DNA repair. Nat Struct Mol Biol. 23 (8), 714-721 (2016).
- Raderschall, E., Golub, E. I., Haaf, T. Nuclear foci of mammalian recombination proteins are located at single-stranded DNA regions formed after DNA damage. Proc Natl Acad Sci U S A. 96 (5), 1921-1926 (1999).
- Forment, J. V., Walker, R. V., Jackson, S. P. A high-throughput, flow cytometry-based method to quantify DNA-end resection in mammalian cells. Cytometry A. 81 (10), 922-928 (2012).
- Mistrik, M., et al. Cells and stripes: A novel quantitative photo-manipulation technique. Sci Rep. 6, 19567 (2016).
- Aten, J. A., Bakker, P. J., Stap, J., Boschman, G. A., Veenhof, C. H. DNA double labelling with IdUrd and CldUrd for spatial and temporal analysis of cell proliferation and DNA replication. Histochem J. 24 (5), 251-259 (1992).
- Podgorny, O., Peunova, N., Park, J. H., Enikolopov, G. Triple S-phase labeling of dividing stem cells. Stem Cell Reports. 10 (2), 615-626 (2018).
- Cappella, P., Gasparri, F., Pulici, M., Moll, J. Cell proliferation method: click chemistry based on BrdU coupling for multiplex antibody staining. Curr Protoc Cytom. Chapter 7, (2008).
- Ligasova, A., Koberna, K. Strengths and weaknesses of cell synchronization protocols based on inhibition of DNA synthesis. Int J Mol Sci. 22 (19), 10759 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유