Method Article
Um dia-Esquema de fluxo de trabalho para detecção de patógenos de bactérias e de resistência antimicrobiana de hemoculturas
Neste Artigo
Resumo
O desenho de um esquema de fluxo de trabalho simples de um dia para diagnóstico de patógenos bacterianos permite o reconhecimento rápido de infecções da corrente sanguínea. A inclusão de oito clinicamente relevantes alvos bacterianas e os seus perfis de resistência a antibióticos oferece a percepção um clínico inicial no mesmo dia, o que pode levar a uma terapia mais adequada.
Resumo
Infecções da corrente sanguínea estão associados a altas taxas de mortalidade por causa da manifestação provável de sepse, sepse grave e choque séptico 1. Assim, a administração rápida de terapia antibiótica adequada é de grande importância no tratamento de infecções da corrente sanguínea. O elemento crítico neste processo é o tempo, fortemente dependente dos resultados da identificação bacteriana e testes de susceptibilidade aos antibióticos. Ambos estes parâmetros são rotineiramente obtido por cultura com base em testes, o que é demorado e leva em média 24-48 horas 2, 4. O objectivo do estudo foi o de desenvolver baseados em DNA ensaios para a rápida identificação de infecções da corrente sanguínea, bem como testes de susceptibilidade antimicrobiana rápida. O primeiro ensaio é um 16S eubacterianas ADNr baseado em tempo real de ensaio PCR, complementados com espécies-ou género sondas específicas 5. Usando estas sondas, bactérias gram-negativas, incluindo Pseudomonas spp., Pseudomonas aeruginosae Escherichia coli, bem como bactérias Gram-positivas, incluindo Staphylococcus spp., Staphylococcus aureus, Enterococcus spp., Streptococcus spp., e Streptococcus pneumoniae poderiam ser distinguidas. Utilizando este ensaio Multiprobe, uma primeira identificação do micro-organismo causador foi dado após 2 h.
Em segundo lugar, nós desenvolvemos um ensaio semi-molecular para testes de susceptibilidade a antibióticos de S. aureus, Enterococcus spp. e (facultativo) aeróbios Gram-negativos hastes 6. Este ensaio baseou-se um estudo em que a PCR foi utilizado para medir o crescimento de bactérias 7. As bactérias colhidas directamente a partir de culturas de sangue são incubadas durante 6 h, com uma selecção de antibióticos, e seguindo um Sybr Verde baseado em tempo real ensaio de PCR determina a inibição do crescimento. A combinação destes dois métodos poderiam dirigir a escolha de uma terapia adequada antibiótico no mesmo dia (Figura 1). Em conclusão, A análise molecular de ambos identificação e antibiograma oferece uma alternativa mais rápida para a detecção de patógenos e pode melhorar o diagnóstico de infecções da corrente sanguínea.
Protocolo
PARTE I: identificação do patógeno
1. Preparação da Amostra
Nota: O fluxo de trabalho inteiro molecular como é descrito no protocolo seguinte deve ser realizada de acordo com as recomendações para garantia da qualidade do diagnóstico molecular 3.
- Adicionar uma alíquota de 0,1 ml de cultura de sangue a 0,9 ml de NaCl 0,9% para um tubo de reacção 1,5 ml a tornar-se uma amostra 1:10 diluído. (Diluir a fim de evitar a inibição qPCR).
- Centrifugar amostra a 13.400 xg durante 5 min a pelete o ADN bacteriano.
- Ressuspender o sedimento bacteriano em 100 uL estéril desmineralizada H 2 O.
- Armazenar a amostra de ADN a 4 ° C até uso posterior.
2. Ensaio de Identificação: Real-Time PCR rDNA 16s
- Preparar as misturas de reacção como se segue. O ensaio consiste em quatro reacções separadas por amostra. Cada mistura inclui 12,50 mestre ulmistura, 0,9 uM para a frente de iniciadores (5-TCCTACGGGAGGCAGCAGT-3) 7, 0,6 mM iniciador reverso (5-GGACTACCAGGGTATCTAATCCTGTT-3) 8 e um painel de sondas. A quantidade de sondas é dado para cada um dos quatro reacções separadas em baixo.
- A primeira reacção inclui:
- 0,2 mM da sonda universal (5-FAM-CGTATTACCGCGGCTGCTGGCAC-3-BHQ1) 8
- 0,2 uM P. sonda aeruginosa (5-JOE-CCAAAACTACTGAGCTAGAGTACG-3-BHQ1)
- A segunda reacção inclui:
- 0,2 uM E. sonda coli (5-JOE-GGAGTAAAGTTAATACCTTTGCTCATT-3-BHQ1)
- 0,2 uM de Pseudomonas spp. sonda (5-NED-CCTTCCTCCCAACTTAAAGTGCTT-3-MGBNFQ)
- A reacção terceira inclui:
- 0,2 mM Staphylococcus spp. sonda (5-NED-AATCTTCCGCAATGGGCGAAAGC-3-MGBNFQ)
- 0,2 uM de S. sonda aureus (5-FAM-AGATGTGCACAGTTACTTACACATAT-3-BHQ1)
- 0,2 uM Enterococcus spp. (5-JOE-TCCTTGTTCTTCTCTAACAACAGAG-3-BHQ1)
- A reacção quarto inclui:
- 0,2 mM da sonda universal (5-FAM-CGTATTACCGCGGCTGCTGGCAC-3-BHQ1)
- 0,3 uM Streptococcus spp. sonda (5-NED-CCAGAAAGGGACSGCTAACT-3-MGBNFQ)
- 0,2 uM de S. pneumoniae sonda (5-JOE-CCAAAGCCTACTATGGTTAAGCCA-3-BHQ1)
- Adicionar estéril desmineralizada H2O para atingir um volume total de 20 uL. Adicionar 20 ul de cada mistura de reacção para os poços de uma placa de PCR de 96 poços.
- Adicionar 5 ul de amostra a cada poço.
- Use uma película adesiva para selar a placa de 96 poços de PCR.
- Execute o prato no PRISM 7900HT ABI Real Time PCR System usando as seguintes condições óptimas de ciclos térmicos:
- Pré-aquecimento a 50 ° C durante 10 min
- Desnaturação inicial a 95 ° C durante 15 min
- 42 ciclos de
- Desnaturação a 95 ° C durante 15 s
- Recozimento a 60 ° C durante 1 min
3. Análise dos Resultados
Ajuste o limiar da Análise de Ct para 0,1 nas configurações de análise de tabulação. Estreitar as configurações de base para iniciar (Ciclo): 6 e End (ciclo): 15.
- Registre o ciclo limiar (Ct) para todas as amostras. O valor de corte para considerar um resultado PCR positivo como pode ser definido como um Ct valor de 35. A quantidade de bactérias presentes em culturas de sangue variou de julho 10-novembro 10 CFU / ml, gerando Ct-valores abaixo de 35.
PARTE II: TESTES susceptibilidade a antimicrobianos
4. Isolamento de bactérias a partir de hemoculturas positivas 9
- Aspirar 5 ml de caldo de uma garrafa de sangue positivo cultura e transferi-lo para um tubo de separador de soro.
- Centrifugar o tubo separador de soro a 2000 xg durante 10 min.
- Descartar o sobrenadante do tubo separador de soro.
- Transferência de bactérios a partir da camada de gel do tubo com uma mecha de algodão estéril em solução salina a 0,9% até 0,5 de McFarland uma suspensão padrão é obtida.
5. Inoculação de placas de Micro Titre
- Dilui-se a suspensão 0,5 McFarland em duplo concentrado Mueller Hinton II caldo para formar uma suspensão de 5 x 10 5 UFC / ml.
- Adicionar esta suspensão para os poços de uma placa de micro titulação, contendo uma selecção de antibióticos (Tabela 1).
- Incubar a placa de micro titulação, a 37 ° C durante 6 h.
- Armazenar uma alíquota da suspensão a 4 ° C (como controlo de crescimento negativo).
- Após 6 h de incubação, transferir o conteúdo de cada poço para um tubo de estéril, bem como a amostra de controlo negativo de crescimento que foi armazenada a 4 ° C.
- Centrifugar os tubos a 16.000 xg durante 5 min.
- Cuidadosamente remover o sobrenadante, sem perturbar o sedimento bacteriano.
- Ressuspender o sedimento em H desmineralizada esterilizada 2 O.
- Diluir as amostras de 10 vezes na estéril desmineralizada H 2 O.
6. Real-time PCR rDNA 16s 10
- Preparar a mistura de PCR como se segue:
- 12,50 ul iQ SYBR Green Supermix
- 0,5 uM primer dianteiro 16S-1 (5-TGGAGAGTTTGATCCTGGCTCAG-3) 11
- 0,25 iM reversa 16S-2 primer (5-TACCGCGGCTGCTGGCAC-3) 11
- estéril desmineralizada H2O para um volume total de 20 uL
- Adicionar 20 ul de mistura de PCR para os poços de uma placa de PCR de 96 poços.
- Adicionar 5 ul de amostra a cada poço.
- Use uma película adesiva para selar a placa de 96 poços de PCR.
- Execute o prato no Single-Cor MyiQ Sistema de Detecção de Real-Time PCR, utilizando os seguintes óptimas condições de ciclos térmicos:
- Desnaturação inicial a 95 ° C durante 4 min
- Inicial de recozimento, a 65 ° C durante 30 s
- 35 ciclos de
- Desnaturação a 95 ° C durante 15 s
- Recozimento a 60 ° C durante 1 min
- Derreter análise da curva (60-95 ° C em 20 min com incrementos de 0,57 ° C)
7. Análise dos Resultados
- Calcular o valor de corte Ct, utilizando uma das seguintes fórmulas (dependendo do tipo de antibiótico).
- Em geral:
Cut-off Ct valor = valor Ct controle do crescimento positivo + 0,5 x (valor Ct controle do crescimento negativo - valor Ct controle de crescimento positivo) - Piperacilina, piperacilina / tazobactam e ceftazidima em bacilos Gram-negativos; amoxicilina, oxacilina e trimetoprim / sulfametoxazol em S.aureus; Amoxicilina em Enterococcus spp:. Cut-off Ct valor = valor Ct controle do crescimento positivo + 0,25 x (Ct crescimento valor negativo controle - valor Ct controle de crescimento positivo)
- Em geral:
- Use a amostra incubada com desmineralizada estéril H 2 O, como controle de crescimento positivo.
- Dependendo do microorganismo, utilizar o controlo de crescimento negativa adequada:
- Gram-negativas Amostra hastes incubadas com uma mistura de antibióticos
- Enterococcus spp Amostra. Armazenado a 4 ° C
- Exemplo S.aureus armazenado a 4 ° C
- Determinar a susceptibilidade (S) ou a resistência (R) da estirpe para o antibiótico testado como se segue:
- Um valor Ct superior ao valor Ct de corte indica susceptibilidade
- Um valor Ct inferior ao valor Ct de corte indica resistência
8. Os resultados representativos
Dois organismos modelo, ou seja, uma E. Gram-negativas coli e S. uma Gram-positivos aureus, são escolhidos para visualizar o procedimento combinado para a detecção e identificação de agentes patogénicos bacterianos e de determinação do seu perfil de antimicrobiana. A primeira parte do protocolo compreende a identificação do patógeno. Spesondas específicos são concebidos para a detecção de oito microrganismos clinicamente relevantes. Em presença de um alvo incluídos no painel de bacteriana, as curvas de amplificação são gerados e valores Ct são calculados (Figura 2). O valor de corte para considerar um resultado positivo de PCR como está definido para um valor de Ct de 35. Na Figura 2A, o perfil de identificação de uma cultura de E. coli sangue infectado é mostrado. A sonda 16S universal está incluído em duas misturas de reação separadas e, conseqüentemente, gera duas curvas de amplificação (Ct de 25,20 e 25,95). O terceiro sinal é derivado da sonda específica para E. coli (Ct de 27,04). A identificação de um S. aureus infectado cultura de sangue é mostrado na Figura 2B. A sonda 16S universal tem sinais de amplificação de 33,35 e 33,71. Os dois sinais restantes são derivadas das sondas específicas para Staphylococcus spp. e S. aureus (Ct de 32,48 e 30,59).
Após a primeira parte do protocolo, o microorganismo causador é conhecida eo perfil antimicrobiana pode ser determinada. A Figura 3 é um exemplo de um antibiótico trama amplificação susceptibilidade de teste, que representa a estirpe de E. coli que foi também mostrado na Figura 2A. Cada linha representa um antibiótico que a amostra bacteriana foi incubada com. Uma amostra com um valor baixo Ct é uma amostra na qual o crescimento ocorreu na presença de uma resistência, antibiótico indicando ao antibiótico testado. Pelo contrário, um valor elevado Ct representa uma amostra na qual nenhum crescimento ocorreu por causa de o funcionamento eficaz da susceptibilidade, antibiótico indicando ao antibiótico testado. A Tabela 1 ilustra a determinação do perfil de antimicrobiana do E. coli e S. isolados aureus. Todos os valores são relatados Ct e, usando as fórmulas mencionadas no texto protocolo (7,1), dois valores de corte Ct são calculadosisoladas de distinguir entre a resistência e susceptibilidade. A estirpe é resistente ao antibiótico se o valor relatado Ct é menor do que o valor calculado Ct cut-off (e vice-versa).
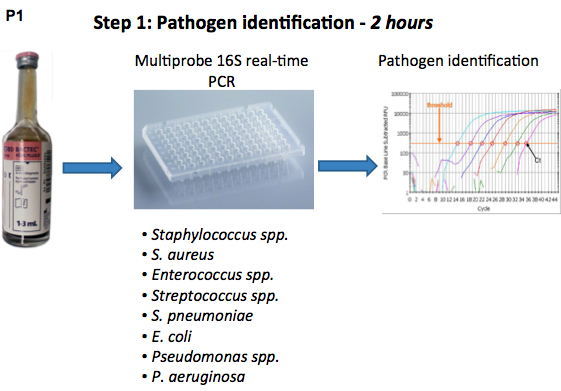
Figura Fluxograma 1. Da identificação do patógeno e procedimento de teste de sensibilidade aos antibióticos em tempo real usando 16S rDNA PCR.
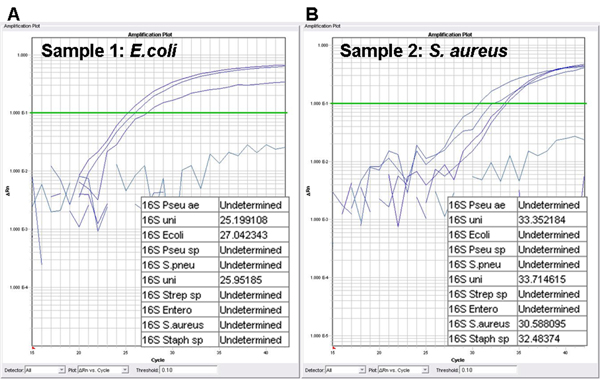
A Figura 2 ensaio Identificação:. Gráficos de amplificação e os valores de limiar de ciclo (valores Ct) Uma cultura de sangue positivo é detectado pelo universal 16S rDNA sonda, enquanto as sondas específicas são utilizados para a identificação do agente patogénico causal.. A. enredo ampliação da cultura de sangue contendo E. coli, B. enredo ampliação da cultura de sangue contendo S. aureus; Pseudomonas ae, Pseudomoncomo aeruginosa; uni, 16S sonda universal; Ecoli, Escherichia coli sonda; Pseudomonas sp, Pseudomonas spp. sonda; S. pneumoniae, Streptococcus pneumoniae sonda; Strep sp, Streptococcus spp. sonda; Entero, Enterococcus spp. sonda; S. aureus, Staphylococcus aureus sonda; Staph sp, Staphylococcus spp. sonda.
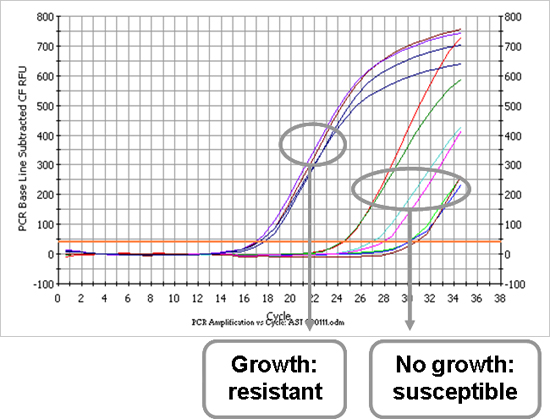
Figura enredo Amplificação 3. Dos testes de susceptibilidade a antibióticos de um E. coli isolado (amostra 1). Cada curva representa um antibiótico que a estirpe foi incubada com. Um sinal precoce é causada por uma carga elevada de bactérias, o que significa que a estirpe cresceu na presença do antibiótico testado e é, portanto, resistente ao antibiótico. Sinais tardios indicam que a estirpe não cresceu na presença do antibiótico, por outras palavras, ela é susceptível.
| Exemplo 1: E. coli | Exemplo 2: S. aureus | ||||
| AST | Ct | R / S | AST | Ct | R / S |
| A amoxicilina de 8 mg / L | 16,83 | R | A amoxicilina de 0,25 mg / L | 21,03 | R |
| Amoxicilina-clavulanato mg 8/4 / L | 17,36 | R | Oxacilina 2 mg / L | 25,80 | S |
| Piperacilina 16 mg / L | 16,67 | R | A vancomicina 2 mg / L | 25,20 | S |
| Piperacilina-tazobactam 16 /4 mg / L | 24,15 | S | Gentamicina 4 mg / L | 25,86 | S |
| Ciprofloxacina 1 mg / L | 29,72 | S | Trimetoprima-sulfametoxazol mg 2/38 / L | 24,62 | S |
| A ceftazidima 1 mg / L | 24,03 | S | |||
| A ceftazidima 8 mg / L | 26,58 | S | |||
| Gentamicina 4 mg / L | 29,83 | S | |||
| Trimetoprima-sulfametoxazol mg 2/38 / L | 27,60 | S | |||
| Controlo de crescimento negativo (mistura de antibióticos) | 30,41 | Controlo de crescimento negativo (amostra armazenada a 4 ° C) | 27,42 | ||
| Controle de crescimento positiva | 16,90 | Controle de crescimento positiva | 20,22 | ||
| Cut-off valor Ct-1 * | 21,76 | Cut-off valor Ct-1 *** | 23,82 | ||
| Cut-off Ct-valor 2 ** | 18,75 | Cut-off Ct-valor 2 **** | 22,02 | ||
| * Para amoxicilina, amoxicilina-clavulanato, ciprofloxacina, gentamicna, sulfametoxazol-trimetoprim ** Para piperacilina, piperacilina-tazobactam, ceftazidima | *** Para vancomicina e gentamicina Para **** amoxicilina, oxacilina e sulfametoxazol-trimetoprim |
Tabela 1. Determinação dos testes de susceptibilidade aos antibióticos das duas amostras (E.coli e S.aureus). Os valores de TC do PCR-ensaio foram copiados para este ficheiro Excel, como o que pode automaticamente calcula os dois alores de corte Ct a partir do controlo de crescimento positivo e negativo, usando as fórmulas mostradas no texto de protocolo. Se um antibiótico mostra um valor Ct inferior ao valor Ct de corte, a estirpe é resistente ao antibiótico, se o valor Ct foi maior do que o ponto de corte, a estirpe é susceptível.
Discussão
O protocolo descrito aqui permite a rápida identificação de patógenos e fornece um perfil funcional antimicrobiano que poderia conduzir a administração precoce de antibióticos adequados, melhorando o prognóstico de pacientes com infecções da corrente sanguínea. Dependendo das condições requeridas de um teste, isto é, baixo custo, com rendimento elevado, o mínimo tempo de rotação, as condições de ensaio pode ser ajustada. Todo o procedimento pode ser realizado dentro de um dia útil. Além disso, as duas partes do protocolo pode ser realizada simultaneamente, o que reduz o tempo de rotação de forma significativa. Como apresentado aqui, o painel de identificação é uma seleção das bactérias mais clinicamente relevantes em nosso hospital. Uma vez que o principal princípio tem como alvo a região do gene 16S, as sondas específicas para outros microorganismos podem ser concebidos e adicionado ao ensaio. O ensaio completo foi originalmente destinado para a análise rápida de culturas de sangue, mas também pode ser usado para o processamento de Oamostra materiais Ther. Este é também o caso para os antibióticos que foram usados para os testes de susceptibilidade aos antibióticos: mais antibióticos ou outras podem ser adicionados, com base em padrões de resistência locais e orientações.
Divulgações
Não temos nada a divulgar.
Agradecimentos
Este trabalho foi financiado pelo azm Profileringsfonds (PF245).
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| Cloreto de sódio (NaCl) | Merck Chemicals | 106404 | 0,9% em água |
| Vacutainer SST tubo separador de soro de 5 ml | BD Diagnostic Systems | 367986 | |
| Mueller Hinton II caldo | BD Diagnostic Dystems | 212322 | 44 g / L em água |
| Centrifugar Rotixa 50 rs | Andreas Hettich GmbH & Co. KG | 4910 | |
| Centrífuga 5415 D | Eppendorf | Descontinuado | |
| Primers | Sigma-Aldrich | nd | |
| Sondas | Sigma-Aldrich/Applied Biosystems | nd | |
| TaqManEnvironmental mestre mix 2,0 | Applied Biosystems | 4396838 | |
| iQ SYBRGreen Supermix | Bio-Rad Laboratories BV | 170-8880 | |
| MicroAmp Optical 96-Bem Placa de Reacção | Applied Biosystems | N8010560 | |
| Película adesiva MicroAmp Optical | Applied Biosystems | 4311971 | |
| iQ placas de 96 poços de PCR | Bio-Rad Laboratories BV | 223-9441 | |
| Microseal B Adesivo Selos | Bio-Rad Trabalhoatories BV | MSB-1001 | |
| Real-time Sistema de Detecção de PCR | Applied Biosystems | ABI PRISM 7900HT | |
| Real-Time PCR Detection System | Bio-Rad Laboratories BV | MyiQ Single-Cor |
Referências
- Wallet, F. Preliminary clinical study using a multiplex real-time PCR test for the detection of bacterial and fungal DNA directly in blood. Clin. Microbiol. Infect. 16, 774 (2010).
- Beekmann, S. E., Diekema, D. J., Chapin, K. C., Doern, G. V. Effects of rapid detection of bloodstream infections on length of hospitalization and hospital charges. J. Clin. Microbiol. 41, 3119 (2003).
- Raymaekers, M., Bakkus, M., Boone, E., de Rijke, B., Housni, H. E. l., Descheemaeker, P., De Schouwer, P., Franke, S., Hillen, F., Nollet, F., Soetens, O., Vankeerberghen, A. Molecular Diagnostics working group. Reflections and proposals to assure quality in molecular diagnostics. Acta. Clin. Belg. 66, 33 (2011).
- Peters, R. P. New developments in the diagnosis of bloodstream infections. Lancet Infect. Dis. 4, 751 (2004).
- Hansen, W. L., Beuving, J., Bruggeman, C. A., Wolffs, P. F. Molecular probes for the diagnosis of clinically relevant bacterial infections in blood cultures. J. Clin. Microbiol. 48, 4432-4432 (2010).
- Beuving, J. Antibiotic susceptibility testing of grown blood cultures by combining culture and real-time polymerase chain reaction is rapid and effective. PLoS ONE. 6, (2011).
- Rolain, J. M., Mallet, M. N., Fournier, P. E., Raoult, D. Real-time PCR for universal antibiotic susceptibility testing. J. Antimicrob. Chemother. 54, 538 (2004).
- Nadkarni, M. A., Martin, F. E., Jacques, N. A., Hunter, N. Determination of bacterial load by real-time PCR using a broad-range (universal) probe and primers set. Microbiol. 148, 257 (2002).
- Waites, K. B., Brookings, E. S., Moser, S. A., Zimmer, B. L. Direct susceptibility testing with positive BacT/Alert blood cultures by using MicroScan overnight and rapid panels. J. Clin. Microbiol. 36, 2052 ( ).
- Vliegen, I. Rapid identification of bacteria by real-time amplification and sequencing of the 16S rRNA gene. J. Microbiol. Meth. 66, 156 (2006).
- Hall, L., Doerr, K. A., Wohlfiel, S. L., Roberts, G. D. Evaluation of the MicroSeq system for identification of mycobacteria by 16S ribosomal DNA sequencing and its integration into a routine clinical mycobacteriology laboratory. J. Clin. Microbiol. 41, 1447 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados