Method Article
Un giorno Scheme flusso di lavoro per batterica Rilevazione dei patogeni e test di resistenza antimicrobica dalle emocolture
In questo articolo
Riepilogo
La progettazione di un semplice schema di un giorno di workflow per la diagnostica dei patogeni batterici permette il rapido riconoscimento di infezioni del sangue. L'inclusione di otto obiettivi clinicamente rilevanti batteriche ed i loro profili di resistenza agli antibiotici offre al medico una prima idea il giorno stesso, che può portare ad una terapia più adeguata.
Abstract
Infezioni del sangue sono associati con alti tassi di mortalità a causa della manifestazione probabile di sepsi, sepsi grave e shock settico 1. Pertanto, la somministrazione rapida di una terapia antibiotica è di primaria importanza nel trattamento delle infezioni del sangue. L'elemento fondamentale in questo processo è di temporizzazione, fortemente dipendente dai risultati di identificazione batterica e test di sensibilità agli antibiotici. Entrambi questi parametri sono comunemente ottenuti dalla cultura test basato, che richiede molto tempo e richiede in media 24-48 ore 2, 4. Lo scopo dello studio era di sviluppare saggi a base di DNA di identificazione rapida di infezioni del sangue, nonché un rapido test di suscettibilità antimicrobica. Il primo saggio è una 16S rDNA eubacterial-based real-time PCR integrate con specie o dei generi di sonde specifiche 5. Utilizzando queste sonde, i batteri Gram-negativi compreso Pseudomonas spp., Pseudomonas aeruginosae Escherichia coli, nonché batteri gram-positivi compresi Staphylococcus spp., Staphylococcus aureus, Enterococcus spp., Streptococcus spp., e Streptococcus pneumoniae potrebbero essere distinti. Usando questo saggio multisonda, una identificazione prima della causativo microrganismo è stato dato dopo 2 ore.
In secondo luogo, abbiamo sviluppato un semi-test molecolare per i test di sensibilità agli antibiotici di S. aureus, Enterococcus spp. e (facoltativo) aerobi gram-negativi 6. Questo saggio è stato basato su uno studio in cui venne usato PCR per misurare la crescita di batteri 7. I batteri raccolti direttamente da colture di sangue vengono incubate per 6 h con una selezione di antibiotici, e dopo un SYBR Green basato real-time PCR determina inibizione della crescita. La combinazione di questi due metodi possono indirizzare la scelta di una terapia antibiotica idonea lo stesso giorno (Figura 1). Insomma, L'analisi molecolare sia di identificazione e sensibilità agli antibiotici offre una alternativa più veloce per il rilevamento degli agenti patogeni e potrebbe migliorare la diagnosi delle infezioni del sangue.
Protocollo
PARTE I: IDENTIFICAZIONE PATHOGEN
1. Preparazione del campione
Nota: L'intero flusso di lavoro molecolare, come è descritto nel seguente protocollo deve essere eseguita secondo le raccomandazioni per la garanzia della qualità nella diagnostica molecolare 3.
- Aggiungere una aliquota 0,1 ml di coltura di sangue a 0,9 ml di NaCl allo 0,9% in una provetta da 1,5 ml di reazione per diventare un campione diluito 1:10. (Diluire al fine di impedire l'inibizione qPCR).
- Centrifugare il campione a 13.400 xg per 5 minuti a pellet del DNA batterico.
- Risospendere il pellet batterico in 100 microlitri sterile demineralizzata H 2 O.
- Conservare il campione di DNA a 4 ° C fino ad ulteriore utilizzo.
2. Identificazione Assay: Real-time PCR 16S rDNA
- Preparare le miscele di reazione come segue. Il test consiste di quattro reazioni separate per campione. Ogni miscela contiene 12,50 maestro plmix, 0,9 uM avanti di primer (5-TCCTACGGGAGGCAGCAGT-3) 7, primer 0,6 micron inverso (5-GGACTACCAGGGTATCTAATCCTGTT-3) 8 e un pannello di sonde. La quantità di sonde è dato per ciascuno dei quattro reazioni separate in seguito.
- La prima reazione comprende:
- 0,2 pM sonda universale (5-FAM-CGTATTACCGCGGCTGCTGGCAC-3-BHQ1) 8
- 0,2 pM P. aeruginosa sonda (5-JOE-CCAAAACTACTGAGCTAGAGTACG-3-BHQ1)
- La seconda reazione comprende:
- 0,2 pM E. coli sonda (5-JOE-GGAGTAAAGTTAATACCTTTGCTCATT-3-BHQ1)
- 0,2 pM Pseudomonas spp. Sonda (5-NED-CCTTCCTCCCAACTTAAAGTGCTT-3-MGBNFQ)
- La terza reazione comprende:
- 0,2 pM Staphylococcus spp. Sonda (5-NED-AATCTTCCGCAATGGGCGAAAGC-3-MGBNFQ)
- 0,2 pM S. aureus sonda (5-FAM-AGATGTGCACAGTTACTTACACATAT-3-BHQ1)
- 0,2 uM Enterococcus spp. (5-JOE-TCCTTGTTCTTCTCTAACAACAGAG-3-BHQ1)
- La quarta reazione comprende:
- 0,2 pM sonda universale (5-FAM-CGTATTACCGCGGCTGCTGGCAC-3-BHQ1)
- 0,3 pM Streptococcus spp. Sonda (5-NED-CCAGAAAGGGACSGCTAACT-3-MGBNFQ)
- 0,2 pM S. pneumoniae sonda (5-JOE-CCAAAGCCTACTATGGTTAAGCCA-3-BHQ1)
- Aggiungere demineralizzata sterile H 2 O a raggiungere un volume totale di 20 pl. Aggiungere 20 pl di ciascuna miscela di reazione ai pozzetti di una piastra a 96 pozzetti PCR.
- Aggiungere 5 pl di campione in ciascun pozzetto.
- Usare un film adesivo per sigillare la piastra a 96 pozzetti PCR.
- Eseguire la piastra sul ABI PRISM 7900HT sistema in tempo reale PCR utilizzando le seguenti condizioni ottimali cicli termici:
- Pre-riscaldamento a 50 ° C per 10 min
- Denaturazione iniziale a 95 ° C per 15 min
- 42 cicli di
- Denaturazione a 95 ° C per 15 s
- Ricottura a 60 ° C per 1 min
3. Analisi dei risultati
Regolare la soglia del Analysis Ct a 0,1 nelle impostazioni di analisi della scheda. Limitare le configurazioni di base per iniziare (ciclo): 6 e End (Cycle): 15.
- Registrare il ciclo soglia (Ct) il valore per tutti i campioni. Il valore di cut-off di considerare un risultato positivo come PCR può essere impostato ad un valore di Ct-35. La quantità di batteri presenti in colture di sangue variava da 10 luglio - 10 Novembre CFU / ml, generando Ct valori inferiore a 35.
PARTE II: PROVE DI SENSIBILITA 'AGLI ANTIBIOTICI
4. Isolamento di batteri da emocolture positive 9
- Aspirare 5 ml di brodo da una bottiglia coltura positiva sangue e trasferirlo in un tubo separatore siero.
- Centrifugare la provetta siero separatore a 2000 xg per 10 min.
- Eliminare il surnatante dal tubo separatore siero.
- Transfer bacbatteri dallo strato di gel del tubo con un tampone di cotone sterile in soluzione fisiologica 0,9% fino a McFarland 0,5 sospensione titolata è ottenuta.
5. Inoculazione di Micro Piastre Titre
- Diluire il 0,5 sospensione McFarland in doppia concentrato Mueller Hinton II brodo per formare una sospensione di 5 x 10 5 CFU / ml.
- Aggiungere questa sospensione ai pozzetti di una piastra di micro titolo contenente una selezione di antibiotici (Tabella 1).
- Incubare la piastra micro titolo a 37 ° C per 6 h.
- Conservare una aliquota della sospensione a 4 ° C (come controllo negativo della crescita).
- Dopo 6 ore di incubazione, trasferire il contenuto di ciascun pozzetto in una provetta sterile, così come il campione negativo di controllo della crescita che è stato conservato a 4 ° C.
- Centrifugare le provette a 16000 xg per 5 min.
- Rimuovere con cautela il surnatante, senza disturbare il pellet batterico.
- Risospendere il pellet in H sterile demineralizzata 2 O.
- Diluire i campioni di 10 volte in sterile demineralizzata H 2 O.
6. Real-time PCR 16S rDNA 10
- Preparare la miscela di PCR come segue:
- 12,50 ul iQ SYBR Green Supermix
- 0,5 pM innesco in avanti 16S-1 (5-TGGAGAGTTTGATCCTGGCTCAG-3) 11
- 0,25 16S-2 pM primer reverse (5-TACCGCGGCTGCTGGCAC-3) 11
- demineralizzata sterile H 2 O a un volume totale di 20 pl
- Aggiungere 20 pl di miscela PCR ai pozzetti di una piastra a 96 pozzetti PCR.
- Aggiungere 5 pl di campione in ciascun pozzetto.
- Usare un film adesivo per sigillare la piastra a 96 pozzetti PCR.
- Esegui il piatto sul singolo colore MyiQ Real-Time PCR Detection System, usando le seguenti condizioni ottimali cicli termici:
- Denaturazione iniziale a 95 ° C per 4 min
- Ricottura iniziale a 65 ° C per 30 s
- 35 cicli di
- Denaturazione a 95 ° C per 15 s
- Ricottura a 60 ° C per 1 min
- Melt analisi della curva (60-95 ° C in 20 min con incrementi di 0,57 ° C)
7. Analisi dei risultati
- Calcolare il cut-off Ct utilizzando una delle seguenti formule (a seconda del tipo di antibiotico).
- In generale:
Cut-off Ct = valore Ct valore positivo controllo della crescita + 0,5 x (valore negativo Ct controllo della crescita - il valore Ct positivo controllo della crescita) - Piperacillina, piperacillina / tazobactam e ceftazidime in gram-negativi, Amoxicillina, oxacillina e trimetoprim / sulfametossazolo in S.aureus, amoxicillina in Enterococcus spp. Cut-off Ct = valore Ct positivo controllo della crescita + 0,25 x (Ct crescita del valore negativo controllo - il valore Ct positivo controllo della crescita)
- In generale:
- Utilizzare il campione incubato con sterile demineralizzata H 2 O, come controllo della crescita positivo.
- A seconda del microrganismo, utilizzare il controllo appropriato crescita negativa:
- Gram-negativi Esempio aste incubate con miscela di antibiotici
- Enterococcus spp. Campione conservato a 4 ° C
- Esempio S.aureus conservati a 4 ° C
- Determinare la suscettibilità (S) o resistenza (R) del ceppo per l'antibiotico testate come segue:
- Un valore Ct superiore al valore di cut-off Ct indica suscettibilità
- Un valore Ct inferiore al valore di cut-off Ct indica la resistenza
8. Risultati rappresentativi
Due organismi modello, cioè un Gram-negativo E. coli e Gram-positivi S. aureus, vengono scelti per visualizzare la procedura combinata per il rilevamento e l'identificazione di patogeni batterici e la determinazione del loro profilo antimicrobica. La prima parte del protocollo comprende l'identificazione patogeno. Spesonde specifici sono progettate per il rilevamento di otto microrganismi clinicamente rilevanti. In presenza di un bersaglio incluso nel pannello batterica, curve di amplificazione vengono generati e valori di Ct sono calcolati (Figura 2). Il valore di cut-off di considerare un risultato positivo PCR come è impostata su un valore di Ct 35. In figura 2A, il profilo di identificazione di un E.coli cultura infettata ematico. La sonda 16S universale è incluso in due miscele di reazione separati e genera di conseguenza due curve di amplificazione (Ct di 25,20 e 25,95). Il terzo segnale è derivato dalla sonda specifica per E. coli (Ct di 27,04). L'identificazione di un S. aureus-cultura sangue infetto è mostrato nella figura 2B. La sonda universale 16S ha acquisito il segnale di amplificazione di 33,35 e 33,71. I due segnali rimanenti sono derivate dalle sonde specifiche per Staphylococcus spp. e S. aureus (Ct di 32,48 e 30,59).
"jove_content" Dopo la prima parte del protocollo, il microrganismo responsabile è nota ed il profilo antimicrobica può essere determinata. Figura 3 è un esempio di un antibiotico trama amplificazione test di sensibilità, che rappresenta il ceppo E. coli che è stato anche dimostrato nella Figura 2A. Ogni riga rappresenta un antibiotico che il campione è stato incubato con batterica. Un campione con un valore basso Ct è un esempio in cui la crescita si è verificata in presenza di un antibiotico, indicando resistenza all'antibiotico testato. Al contrario, un valore elevato Ct rappresenta un campione in cui si è verificata alcuna crescita a causa del funzionamento efficace del antibiotico, indicando suscettibilità all'antibiotico testato. La Tabella 1 illustra la determinazione del profilo antimicrobica della E. coli e S. aureus isolati. Tutti i valori sono riportati Ct e, utilizzando le formule indicate nel testo del protocollo (7.1), due cut-off valori Ct sono calcolatiregolamentato di distinguere tra resistenza e suscettibilità. Il ceppo è resistente all'antibiotico se il valore riportato Ct è inferiore al calcolato cut-off Ct (e viceversa).
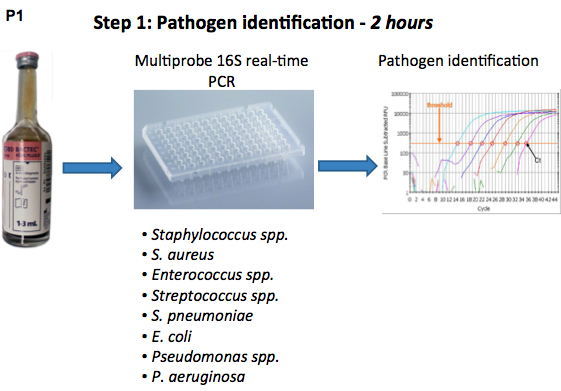
Figura 1. Diagramma di flusso del patogeno identificazione e test di suscettibilità agli antibiotici procedure utilizzando real-time PCR 16S rDNA.
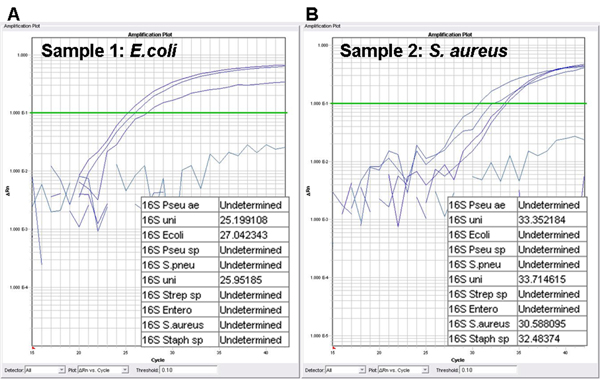
Figura 2 Identificazione dosaggio:. Trame di amplificazione e valori ciclo soglia (valori di Ct) Una coltura di sangue positiva viene rilevata dal universale 16S rDNA sonda, mentre le sonde specifiche vengono utilizzati per l'identificazione del patogeno causale.. Amplificazione plot A. della cultura sangue contenente E. coli; amplificazione trama B. della cultura sangue contenente S. aureus, pseudo ae, Pseudomoncome aeruginosa; uni, 16S sonda universale; Ecoli, Escherichia coli sonda; pseudo sp, Pseudomonas spp. sonda; S. pneu, Streptococcus pneumoniae sonda; Strep sp, Streptococcus spp. sonda; Entero, Enterococcus spp. sonda; S. aureus, Staphylococcus aureus sonda; Staph sp, Staphylococcus spp. sonda.
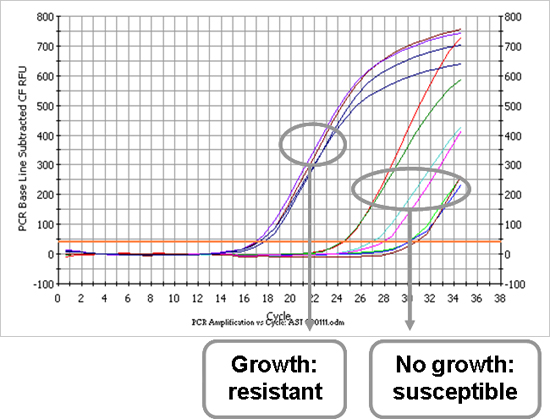
Figura 3. Plot di amplificazione delle prove di suscettibilità agli antibiotici di E. coli isolare (campione 1). Ciascuna curva rappresenta un antibiotico che il ceppo è stato incubato con. Un primo segnale è causata da un elevato carico batterico, il che significa che il ceppo è cresciuto in presenza dell'antibiotico testato ed è quindi resistente all'antibiotico. Segnali tardiva indicano che il ceppo non è cresciuto in presenza dell'antibiotico, in altre parole, esso è suscettibile.
| Esempio 1: E. coli | Esempio 2: S. aureus | ||||
| AST | Ct | R / S | AST | Ct | R / S |
| Amoxicillina 8 mg / L | 16,83 | R | Amoxicillina 0,25 mg / L | 21,03 | R |
| Amoxicillina-acido clavulanico 8/4 mg / L | 17,36 | R | Oxacillin 2 mg / L | 25,80 | S |
| Piperacillina 16 mg / L | 16,67 | R | Vancomicina 2 mg / L | 25,20 | S |
| Piperacillina-tazobactam 16 /4 mg / L | 24,15 | S | Gentamicina 4 mg / L | 25,86 | S |
| Ciprofloxacina 1 mg / L | 29,72 | S | Trimetoprim-sulfametossazolo 2/38 mg / L | 24,62 | S |
| Ceftazidime 1 mg / L | 24,03 | S | |||
| Ceftazidime 8 mg / L | 26,58 | S | |||
| Gentamicina 4 mg / L | 29,83 | S | |||
| Trimetoprim-sulfametossazolo 2/38 mg / L | 27,60 | S | |||
| Controllo della crescita negativo (miscela di antibiotici) | 30,41 | Controllo della crescita negativo (campione conservato a 4 ° C) | 27,42 | ||
| Controllo positivo di crescita | 16,90 | Controllo positivo di crescita | 20,22 | ||
| Cut-off Ct-valore 1 * | 21,76 | Cut-off Ct-valore 1 *** | 23,82 | ||
| Cut-off Ct-valore 2 ** | 18,75 | Cut-off Ct-valore 2 **** | 22,02 | ||
| * Per amoxicillina, amoxicillina-acido clavulanico, ciprofloxacina, gentamicin, trimetoprim-sulfametossazolo ** Per piperacillina, piperacillina-tazobactam, ceftazidime | *** Per vancomicina e gentamicina **** Per amoxicillina, oxacillina e trimetoprim-sulfametossazolo |
Tabella 1. Determinazione del test di sensibilità agli antibiotici dei due campioni (E. coli e S. aureus). Valori Ct della PCR-analisi sono stati copiati a questo file excel, in quanto in grado di calcolare automaticamente i due cut-off ALORI Ct dal controllo di crescita positivo e negativo, utilizzando le formule indicate nel testo del protocollo. Se un antibiotico mostra un valore Ct inferiore al valore soglia Ct, il ceppo è resistente all'antibiotico, se il valore Ct è maggiore del cut-off, il ceppo è sensibile.
Discussione
Il protocollo qui descritto permette la rapida identificazione di agenti patogeni e fornisce un profilo funzionale antimicrobico che potrebbe portare alla somministrazione precoce di antibiotici adeguati, migliorando così la prognosi dei pazienti con infezioni del sangue. A seconda delle condizioni richieste di test, cioè a basso costo, velocità elevata, minimo tempo di ritorno, condizioni di prova può essere regolata. L'intera procedura può essere eseguita entro un giorno lavorativo. Inoltre, le due parti del protocollo possono essere eseguite contemporaneamente, che riduce il tempo di ritorno in modo significativo. Come qui presentata, il pannello di identificazione è una selezione dei batteri più clinicamente rilevanti nel nostro ospedale. Poiché il principio fondamentale è il targeting del gene 16S regione, sonde specifiche per altri microrganismi possono essere progettati e aggiunte al saggio. Il dosaggio completo era originariamente prevista per l'analisi rapida di emocolture, ma può anche essere utilizzato per la trasformazione di oltre campioni di materiali. Questo è anche il caso per gli antibiotici che sono stati utilizzati per i test di sensibilità agli antibiotici: antibiotici più o di altro tipo possono essere aggiunti, in base a pattern di resistenza locali e le linee guida.
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato dal azm Profileringsfonds (PF245).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| Cloruro di sodio (NaCl) | Merck Chemicals | 106404 | 0,9% in acqua |
| Vacutainer SST Siero Tubo Separator 5 ml | BD Diagnostic Systems | 367986 | |
| Mueller Hinton II brodo | BD Diagnostic Dystems | 212322 | 44 g / L in acqua |
| Centrifugare Rotixa 50 rs | Andreas Hettich GmbH & Co. KG | 4910 | |
| Centrifuga 5415 D | Eppendorf | Fuori | |
| Primers | Sigma-Aldrich | na | |
| Sonde | Sigma-Aldrich/Applied Biosystems | na | |
| TaqManEnvironmental master mix 2,0 | Applied Biosystems | 4396838 | |
| iQ SYBRGreen Supermix | Bio-Rad Laboratories BV | 170-8880 | |
| MicroAmp ottica piastra a 96 pozzetti di reazione | Applied Biosystems | N8010560 | |
| MicroAmp Film adesivo ottico | Applied Biosystems | 4311971 | |
| iQ 96 pozzetti PCR Piastre | Bio-Rad Laboratories BV | 223-9441 | |
| Microseal B Adesivo Seals | Bio-Rad Laboratories BV | MSB-1001 | |
| Real-time PCR Detection System | Applied Biosystems | ABI PRISM 7900HT | |
| Real-Time PCR Detection System | Bio-Rad Laboratories BV | MyiQ singolo-Color |
Riferimenti
- Wallet, F. Preliminary clinical study using a multiplex real-time PCR test for the detection of bacterial and fungal DNA directly in blood. Clin. Microbiol. Infect. 16, 774 (2010).
- Beekmann, S. E., Diekema, D. J., Chapin, K. C., Doern, G. V. Effects of rapid detection of bloodstream infections on length of hospitalization and hospital charges. J. Clin. Microbiol. 41, 3119 (2003).
- Raymaekers, M., Bakkus, M., Boone, E., de Rijke, B., Housni, H. E. l., Descheemaeker, P., De Schouwer, P., Franke, S., Hillen, F., Nollet, F., Soetens, O., Vankeerberghen, A. Molecular Diagnostics working group. Reflections and proposals to assure quality in molecular diagnostics. Acta. Clin. Belg. 66, 33 (2011).
- Peters, R. P. New developments in the diagnosis of bloodstream infections. Lancet Infect. Dis. 4, 751 (2004).
- Hansen, W. L., Beuving, J., Bruggeman, C. A., Wolffs, P. F. Molecular probes for the diagnosis of clinically relevant bacterial infections in blood cultures. J. Clin. Microbiol. 48, 4432-4432 (2010).
- Beuving, J. Antibiotic susceptibility testing of grown blood cultures by combining culture and real-time polymerase chain reaction is rapid and effective. PLoS ONE. 6, (2011).
- Rolain, J. M., Mallet, M. N., Fournier, P. E., Raoult, D. Real-time PCR for universal antibiotic susceptibility testing. J. Antimicrob. Chemother. 54, 538 (2004).
- Nadkarni, M. A., Martin, F. E., Jacques, N. A., Hunter, N. Determination of bacterial load by real-time PCR using a broad-range (universal) probe and primers set. Microbiol. 148, 257 (2002).
- Waites, K. B., Brookings, E. S., Moser, S. A., Zimmer, B. L. Direct susceptibility testing with positive BacT/Alert blood cultures by using MicroScan overnight and rapid panels. J. Clin. Microbiol. 36, 2052 ( ).
- Vliegen, I. Rapid identification of bacteria by real-time amplification and sequencing of the 16S rRNA gene. J. Microbiol. Meth. 66, 156 (2006).
- Hall, L., Doerr, K. A., Wohlfiel, S. L., Roberts, G. D. Evaluation of the MicroSeq system for identification of mycobacteria by 16S ribosomal DNA sequencing and its integration into a routine clinical mycobacteriology laboratory. J. Clin. Microbiol. 41, 1447 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon