É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
RNAi de triagem para identificar fenótipos pós-embrionário em C. elegans
Neste Artigo
Resumo
Nós descrevemos um método sensibilizada para identificar os reguladores pós-embrionário de expressão da proteína e localização no C. elegans Usando uma tela de genoma baseada em RNAi e um transgene integrado que expressa uma proteína fluorescente funcional marcada.
Resumo
C. elegans tem provado ser um sistema de modelo valioso para a descoberta e caracterização funcional de muitos genes e vias gene 1. Instrumentos mais sofisticados e recursos para estudos neste sistema estão facilitando a descoberta contínua de genes com fenótipos mais sutis ou funções.
Aqui nós apresentamos um protocolo generalizada, adaptado para a identificação de C. elegans genes com fenótipos pós-embrionário de interesse usando RNAi 2. Este procedimento é facilmente modificado para ensaio do fenótipo de escolha, seja por óptica de luz ou de fluorescência em um microscópio de dissecação ou composto. Este protocolo de triagem capitaliza os ativos físicos do organismo e ferramentas moleculares a C. elegans comunidade científica tem produzido. Como um exemplo, que demonstram o uso de um transgene integrado que expressa um produto fluorescente em uma tela RNAi para identificar genes necessários para a localização normal desteproduto em larvas fase tardia e adultos. Em primeiro lugar, utilizou-se uma biblioteca genómica comercialmente disponível RNAi com full-length inserções de ADNc. Esta biblioteca facilita a rápida identificação de vários candidatos por RNAi redução do produto do gene candidato. Em segundo lugar, gerou um transgene integrado que expressa nossa proteína fluorecently marcados de interesse em um fundo RNAi-sensível. Em terceiro lugar, por exposição dos animais a eclodidos RNAi, esta tela permitir a identificação de produtos de genes que desempenham um papel vital embrionário que de outra forma mascarar um papel pós-embrionário na regulação da proteína de interesse. Por último, este ecrã utiliza um microscópio composto equipado para a resolução de célula única.
Protocolo
1. Construção estirpe de Triagem
O desenho cuidadoso da estirpe de rastreio é crítica para o sucesso da tela e tem sido descrito em outro lugar 3. Para alguns investigadores, utilizando uma estirpe que expressa um produto visível a partir de um transgene é necessária para a experiência. Muitas cepas abrigando transgenes integrados estão disponíveis os pesquisadores CGC ou individual. Se uma estirpe transgénica é necessário para a tela, mas não estiver disponível, então ele pode ser gerada utilizando um método publicado como bombardeamento 4, UV / TMP 5, ou transposão Mos inserção 6. A fim de visualizar a nossa proteína de interesse, inseriu-se o sequência de codificação da GFP-em moldura com a sequência madura de cDNA (usamos dbl-1 sequência). Porque esta proteína de fusão GFP não é visível através da dissecção âmbito, foi utilizado um marcador coinjection que era visível e não afetou a visualização de nossa proteína de interesse (TTX-3p :: rfp , para uma revisão de vários outros marcadores padrão, ver 7). Criamos, então, um transgene integrado da matriz extracromossómico. Nós descobrimos que o bombardeio do transgene rendeu um baixo número de cópia linha integrado que nem resgatou o fenótipo estereomicroscópio nem produziu níveis visíveis de produto GFP-tagged (Beifuss e Gumienny, inédito). UV / TMP integração de uma matriz de múltiplas cópias extracromossómico originalmente feito por injecção 8,9 rendeu visível, emergência níveis de produto do transgene (Figura 3A). Western blot com anti-GFP anticorpo confirmou que o transgene (nome alelo texIs100) é expressa e processados correctamente (Beifuss e Gumienny, não publicado). Transgenes integrados deverão ser outcrossed cinco vezes para remover mutações fundo estranhos, independentemente da fonte.
Para nossa tela, queríamos a única proteína de interesse a ser a forma, transgênicos marcados. Portanto, nós removemos o g funcional endógenaeno através da introdução de uma perda de função-mutante dbl-1 alelo.
Finalmente, a estirpe sensibilizados para os efeitos de RNAi. O RNAi a partir da biblioteca, por muitos genes, produzem uma redução mais grave no produto do gene se os animais conter uma mutação que sensibiliza os animais aos efeitos da RNAi 10,11 O tecido (s) de interesse deverá ser considerada quando se escolhe um adequado RNAi sensibilizar fundo 11. Utilizou-se o alelo RRF-3 (pk1426) canônico para tornar a nossa estirpe de triagem. RRF-3 é um RNA-polimerase dirigida RNA homólogo (RdRp) que normalmente inibe somática RNAi 10. Mutações em outros genes RNAi hypersensitizing como eri-1 ou eri-1 lin-15b pode ser usado em vez de aumentar a eficácia do RNAi 11-13.
A estirpe de rastreio que fizemos tem o genótipo RRF-3 (pk1426); texIs100; dbl-1 (NK3).
2. Escolha e preparação doRNAi biblioteca
C. comercialmente disponível bibliotecas de cDNA elegans representam cerca de 55% ou 87% dos genes previstos em C. elegans individualmente (Vidal laboratório ou bibliotecas Ahringer laboratório, respectivamente). Clones individuais estão disponíveis para compra (Open Biosystems, Geneservice, Ltd.). Nós escolhemos a biblioteca ORF-RNAi construído pelo laboratório Vidal (Aberto Biosystems) porque os seus clones são na sua maioria de comprimento completo do Gateway cDNAs clonados e pronto para aplicações a jusante (Invitrogen Corporation, Carlsbad, CA). O original Ahringer biblioteca genômica laboratório cDNA contém fragmentos de cDNA que não são tão amplamente útil para experimentos de caracterização a jusante 14,15. Ambas as bibliotecas utilizar um vector que contém dois promotores T7 que flanqueiam o inserto (Figura 1). As construções são cultivadas em estirpe bacteriana HT115, que expressa a polimerase de T7 indução por isopropil-β-D-tiogalactopiranósido (IPTG). As bactérias contendo clones da biblioteca são induzidas com IPTG paraproduzir dsRNA (ver passo 3.4).
Duplicar a biblioteca inteira após o recebimento e usar o duplicado para todos os experimentos. As bibliotecas originais e duplicados devem ser armazenados em freezers diferentes -80 ° C que estão conectados para separar as linhas elétricas.
3. Preparação de bactérias com biblioteca de clones
- (Dentro de dois meses do experimento) Streak bactérias a partir de clones selecionados de alimentação da biblioteca, bem como controles positivos e negativos, sobre rotulados LB-carbenicilina / tetraciclina placas (nós crescemos 8 culturas por placa de 100 mm) e incubar a 37 ° C durante a noite ( > 16 horas) (Figura 1). Para cada placa de 24 cavidades, usamos dois controles. O controlo positivo, bli-4 (K04F10.4), produz uma dose-dependente pheontype pós-embrionário 16,17. O controlo negativo, C06C3.5, é um pseudogene predito ( WormBase.org ) que faz com que não fenótipos observados (Beifuss e Gumienny, não publicado).
- (Dentro de dois meses de experimento, de preferência cedo) Preparar 24-bem Nematóides Crescimento Médio (NGM) placas contendo 25 - 50 mcg / ml carbenicilina e 1 mM IPTG. Armazenar a 4 ° C.
- (Dia 1) Para cada clone, inocular 2 ml meio LB contendo 50 ug / ml de carbenicilina com uma única colónia da placa de riscado (s) (no Passo 3,1), utilizando uma técnica estéril padrão. Incubar a 37 ° C durante a noite (> 16 horas) em um conjunto agitador para 220-240 rpm (Figura 1). A solução deve ser turvo, após 16 horas.
- (Dia 2) Na manhã seguinte, induzir a produção de RNA de cadeia dupla (dsRNA) pela adição de IPTG para as culturas a uma concentração final de 1 mM. Incubar as culturas a 37 ° C, 220 - 240 rpm, para um adicional de 4 - 5 horas. Este passo garante uma mais consistente e robusto knockdown RNAi induzida do produto do gene de contar apenas com o IPTG no agar da placa NGM (no Passo 3,5).
- Spot de 30 ul de cada cu induzidas bacterianasLTURE em poços separados de uma placa de 24 poços RNAi NGM contendo 25 - 50 ug / ml de carbenicilina e 1 mM de IPTG (Figura 1, ver Figura 4 para o modelo utilizado para rastrear experiências RNAi). Coloque placas, descoberto, em uma câmara de fluxo estéril por 20 minutos ou até que as manchas bacterianas estão secos. Não overdry. Overdrying faz com que as bordas do agar para se afastar da placa e os animais serão irrecuperável se rastejar nas fendas resultantes.
4. Preparação dos nemátodos
Comece com uma população de nematóides encenado. Estadiamento animais diminui a possibilidade de que as diferenças observadas entre o controlo e os ensaios experimentais RNAi são simplesmente devido a diferenças no estágio de desenvolvimento dos animais (no entanto, se o RNAi provoca um atraso no desenvolvimento, isto deve ser notado pelo screener (Figura 2)) . Além disso, do início do experimento RNAi com larvas L1 elimina os potenciais efeitos de confusão de letalidade embrionária por RNAi de genes que também podem desempenhar papéis pós-embrionário de interesse.
De teste é realizado por primeiro branqueamento uma população mista de estágio da estirpe de triagem, o que apenas casca de ovo-protegidas embriões sobreviver. Este fases animais para dentro de cerca de 12 horas a 20 ° C ou cerca de 18 horas a 16 ° C 18. Para mais firmemente animais fase, animais de prisão na primeira fase larval (L1), deixando escotilha embriões em meio M9 sem alimentos. Animais L1 Starved colocado sobre o crescimento resumo alimento da mesma idade de partida 19.
- (Dia 1) Use três placas de 100 mm de confinamento, os animais de triagem limpas tensão que contêm adultos grávidos e embriões. Lavar os animais a partir de placas utilizando M9 estéril por pipetagem líquido através da superfície da placa com cuidado para remover os animais e embriões. Transferir a solução de lavagem para um tubo de 15 ml estéril com tampa de rosca.
- Se a solução de lavagem é maior do que 3,5 ml, girar para baixo os animais (~ 3000 rpm durante 30 segundos) e remover o líquido para 3,5 ml. Se a solução de lavagem é lenos de 3,5 ml, adicionar H 2 0 ou M9 ao tubo para totalizar 3,5 ml.
- Usar equipamento de proteção adequado (jaleco, luvas e óculos de proteção) ao manusear produtos químicos. Misturar 0,5 ml NaOH 5 N com 1 ml de lixívia fresco (hipoclorito de sódio a 5%, menos de 1 ano de idade). Adicionar esta mistura para a lavagem de nemátodos 3,5 ml. Iniciar um cronômetro em contagem acima. Este processo não deve demorar mais de 10 minutos. Se isso acontecer, então embriões poderá ser comprometido eo rendimento pode ser reduzido, ou a lixívia é velho e deve ser substituído.
- O tubo de vórtice durante cerca de 10 segundos. Repita a vórtex a cada dois minutos para 4 a 10 minutos. Após cada centrifugação, observar a solução em um escopo de dissecação de cadáveres de animais.
- Quando os embriões são libertados e adultos são fragmentados (6 - 8 minutos), os embriões pelete por centrifugação do tubo numa centrífuga tampo da mesa (~ 3000 rpm, 30 segundos).
- Aspirar o máximo do sobrenadante quanto possível, sem perturbar o sedimento. Tenha cuidado, nãoganancioso.
- Adicionar M9 estéril para ~ 10 ml. Brevemente Vortex ou agite bem para ressuspender o sedimento.
- Spin e remover o sobrenadante novamente. Se o cheiro de lixívia pode ser detectado no tubo, repetir lavagem conforme necessário.
- Ressuspender embriões em 10 - 15 ml estéril M9 e transferência para um pequeno frasco de Erlenmeyer (25 ml) (para aeração). Agitar à temperatura adequada para a estirpe e experiência (15 - 25 ° C) durante a noite (> 16 horas a 20 ° C). Neste tempo, os animais serão todos eclodem e parada na diapausa L1.
- (Dia 2) girar para baixo L1 animais fase de um tubo de 15 ml e ressuspender em 0,5 - 1 ml M9 (~ 3000 rpm, 30 seg.). Coloque 5 uL solução sobre uma placa separada e contar o número de animais. Faça ajustes de volume, se necessário, para produzir 30 worms / 3 - 10 uL de solução.
- Manchar aproximadamente 30 hipoclorito animais L1 sincronizados fase para as bactérias em cada poço da placa de 24 cavidades preparada. Mo muitosre de 30 animais por poço pode resultar na população faminta antes de atingir a maturidade. Deixe a placa seca com o askew tampas. Recoloque a tampa e prenda com um elástico. Inverta a placa e colocá-lo à temperatura adequada durante o tempo necessário para marcar na fase desejada. Devido RRF-3 (pk1426) é um alelo sensível à temperatura, os nossos animais da linhagem de rastreio foram crescidas a 15-17 ° C durante três a quatro dias para observar L4 para animais fase adulta (Figura 1).
5. Observação de nematóides
(Dia 5) Uma vez que os nemátodos ter crescido para a fase desejado (Figura 2A), confirmar que os controlos positivos e negativos produzem os fenótipos esperados usando um microscópio de dissecação. Usamos bli-4 como um controlo para a eficácia RNAi, porque produz dependentes da dose pós-embrionárias defeitos que variam de adultos bolhas (fenótipo RNAi leve, não mostrado) para developmentally preso larvas, minúscula (e, fortexpected fenótipo RNAi) (Figura 2B). Em seguida, observar os animais em cada experimental bem usando um microscópio de dissecação e nota óbvias fenótipos anormais induzida por RNAi (Figura 2C). Depois que os controles são confirmadas e fenótipos bruta observado, filtrar os experimentos de RNAi para fenótipos de interesse. Usamos um microscópio composto equipado com fluorescência e um objectivo 63x observar pelo menos cinco animais de cada experimento RNAi, começando com os controlos (Figura 3).
- Prepara-se uma corrediça (padrão 75 x 25 mm), fazendo uma almofada de agar 4% (4% de agar em H2O). Para assegurar uma espessura uniforme de almofada de ágar, colocar uma lâmina de vidro limpa entre dois espaçadores (cada espaçador é feita de uma lâmina de microscópio com duas camadas de fita de laboratório sobre ele). Pipetar cerca de 100 uL (duas a três gotas de uma pipeta de Pasteur de vidro) de ágar-ágar 4% fundido sobre o centro da lâmina de vidro limpo. Formar uma almofada de ágar, cobrindo o agar fundido usando uma lâmina adicional colocada rapidamente, mas suavemente em cima dos espaçadores umnd o agar fundido. Exponha o bloco de ágar, deslizando as lâminas de vidro à parte, deixando o pad aderir a um slide.
- Coloque um ~ 5 uL gota de anestésico (levamisole ou azida de sódio, por exemplo) sobre a almofada de ágar. Mount 8 - 12 animais do anestésico. Cubra com um 22 x 22 mm # 1,5 lamela (ou a espessura da lamela mais adequado para o microscópio composto) e observar animais utilizando um microscópio composto.
- Genes Record, número de animais observados, e fenótipo resultados para cada experimento RNAi. Para uma tela, fazer e usar um modelo padrão (ver exemplo, a Figura 5).
- Verificar o fenótipo RNAi produzida por um outro fenótipo, secundário, se possível, e, repetindo a experiência. (Por exemplo, a tela exemplo mostrado utilizado alteração da GFP-tagged localização DBL-1 como um ecrã primário eo tamanho do corpo como um ecrã secundário 20.) As bactérias podem ser utilizadas a partir da mesma raia (armazenada a 4 ° C) durante até dois meses. Estrias mais velhas podem result no crescimento pobre em líquidos culturas durante a noite e deve ser restreaked primeiro.
- Sequência, pelo menos, uma extremidade da inserção de cDNA para confirmar que o inserto corresponde à etiqueta biblioteca (a biblioteca Ahringer teve uma análise realizada fiabilidade; ver http://biocompute.bmi.ac.cn/CelRNAi/ ) 21. Primers recomendado (para pastilhas a partir de qualquer biblioteca) anneal em sites da L4440 derivado vector e flanqueiam o sítio de inserção: 5'-AGCGAGTCAGTGAGCGAG-3 'e 5'-GTAAAACGACGGCCAGT-3' (iniciador M13f20).
Usando este método, uma única pessoa pode razoavelmente palco e observar de duas a quatro conjuntos de experimentos a cada semana. Por exemplo, os animais encenado na segunda-feira e terça-feira pode ser observado quinta-feira e sexta-feira, respectivamente, e podem voltar a ser encenada nesta sexta-feira e sábado para segunda-feira e observação terça-feira. Dependendo do fenótipo (s) rastreados cada dia, um conjunto pode compreender uma placa de 24 poços por micro compostoscopy ou mais, se o fenótipo é rapidamente identificados. Assim, pelo menos, 88 experiências diferentes RNAi pode ser observado em uma semana, tendo em consideração controlos positivos e negativos e taxa de rastreio. Uma tela âmbito dissecando poderia ser realizada muito mais rápido, sem a necessidade de montar os animais em lâminas para visualização. Várias pessoas também pode aumentar o rendimento de um laboratório por programações incríveis e / ou utilizando microscópios múltiplas. Um método alternativo para o crescimento de animais de uma fina película de agar e transferência de uma fatia do agar contendo animais directamente para um slide para visualização pode economizar tempo. Esta variação foi utilizado com sucesso para o rastreamento de anomalias da cauda do sexo masculino na ampliação de 400x 22.
6. Os resultados representativos
Exemplos de localização normal e alterada de GFP-tagged DBL-1 são mostrados na Figura 3. Expressão normal de GFP-tagged DBL-1 inclui ventral da medula nervosas corpos celulares e uma fileira de punctae (Figura 3A). DBL-1 is severamente atenuada quando os animais são alimentados com RNA que impede a tradução do mRNA dbl-1 (Figura 3B). dbl-1 (RNAi) também produz pequenos animais, o ecrã secundário neste exemplo (dados não mostrados). Os genes que afectam DBL-1 localização são prontamente identificados por RNAi usando uma estirpe concebido para este ecrã (Figura 3C).

Figura 1. Esquema de tela para identificar reguladores extracelulares de DBL-1 de sinalização. As bactérias a partir da biblioteca de alimentação são cultivadas durante a noite, induzida com IPTG e incubados a 37 ° C durante um período adicional de 4 horas para permitir que as bactérias para produzir ARN de cadeia dupla (dsRNA). 30 ul da cultura bacteriana é manchado induzida por poço para um 24-bem NGM placa (meio de crescimento de nemátodos) contendo 25 ug / ml de carbenicilina e 1 mM de IPTG e deixou-se secar numa câmara de fluxo estéril. Nós primeira fase larval fase de larva (L1), permitindo que o hipoclorito tratada com EMBRyos eclodir na mídia sem alimentos, o que induz uma diapausa L1 (Arrested Development). Cerca de 30 animais fase sincronizados L1 são plaqueadas em cada poço contendo bactérias a partir da biblioteca RNAi, o que permite encenado animais de retomar o crescimento e para consumir o ARNdc criado pelos 23 bactérias. Após 72 hr a 15 ° C, os vermes adultos jovens são rastreados para um fenótipo visível. Nessa idade, os defeitos de tamanho corporal são evidentes e fluorescência é brilhante de texIs100 expressão do transgene.
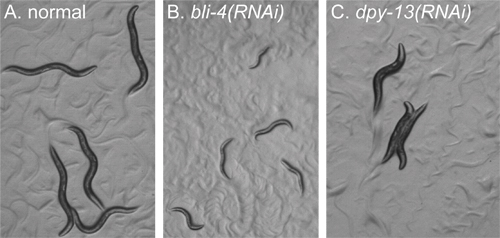
Figura 2. Exemplos de fenótipos para identificar antes do exame. Todas as imagens dos animais em uma placa de poços foram tomadas com a mesma ampliação usando um microscópio de dissecação. Os animais foram todas feitas cerca de 72 horas após o plaqueamento como larvas L1 fome. Todas as imagens foram tratados de forma idêntica. A) RNAi de um gene que confere sem defeitos brutos morfológicas. Animais aparecem tipo selvagem. B) RNAi de bli-4. Animais exibir Arrested Development, e são pequenos em comparação com os animais no painel A. C) RNAi de dpy-13. Animais estão no mesmo estágio de desenvolvimento como animais no painel A, mas apresentam uma morfologia corporal "atarracado".
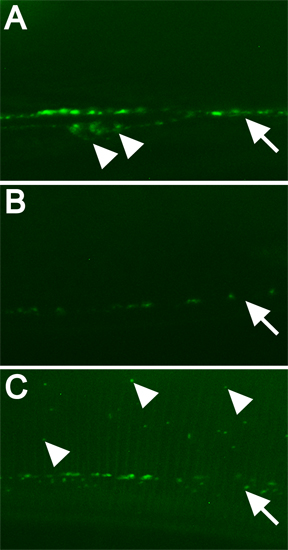
Figura 3. Exemplos de proteína fluorescente etiquetado e como RNAi de genes específicos altera padrão de localização. Todas as imagens foram tomadas com uma ampliação de 630x com microscopia confocal disco giratório, 5 seg. exposição. Barra de escala = 10 mM. Todas as imagens foram tratados de forma idêntica. Pontas de seta abertos indicam corpos celulares. Setas marcar linha de punctae. Setas cheias indicam alguns GFP-tagged aberrante localizada DBL-1. A) RNAi-fed pseudogene C06C3.5 ("tipo selvagem"). B) dbl-1 controle (RNAi). C) RNAi de um gene necessário para a localização normal de GFP-tagged DBL-1.
Figura 4. Exemplo de modelo para rastreamento experimentos de RNAi (clones de bibliotecas) em placas de 24 poços. Este modelo pode ser usado para criar um registo permanente das experiências, ao contrário directamente etiquetagem placas.
Figura 5. Exemplo de modelo para fenótipos de gravação de RNAi. Expandir conforme necessário.
Discussão
O método de triagem RNAi aqui apresentado permite uma análise rápida e sensível de produtos de genes necessários para um fenótipo (ou transgénico) normal pós-embrionário. O exemplo mostrado é um ecrã para genes envolvidos na localização subcelular de uma proteína fluorescentemente marcado. No entanto, este protocolo pode ser modificado para identificar genes que afectam outros fenótipos pós-embrionário de interesse.
Este método tira proveito de uma abordagem do gene candida...
Divulgações
Não temos nada a divulgar.
Agradecimentos
Os autores gostariam de agradecer ao Dr. Rick Padgett (Instituto Waksman, Rutgers University, NJ) para o dom do dbl-1 cDNA e Christopher Dr. Rongo (Instituto Waksman, Rutgers University, NJ) para um marcador de injeção. O laboratório do Dr. Barth Grant realizou o bombardeio arma gene para a integração baixo número de cópias do GFP dbl-1 construto. O René Garcia laboratório forneceu assistência técnica durante a criação de texIs100. O René Garcia, lints Robyn, e Hongmin Qin laboratórios prestou assessoria produtivo. Este trabalho foi financiado por fundos start-up do Departamento TAMHSC de Medicina Molecular e Celular. O escopo composto e confocal girando do disco foram adquiridos com recursos fornecidos pelo departamento e do Colégio TAMHSC do Office Medicina do Dean.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| Agar NGM | Meio de crescimento de nemátodos | IPM Scientific, Inc | Pode ser preparado seguindo NGM protocolo ágar 25 |
| Meio M9 | 22mm KH 2 PO 4, 42mm de Na 2 HPO 4, 86mm de NaCl, 1 mM de MgSO4 | 26 | |
| Agar-Agar | Merck | 1.01614.1000 | 2% em água para placas NGM. 4% em água para almofadas de lâmina de microscópio (autoclave inicialmente e de microondas para derreter a seguir). |
| Bacto Peptona | Becton Dickinson - Difco CP | 211677 | 0,25% |
| IPTG | Research Products International Corp | I56000-5.0 | 1 mM de concentração final, |
| carbenicilina | Research Products International Corp | C46000-5.0 | 50 ug / ml de diluição de trabalho |
| LB Broth Lennox | Becton Dickinson - Difco CP | 240230 | 20 g / litro |
| tetraciclina | Sigma | 268054 | 12,5 ug / ml de diluição de trabalho |
| hipoclorito de sódio | Qualquer marca | Lixívia a 5% | Use água sanitária fresco. |
| de hidróxido de sódio | Qualquer Marca | CAS 1310-73-2 | 5 estoque de N |
| Meio M9 | Wormlab Livro de Receitas | http://130.15.90.245/wormlab_recipe_book.htm # Commonlab | 26 |
| levamisol | Sigma | 31742 | 100 uM - 1 mM de diluição de trabalho |
| de azida de sódio | Fisher Scientific | S227 | 10 mM em M9 diluição de trabalho |
| 24-bem placa | Greiner Bio-One | 662160 | Distribuidor VWR |
| lâminas de microscópio | Qualquer marca | 75 x 25 x 1 mm | |
| cobertura deslizamentos de microscópio | Qualquer marca | 22 x 22 mm No.1.5 | Utilizar a espessura recomendada pelo fabricante microscópio. |
| microscópio composto | Carl Zeiss, Inc. | A1m | Use objetivos e filtros para atender às necessidades do experimento. |
| bomba de mídia | Manómetro da bomba Varistaltic | Kate modelo # 72-620-000 | Utilize tubos e configurações adequadas para a máquina |
Referências
- Giacomotto, J., Segalat, L. High-throughput screening and small animal models, where are we. Br. J. Pharmacol. 160, 204-216 (2010).
- Lamitina, T. Functional genomic approaches in C. elegans. Methods in Molecular Biology. 351, 127-138 (2006).
- Boutros, M., Ahringer, J. The art and design of genetic screens: RNA interference. Nat. Rev. Genet. 9, 554-566 (2008).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Yandell, M. D., Edgar, L. G., Wood, W. B. Trimethylpsoralen induces small deletion mutations in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 91, 1381-1385 (1994).
- Frokjaer-Jensen, C. Single-copy insertion of transgenes in Caenorhabditis elegans. Nature Genetics. 40, 1375-1383 (2008).
- Giordano-Santini, R., Dupuy, D. Selectable genetic markers for nematode transgenesis. Cell. Mol. Life. Sci. , (2011).
- Berkowitz, L. A., Knight, A. L., Caldwell, G. A., Caldwell, K. A. Generation of Stable Transgenic C. elegans Using Microinjection. J. Vis. Exp. (18), e833 (2008).
- Mello, C., Fire, A. DNA transformation. Methods in Cell Biology. 48, 451-482 (1995).
- Simmer, F. Loss of the putative RNA-directed RNA polymerase RRF-3 makes C. elegans hypersensitive to RNAi. Curr. Biol. 12, 1317-1319 (2002).
- Zhuang, J. J., Hunter, C. P. Tissue-specificity of Caenorhabditis elegans enhanced RNAi mutants. Genetics. , (2011).
- Samuelson, A. V., Klimczak, R. R., Thompson, D. B., Carr, C. E., Ruvkun, G. Identification of Caenorhabditis elegans genes regulating longevity using enhanced RNAi-sensitive strains. Cold Spring Harbor Symposia on Quantitative Biology. 72, 489-497 (2007).
- Wang, D. Somatic misexpression of germline P granules and enhanced RNA interference in retinoblastoma pathway mutants. Nature. 436, 593-597 (2005).
- Fraser, A. G. Functional genomic analysis of C. elegans chromosome I by systematic RNA interference. Nature. 408, 325-330 (2000).
- Kamath, R. S. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421, 231-237 (2003).
- Peters, K., McDowall, J., Rose, A. M. Mutations in the bli-4 (I) locus of Caenorhabditis elegans disrupt both adult cuticle and early larval development. Genetics. , 129-195 (1991).
- Thacker, C., Peters, K., Srayko, M., Rose, A. M. The bli-4 locus of Caenorhabditis elegans encodes structurally distinct kex2/subtilisin-like endoproteases essential for early development and adult morphology. Genes & Development. 9, 956-971 (1995).
- Byerly, L., Cassada, R. C., Russell, R. L. The life cycle of the nematode Caenorhabditis elegans. I. Wild-type growth and reproduction. Dev. Biol. 51, 23-33 (1976).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Savage-Dunn, C. Genetic screen for small body size mutants in C. elegans reveals many TGFbeta pathway components. Genesis. 35, 239-247 (2003).
- Qu, W. Reliability analysis of the Ahringer Caenorhabditis elegans RNAi feeding library: a guide for genome-wide screens. BMC Genomics. 12, 1471-2164 (2011).
- Nelson, M. D. A Bow-Tie Genetic Architecture for Morphogenesis Suggested by a Genome-Wide RNAi Screen in Caenorhabditis elegans. PLoS Genetics. 7, e1002010 (2011).
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854 (1998).
- Sarin, S., Prabhu, S., O'Meara, M. M., Pe'er, I., Hobert, O. Caenorhabditis elegans mutant allele identification by whole-genome sequencing. Nature Methods. 5, 865-867 (2008).
- Lewis, J. A., Fleming, J. T. Basic culture methods. Methods in Cell Biology. 48, 3-29 (1995).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77, 71-94 (1974).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados