Method Article
Fluxo de ácido nucléico bloqueado Citometria de fluorescência In situ Hibridação (LNA fluxo FISH): um método para detecção de RNA bacteriano Pequenas
Neste Artigo
Resumo
Um método high-throughput romance é descrito que permite a detecção e quantificação relativa de RNA pequeno e expressão do RNAm da única células bacterianas usando bloqueado sondas de ácidos nucléicos e citometria de fluxo por fluorescência In situ.
Resumo
Hibridização fluorescente in situ (FISH) é uma poderosa técnica que é usada para detectar e localizar seqüências específicas de ácidos nucleicos no ambiente celular. A fim de aumentar o rendimento, FISH pode ser combinado com a citometria de fluxo (fluxo FISH) para permitir a detecção de alvo sequências de ácidos nucleicos em milhares de células individuais. Como resultado, o fluxo de FISH oferece uma vantagem distinta sobre lisado / ensemble com base em métodos de detecção de ácidos nucleicos, porque cada célula é tratada como uma observação independente, permitindo assim mais forte análises estatísticas e variância. Esses atributos têm solicitado o uso de métodos FISH e fluxo de FISH em um número de diferentes aplicações ea utilidade desses métodos tem sido demonstrado com sucesso em telomere determinação identificação 1,2 comprimento, celular e expressão gênica 3,4, monitoramento de multiplicação viral em células infectadas 5, e análise da comunidade bacteriana e enumeração6.
Tradicionalmente, a especificidade dos métodos de FISH e fluxo de FISH tem sido transmitida por sondas de oligonucleotídeos de DNA. Recentemente, no entanto, a substituição de sondas de DNA oligonucleotídeo com análogos de ácidos nucléicos como sondas FISH FISH e fluxo tem aumentado a sensibilidade e especificidade de cada técnica devido às altas temperaturas de fusão (T m) destes análogos de ácidos nucléicos naturais 7,8 . Bloqueado ácidos nucleicos (LNA) sondas são um tipo de ácido nucléico analógicos que contêm nucleotídeos LNA spiked ao longo de um DNA ou RNA seqüência 9,10. Quando acoplado com fluxo de FISH, sondas LNA já foi mostrado para outperform sondas de DNA convencional 7,11 e tem sido utilizado com sucesso para detectar mRNA eucarióticas 12 e RNA viral em células de mamíferos 5.
Aqui nós expandir essa capacidade e descrever um método de fluxo de LNA FISH, que permite a detecção específica de RNA na célula bacterianas (Figura 1). Especificamente, estamos interessados na detecção de pequenos não-codificantes reguladoras RNA (sRNA), que têm atraído um interesse considerável nos últimos anos, pois foram encontrados para servir como elementos reguladores-chave em muitos processos celulares críticos 13. No entanto, existem ferramentas limitadas para estudar sRNAs e os desafios de detectar sRNA em células bacterianas é devido em parte ao tamanho relativamente pequeno (tipicamente 5-30 nucleotídeos de comprimento) e baixa abundância de moléculas sRNA, bem como a dificuldade geral em trabalhar com menor células biológicas com diferentes membranas celulares. Neste método, descrevemos fixação e permeabilzation condições que preservam a estrutura das células bacterianas e permitir a penetração de sondas LNA, bem como as etapas de amplificação de sinal que permitem a detecção específica de baixa abundância sRNA (Figura 2).
Protocolo
1. Sondas LNA & design experimental
- Sondas de design LNA que são o complemento reverso de seu sRNA transcrito / mRNA ou tê-los personalizados em www.exiqon.com . Se estiver usando a opção de design personalizado, você vai saber a seqüência, mas não o padrão spiking LNA. A adição de cada resíduo LNA em um DNA ou RNA resultados oligonucleotídeo em um aumento na T m de entre dois e 10 ° C por um duplex de hibridização LNA-RNA 14. Idealmente, as sondas LNA projetado deve ser entre 20-25 nucleotídeos de comprimento (mais sondas são mais difíceis de sintetizar) e ter um T m de entre 85-90 ° C para a RNA hibridização.
- Depois de projetar a seqüência, use BLAST http://blast.ncbi.nlm.nih.gov/Blast.cgi ou comparar a seqüência de sonda para o seu banco de dados local para garantir a sonda é específico apenas para o seu sRNA / mRNA do interesse.
- Ao encomendar a sonda LNA, adicione uma modificação biotina-TEG para terminar a 5 'do oligonucleotídeo LNA. O Biotinilação é necessário para o pós-coloração hibridização com um conjugado de estreptavidina-dye. É possível adicionar esta modificação para a extremidade 3 ', se necessário, mas a adição à extremidade 5' é menos caro e deve resultar em rendimentos mais elevados.
- Três controles negativos devem ser incluídos no método: (i) um controle "não LNA", no qual uma sonda LNA não é adicionado durante a etapa de hibridização, (ii) um controle "não dye", na qual a sonda LNA é hibridizada com o alvo sRNA mas o evento de hibridização não é detectado, devido à ausência da mancha fluorescente, e controle (iii) 'mRNA sRNA não-expresso /' a que utiliza uma sonda LNA que tem como alvo um sRNA inexistente ou não-expresso, a fim para monitorar a hibridação não específica.
- A sonda LNA específico aqui é complementar à CsrB sRNA e tem a seqüência 5'-biotina-TEG-gTccAttTccCgtCctTagCagC-3 '(monômeros LNA emletras maiúsculas).
- Após a recepção do LNA liofilizado, ressuspender com nuclease-free de água a 50 mcg / mL e armazenar em 10-20 mL alíquotas a -20 ° C.
Soluções
- 8% paraformaldehye (PFA), 10% de ácido acético em PBS: Misture 1 mL pfa 32%, 400 mL de ácido acético, 400 mL PBS 10X, pH = 7,4 e 2,2 mL de água nuclease-free. Para alíquotas congeladas, congelar em alíquotas de uso único a -20 ° C sem ácido acético.
- 60% solução de sulfato de dextran: adicionar 6,0 g de sulfato de dextran a um tubo cônico de 50 mL e adicionar água a um volume final de 10 mL. Aqueça esta mistura a 65 ° C em banho-maria até que o sulfato de dextran é dissolvido (pode demorar 1-2 horas). Água adicional pode precisam ser adicionados durante o processo de solubilização para manter um volume de 10 mL. Alíquota da solução de sulfato de dextran 60% e armazenar a -20 ° C até o uso.
2. Fixação
- Colheita 1x10 8 células de bioluminescent Vibrio campbellii ou suas bactérias de interesse e transferi-los para um tubo de microcentrífuga de 1,5 mL. Esta quantidade de células fornece bastante material de partida para quatro fluxo-FISH amostras (uma amostra de sonda específica LNA e três controles negativos). Adicional de 1,5 mL alíquotas tubo de microcentrífuga devem ser colhidas se outras sRNA / mRNA espécies precisam ser testados.
- Agregar as células bacterianas por centrifugação a 2300 xg por cinco minutos. Descartar o sobrenadante por pipetagem e ressuspender as células em 400 mL de 1X PBS.
- A esta mistura, adicione 400 mL de uma paraformaldehye 8% (PFA) e 10% de ácido acético em solução salina tamponada com fosfato de 1X (1X PBS) solução, misture bem e incubar por 10 minutos a 25 ° C a mistura uma vez depois de cinco minutos. Todas as incubações, salvo indicação em contrário, são executadas em um suporte para tubo aquecido. A solução pfa devem estar preparados frescos ou utilizados a partir de alíquotas congeladas após a adição de ácido acético. Paraformaldeído é um fixador de reticulação que hELPS para preservar a morfologia das células e reter RNA intracelular.
- Agregar as células a partir desta mistura por centrifugação a 4500 xg por dois minutos e remover o sobrenadante por pipetagem. Todos os passos futuros que requerem centrifugação deve usar as mesmas configurações (7K, 2 min). É importante notar que após a centrifugação sobrenadantes deve ser removido por pipetagem e não de decantação.
- Lave as células duas vezes com 400 mL PBS 1X e ressuspender o pellet celular final, em 400 mL PBS 1X. As células são fixos e podem ser armazenados a 4 ° C em 400 mL 1X PBS por até sete dias.
3. Dietil tratamento pyrocarbonate
- Prepare 400 mL de uma solução 0,1% de dietil pyrocarbonate (DEPC) em 1X PBS (400 mL desta solução é necessário para cada amostra a ser testada assim que a escala em conformidade). A solução DEPC deve ser preparado a partir de um estoque DEPC. Tratamento DEPC inativa RNases por carbethoxylation e diminui asinal de fundo.
- Adicionar 400 mL da solução 0,1% DEPC para cada amostra de células bacterianas fixa e misture bem por pipetagem. Incubar esta mistura a 25 ° C por 12 minutos. Lave as células duas vezes com 400 mL de 1X PBS, agregar as células (7K, 2 min) e remover o sobrenadante.
4. Permeabilização
- Adicionar 1,0 mg de lisozima a 1 mL tampão Tris-EDTA (10 mM Tris, 1 mM EDTA, pH 8,0) para uma solução de 1,0 mg / mL. Esta solução deve ser preparada. Adicionar 800 mL da solução de lisozima 1 mg / mL para as células, misture bem por pipetagem e incubar a 25 ° C por 30 minutos com a mistura ocasional. Agregar as células (7K, 2 min) e remover o sobrenadante. Lisozima atua como um reagente permeabilização hidrolisando o presente peptidoglicano na parede celular bacteriana.
- Prepare uma solução de 3,0 mg / mL de proteinase K em tampão TE. Esta solução deve ser preparada a partir de um mg / mL proteinase K 20 ações que está armazenada a -20 ° C.Proteinase K é uma serina protease que digere uma variedade de proteínas e ajuda a acessibilidade das sondas e reagentes para o RNA.
- Adicionar 800 mL de 3,0 mcg / mL proteinase K solução para o pellet celular e misture bem por pipetagem. Incubar a 25 ° C por 15 minutos com vortex no meio da incubação.
- Agregar as células (7K, 2 min), remover o sobrenadante e lavar as células uma vez com 400 mL PBS 1X. Depois de remover o sobrenadante lavagem, ressuspender as células em 400 mL PBS 1X e adicionar 100 mL da ressuspensão em quatro novos tubos de 1,5 mL de microcentrífuga. Agregar as células (7K, 2 min) e remover o sobrenadante.
5. Hibridização
- Preparar 100 mL de tampão de hibridação (HB) para cada amostra. O HB é composta de formamida 50% em volume, 10% sulfato de dextran em massa (acrescente 16,7 mL da solução 60% de sulfato de dextran para cada mL 100), a solução de 1X Denhardt, 50 mM fosfato de sódio pH 7,0,Citrato de sódio 2X (SSC), 20 mg de DNA cortado salmão esperma (SADE) e 20 g de fermento tRNA.
- Cada um dos componentes do HB tem uma finalidade diferente. Formamida reduz a temperatura de fusão do ácido nucléico duplexes contribuindo com a desnaturação do RNA e minimizando os danos celulares. Sulfato de dextrano é usado como um volume de exclusão de polímero de concentrar a sonda e aumentar a taxa de hibridação. Solução Denhardts, levedura e tRNA SSSD servir de agentes bloqueadores para reduzir a ligação não específica.
- Em cada uma das células-ressuspensão contendo 1,5 mL tubos de microcentrífuga, adicionar 95 mL HB e 5,0 mL da sRNA / mRNA específicos LNA [20 pmol de concentração LNA específicos final] ou água (para o controle LNA não negativo). Por exemplo:
- Tubo 1-95 HB mL + 5,0 mL sRNA / sonda mRNA específicos
- Tubo de 2-95 mL HB + 5,0 mL sRNA / sonda mRNA específicas (controle negativo "não dye ')
- Tubo de 3-95 mL HB + 5,0 mL sRNA/ MRNA sonda ("não-expresso sRNA / mRNA 'controle negativo)
- Tubo de 4-95 mL HB + 5,0 mL de água (controle "não LNA" negativo)
- Incubar todas as quatro amostras a 60 ° C por 60 minutos. A temperatura de hibridização depende da sonda LNA (s) T m e deve ser fixado em aproximadamente 30 ° C abaixo do previsto T m de RNA de recozimento. Tanto a temperatura de hibridização elevado e formamida na HB garante a desnaturação da estrutura secundária do mRNA sRNA ou de interesse.
6. Pós-hibridação lava
- Após a hibridização, adicionar 1,0 mL 0,1 x SSC com 0,1% tween-20 (SSCT) para a mistura de hibridização. Isto é necessário para permitir que as células a serem removidas da solução de hibridização viscoso. Agregar as células (7K, 2 min) e remover o sobrenadante.
- Adicionar 200 mL de formamida 50%, 2X SSC, 0,1% tween-20 para as pelotas de células, misturar bem e incubcomeu durante 30 minutos a 65 ° C em um bloco de aquecimento.
- Adicione 1 mL 0,1 x SSCT à mistura, agregar as células (7K, 2 min) e remover o sobrenadante.
- Adicionar 500 mL SSCT 0,1 x para ressuspender as células e incubar por 40 minutos a 65 ° C em um bloco de calor. Agregar as células (7K, 2 min) e remover o sobrenadante.
7. Bloqueio e coloração
- Preparar o tampão de bloqueio (BB) e da solução de coloração estreptavidina-dye. Prepare uma solução de 1X BB a partir de um estoque de 5X (comercialmente disponíveis, consulte Reagentes). Para cada conjunto de quatro tubos, prepare 1,2 mL 1X BB.
- Adicionar 200 mL BB 1X para as pelotas de células, ressuspender e incubar por 30 minutos a 25 ° C.
- Um corante estreptavidina conjugada é usado para detectar as sondas biotiniladas LNA. Enquanto bloqueia, prepare uma solução de 2 mg / mL DyLight 488 estreptavidina ou o seu desejado conjugado estreptavidina-dye em 1X BB por 30 min (para três amostras, fazer 300 mL).
- Depois incubation com 1X BB, agregar as células (7K, 2 min) e remover o sobrenadante. Adicionar 50 mL da solução de estreptavidina-dye para o pellets de células, misturar bem e incubar por 12 minutos a 25 ° C, com mistura constante em um vórtice ou em um Thermomixer. Para o controle "não dye ', adicionar 50 mL tampão 1X bloqueio apenas. Para o resto do protocolo, proteger os tubos de luz usando uma folha de alumínio.
- Lave as células uma vez com 500 mL de buffer SSCT 0,1 x e uma vez com 400 mL 1X PBS com 0,1% tween-20 (PBST). Agregar as células (7K, 2 min) e remover o sobrenadante.
- Após a coloração estreptavidina-conjugado inicial, o sinal é amplificado através da introdução de um anticorpo anti-estreptavidina biotina para o conjugado estreptavidina-dye e coloração pela segunda vez com a estreptavidina-dye, aumentando assim a saída do sinal por hybridized LNA sonda (Figura 2) .
- Adicionar 100 mL de 1 mg / mL biotinilado anti-estreptavidina anticorpos em 1X PBS, resuspend as células, e incubar a 25 ° C por 30 minutos com a mistura ocasional. Agregar as células (7K, 2 min), remover o sobrenadante e lavar duas vezes com 400 mL PBST 1X.
- Adicionar 50 mL da 2 mg / ml de solução de estreptavidina-dye para as células, misturar bem e incubar por 12 minutos a 25 ° C, com mistura constante em um vórtice ou em um Thermomixer. Para o controle "não dye ', adicionar 50 mL tampão 1X bloqueio apenas.
- Lave as células uma vez com 500 mL de buffer SSCT 0,1 x e uma vez com 400 mL PBST 1X.
- Ressuspender as células em 200 mL 1X PBST e armazenar a 4 ° C até que esteja pronto para análise de citometria de fluxo. Para menores células bacterianas definir a taxa de fluxo para retardar para diminuir o tamanho do núcleo. Além disso, para citómetros de fluxo com cortes limite para a frente de dispersão, o corte deve ser definido o mais baixo possível para garantir a pequenas células bacterianas são detectados.
- Para detecção DyLight 488, o citômetro de fluxo deve ser equipado com um laser de 488 nm e emissão padrãofiltros para FITC. Pelo menos 2x10 4 eventos deverão ser recolhidos. Para compatibilidade com citómetros de fluxo equipado com lasers diferentes, outras estreptavidina conjugada fluoróforos pode ser usado para este protocolo.
8. Resultados representativos
Um exemplo de células que são fixadas e permeabilizadas efetivamente é mostrada no gráfico de pontos de citometria de fluxo na Figura 3A. Ao usar as condições descritas acima para permeabilização, a população de células é pequena e homogênea, indicando alguns agregados celulares. Quando maior (5 mg / mL) as concentrações de lisozima são usados, as células são mais propensos a agregar e mostrar os valores de dispersão aumentou para a frente, indicativos de partículas maiores (Figura 3B). Um exemplo de dados de citometria de fluxo a partir de uma bem sucedida experiência de fluxo LNA-FISH é mostrado na Figura 4. O histograma demonstra a detecção específica de um sRNA alvo, juntamente com três controles negativos.O controle "não dye" negativo produz o mínimo de fluorescência, seguido pelo "não LNA 'e' não-expresso sRNA" controles negativos. Finalmente, a amostra com o sRNA específicos LNA sonda produz a maior fluorescência. Depois que o método e sondas LNA são validados desta forma, experimentos adicionais podem ser projetados para monitorar as alterações no sinal sRNA ao longo do tempo, em resposta às mutações ou condições de cultura variada, etc
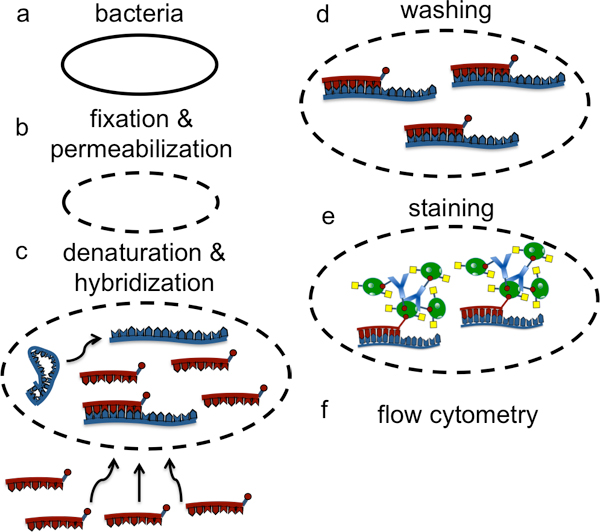
Figura 1. Representação do esquema geral de uma experiência de fluxo LNA-FISH para a detecção de sRNA bacteriana.

Figura 2. Coloração e amplificação do sinal de fluxo LNA-FISH. a) Hibridação da sonda LNA biotinilado. b) A coloração fluorescente com o DyLight conjugado de estreptavidina 488. c) Vinculação da biotinilado anti-streptavidin de anticorpos para a estreptavidina 488 DyLight. O anticorpo pode se ligar estreptavidina através de seu site de ligação de antígenos ou pode ser ligado por estreptavidina através de seus resíduos biotina. d) A amplificação do sinal através da coloração ainda mais com as fluorescentes DyLight conjugado de estreptavidina 488.
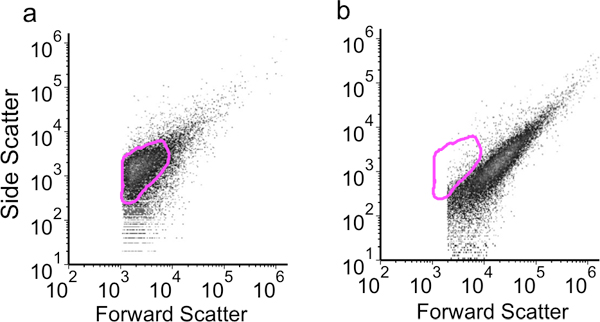
Figura 3. Parcelas Citometria de fluxo dot mostrando um lado versus resultados de dispersão para a) 1 mg / mL de lisozima, 3 mg / mL proteinase K permeabilização e b) 5 mg / mL de lisozima, 3 mg / ml proteinase K permeabilização.
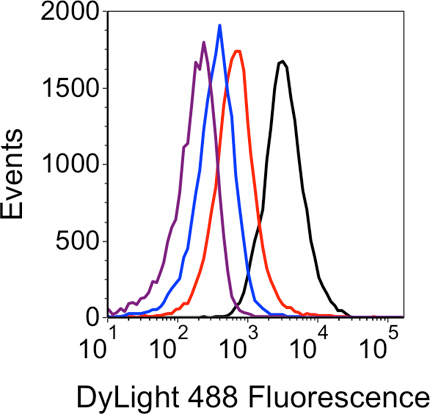
Figura 4. Histograma análise do fluxo de FISH-LNA resultados. O sinal fluorescente gerada a partir de cada amostra é mostrado pelos quatro traços: preto - sRNA específicos LNA sonda; vermelho - controle negativo "não expressa sRNA '; azul - controle negativo' no LNA; roxo -Controle "não dye 'negativo.
Discussão
O método do fluxo de LNA-FISH apresentada aqui foi usado para detectar a expressão de um sRNA do Gram-negativos marinhos bactéria Vibrio campbellii cuja expressão já havia sido confirmado através de microarray baseado em perfis de expressão e transcrição reversa reação em cadeia da polimerase 16. Até o momento, têm utilizado este método para monitorar a expressão de uma variedade de RNAs (por exemplo, trans-codificado sRNA, sRNA que modulam a atividade de proteína, riboswitches e mRNA). Como tal, estamos confiantes de que o método é adaptável e pode ser usado para detectar qualquer alvo sRNA ou mRNA e podem ser modificados para utilização em qualquer espécie bacteriana ou tipo de célula. Se a modificação deste protocolo é necessário para outros tipos de células, a variável mais importante a considerar é o passo manipular permeabilização. Por exemplo, ao modificar as condições de permeabilização aqui descritos, as mudanças nas concentrações de lisozima e proteinase K deve ser testado. Descobrimos que duplicar outriplicando a quantidade de lisozima e proteinase K resultou em mudanças importantes no fluxo de FISH-LNA resultados. Além disso, quando testar as condições de permeabilização adicionais, as células devem ser analisados para a aglomeração, a perda de células, e obter o melhor sinal à relação de fundo de fluorescência. A extensão da aglomeração de células podem ser testadas por microscopia e / ou citometria de fluxo. Por citometria de fluxo, aglomerados de células estão presentes como caudas em uma frente contra trama lado dot dispersão eo método de permeabilização correta minimizar esse efeito de rejeitos. Este método também é passível de detecção multiplex sRNA sem grandes modificações com o protocolo descrito como sondas adicionais LNA rotulados com haptenos, tais como digoxigenina poderia ser adicionado durante a etapa de hibridização e, então, detectado com um anticorpo anti-digoxigenina conjugado anticorpo e fluoróforo-secundário. Atualmente, a única limitação que encontramos com este método é a sua incapacidade para gerar sinal suficiente fracamente exespécies sRNA pressionado e esforços estão sendo feitos para determinar o limiar de detecção (em número absoluto de cópia por célula).
No geral, o método do fluxo de FISH LNA oferece a oportunidade de medir sRNA bacteriana ou expressão de mRNA no único nível de célula de uma forma de alto rendimento para fornecer insights sobre a presença e abundância relativa de uma espécie sRNA eo grau em que sua expressão varia em uma população.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi financiado pelo Office of Naval Research via EUA Naval Research Laboratory fundos básicos.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| Fosfato 10X solução salina tamponada (PBS), pH 7,4 | Applied Biosystems / Ambion | AM9625 | |
| Nuclease água livre de | Applied Biosystems / Ambion | AM9932 | (Não tratada DEPC) |
| Paraformaldeído 32% | Microscopia Eletrônica de Ciências | RT 15714 | Etanol livre |
| Ácido acético | Acros Organics | 327290010 | |
| Dietil pyrocarbonate (DEPC) | Sigma Aldrich | D5758 | Mantenha desidratado |
| Lisozima | Sigma Aldrich | L2879 | |
| Proteinase K (20 mg / mL) | Applied Biosystems / Ambion | AM2546 | |
| Formamida | Applied Biosystems / Ambion | AM9342 | Alíquota, deionizada e freeze |
| Sulfato de dextrano MW> 500000 | Sigma Aldrich | D8906 | Solução alíquota de 60% e congelar |
| Solução Denhardt nos concentrar 50X | Sigma Aldrich | D2532 | Alíquotas e congelar |
| Tampão de citrato de sódio 20X | Applied Biosystems / Ambion | AM9770 | |
| DNA de esperma de salmão (cortado) 10 mg / mL | Applied Biosystems / Ambion | AM9680 | Alíquotas e congelar |
| TRNA de levedura (10 mg / mL) | Life Technologies / Invitrogen | 15401-011 | Alíquotas e congelar |
| Tween-20 | Sigma Aldrich | P9416 | |
| 5X em solução de hibridização in situ bloqueio | Vector Laboratories | MB-1220 | |
| DyLight 488 estreptavidina | Vector Laboratories | SA-5488-1 | |
| Biotinilado anti-estreptavidina | Vector Laboratories | BA-0500 | Alíquotas e congelar |
Referências
- Rufer, N., Dragowska, W., Thornbury, G., Roosnek, E., Lansdorp, P. M. Telomere length dynamics in human lymphocyte subpopulations measured by flow cytometry. Nat. Biotechnol. 16, 743-747 (1998).
- Lansdorp, P. M. Heterogeneity in telomere length of human chromosomes. Hum. Mol. Genet. 5, 685-691 (1996).
- Pernthaler, A., Amann, R. Simultaneous Fluorescence In Situ Hybridization of mRNA and rRNA in Environmental Bacteria. Appl. Environ. Microbiol. 70, 5426-5433 (2004).
- Jen, C. J. Flow-FISH analysis and isolation of clostridial strains in an anaerobic semi-solid bio-hydrogen producing system by hydrogenase gene target. Appl. Microbiol. Biotechnol. 74, 1126-1134 (2007).
- Robertson, K. L., Verhoeven, A. B., Thach, D., Chang, E. Monitoring Viral RNA in Infected Cells with LNA Flow-FISH. RNA. 16, 1679-1685 (2010).
- Friedrich, U., Lenke, J. Improved Enumeration of Lactic Acid Bacteria in Mesophilic Dairy Starter Cultures by Using Multiplex Quantitative Real-Time PCR and Flow Cytometry-Fluorescence In Situ Hybridization. Appl. Environ. Microbiol. 72, 4163-4171 (2006).
- Thomsen, R., Nielsen, P. S., Jensen, T. H. Dramatically improved RNA in situ hybridization signals using LNA-modified probes. RNA. 11, 1745-1748 (2005).
- Silahtaroglu, A. N., Tommerup, N., Vissing, H. FISHing with locked nucleic acids (LNA): evaulation of different LNA/DNA mixmers. Mol. Cell. Probes. 17, 165-169 (2003).
- Obika, S. Synthesis of 2'-O, 4'-C-methyleneuridine and -cytidine. Novel bicyclic nucleosides having a fixed C3'-endo sugar puckering. Tetrahedron. Lett. 38, 8735-8738 (1997).
- Koshkin, A. A. LNA (locked nucleic acids): synthesis of the adenine, cytosine, guanine, 5-methylcytosine, thymine, and uracil bicyclonucleoside monomers oligomerisation, and unprecedented nucleic acid recognition. Tetrahedron. 54, 3607-3630 (1998).
- Kubota, K., Ohashi, A., Imachi, H., Harada, H. Improved in situ hybridization efficiency with locked-nucleic-acid-incorporated DNA probes. Appl. Environ. Microbiol. 72, 5311-5317 (2006).
- Robertson, K. L., Thach, D. C. LNA flow-FISH: A flow cytometry-fluorescence in situ hybridization method to detect messenger RNA using locked nucleic acid probes. Anal. Biochem. 390, 109-114 (2009).
- Waters, L., Storz, G. Regulatory RNAs in bacteria. Cell. 136, 615-628 (2009).
- Petersen, M., Wengel, J. LNA: a versatile tool for therapeutics and genomics. Trends. Biotechnol. 21, 74-81 (2003).
- Corput, M. P. C. v. d., Grosveld, F. G. Fluorescence in Situ Hybridization Analysis of Transcript Dynamics in Cells. Methods. 25, 111-118 (2001).
- Silveira, A. C. G. Identification of non-coding RNAs in environmental vibrios. Microbiology. 156, 2452-2458 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados