Method Article
Um fluxo de trabalho Análise Quantitativa de Fitness
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Análise Quantitativa de Fitness (QFA) é uma série complementar de métodos experimentais e computacionais para estimar as aptidões cultura microbiana. QFA estima o efeito de mutações genéticas, drogas ou outros tratamentos aplicados sobre o crescimento micróbio. Experimentos de escala a partir da análise focalizada de uma única cultura a milhares de culturas paralelas podem ser projetados.
Resumo
Análise Quantitativa de Fitness (QFA) é um fluxo de trabalho experimental e computacional para comparar as aptidões de culturas microbianas cultivadas em 1,2,3,4 paralelo. QFA pode ser aplicado a observações orientadas de culturas individuais, mas é mais útil para genoma interacção genética ou telas de drogas investigar até milhares de culturas independentes. O método central experimental é a inoculação de independentes, diluir líquidos culturas microbianas em placas de agar sólidos que são incubadas e regularmente fotografadas. Fotografias de cada ponto do tempo são analisados, produzindo estimativas quantitativas de células de densidade, que são utilizados para construir curvas de crescimento, permitindo que as medidas quantitativas aptidão para ser derivado. Aptidões de cultura podem ser comparados para quantificar e classificar forças de interacção genética ou sensibilidades de drogas. O efeito sobre a aptidão da cultura de quaisquer tratamentos adicionados em agar de substrato (por exemplo, moléculas pequenas, antibióticos ou nutrientes) ou aplicadas a placas de externoly (por exemplo, irradiação UV, temperatura) pode ser quantificado por QFA.
O fluxo de trabalho QFA produz taxa de crescimento estima análoga aos obtidos por medição espectrofotométrica da paralelas culturas líquidas em leitores de placas de 96 poços ou de 200 poços. Importante, QFA tem rendimento significativamente superior em comparação com esses métodos. Culturas QFA crescer sobre uma superfície de agar sólido e são, portanto, bem arejadas durante o crescimento sem a necessidade de agitação ou agitação.
Rendimento QFA não é tão alto quanto o de alguns matriz genético sintético (SGA) métodos de triagem 5,6. No entanto, uma vez que as culturas QFA são fortemente diluído antes de ser inoculadas em ágar, QFA pode capturar as curvas de crescimento mais completos, incluindo exponencial e fases de saturação 3. Por exemplo, as observações da curva de crescimento permitir tempos de duplicação de cultura a ser estimado directamente com alta precisão, como discutido anteriormente 1.
Aqui apresentamos umaespecífica QFA protocolo aplicado a milhares de S. cerevisiae culturas que são automaticamente tratados por robôs durante a incubação a inoculação, e de imagem. Qualquer um destes passos automatizados pode ser substituído por um procedimento, o equivalente manual, com uma redução na taxa de transferência associada, e também apresenta um protocolo manual inferior de transferência. As mesmas ferramentas de software QFA pode ser aplicado a imagens capturadas em qualquer fluxo de trabalho.
Temos uma vasta experiência QFA aplicando às culturas da levedura de brotamento S. cerevisiae, mas esperamos que QFA provará igualmente útil para examinar as culturas da levedura S. pombe e as culturas bacterianas.
Protocolo
Manual do QFA Protocol (para as estirpes de S. cerevisiae)
1. Levedura Cepas de cultura
- Até 96 estirpes de levedura independentes são cultivadas em 200 uL de meios líquidos ricos em um prato de cultura de 96 poços. As estirpes são cultivadas até à saturação, em uma temperatura controlada incubadora.
- Um pino 96, 1/8 "diâmetro manual do réplica Plater (SIGMA R-2508) é esterilizada por imersão do primeiro Plater réplica em etanol a 100%, à chama e, em seguida, deixando-se arrefecer.
- 200 uL de água estéril é vestida de uma placa de cultura de 96 poços. As culturas são agitadas saturados (Eppendorf MixMate, 30 seg, 750 rpm) e diluiu-se por imersão a ferramenta pino esterilizada para as estirpes saturados de três vezes, em seguida, na placa que contém a água uma vez.
- A ferramenta de pino é re-esterilizados como por 1,2.
- A ferramenta de pino esterilizado é usado para detectar a cultura diluída em placas de agar vendidos por imersão na placa contendo a cultura diluída três vezes asn transferir a ferramenta de pino para uma placa de agar rectangular sólida.
- As placas de agar rectangulares sólidos são incubadas a uma temperatura apropriada, antes de ser trabalhada manualmente utilizando um S e P Robotics spImager. Métodos de captura de imagem alternativos pode também ser usado como uma FujiFilm LAS4000, ou uma mais barata câmera digital SLR de cuidados forem tomados para garantir uma iluminação uniforme e consistente posicionamento da placa em relação à câmara de imagens.
- O método é adaptável a um número menor de culturas paralelas, tais como 48, que podem ser montados em placas de petri redondos se o cuidado de assegurar o ângulo relativo mesmo para a câmara de imagem durante cada fotografia.
Totalmente Automatizado QFA Protocol (para as estirpes de S. cerevisiae)
2. Levedura Cepas de cultura
- Comece com uma matriz retangular de leveduras independentes que crescem em agar sólido. Neste exemplo, as estirpes de partida são o resultado do cruzamento de uma temperaturamutação consulta sensível com a coleção de exclusão levedura (YDC) pela matriz genética sintética (SGA). Placas de finais PIG estão em formato 1536. Esta é a 16 linhas por 24 colunas de colônias e culturas que representam quatro repetições de 384 cepas de leveduras independentes. Veja a figura 1 para layouts de placas.
- 384 estirpes fora de 1536 são transferidos roboticamente usando um S e P Robotics Inc robô BM3-SC com um pino 96, 1-milímetros pintool diâmetro do pino estéril em quatro placas de cultura de 96 poços contendo 200 uL de meios selectivos (Figura 1). Pintool limpeza e esterilidade é conseguido através da sequencialmente lavar duas vezes em água estéril (uma vez com uma escova rotativa para remover detritos), etanol a 70% (com sonicação) e, finalmente, etanol a 100%.
- As culturas são crescidas até à saturação (durante três dias a 20 ° C neste exemplo, ver Figura 1).
3. Spotting culturas de leveduras
- Usando um Beckman Biomek FX, as culturas saturadas umare ressuspensas em vórtice em um Teleshake Variomag (sentido anti-horário em 1000rpm por 20 segundos) e dilui-se cerca 1:70 fixando em 200 ul de água estéril utilizando uma agulha estéril 96 pin pin ferramenta robótica (V & P Científico magnética montagem pintool, 2 diâmetro do pino mm). Limpeza pino da ferramenta e esterilidade é conseguida por lavagem em água estéril, lavagem em etanol a 70% (com um pincel para remover detritos) e, finalmente, imersão em etanol a 70% seguido de secagem.
- Culturas diluídas são manchados em placas de agar sólidos em 384 formato usando a ferramenta mesmo pino robótico (ver Figura 1) após a limpeza e esterilização.
- Após fixação, as placas são transferidos para uma temperatura Cytomat controlada, incubadora humidificada com automático carrossel e portinhola de acesso.
4. Captura de Imagem
- Um S & P Robotics automatizado imager repetidamente remove as placas da incubadora Cytomat, remove a tampa do prato, coloca-los com precisão em uma anexard, o espaço uniformemente iluminado por baixo de uma Canon EOS Rebel Ti 35 milímetros DSLR, captura uma imagem de 5184 x 3456 px resolução, substitui a tampa da placa, e retorna para o carrossel incubadora. Uma imagem é captada imediatamente após transferência inicial para a incubadora para permitir uma medição de zero crescimento tempo ponto (de fundo). Manipulação do robô e fotografia leva 2 minutos por placa.
- Com um carrossel completamente carregado, o máximo alcançável placa de frequência de captura de imagem é uma fotografia de seis em seis horas. As placas são incubadas durante até seis dias (até à saturação de crescimento de colónias. A Figura 1 mostra três imagens características a partir de um tempo-curso a 20 ° C.
- Para fins de controle, as placas são denominados de acordo com o nome incubadora, a temperatura número de lote, única e sequencial e posição na incubadora. Nomes das imagens são uma concatenação do nome de prato e timestamp e são automaticamente construído sobre a captura de imagem (por exemplo K000011_030_010_2011-09-22_09-47-54.jpg, FT Tipo de Arquivo1, Tabela 1).
5. Captura de Metadados Experimental
Os arquivos de metadados descritos nesta seção são arquivos de texto delimitados, que são gerados manualmente (por exemplo, usando o software de planilha eletrônica). O ficheiro de descrição experimental (Tabela, FT2 1) é única para cada experiência, mas a descrição da biblioteca (FT3, Tabela 1) pode ser re-utilizado extensivamente uma vez construído.
- O experimento está descrito no arquivo Descrição Experimental (FT2, Tabela 1) contendo colunas para código de barras (ou o nome do prato gerada automaticamente), timestamp experimento inicial, tratamento com placa, o conteúdo do meio de agar sólido, nome de tela, biblioteca, número da placa (para bibliotecas com múltiplas placas) e um número de repetição quadrantes (ver Figura 1) para o dimensionamento para baixo a partir de 1536 para 384 formato formato.
- A biblioteca cepa de levedura é descrito com um arquivo Descrição Library (FT3, Tabela1), indicando o genótipo cultivadas em cada local de cultura em cada prato de uma biblioteca. Ele contém colunas para: nome da biblioteca, ORF, número da placa, placa de linha, coluna placa e uma coluna de notas opcional.
- Um arquivo de nome opcional padrão Gene (FT4, Tabela 1) descrevendo o nome gene padrão (por exemplo RAD9) associado a cada ORF sistemática y-número (por exemplo YDR217C) identificação de cepas sendo rastreadas podem ser fornecidas. Este arquivo contém duas colunas: nome ORF & Gene.
6. Análise de Dados
O fluxo de trabalho QFA computacional requer acesso a uma estação de trabalho do computador razoavelmente poderoso multicore (por exemplo, Precision T3500 com um Dell Xeon quad-core 2,67 GHz de processador e RAM 12Gb) no qual está instalado o Colonyzer imagem ferramenta de software de análise de 3,4 eo QFA pacote R 7 , sendo que ambos estão documentadas on-line, estão disponíveis gratuitamente e rodar em uma variedade de sistemas operacionais.
- Executar Colonyzer com cada uma das imagens captadas placa como entrada, gerando um ficheiro de saída Colonyzer (Tabela FT5 1) para cada imagem capturada. Arquivos de saída Colonyzer especificar estimativas de cultura de densidade, a área de cultura, forma e cor para cada uma das localizações 384 na placa de imagens. Arquivo de saída é automaticamente copiado o nome do arquivo imagem de origem (por exemplo, a fotografia da placa K000011_030_010_2011-09-22_09-47-54.jpg (FT1, Tabela 1) corresponde ao arquivo de saída Colonyzer K000011_030_010_2011-09-22_09-47-54.dat ( FT5, Tabela 1)).
- Usando o pacote de R QFA, coloque metadados experimental (FT2, Tabela 1) e arquivos de saída Colonyzer (Tabela FT5 1). Estes dados são mesclados em quadros I de dados (que pode ser exportado como QFA-primas arquivos dados de texto (FT6, Tabela 1)), para posterior análise.
- O QFA pacote de R contém funções para coletar timecourses densidade celular para cada cultura, para caber lo generalizadamodelos populacionais gistic para observações e para tanto enredo. (ver Figuras 2 e 3 para exemplos) Valores de parâmetros embutidos são gravadas em arquivos de parâmetros QFA logística (FT7 Tabela 1). Veja QFA documentação do pacote R para obter mais detalhes.
- O QFA pacote de R também contém funções para controle de qualidade: colônias de ponta são descartados devido à maior disponibilidade de nutrientes para as bordas da placa e dificuldade de análise de imagem perto de paredes de chapa, culturas que falharam na SGA e genótipos exibindo ligação com genes específicos tela de marcadores são removidos a partir de análise.
- Várias medidas aptidão quantitativos são derivados a partir dos parâmetros da população modelo logístico para cada cultura. Estes incluem as taxas máximas de população de duplicação (MDR), o número de divisões de inoculação à saturação (MDP). Para cada conjunto de culturas em duplicado (do genótipo original por exemplo) e para cada definição de fitness, diversas estatísticas de resumo das estimativas de fitness são compbuído: média e mediana de fitness desvio de fitness, padrão e número de repetições, observou. Estatísticas sumárias são a saída para arquivos QFA Resumo ginástica (FT8, Tabela 1) para posterior análise, por exemplo, o cálculo dos escores de interação genéticos (FT9 Tabela 1).
7. Os resultados representativos
A Figura 2 mostra uma curva de crescimento típica para fora de 384 culturas em crescimento QFA a 20 ° C em agar sólido como quantificado por Colonyzer, com o aumento da densidade celular primeiro tornar-se detectável após cerca de 2 dias. Este atraso é provável um efeito combinado de uma fase de latência cultura após a inoculação em meio de cultura sólido e um limite inferior de detecção para a densidade celular. A Figura 3 mostra as curvas de crescimento semelhantes 308 capturados a partir de uma única placa. A Figura 4 demonstra a comparação de dois de todo o genoma telas QFA para inferir genéticaforça de interação (adaptado a partir de 1). O QFA pacote R 7 também inclui funções para produzir a Figura 3 e para a saída de listas ordenadas dos genótipos analisados e as estimativas de força de interação genética (GIS), juntamente com um valor q (taxa de detecção False (FDR) corrigido p-valor) para o importância do GIS observado.

Figura 1. Esquema para inoculação robótico de estirpes de levedura em 384 spot-formato. Este procedimento começa com 1536 culturas independentes por placa (à esquerda). Neste exemplo típico, as colónias nas posições 1,1; 1,2; 2,1 e 2,2 (cor vermelha) são quatro repetições de o mesmo genótipo his3 :: culturas KANMX em amarelo, crescendo sobre a borda da placa. , tem uma vantagem de crescimento devido à falta de concorrência e portanto, não são examinados pelo QFA. Um destes repetições (por exemplo 1,1) é inoculada em meio de crescimento líquidos em placa de 96 poços s usando uma ferramenta de 96 pinos que inocula 96 de 1536 colônias de cada vez. A fim de inocular um idêntico para cada um de 384 deleções de genes, quatro diferentes quadrantes "" (indicado como vermelho, azul, verde e púrpura) são inoculados em quatro diferentes placas de 96 poços contendo meio de crescimento. Após crescimento até à saturação (por exemplo 3 dias a 20 ° C), as culturas são diluídas em água, em seguida, os quatro quadrantes de uma repetição são vistos em 384 formato sobre uma placa de agar sólido (direita) no mesmo padrão que a placa de PIG inicial (como indicado pela cor). O processo pode ser repetido para testar outros repetições: 1,2; 2,1 e 2,2. Exemplo lapso de tempo imagens à direita foram capturados 0,5, 2 e 3,5 dias após a inoculação. Adaptado a partir da Figura Complementar 1 2. Clique aqui para ver maior figura .
018/4018fig2.jpg "/>
Figura 2. Um único robô-capturado curva de crescimento QFA. A) curva de crescimento QFA de uma estirpe de levedura que cresce em agar contendo galactose a 20 ° C. As imagens foram capturadas roboticamente aproximadamente a cada 2 horas. Fase exponencial a esta temperatura foi observável para cerca de 1,5 dias, com o aumento da densidade primeira cultura tornar-se detectável cerca de 2 dias após a inoculação. Um modelo generalizado logística foi ajustado aos dados observados (curva cinza). Os parâmetros do modelo automaticamente montados pelo pacote R QFA são apresentados: K (capacidade de carga (AU)), r (taxa de crescimento (d -1)), g (inoculo densidade (AU)), v (simetria de crescimento). B) Quanto ao painel A, traçado com densidade de célula em uma escala logarítmica. Clique aqui para ver maior figura .
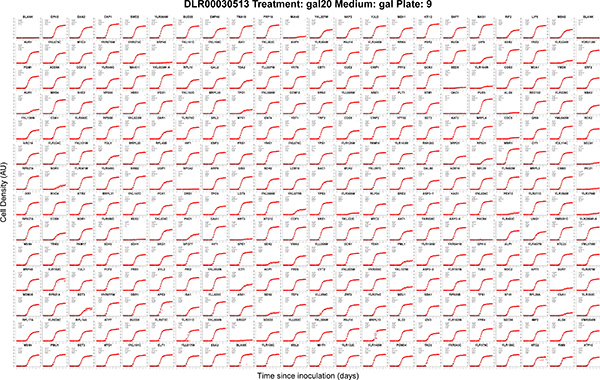
Figura 3. Automaticamente gene curvas de crescimento nominal capturado em paralelo de uma placa de 384 formato único. Cada painel mostra as estimativas de densidade de células (cruzes vermelhas) e ajuste do modelo (curvas pretas) para as culturas independentes rotulados pelo nome gene ou ORF y número cresceu em um procedimento QFA. Esta placa foi fotografada exemplo por robô e cultivadas numa incubadora automatizado a 20 ° C. Metadados Experimental são incluídos automaticamente no título figura: Nome do prato, tratamento com placa, ágar e número da placa da biblioteca. Os valores dos parâmetros do modelo são equipadas também automaticamente impresso em cada painel (ver Figura 2). Culturas de ponta foram retirados da análise QFA, deixando os 308 culturas neste lote (ver QFA Protocolo 5.4). Uma vez que as culturas de borda não são analisadas em QFA, estes locais de cultura são tipicamente preenchido com estirpes de controlo neutros (PGAL-HIS3, neste caso). A versão original desta figura, a saída pelo pacote de R QFA, é em formato. Pdf e é, portanto, infinitamente zoomable e pesquisável por consultas de texto.:/ / Www.jove.com/files/ftp_upload/4018/4018fig3.pdf "target =" _blank "> Clique aqui para ver maior figura.

Figura 4. Comparando adequação de duas telas QFA para deduzir interação genética. Este gráfico mostra a comparação da aptidão de eliminações levedura de brotamento (yfgΔ), em conjunto com a mutação ura3Δ neutro ou a temperatura mutação CDC13-1 sensível e cresceu na temperatura semi-permissivo para CDC13- 1 (27 ° C). Aptidão foi calculada como o produto da taxa de duplicação máximo e potencial máximo de duplicação 1. A maioria das estirpes de deleção crescer bem quando combinada com a mutação ura3Δ neutro, tal como esperado e têm aptidão elevada, mas combinadas com deleções CDC13-1 têm uma ampla gama de aptidões. As linhas de luz azul cruzar-se em dialocalização e da mutação his3Δ neutro, a linha sólida é um modelo de regressão linear de independência genética (aptidão esperado de CDC13-1 yfgΔ mutantes dadas aptidão de ura3Δ yfgΔ mutantes) ea linha a tracejado é a linha de aptidão iguais. Exclusões de cor verde melhorar significativamente o defeito de fitness de CDC13-1. Exclusões de cor vermelha significativamente suprimir o mesmo defeito. Distância vertical de qualquer mutação CDC13-1 yfgΔ a partir da linha sólido indica a força de interacção genética entre CDC13-1 e yfgΔ sob as condições de placas. Note-se que alguns dos mais fortes supressores de CDC13-1 são previamente identificados genes de ponto de verificação Rad17, DDC1 e RAD24 ea nuclease EXO1 perto do canto superior direito. Genes cuja exclusão reduz de fitness incluem YKU80 (que codifica para o reparo do DNA e dos telômeros nivelamento proteína YKU80) perto da canto inferior direito. Observe também que alguns grupos de genes que funcionam em conjunto tendem a ser co-localizado neste enredo. Três clusters exemplo são destacados em azul SPE1, SPE2 e SPE3: genes de síntese espermidina, Rad17, DDC1 e RAD24: componentes de codificação da pinça deslizante checkpoint e carregador de checkpoint grampo, MRE11, RAD50 e XRS2: o complexo MRX, envolvidos em DDR e Est1 & EST3: genes envolvidos na regulação do comprimento dos telômeros. Adaptado a partir de 1B Complementar Figura 1, os dados brutos a partir do qual este gráfico foi gerado pode ser baixado aqui: http://research.ncl.ac.uk/colonyzer/AddinallQFA/ . Clique aqui para ver maior figura .
Tabela 1. Arquivos eletrônicos QFA. Todos os arquivos listados (com exceção FT1) são arquivos de texto delimitados e pode ser construída ou ler usando qualquer editor de texto ou software de planilha eletrônica. Para mais detalhes sobre a construção de arquivos de metadados QFA e interpretação precisa da saída QFA consulte o QFA documentação do pacote R 7 e a documentação do software Colonyzer 3.
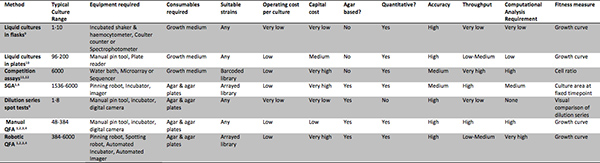
Tabela 2. Métodos para avaliar a aptidão da cultura. Um resumo das características e requeremmentos de um certo número de métodos para avaliar aptidão cultura microbiana. Uma revisão de Blomberg 8 contém comparação mais detalhada de alguns deles. Clique aqui para ver maior figura .
Discussão
QFA é em muitos sentidos, um descendente directo de três estabelecidas bancada em escala de técnicas microbiológicas: cultura de diluição testes da série 9 no local, a determinação da curva de crescimento em culturas líquidas gasosas 9 e revestimento réplica 13. Estes três métodos são resumidos e comparados com QFA e outras técnicas de alto rendimento na Tabela 2. Considerando que os ensaios de diluição no local da série definir aptidão uma estirpe como a sua capacidade para formar colónias em uma série de culturas crescidas a partir de uma gama de diluições de inóculo, QFA aptidão estirpe medidas por observações repetidas de uma única cultura para construir uma curva de crescimento. A quantificação aptidão a partir de culturas única permite muitas estirpes mais a ser examinados simultaneamente, sob condições idênticas. Replicando matrizes de culturas permite testes repetidos de coleções de tensão, mais útil ao testar as diferenças de cultura de fitness em ambientes diferentes ou fundos genéticos. O protocolo apresentado QFAaqui usa equipamento caro robótico para replicar, inocular, incubar e placas de agar de imagem, apropriado para a observação de crescimento de milhares de culturas independentes (QFA robótico, Tabela 2). No entanto, desde QFA baseia-se na estabelecidas, técnicas de laboratório tradicionais, pode também ser efectuada muito mais barato, substituindo assistência robô com passos manuais (Manual QFA, Tabela 2). Manual do QFA envolve replicação e inoculação de culturas em ágar usando uma ferramenta de pino manual e transferência manual de placas de e para uma incubadora para a fotografia. O fluxo de trabalho mesma análise computacional pode ser aplicado para gerar curvas de crescimento a partir de qualquer desenho experimental.
Estimativas QFA aptidão parecem ser relativamente preciso. Na Figura 4, as aptidões médios de quatro exemplo, agregados de funcionalmente deleções de genes relacionados são realçados. A proximidade de membros de cada classe de genes funcionalmente relacionados em duas inderentes origens genéticas (ura3Δ e CDC13-1) indica a reprodutibilidade das estimativas QFA fitness. Por exemplo, apenas 3 a deleção do gene (de um 4.300 possível) separar os três membros do complexo MRX conservada.
Alto rendimento alternativas para QFA para testar as características de crescimento de estirpes microbianas incluem PIG 5,6, telas de concorrência em bibliotecas de código de barras 11,12 e telas de captura cinética de densidade óptica em leitores espectrofotométricos placa 10, que estão resumidos na Tabela 2 e descrito mais completamente em outro lugar 8 . QFA e placas de PIG, onde as culturas crescem em superfícies sólidas de ágar (ágar métodos baseados, Tabela 2) pode ser tratada de forma rápida e fácil pelo robô. Culturas de superfície sólida de agar são bem arejadas todo o crescimento e as células podem crescer em culturas fixas, permitindo que os micróbios sociáveis a crescer em uma comunidade 14. Deixado intacto, comunidades microbianas umffect suas próprias micro-ambientes, toxinas secretoras, tais como etanol, e possivelmente de sinalização entre as células. No entanto, contínua mistura de culturas líquidas, necessárias para alcançar aeração adequada, artificialmente perturba comunidades microbianas e os seus micro-ambientes que possam afetar seus modos de crescimento. A contaminação cruzada é uma preocupação menor em métodos de ágar sólido uma vez que há menos oportunidades para líquidos salpicados transportando células entre as culturas. Se ocorrer contaminação por estrangeiros transportados pelo ar micróbios em ensaios de agar sólidos, que muitas vezes podem ser detectado por inspecção visual das placas de agar sólido e representaram ou removido.
Rendimento QFA é significativamente mais elevada do que a de paralelas métodos líquidos de duas maneiras. Primeiro de tudo, no QFA (e SGA) placas, culturas inoculadas são embalados em conjunto de forma mais densa, dando culturas mais independentes por placa. Em QFA existem normalmente 308 culturas, não contando com culturas não experimentais de ponta (Figura 1) COMcomparação em paralelo culturas líquidas com 96 ou 100 culturas por placa. Em segundo lugar, as experiências QFA pode ser dimensionada para um número muito maior de pratos. Considerando que o número de placas analisados numa experiência QFA único é limitada apenas pelo espaço disponível na sala de incubadora ou quente, a frequência de captura de imagem e mínima permitida a frequência de captura máxima alcançável, o número de placas em uma experiência de crescimento líquido é fortemente limitada pela capacidade do leitor de placas (tipicamente um ou dois pratos), ou do empilhador ligado ao leitor de placas (tipicamente 25-50 placas). Recentemente, realizou uma experiência totalmente automatizado QFA em 123 placas, cada uma com 308 culturas experimentais, dando 37,884 culturas simultâneas. Nós estimamos que o número máximo possível de culturas independentes que crescem em líquido é 96 (culturas / placa) x 50 (placas / empilhador) = 4800, que é de cerca de dez vezes menor do que o nosso rendimento QFA. Uma alternativa para as telas de cultura líquido é utilizar muitos automatizado grodispositivos wth em paralelo (Tabela 1 a partir da revisão por Blomberg 8), cada um tendo uma capacidade de um ou dois pratos. Controle de temperatura em cada dispositivo é independente, e assim as condições não são idênticas, mas assumindo que eles são, este fluxo de trabalho exigiria pelo menos 190 desses dispositivos para combinar QFA rendimento (assumindo 200 culturas líquidas por dispositivo).
Em resumo, QFA é um fluxo de trabalho de alta qualidade que podem ser utilmente aplicadas para reunir fenótipos quantitativos de crescimento em ambos os experimentos de pequena escala e centraram-se alto rendimento telas. É flexível o suficiente para ser aplicado de forma eficaz com diferentes requisitos para o equipamento robótico. O componente computacional do fluxo de trabalho QFA baseia-se livremente disponível código fonte, aberto.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Agradecemos a todos os membros de nosso laboratório e do Centro de Biologia do Sistema Integrado de Envelhecimento e Nutrição (CISBAN) para apoio e discussões úteis. Este estudo foi apoiado pelo Biotechnology and Biological Sciences Research Council (BBSRC) (BB/C008200/1) e do Wellcome Trust (075294, 093088).
Materiais
| Name | Company | Catalog Number | Comments |
| Nome de reagente / equipamentos | Companhia | Número de catálogo | Comentários |
| Replica Plater | Sigma | R-2508 | 96 pinos Manual pintool com 1/8 "de diâmetro pinos |
| Misture-Mate | Eppendorf | 5353 000.014 | |
| Biomek FX | Beckman | A31842 | |
| Teleshake | Thermo Scientific | 50095890 | Instalado em Biomek FX |
| BM3-SC | S & P Robotics Inc | BM3-SC | 192 prateleira rotativa carrossel |
| spImager | S & P Robotics Inc | spImager | Alta resolução manual de imagem |
| spImager com Cytomem | S & P Robotics Inc | Personalizado | Temperatura controlada alta resolução de imagem automatizado |
| Cytomat 6001 com trocador de calor | Thermo Scientific | 51022222 | Anexado ao spImager |
| Ecoline RE207 | Lauda | RE207 | Anexado ao Cytomat |
| spImager com carrossel | S & P Robotics Inc | Personalizado | Resolução de imagem automatizado de alta |
| Robot fixação ferramenta pinos para FP12pins | V & P Científico | AFIX96FP12 | N / A |
| 96 x FP12 pinos | V & P Científico | FP12 | 50,4 mm de comprimento, 17 mm de comprimento exposto pin |
| Docking Station para a Ferramenta de Pin | V & P Científico | VP425 | Encaixe de estação de base removido para permitir a secagem do ventilador de pinos |
| Pin Escova de limpeza | V & P Científico | VP425 | N / A |
| Replica Plater | Sigma | R-2508 | Ferramenta manual de pinos |
| Nunc OmniTray com Tampa | Nunc | 734-0490 | N / A |
| 96 bem estéreis placas de poliestireno | Greiner BioOne | 655161 | N / A |
| 96 bem estéreis tampas de poliestireno | Greiner BioOne | 656171 | N / A |
Tabela 3. Reagentes específicos e equipamentos.
Referências
- Addinall, S. G., Holstein, E., Lawless, C., Yu, M., Chapman, K., et al. Quantitative Fitness Analysis shows that NMD proteins and many other protein complexes suppress or enhance distinct telomere cap defects. PLoS Genetics. 7 (4), e1001362 (2011).
- Addinall, S. G., Downey, M., Yu, M., Zubko, M. K., Dewar, J., et al. A genomewide suppressor and enhancer analysis of cdc13-1 reveals varied cellular processes influencing telomere capping in Saccharomyces cerevisiae. Genetics. 180, 2251-2266 (2008).
- Lawless, C., Wilkinson, D., Young, A., Addinall, S., Lydall, D. Colonyzer: automated quantification of micro-organism growth characteristics on solid agar. BMC Bioinformatics. 11, 287-28 (2010).
- Tong, A. H., Evangelista, M., Parsons, A. B., Xu, H., Bader, G. D., et al. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Costanzo, M., Baryshnikova, A., Bellay, J., Kim, Y., Spear, E. D., et al. The genetic landscape of a cell. Science. 327, 425-431 (2010).
- Blomberg, A. Measuring growth rate in high-throughput growth phenotyping. Curr. Opin. Biotechnol. 22 (1), 94-102 (2011).
- Maringele, L., Lydall, D. EXO1-dependent single-stranded DNA at telomeres activates subsets of DNA damage and spindle checkpoint pathways in budding yeast yku70Δmutants. Genes and Dev. 16 (15), 1919-1933 (2002).
- Warringer, J., Blomberg, A. Automated screening in environmental arrays allows analysis of quantitative phenotypic profiles in Saccharomyces cerevisiae. Yeast. 20, 53-67 (2003).
- Hillenmeyer, M., Fung, E., Wildenhain, J., Pierce, S., Hoon, S., Lee, W., Proctor, M., St. Onge, R., Tyers, M., Koller, D., Altman, R., Davis, R., Nislow, C., Giaever, G. The chemical genomic portrait of yeast: uncovering a phenotype for all genes. Science. 320 (5874), 362-365 (2008).
- Smith, A. M., Durbic, T., Oh, J., Urbanus, M., Proctor, M., Heisler, L. E., Giaever, G., Nislow, C. Competitive Genomic Screens of Barcoded Yeast Libraries. J. Vis. Exp. (54), e2864 (2011).
- Lederberg, J., Lederberg, E. M. Replica plating and indirect selection of bacterial mutants. J. Bacteriol. 63, 399-406 (1952).
- Queller, C. Behavioural ecology: The social side of wild yeast. Nature. 456, 589-590 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados