É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Estudar proteólise de ciclina B no nível da célula individual em populações de células inteiras
Neste Artigo
Resumo
Metáfase para anáfase transição é disparada através anáfase-promover complexo (APC / C) dependente de ubiquitinação e subsequente destruição da ciclina B. Aqui, nós estabelecemos um sistema que, na sequência de pulso-caça, rotulagem permite monitorar proteólise ciclina B em populações de células inteiras e facilita a detecção de interferência do checkpoint mitótico.
Resumo
Distribuição igual dos cromossomas entre duas células filhas durante a divisão celular é um pré-requisito para garantir a estabilidade genética 1. Imprecisões durante a separação de cromossomos são uma marca registrada de malignidade e associada com doença progressiva 2-4. O posto de controle conjunto do eixo (SAC) é um mecanismo de vigilância mitótico que traz de volta as células em metáfase até que cada único cromossomo estabeleceu uma ligação bipolar estável ao fuso mitótico 1. O SAC exerce a sua função através da interferência com a activação da APC / C subunidade CDC20 para bloquear a proteólise de ciclina securin e B e, assim, a separação dos cromossomas e de saída mitótico. Penhora incorreta de cromossomos impede o silenciamento de SAC sinalização e provoca a inibição continuou da APC / C CDC20 até o problema ser resolvido para evitar missegregation cromossômica, a aneuploidia e tumores malignos 1.
A maioria dos estudos que tratam da influence cromossômica de apego indevido sobre a APC / C-dependente proteólise aproveitou a interrupção eixo usando despolimerizando ou microtúbulos drogas estabilizadoras de interferir com apego cromossômica aos microtúbulos. Uma vez que a interferência com a cinética de microtúbulos pode afectar o transporte e localização de reguladores críticos, estes procedimentos de suportar o risco de induzir efeitos artificiais 5.
Para estudar como o SAC interfere com a APC / C-dependente proteólise da ciclina B durante a mitose em imperturbável populações de células, foi estabelecido um sistema de histona H2-GFP-base que permitiu o monitoramento simultâneo de alinhamento metáfase de cromossomos mitóticos e proteólise da ciclina B 6 .
Para descrever os perfis proteolíticos, geramos um quimérico ciclina B molécula repórter com uma porção C-terminal de SNAP 6 (Figura 1). Em uma reacção de auto-rotulagem, o SNAP-porção é capaz de formar ligações covalentes comalquilguanina-transportadores (substrato SNAP) 7,8 (Figura 1). Moléculas de substrato SNAP estão prontamente disponíveis e realizar um amplo espectro de diferentes fluorocromos. Quiméricos ciclina B SNAP-moléculas tornam-se rotuladas por adição do substrato de SNAP-membrana permeável ao meio de crescimento de 7 (Figura 1). Seguindo a reacção de marcação, a intensidade de ciclina B-SNAP fluorescência cai numa reacção do tipo de pulso-caça forma e intensidades de fluorescência reflectem os níveis de degradação da ciclina B 6 (Figura 1). O nosso sistema facilita o controlo da proteólise mitótico APC / C-dependente em grande número de células (ou várias populações de células) em paralelo. Desse modo, o sistema pode ser uma ferramenta valiosa para a identificação de agentes / moléculas pequenas que são capazes de interferir com a actividade proteolítica na metafase para a anafase transição. Além disso, como a síntese de ciclina B durante a mitose foi recentemente sugerida como uma mechanis importantesm na promoção de um bloco mitótica em camundongos e humanos, mantendo os níveis de ciclina expressão B estáveis 9,10, este sistema permitiu-nos analisar ciclina proteólise B como um elemento de um equilíbrio harmonioso 6.
Protocolo
1. Semeadura de U2OS baseados em células ciclina B SNAP-Repórter (Clone 11 Células 6) em lâminas de microscopia Câmara
- Tripsinizar as células subconfluentes SNAP repórter que foram deixadas a crescer de forma assíncrona em fase log durante pelo menos 48 h.
- Trabalhando com 8 poços (câmaras de microscópio distribuição constante de células).
Para propagação de células em câmaras de 8 poços a microscópio uma distribuição constante por toda a superfície da câmara de microscópio, de centrifugação 10.000 células e ressuspender em 350 ul de fenol de meio de crescimento isento de vermelho de normal (suplementado com 10% de soro fetal bovino, penicilina / estreptomicina e piruvato de sódio). Transferir a suspensão de células na câmara de microscópio (Figura 2).
Trabalhando com 8 câmaras microscópio poços (densidade celular máxima no centro).
Para propagação de células a uma densidade mais elevada no centro do microscópio Chamber, carregar a câmara com 300 ul de fenol de meio de crescimento isento de vermelho de normal. Adicionar 5000 células cuidadosamente para o centro da câmara de microscópio (Figura 2).
Trabalhando com placas de 96 cavidades especiais óptica (distribuição constante de células).
Para propagação de células em placas de 96 poços a uma constante de distribuição por toda a superfície do poço, centrifugar 5000 células e ressuspender em 300 ul de fenol de meio de crescimento isento de vermelho de normal. Transferir a suspensão de células para uma placa de 96 poços. Dependendo do número total de células contendo poços necessário, ajustar o número de células e o volume total do meio de suspensão (Figura 2).
Trabalhando com placas de 96 cavidades especiais óptica (densidade celular máxima no centro).
Para propagação de células em placas de 96 poços, cuidadosamente adicionar 1.500 células em 15 ul de meio de crescimento de fenol livre de vermelho normal num smalgota l para o centro de cada poço para obter restrição do crescimento celular para o centro do poço (Figura 2).
- Permitir que as células semeadas a crescer durante pelo menos 18 horas em condições normais de cultura de células (37 ° C, humidade relativa do ar de 100%, 5% CO 2).
2. A coloração de células Reporter com substrato SNAP
- 30 min antes do início do procedimento de coloração permitir alíquotas de meio de crescimento de fenol vermelho normal, livre de se aquecer até 37 ° C.
- Para um manuseamento fácil do substrato SNAP (no nosso caso TMR Star) dissolver Estrela TMR em DMSO para se obter uma concentração na solução-mãe de 400 uM, a qual pode ser armazenada a -20 ° C.
- Antes da coloração, diluir 0,5 ul de solução-mãe da estrela TMR em 200 ul de fenol de meio de crescimento isento de vermelho de normal (37 ° C) para obter uma concentração final de rotulagem de 1 uM.
- Remover meio de crescimento normal das células em crescimento de forma assíncrona e incubar em lAbeling meio durante 25 minutos sob condições de cultura padrão.
- Remover marcação de células de média e de lavagem por quatro vezes com fenol meio de crescimento isento de vermelho de normal (37 ° C). Incubar as células em 300 ul de fenol meio de crescimento isento de vermelho de normal (37 ° C) durante 30 min. Antes de transportar para o microscópio de substituir o meio com meio de crescimento fresco fenol livre de vermelho de normal (37 ° C) para remover o substrato SNAP residual não ligado.
- Células de transporte para o microscópio de uma caixa de poliestireno expandido, uma relação pré-aquecido (37 ° C) do bloco de aquecimento para minimizar a variação de temperatura.
3. Medição da Intensidade de Fluorescência
- Duas horas antes da análise de ajustar a temperatura do ar da câmara climática a 37 ° C em modo de secagem, a fim de trazer o microscópio inteiro com todos os seus componentes para a temperatura desejada. O pré-aquecimento antes de definir a humidade é importante para evitar a condensação e subsequentes danos ao microscópio.
- Ajuste t umidade do aro 60% de% de CO 2-5 antes do início da análise.
- Inicie a digitalização ^ software de aquisição de R e definir configurações padrão (ver quadro 1).
- Definem as posições dos poços a serem analisados.
- Definir a Dt (tempo do ciclo de aquisição) e o número absoluto de ciclos de aquisição.
- Se a análise de um número maior de poços é desejado, seleccionar a focagem automática de hardware, de outra forma, é suficiente utilizar autofocus software sozinho.
- Iniciar e supervisionar a aquisição para os primeiros dois ciclos de aquisição. O microscópio focará o sinal H2-GFP histona, com subsequente aquisição de uma primeira imagem de que o canal antes de o filtro é alterado e a imagem correspondente da estrela TMR for adquirida (Figura 3). Este é, então, repetido para todas as posições dentro de um poço e para cada um dos poços a serem examinados, antes de repetir novamente o ciclo seguinte.
4. Análise de u Perfis ProteolyticDigitalização cantar ^ R
- Inicie a digitalização ^ software de Análise de R e analisar as imagens com os núcleos das células, tal como visualizado por histona H2-GFP, definido como o objecto principal, usando um limiar baseado na intensidade do sinal e um algoritmo de bacia para auxiliar na separação de células vizinhas. Um subobject consistindo de um núcleo com citoplasma deve ser criada para análise TMRstar. (Propriedades importantes do objeto principal são posições X e Y, o tempo, e máxima e média intensidade de GFP para o objecto principal e a intensidade total dividido pela área do subobject TMRstar.) Este processo de análise pode demorar várias horas, devido a grandes volumes de dados quantidades.
- Mudar para rastrear modo a visualizar o TMR subobject média intensidade de fluorescência ao longo do tempo da estrela (vestígios de células), atribuídas aos objectos analisados principais (Figura 4). O número de células a ser examinada pode ser reduzida por gating nessas medições de duração de pelo menos 140 ciclos e com uma elevada intensidade máxima de H2-GFP. Olhandoem números maiores de células permite que um representante da primeira e vista objectivo (Figura 4).
- Seleccionar um rastreio celular de interesse para visualizar histona H2-GFP e TMR Estrela fluorescência simultaneamente ao nível da célula individual (Figura 5A).
- Usando o botão direito do mouse em uma célula de interesse e gerar uma galeria de imagem exportável para ilustração de histona H2-GFP e ciclina B-SNAP TMR estrela para cada ponto de tempo único.
- Mudar para o modo de população do software de Análise de digitalização R ^ e portão na região em que a célula de interesse é representada no vs Y X gráfico de pontos (Figura 5B).
- Aplicar uma janela nova parcela de ponto para a região e visualizar fechado significa TMR Estrela intensidade de fluorescência ao longo do tempo (Figura 5C).
- Exportação de dados (tempo e intensidade de fluorescência) para o Microsoft Excel para cálculo.
5. Resultados representativos
Figoure 5D e 5E mostram a ciclina B cinética, representada por uma curva de intensidade de fluorescência TMR estrela, de uma célula que prossegue através de uma mitose normal sem sinais de desalinhamento cromossómica (Figura 5E). Após a compactação citoplasmática após ruptura do envelope nuclear (NEBD, como indicado pelo triângulo vermelho), TMR Estrela intensidade de fluorescência mostra um aumento abrupto até que a janela isomorfos (área mais clara no diagrama) é alcançado quando a célula entra na prometafase 6. A intensidade de fluorescência permanece a um nível estável, enquanto os rendimentos celulares através prófase e metáfase e então começa a cair rapidamente uma vez que todos os cromossomas estabeleceram uma placa metafásica estável (Figura 5D e 5E). Esta queda precede a separação dos cromossomos durante a anáfase (ponto azul na curva). Na mitose tarde a cromatina começa a decondense (barras azuis) e a célula adota morfologia interfase enquanto a curva de intensidade de fluorescência, se aproxima de umplanalto que é mais baixo do que o planalto antes da mitose (Figura 5D e 5E).
| Autofoco configurações | Histona H2-GFP (configurações principal aquisição do objeto) | Ciclina B marcada com SNAP-TMR Estrela (Configurações de aquisição) | As repetições de aquisição do tempo de ciclo |
| Grosseiras autofocus + / -39 24 uM Camadas Autofoco multa + / -5,4 UM 14 Camadas | GFP filtro definido: Tempo de exposição: 100 mseg Intensidade luminosa: 25% | TRITC filtro definido: Tempo de exposição: 150 msec A intensidade da luz: 33,3% | 2 a 5 min. |
| GFP filtro definido: Tempo de exposição: 12 mseg A intensidade da luz: 12,5% | Até 48 h de análise continuada (limitada pela humidade do ar reduzida de 60%) |
Tabela 1. Configurações padrão conforme utilizadas para análise da ciclina B proteólise. Histona H2-GFP foi utilizado como uma estrutura de referência que definem o plano de focagem para a medição da ciclina B-SNAP intensidade de fluorescência. Intensidades de luz durante o processo de focagem são menores em comparação com as configurações de aquisição de imagem para evitar fototoxicidade cumulativo.
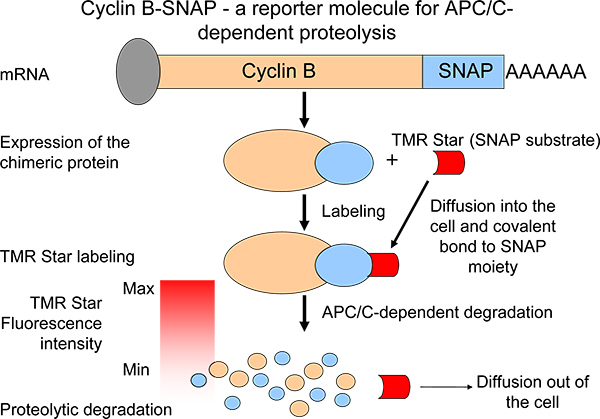
Figura 1. Esquemática da medição da ciclina B proteólise através da expressão de um derivado quimérico ciclina B com características de degradação semelhantes à proteína endógena. A diminuição na intensidade de fluorescência após marcação de pulso-caça é uma medida da APC / C-actividade.
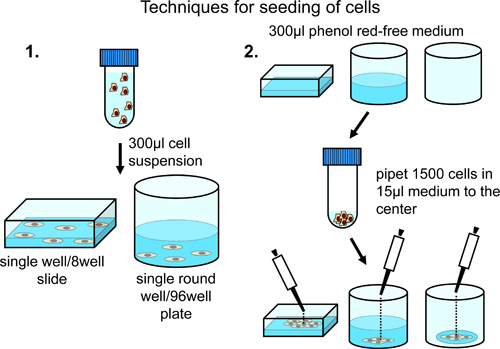
Figura 2. Análise de células em oito lâminas bem ou placas de 96 poços. Distribuição regular docélulas em toda a superfície do poço é conseguida por ressuspensão num volume final de 300 ul, e é recomendada, se apenas um único ou poucos posições são definidas manualmente para análise. O enriquecimento de células na região central é conseguida pela adição de células ao centro dos poços pré-carregadas ou vazias. Esta técnica é recomendado no caso de aquisição de imagem automatizado em poços diferentes.

Figura 3. Seqüência de aquisição de dados. A) Detecção de histona H2-GFP é usada para a definição de foco avião e monitoramento de alinhamento cromossômica durante a mitose. B) A intensidade da fluorescência da intensidade de fluorescência TMR estrela é uma medida para a quantidade absoluta de pulse-chase rotulado ciclina B SNAP-.
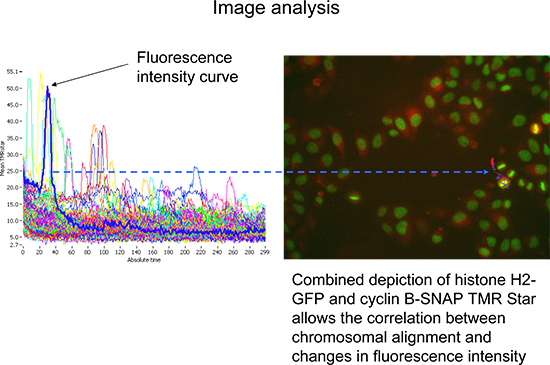
Figura 4. Representante representação digitalização ^ Análise R baseado em software de média TMR Estrelaintensidades de fluorescência (intensidade TMRstar total dividido por área) ao longo do tempo. Seleção de um traço célula representativa (azul). As imagens correspondentes (mostrado à direita) permitem a visualização simultânea de histona H2-GFP (verde) e ciclina B-SNAP (vermelho). Clique aqui para ver maior figura .
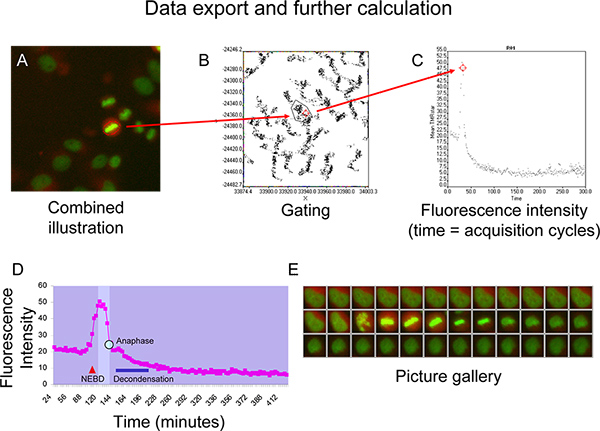
Figura 5. A + B) Exemplo de propagação de pontos representando uma célula de juros sobre o enredo XY-ponto. C) A visualização da intensidade média de fluorescência TMRstar (intensidade TMRstar total dividido pela área) ao longo do tempo utilizando o gráfico de pontos. D + E) curva de intensidade de fluorescência de Representante ciclina B SNAP-com janela isomórfica (brigher campo no diagrama) entre descondensação NEBD (quebra envelope nuclear) e cromatina (barra azul) como gerado no Microsoft Excel. Anafase é indicado por tele ponto azul na curva. Clique aqui para ver maior figura .
Filme 1. Clique aqui para ver filme suplementar .
Discussão
Apresentamos aqui uma abordagem live-célula de imagem baseada em facilitar o monitoramento simultâneo de ciclina B proteólise e alinhamento de cromossomas. Esta abordagem permite o estudo de controlo mitótico em populações de células não perturbados ao nível da célula individual. As curvas de degradação de ciclina B facilitar percepções directas para a actividade do APC / C e assim, indirectamente, reflectem o estado do SAC 6.
Esta abordagem, apesar de estabelecidos ...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Somos gratos a S. Taylor para fornecer o pLPCX-histona H2-GFP plasmídeo. Agradecemos R. Mertelsmann de apoio contínuo. Este trabalho foi financiado pelo Deutsche Forschungsgemeinschaft.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do produto | Companhia | Número de catálogo | Comentários (opcional) |
| Linha de células repórter | gerado em casa, como descrito 6 | clone 11 células repórter (U2OS baseados ciclina B-SNAP células que expressam) | |
| Retroviral ciclina vetor de expressão B SNAP- | gerado em casa, como descrito 6 | pLNCX2-ciclina B mut5 SNAP- | |
| Fenol livre de DMEM | Gibco | 21063-029 | A suplementação com FCS, piruvato de sódio, penicilina / estrepto-mycin exigido |
| SNAP-Cell TMR-Star | New England Biolabs | S9105S | Solução estoque 400 mM em DMSO |
| Placa Opstics especial, de 96 poços | Costar | 3720 | |
| μ Slide-8 bem, ibiTreat | Ibidi | 80826 | |
| Unidade de Microscopia | Olimpo | IX-81 microscópio invertido com câmara climática | |
| Objetivo | Olimpo | UPLSAPO objetiva de 20x (NA 0,75) | |
| Software de aquisição | Olimpo | Digitalização ^ Aquisição R software (v.2.2.09) | |
| Software de análise de | Olimpo | Digitalização ^ Análise R software (v.1.2.0.6) |
Referências
- Musacchio, A., Salmon, E. D. The spindle-assembly checkpoint in space and time. Nat. Rev. Mol. Cell Biol. 8, 379-393 (2007).
- Nasmyth, K. Segregating sister genomes: the molecular biology of chromosome separation. Science. 297, 559-565 (2002).
- Cahill, D. P. Mutations of mitotic checkpoint genes in human cancers. Nature. 392, 300-303 (1998).
- Lengauer, C., Kinzler, K. W., Vogelstein, B. Genetic instabilities in human cancers. Nature. 396, 643-649 (1998).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463, 485-4892 (2010).
- Schnerch, D. Monitoring APC/C activity in the presence of chromosomal misalignment in unperturbed cell populations. Cell Cycle. 11, (2012).
- Keppler, A. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat. Biotechnol. 21, 86-869 (2003).
- Jansen, L. E., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J. Cell. Biol. 176, 795-805 (2007).
- Malureanu, L. Cdc20 hypomorphic mice fail to counteract de novo synthesis of cyclin B1 in mitosis. J. Cell Biol. 191, 313-329 (2010).
- Mena, A. L., Lam, E. W., Chatterjee, S. Sustained spindle-assembly checkpoint response requires de novo transcription and translation of cyclin B1. PLoS One. 5, (2010).
- Wolthuis, R. Cdc20 and Cks direct the spindle checkpoint-independent destruction of cyclin. A. Mol. Cell. 30, 290-302 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados