È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Studiare proteolisi della ciclina B a livello di singola cellula in popolazioni di cellule intere
In questo articolo
Riepilogo
Metafase di anafase transizione viene attivato attraverso la promozione anafase-complesso (APC / C)-dipendente ubiquitinazione e la successiva distruzione della ciclina B. Qui, abbiamo creato un sistema che, a seguito di pulse-chase etichettatura, permette di monitorare ciclina proteolisi B in popolazioni di cellule intere e facilita la rilevazione di interferenza del checkpoint mitotico.
Abstract
Distribuzione uniforme dei cromosomi tra le due cellule figlie durante la divisione cellulare è un prerequisito per garantire la stabilità genetica 1. Imprecisioni in fase di separazione dei cromosomi sono una caratteristica di malignità e associata a progressione della malattia 2-4. Il checkpoint del fuso (SAC) è un meccanismo di controllo mitotico che trattiene le cellule in metafase fino a quando ogni singolo cromosoma ha stabilito un legame stabile bipolare al fuso mitotico 1. La SAC esercita la sua funzione per interferenza con l'attivazione di APC / C subunità Cdc20 di bloccare la proteolisi di securin e ciclina B e quindi la separazione dei cromosomi e uscire mitotico. Attacco improprio dei cromosomi impedisce di mettere a tacere SAC segnalamento e provoca l'inibizione segue da APC / C Cdc20 fino a quando il problema è risolto per evitare missegregation cromosoma, aneuploidia e carcinomi 1.
La maggior parte degli studi che hanno affrontato il influence improprio di attaccamento cromosomica su APC / C-dipendente proteolisi ha approfittato di interruzione del mandrino utilizzando depolimerizzante o microtubuli stabilizzazione farmaci di interferire con l'attaccamento cromosomica ai microtubuli. Poiché interferenze con cinetiche microtubuli può influenzare il trasporto e la localizzazione dei regolatori critici, queste procedure sopportare un rischio di indurre effetti artificiali 5.
Per studiare come la SAC interferisce con APC / C-dipendente della ciclina B proteolisi durante la mitosi in imperturbabile popolazioni cellulari, abbiamo stabilito un sistema di istoni H2-GFP-base che ha permesso il monitoraggio simultaneo di allineamento metafase dei cromosomi mitotici e proteolisi della ciclina B 6 .
Per rappresentare profili proteolitici, abbiamo generato una molecola chimerica B ciclina reporter con una porzione C-terminale SNAP 6 (figura 1). In una reazione di auto-etichettatura, la SNAP-porzione è in grado di formare legami covalenti conalkylguanine-vettori (substrato SNAP) 7,8 (Figura 1). Molecole substrato SNAP sono facilmente disponibili e trasportare un ampio spettro di differenti fluorocromi. Chimerici ciclina B-SNAP molecole diventano etichettati dopo aggiunta della membrana permeabile substrato SNAP al mezzo di crescita 7 (Figura 1). Dopo la reazione di marcatura, l'intensità ciclina B-SNAP fluorescenza gocce in un pulse-chase reazione modo simile e intensità di fluorescenza riflettere livelli di ciclina B degradazione 6 (figura 1). Il nostro sistema facilita il monitoraggio della mitotico APC / C-dipendente proteolisi in un gran numero di cellule (o più popolazioni di cellule) in parallelo. In tal modo, il sistema può essere un valido strumento per identificare agenti / piccole molecole che sono in grado di interferire con l'attività proteolitica in metafase per anafase transizione. Inoltre, come sintesi di ciclina B durante la mitosi è stato recentemente proposto come un mechanis importantim nel promuovere un blocco mitotico nei topi e negli esseri umani, mantenendo stabili i livelli di espressione della ciclina B 9,10, questo sistema ci ha permesso di analizzare proteolisi B ciclina come uno degli elementi di una situazione di equilibrio 6.
Protocollo
1. Semina di U2OS a base di ciclina B-SNAP cellule Reporter (Clone 11 celle 6) su vetrini per microscopio Camera
- Tripsinizzare subconfluenti cellule giornalista SNAP che sono stati autorizzati a crescere in modo asincrono in fase di log per almeno 48 ore.
- Lavorare con 8 camere e microscopio (distribuzione costante di cellule).
Per la semina di cellule su 8 camere microscopio e ad una distribuzione costante su tutta la superficie della camera di microscopio, centrifuga 10.000 cellule e risospendere in 350 pl di rosso fenolo terreno privo crescita normale (integrato con 10% siero fetale bovino, penicillina / streptomicina e sodio piruvato). Trasferire sospensione cellulare alla camera microscopio (Figura 2).
Lavorare con 8 camere e microscopio (densità cellulare massima al centro).
Per la semina di cellule ad una densità superiore al centro del chamb microscopioer, caricare la camera con 300 ml di rosso fenolo terreno privo di una crescita normale. Aggiungere 5.000 cellule attenzione al centro della camera microscopio (Figura 2).
Lavorare con ben 96 piatti speciali ottiche (distribuzione costante di cellule).
Per la semina di cellule su piastre da 96 pozzetti ad una distribuzione costante su tutta la superficie del pozzetto, centrifugare 5000 e risospendere le cellule in 300 microlitri di rosso fenolo terreno privo di crescita normale. Trasferire la sospensione cellulare di una piastra a 96 pozzetti. A seconda del numero totale di cellule contenenti pozzetti necessari, regolare il numero di cellule e il volume totale del mezzo di sospensione (Figura 2).
Lavorare con ben 96 piatti speciali ottiche (densità cellulare massima al centro).
Per la semina di cellule su piastre a 96 pozzetti, aggiungere con cautela 1.500 cellule in 15 ml di rosso fenolo terreno privo di crescita normale in un smalL goccia al centro di ogni pozzetto per ottenere restrizione della crescita cellulare al centro del pozzetto (Figura 2).
- Permettono cellule seminate a crescere per almeno 18 ore in condizioni standard di coltura cellulare (37 ° C, umidità 100%, 5% CO 2).
2. Colorazione di cellule Reporter con substrato SNAP
- 30 min prima dell'inizio della procedura di colorazione permettono aliquote di rosso fenolo terreno privo crescita normale riscaldare fino a 37 ° C.
- Per una facile manipolazione del substrato SNAP (nel nostro caso TMR stella) sciogliere stella TMR in DMSO per ottenere una concentrazione nella soluzione stock di 400 pM, che può essere conservato a -20 ° C.
- Prima della colorazione, diluire 0,5 ml di soluzione TMR magazzino stella in 200 pl di rosso fenolo terreno privo crescita normale (37 ° C) per ottenere una concentrazione finale di 1 pM etichettatura.
- Estrarre il supporto di normale crescita delle cellule in modo asincrono in crescita e incubare in lAbeling media per 25 minuti in condizioni di coltura standard.
- Rimuovere etichettatura celle medie e lavare quattro volte con rosso fenolo mezzo privo crescita normale (37 ° C). Incubare le cellule in 300 microlitri di rosso fenolo terreno privo crescita normale (37 ° C) per 30 min. Prima del trasporto al microscopio sostituire il mezzo fresco con rosso fenolo terreno privo crescita normale (37 ° C) per rimuovere residui di substrato SNAP non legato.
- Cellule di trasporto al microscopio in una scatola di polistirolo su un pre-riscaldato (37 ° C) blocco termico per ridurre al minimo variazione di temperatura.
3. Misurazione dell'intensità di fluorescenza
- Due ore prima dell'analisi regolare la temperatura della camera climatica a 37 ° C in modo secco per portare l'intera microscopio con tutti i suoi componenti alla temperatura desiderata. Pre-riscaldamento prima di impostare l'umidità è importante per evitare danni condensazione e successiva al microscopio.
- Regolare t umidità dell'ariao 60% e CO 2 al 5% prima dell'inizio dell'analisi.
- Avviare il software di acquisizione di scansione ^ R e definire le impostazioni standard (vedi tabella 1).
- Definire le posizioni dei pozzetti da analizzare.
- Definire il At (tempo di ciclo di acquisizione) e il numero assoluto di cicli di acquisizione.
- Se l'analisi di un maggior numero di pozzi è desiderato, selezionare autofocus hardware, altrimenti è sufficiente utilizzare solo software autofocus.
- Avviare l'acquisizione e supervisione per i primi due cicli di acquisizione. Il microscopio si concentrerà sul istoni H2-GFP segnale, con successiva acquisizione di una prima immagine in quel canale prima del filtro viene modificata e la corrispondente immagine viene acquisita stella TMR (Figura 3). Questo viene quindi ripetuto per tutte le posizioni all'interno di un pozzo e per ciascuno dei pozzetti da esaminare, prima di ripetere nuovamente il ciclo successivo.
4. Analisi di Proteolytic u profilicantare Scan ^ R
- Avviare il software di analisi Scan ^ R e analizzare le immagini con i nuclei delle cellule, come visualizzato mediante istoni H2-GFP, definito come l'oggetto principale, tramite una soglia in base all'intensità del segnale e un algoritmo spartiacque per aiutare a separare cellule vicine. Una sotto-oggetto costituito da un nucleo con il citoplasma deve essere creato per l'analisi TMRstar. (Proprietà importanti dell'oggetto principale sono posizioni X e Y, l'ora e la massima e media intensità di BPA per l'oggetto principale e l'intensità totale diviso per area per il subobject TMRstar.) Questo processo di analisi può richiedere diverse ore a causa di dati di grandi dimensioni quantitativi.
- Cambiare modalità di tracciare per visualizzare la media subobject TMR stella intensità di fluorescenza nel tempo (tracce cellulari), assegnato a oggetti analizzati principali (Figura 4). Il numero di cellule da esaminare può essere ridotto dal gating su tali misure di durata almeno 140 cicli e ad alta intensità massima di H2-GFP. Guardandoa un numero maggiore di cellule permette un rappresentante prima e vista obiettivo (Figura 4).
- Selezionare una traccia di cellule di visualizzare istoni H2-GFP e TMR stella fluorescenza simultaneamente a livello di singola cellula (Figura 5A).
- Utilizzando il tasto destro del mouse su una cella di interesse e generare una galleria di foto esportabile per l'illustrazione delle istone H2-GFP e ciclina B-SNAP stella TMR per ogni punto di volta.
- Passare alla modalità di popolazione del software ^ Scan Analisi R e porta la regione in cui è rappresentata la cella di interesse nella proprietà vs X Y punto (Figura 5B).
- Applicare una nuova finestra diagramma a punti per la regione gated e visualizzare significa TMR stella intensità di fluorescenza nel tempo (Figura 5C).
- Esportazione dei dati (tempo e l'intensità di fluorescenza) in Microsoft Excel per il calcolo ulteriore.
5. Risultati rappresentativi
Ficoure 5D e 5E illustrano cinetica ciclina B, rappresentata da una intensità TMR stella curva di fluorescenza, di una cellula che avviene con la mitosi normale senza segni di disallineamento cromosomico (Figura 5E). Su compattazione del citoplasma seguente suddivisione involucro nucleare (NEBD, come indicato dal triangolo rosso), TMR stella intensità di fluorescenza mostra un aumento brusco finché la finestra isomorfi (zona luminosa nel diagramma) viene raggiunta quando la cellula entra prometafase 6. Intensità di fluorescenza rimane ad un livello stabile finché procede cellulari attraverso profase e metafase e quindi inizia a scendere rapidamente una volta tutti i cromosomi hanno stabilito una piastra stabile metafase (Figura 5D e 5E). Questo calo precede la separazione dei cromosomi durante l'anafase (punto blu sulla curva). Nel tardo mitosi la cromatina comincia a decondense (barre blu) e la cellula adotta morfologia interfase mentre la curva si avvicina ad una intensità di fluorescenzaplateau che è inferiore alla prima mitosi plateau (Figura 5D e 5E).
| Autofocus impostazioni | Istoni H2-GFP (principali impostazioni di acquisizione degli oggetti) | Ciclina B-SNAP marcato con TMR stella (Impostazioni di acquisizione) | Tempo di ciclo di acquisizione Ripetizioni |
| Coarse autofocus + / -39 micron 24 strati Belle messa a fuoco automatica + / -5,4 Micron 14 strati | GFP filtro impostato: Tempo di esposizione: 100 msec Intensità luminosa: 25% | TRITC filtro impostato: Tempo di esposizione: 150 msec Intensità luminosa: 33,3% | 2 a 5 min. |
| GFP filtro impostato: Tempo di esposizione: 12 msec Intensità luminosa: 12,5% | Fino a 48 ore di analisi continua (limitata dalla ridotta umidità dell'aria del 60%) |
Tabella 1. Impostazioni standard come utilizzate per l'analisi di proteolisi ciclina B. Histone H2-GFP è stato utilizzato come struttura di riferimento per definire il piano di messa a fuoco per la misurazione della ciclina B-SNAP intensità di fluorescenza. Intensità di luce durante la procedura di messa a fuoco sono inferiori rispetto alle impostazioni di acquisizione delle immagini per evitare di fototossicità cumulativo.
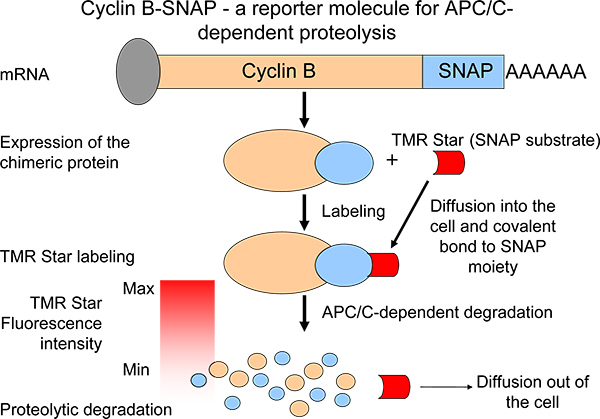
Figura 1. Schematico della misurazione della proteolisi ciclina B attraverso l'espressione di un derivato chimerico ciclina B degradazione con caratteristiche simili alla proteina endogena. Il calo di intensità di fluorescenza dopo pulse-chase etichettatura è una misura di APC / C-attività.
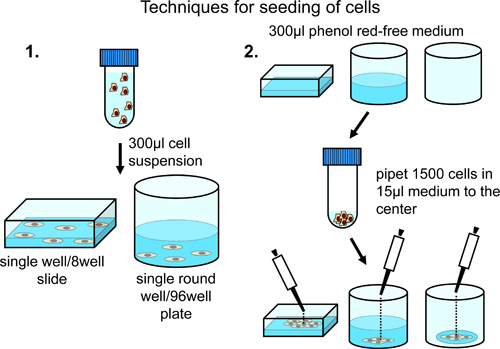
Figura 2. Analisi di celle 8 pozzetti o piastre da 96 pozzetti. Distribuzione regolare deicellule attraverso la superficie del pozzetto è realizzato mediante risospensione in un volume finale di 300 microlitri ed è consigliata, se solo posizioni singola o poche sono definiti manualmente per l'analisi. Arricchimento delle cellule nella regione centrale si ottiene per aggiunta di cellule al centro di pozzetti preriempite o vuoto. Questa tecnica è consigliata in caso di acquisizione di immagini automatizzata in pozzi diversi.

Figura 3. Sequenza di acquisizione dei dati. A) Individuazione di istoni H2-GFP è utilizzato per la definizione del piano di messa a fuoco e il controllo di allineamento dei cromosomi durante la mitosi. B) intensità di fluorescenza delle intensità di fluorescenza TMR stella è una misura per la quantità assoluta di pulse-chase marcato ciclina B-SNAP.
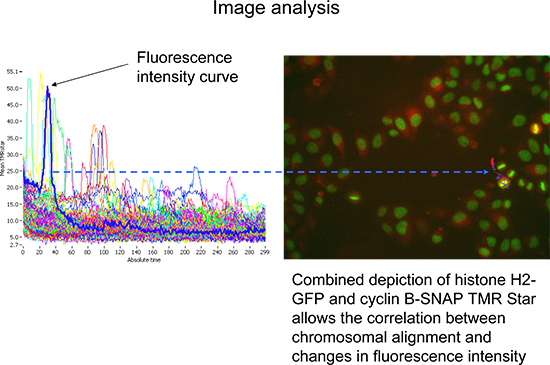
Figura 4. Rappresentante Scan rappresentazione ^ Analisi R basata su software di media TMR Stellaintensità di fluorescenza (intensità TMRstar totale diviso per zona), nel corso del tempo. Selezione di una traccia cella rappresentante (blu). Le immagini corrispondenti (mostrato a destra) consentono la visualizzazione simultanea di istoni H2-GFP (verde) e ciclina B-SNAP (rosso). Clicca qui per ingrandire la figura .
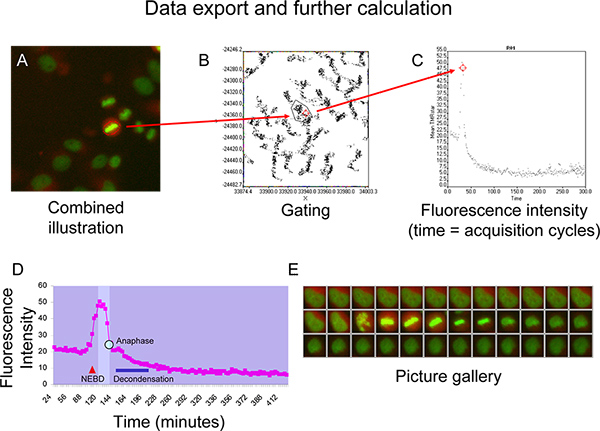
Figura 5. A + B) Esempio di gating dei punti che rappresentano una cella di interesse sulla XY-dot plot. C) Visualizzazione della fluorescenza media TMRstar (intensità TMRstar totale diviso per zona) nel tempo con il dot-plot. D + E) Rappresentante curva di intensità di fluorescenza della ciclina B-SNAP con finestra isomorfi (campo brigher nel diagramma) tra NEBD (rottura involucro nucleare) e cromatina decondensazione (barra blu) come generato in Microsoft Excel. Anafase viene indicato con tegli punto blu sulla curva. Clicca qui per ingrandire la figura .
Movie 1. Clicca qui per visualizzare filmati supplementare .
Discussione
Vi presentiamo qui un live-cell approccio di imaging basata facilitare il monitoraggio simultaneo di proteolisi ciclina B e l'allineamento dei cromosomi. Questo approccio permette lo studio del controllo mitotico negli imperturbabile popolazioni cellulari a livello di singola cellula. Curve di degradazione della ciclina B facilitare intellezioni dirette nell'attività della APC / C e quindi indirettamente riflettere lo stato della SAC 6.
Questo approccio, pur calcolato sul...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Siamo grati a S. Taylor per la prestazione del pLPCX-istoni H2-GFP plasmide. Ringraziamo R. Mertelsmann per il supporto continuo. Questo lavoro è stato finanziato dalla Deutsche Forschungsgemeinschaft.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del prodotto | Azienda | Numero di catalogo | Commenti (opzionale) |
| Reporter linea cellulare | generato in-house, come descritto 6 | clonare 11 celle giornalista (U2OS basati ciclina B-cellule che esprimono SNAP) | |
| Retrovirale ciclina B-SNAP vettore di espressione | generato in-house, come descritto 6 | pLNCX2-ciclina B mut5-SNAP | |
| Phenolred senza DMEM | Gibco | 21063-029 | La supplementazione con FCS, piruvato di sodio, penicillina / strepto-mycin richiesta |
| SNAP-Cell TMR-Star | New England Biolabs | S9105S | Soluzione madre 400 mM in DMSO |
| Speciale Opstics Piatto, 96 pozzetti | Costar | 3720 | |
| μ-Slide 8 bene, ibiTreat | Ibidi | 80826 | |
| Microscopia unità | Olimpo | IX-81 Microscopio inverso con camera climatica | |
| Obiettivo | Olimpo | UPLSAPO obiettivo 20x (NA 0,75 €) | |
| Acquisizione software | Olimpo | Scan ^ R Acquisizione software (v.2.2.09) | |
| Analisi del software | Olimpo | Scan ^ R Software di analisi (v.1.2.0.6) |
Riferimenti
- Musacchio, A., Salmon, E. D. The spindle-assembly checkpoint in space and time. Nat. Rev. Mol. Cell Biol. 8, 379-393 (2007).
- Nasmyth, K. Segregating sister genomes: the molecular biology of chromosome separation. Science. 297, 559-565 (2002).
- Cahill, D. P. Mutations of mitotic checkpoint genes in human cancers. Nature. 392, 300-303 (1998).
- Lengauer, C., Kinzler, K. W., Vogelstein, B. Genetic instabilities in human cancers. Nature. 396, 643-649 (1998).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463, 485-4892 (2010).
- Schnerch, D. Monitoring APC/C activity in the presence of chromosomal misalignment in unperturbed cell populations. Cell Cycle. 11, (2012).
- Keppler, A. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat. Biotechnol. 21, 86-869 (2003).
- Jansen, L. E., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J. Cell. Biol. 176, 795-805 (2007).
- Malureanu, L. Cdc20 hypomorphic mice fail to counteract de novo synthesis of cyclin B1 in mitosis. J. Cell Biol. 191, 313-329 (2010).
- Mena, A. L., Lam, E. W., Chatterjee, S. Sustained spindle-assembly checkpoint response requires de novo transcription and translation of cyclin B1. PLoS One. 5, (2010).
- Wolthuis, R. Cdc20 and Cks direct the spindle checkpoint-independent destruction of cyclin. A. Mol. Cell. 30, 290-302 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon