Method Article
Produção de
Neste Artigo
Resumo
Descrevemos a coleção de não fertilizado Xenopus tropicalis Ovos e produção de um extrato de ovo II-preso meiose. Este extracto de ovo pode ser utilizada para purificar a microtúbulos e RNAs microtúbulos associados.
Resumo
Muitos organismos localizar mRNAs para destinos subcelulares específicas para a expressão do gene de controle espacial e temporalmente. Estudos recentes têm demonstrado que a maior parte do transcriptoma está localizada a uma posição não aleatória em células e de embriões. Uma abordagem para identificar mRNAs localizadas é purificar bioquimicamente uma estrutura celular de interesse e para identificar todos os transcritos associados. Usando tecnologias de sequenciamento de alto rendimento recentemente desenvolvidos agora é fácil identificar todos os RNAs associados com uma estrutura subcelular. Para facilitar a identificação do transcrito, é necessário trabalhar com um organismo com um genoma completamente sequenciado. Um sistema atractivo para a purificação bioquímica das estruturas celulares são extractos de ovos produzidos a partir da rã Xenopus laevis. No entanto, X. laevis atualmente não tem um genoma completamente seqüenciado, o que dificulta a identificação transcrição. Neste artigo descrevemos um métodopara a produção de extratos de ovos de um sapo relacionado, X. tropicalis, que tem um genoma completamente sequenciado. Nós fornecemos detalhes para a polimerização dos microtúbulos, purificação e isolamento transcrição. Embora este artigo descreve um método específico para a identificação dos transcritos associada aos microtúbulos, acreditamos que isso será facilmente aplicada a outras estruturas subcelulares e irá proporcionar um método poderoso para identificação de ARN localizadas.
Introdução
Controle espacial e temporal da expressão do gene é importante para todas as células, e é especialmente importante para o controle do início embrionário tamborilar 1. Controlo espacial da expressão do gene é conseguida através da localização activa de mRNAs para destinos específicos dentro das células ou embriões. Em muitos tipos de células muito grandes, (por exemplo, oócitos, embriões, e neurónios) localização do mRNA é usada para restringir a expressão da proteína para o local de acção da proteína codificada. Uma vez que um ARNm localizada pode catalisar muitas rodadas de produção de proteína é mais eficiente para localizar um para localizar mRNA de moléculas individuais de proteínas. MRNAs localizados são tipicamente translationally reprimido até chegarem ao seu destino, o que serve para limitar ainda mais a localização da proteína codificada 2. Além dos muitos casos bem documentados de localização de RNA para controlar a modelagem embrionária, vários estudos têm demonstrado que os mRNAs são localizadospara o local de ação da proteína codificada. Exemplos importantes incluem a localização da β-actina e Arp2 3/3 4 mRNAs para o bordo de ataque da motilidade de fibroblastos e localização dos mRNAs para muitos reguladores de mitose e meiose para fusos mitóticos 5-7.
Muitos dos exemplos clássicos de mRNAs localizados foram identificados através de telas genéticos para mutações efeito materno e posteriormente foram determinados para codificar RNAs localizadas. No entanto, os últimos estudos do genoma começaram a fornecer uma visão mais ampla no âmbito da RNAs localizadas. Um recente na tela de hibridização in situ em embriões de Drosophila que demonstraram ~ 70% de todos os mRNAs têm uma localização específica, incluindo muitos novos destinos 8. A purificação do pseudopodia a partir de fibroblastos de rato identificado um grupo diverso de mRNAs localizada 9. Trabalhe a partir de nosso grupo usando purificação bioquímica dos microtúbulos de meiose Xenopus ovo extrai identificadas centenas de ARNm que copurify com o fuso de 5,7. O nosso trabalho mostrou que a maioria do ARNm de microtúbulos localizadas codificam proteínas que funcionam no controlo da mitose, apoiando a ideia de que os mRNAs estão localizadas no local de acção da proteína codificada. Além disso, a capacidade de detectar o enriquecimento em ARNm de uma fracção subcelular por purificação bioquímica destaca o poder desta abordagem para a identificação de mRNAs localizadas.
RNAs mais localizadas usar o transporte ativo no citoesqueleto, ou actina ou microtúbulos, para conseguir transporte para o seu destino final 10. Para obter uma melhor compreensão da extensão e tipo de RNAs que estão localizadas a destinos específicos utilizando uma abordagem bioquímica, é necessário dispor de um sistema in vitro que podem recapitular processos do citoesqueleto. Um dos sistemas principais para estudar a biologia do citoesqueleto é extractos de ovos produzidosa partir de ovos não fertilizados do sapo Xenopus laevis. X. laevis extratos de ovos têm sido utilizados há décadas para estudar uma grande variedade de processos do citoesqueleto e têm contribuído muito para a nossa compreensão dos mecanismos e moléculas que controlam a montagem do citoesqueleto e dinâmica 11. Além disso, X. laevis extratos de ovos são passíveis de purificação em larga escala de microtúbulos e proteínas associadas 12,13 e existem métodos bem desenhados para a produção de vários tipos de extratos de ovos 14-16. No entanto, para os estudos genómicos existem várias desvantagens para a utilização de X. laevis como um sistema modelo.
Durante décadas sapos Xenopus laevis foram um poderoso sistema para o estudo da biologia do desenvolvimento celular e, devido ao tamanho grande e de desenvolvimento de oócitos externa robusta 17. Além disso, o desenvolvimento de sistemas de extracto de ovos que podem recapitular muitos processe celulars em um tubo de ensaio fez este sapo um modelo experimental poderosa. No entanto, Xenopus laevis tem sido dificultada pela falta de um genoma completo, o qual foi retardado pela natureza alotetraplóide do contraste genome.In, uma espécie estreitamente relacionadas, Xenopus tropicalis, tem um genoma diplóide que foi sequenciado em 2010, 18. Enquanto X. tropicalis não é tratável como experimentalmente como X. laevis 17, a disponibilidade de um genoma sequenciado torna um sistema de modelo atractivo para realizar análises de genoma.
Neste relatório nós descrevemos um método para tornar-II, citostáticos extratos fator de presos meiose (CSF) de X. tropicalis 19. Em seguida, descrevemos um método simples para purificar microtúbulos e RNAs associados a partir deste extrato. Os RNAs pode então ser convertido em bibliotecas passíveis de sequenciação utilizando tecnologias de sequenciação de alto rendimento recentemente desenvolvidos. Uma vez que as bibliotecassão sequenciadas podem ser alinhados com o genoma da rã para identificar mRNAs específicos que são enriquecidas na amostra em relação ao extracto de microtúbulos total. Isto fornece um método poderoso para detectar a localização mRNA microtúbulos-alvo em uma escala de todo o genoma. Além de ser capaz de detectar ARNm localizadas, a utilização de sequenciação de alto rendimento e um genoma sequenciado oferecem a possibilidade de descobrir novos transcritos que não estão presentes na base de dados pública de anotações.
Protocolo
1. Geração de X. tropicalis Ovos
Todos os Xenopus tropicalis rãs são ordenados de OCSAN. Nossos rãs estão alojados num habitats aquáticos de recirculação do sistema de água mantida a 27 ° C. Há muitas opções para os sistemas de água para atendimento de X. tropicalis. Algumas boas informações gerais sobre essa espécie de rã pode ser encontrada nos sites da Harland and Grainger laboratórios ( http://tropicalis.berkeley.edu/home/ , http://www.faculty.virginia.edu/xtropicalis / ). Nossos sapos são mantidas em tankwater que consiste em (0,4 g Ciclid Lake sais, 0,6 g de sal marinho, 0,625 g de NaHCO3 por litro de água, pH 7,0) 20. Esta receita resulta em uma condutividade de 1,800 mS ~, que é uma alta salinidade para X. tropicalis. No entanto, descobrimos que nossos sapos prosperar em this meio ambiente e qualidade de oócitos é melhorada. Receitas tankwater alternativos podem ser encontrados acima nos recursos listados para o general X. cuidado tropicalis.
- Rãs são injectadas com gonadotropina coriónica humana (hCG), em três dias consecutivos para estimular a postura de ovos: Em primeiro lugar, preparar duas concentrações de solução de hCG. Ressuspender 10.000 U de hCG pó liofilizado em 10 ml estéril, desionizada H 2 O para uma concentração final de 1000 U / ml. Em seguida, dilui-se 1 ml de solução de hCG 1000 U / ml em 9 ml de H 2 O para uma concentração final de 100 U / ml. Armazenar ambas as soluções a 4 ° C.
- No dia 1, 4-6 rãs preparar para a postura de ovos pela injeção de hCG entre 02:00-15:00. Injectar cada rã no saco linfático dorsal, perto da cloaca com 0,2 ml de solução de hCG, 100 U / ml. Tendo as rãs rápido durante os subseqüentes duas injeções irá minimizar a quantidade de resíduos sapo presente durante a postura de ovos, mas é opcional.
- No dia 2, injetar os mesmos saposcom 0,2 ml U / ml solução 100 hCG entre 2:00-15:00.
- No dia 3, injecta-se os mesmos sapos com 0,2 ml de solução de hCG 1000 U / ml, entre as 07:00 - 10:00. Configure rãs põem ovos: encher um balde de plástico de 6 quart com tankwater fresco, adicione as rãs e coloque no escuro, a 25 ° C. Após esta injeção, postura começará após 4 horas e será concluída até 7 horas. Frogs deve pôr ovos em um ambiente que é mantida em um mínimo de 25 ° C.
- Faça extrair soluções e ter equipamentos prontos imediatamente antes de coletar ovos.
MMR 20X: 100 mM HEPES, pH 7,8, 2 mM de EDTA pH 7,8, 2 M de NaCl, 40 mM de KCl, 20 mM de MgCl2, 40 mM de CaCl2. Autoclave e armazenar a temperatura ambiente. Prepare 1 L de 1X MMR imediatamente antes de extrair a preparação.
XB 10X: 100 mM de HEPES, pH7.7, 10 mM MgCl2, 1 mM de CaCl2, KCl 1 M, 500 mM de sacarose. Autoclave e armazenar a 4 ° C. Prepare-se 1 L de 1X XB just antes de extrair a preparação. Solução Dejelly: Preparar 250 ml de solução de cisteína 3% em H2O desionizada e o pH a 7,8-8,0 com NaOH 10N. Preparar imediatamente antes da extracção de preparação.
CSF-XB: tomar 200 ml de 1X XB e adicionar 2 ml de 0,5 M EGTA pH 7,7 e 200 mL 1 M MgCl2. Preparar imediatamente antes da extracção de preparação.
CSF-XB +: recolher 50 ml de CSF-XB e adicionar 50 ul de LPC (10 mg / ml de cada unidade de leupeptina, pepstatina, e quimostatina em DMSO). Adicionar 50 ul de citocalasina D (10 mg / ml em DMSO). Preparar imediatamente antes da extracção de preparação.
Prepara-se uma solução de gelatina a 0,2% em H2O desionizada, microondas para dissolver e filtrar esterilizado. Guarde à temperatura ambiente.
Reserve 2 Beckman 2 x ½ tubos ultracentrífuga polegadas.
Preparar dois ml de tubos de centrifugação de fundo redondo de vidro 15 com 0,5 ml de H2O em cada um para amortecer oultracentrífuga tubo.
Fazer fogo polido Pasteur pipetas de vidro. Tire o final off de 5 ¾ polegadas pipetas de vidro para expor uma grande abertura, e exponha ao fogo para suavizar a nova ponteira exposta.
- Prepara-se uma proveta de vidro de 500 ml para a armazenagem dos ovos agitando uma solução de gelatina a 0,2% em torno de revestir as paredes do recipiente. Descartar a solução de gelatina a partir de proveta após o uso.
- Recolhe ovos do balde de plástico utilizado para o assentamento de 6-7 horas após a terceira injecção no dia 3. Se desejar, esprema delicadamente cada sapo uma vez para obter todos os ovos restantes. Lavar uma vez com ovos tankwater fresco e transferir para a proveta de vidro de 500 ml revestido, com solução de gelatina a 0,2%.
2. Preparação do extrato de X. tropicalis Eggs
Todos os passos de preparação de extracto pode ser realizada à temperatura ambiente, aproximadamente 25 ° C. Durante as lavagens, é importante para manter o ovos submersa líquido para que eles permaneçam molhadas. A exposição ao ar pode fazer com que os ovos para escapar a paragem do ciclo celular ou a lise.
- Decantar tankwater tanto quanto possível, enquanto reservando bastante líquido para manter os ovos molhado. Inclinar a proveta contendo ovos para o lado e adicionar 300 mL de 1X ~ MMR lentamente para a parede do recipiente, de modo que a agitação física dos ovos é minimizado. Vamos resolver os ovos, em seguida, decantar o sobrenadante contendo detritos. X. tropicalis ovos são fibroso nesta etapa, para a retirada de ovos activados é feito após dejellying. Repetir para um total de três lavagens de 1X MMR.
- Dejelly os ovos. Decantar fora tanto MMR quanto possível e add metade da solução dejelly. Swirl continuamente por cerca de 5 min. Dissolvendo geléia casacos será visível no sobrenadante após um par de minutos. Decantar e adicionar a solução dejelly restante. Continue a girar continuamente até que os ovos embalar muito bem e todo oriente com seu pólo vegetal (o pólocom pigmento branco) para o fundo do prato. Decantar rapidamente fora tanto dejelly solução possível. Uma vez que os ovos são dejellied eles são muito sensíveis a manipulações mecânicas.
- Adiciona-se cuidadosamente XB aos ovos. Na primeira lavagem XB, remover os ovos que escaparam CSF prisão removendo lisadas, inchado, ovos brancos, e pseudocleavage. Ativado X. ovos tropicalis tendem a estabelecer-se na parte superior central, então use uma pipeta de plástico para retirar estes para fora. Também remover pedaços de pele e resíduos sapo. Lavar os ovos com um total de três vezes, com ~ 300 ml de solução de XB 1X, agitando suavemente ovos entre lavagens e permitindo-lhes assente no fundo da proveta. Como antes, decantar o máximo de cada solução de lavagem quanto possível, mantendo ovos molhado.
- Lave os ovos duas vezes com CSF-XB e decantar.
- Adicionar CSF-XB + para ovos. Usando um fogo-polido gelatina tratado com pipeta Pasteur, transferir os ovos para tubos de centrífuga com Ultra-CSF-XB +, tomando cuidado para não expor o eGGS ao ar. Coloque dentro dos tubos de centrífuga de 15 ml de vidro com a almofada de água.
- Girar ovos numa centrífuga clínica a 200 xg durante 1 min, para aumentar a velocidade de rotação e 800 xg durante 30 seg.
- Utilização de um aspirador para remover tanto tampão quanto possível de ovos. Eles devem estar quase seco no topo. Mover rapidamente para ovos numa centrífuga Sorvall RC-6 centrífuga equipada com um rotor HB-6 (ou equivalente) e girar 17.000 xg durante 15 min a 20 ° C.
- Retirar a camada citoplasmática amarela entre o pigmento e as camadas lipídicas utilizando uma agulha de calibre 18 ligada a uma seringa de 1 ml. Punção do lado do tubo e puxar o tambor de seringa, lentamente para se obter a camada de extracto citoplasmático. Evite grânulos de pigmento, tanto quanto possível.
- Transferir citoplasma para novo tubo ultracentrifugação. É normal que o extrato a aparecer um pouco nublado nesta etapa. Lugar dentro do tubo de centrífuga de 15 ml de vidro com colchão de água. Girar novamente 17.000 xg durante 10 min a 20 ° C. Repita extraction com agulha de calibre 18.
- Transferir citoplasma para um microtubo de 1,5 ml. Estimar o volume de extracto e diluir citocalasina D e LPC 1:1.000 no extrato. Misture bem com uma pipeta de 1 ml ponta, pipetando para cima e para baixo, muitas vezes sem a introdução de bolhas de ar. A produção típica de uma colônia saudável de rã é de aproximadamente 300-500 ml de extrato / sapo. Para preservar a actividade máxima, é necessário armazenar o extracto e executar manipulações experimentais à temperatura ambiente (20-25 ° C).
3. Purificação Taxol estabilizadas Microtubules de X. tropicalis Extrato
- Adicionar taxol a uma alíquota de 100-200 ul de extracto a uma concentração final de 10 uM e incubar à temperatura ambiente durante 30 min. Para as reacções de controlo, o tratamento de uma quantidade equivalente de extracto com a microtúbulos destablilzing droga Nocodazol (10 uM). Reserva a 100 ul de extracto não tratado, para análise.
- Dilui-se a droga tratadaextrai-se com 10 volumes BRB-80 (80 mM de PIPES, pH 6,8 MgCl2 1 mM, EGTA 1 mM) + 30% de glicerol. Montar 14 tubos de polipropileno de fundo redondo ml contendo 10 ml de BRB-80 + 60% de glicerol almofada. Usando uma pipeta de ponta larga furo, a camada de extracto de reacção tratada com drogas suavemente no topo do BRB-80 + 60% de glicerol almofada. Centrifugar durante 10 minutos a 17000 xg a 20 ° C, numa centrífuga Sorvall RC-6 centrífuga equipada com um rotor HB-6 (ou equivalente) e adaptadores de tubos.
- Aspirar o material contendo extracto unsedimented sobrenadante e lavar duas vezes com a interface desionizada H 2 O. Aspirar o volume almofada restante lentamente, tomando cuidado para não perturbar o sedimento tipo gel contendo microtúbulos, proteínas associadas a microtúbulos, e RNAs associada aos microtúbulos na amostra tratada com taxol. A amostra tratada com Nocodazol não contêm material visível. Ressuspender o sedimento em 1 ml de Trizol e prosseguir com as instruções do fabricante para o isolamento de ARN. Untrextracto eated (até 100 ul) pode ser directamente ressuspensas em 1 ml de Trizol.
- Existem agora kits disponíveis comercialmente para a preparação de bibliotecas transcriptoma adequados para o ARN-seq. Estes podem ser adquiridos através http://www.illumina.com/ e http://www.454.com/ .
Resultados
Para identificar X. transcrições tropicalis associados com os microtúbulos, preparamos um extracto citosólico a partir de ovos não fertilizados presos em metáfase da meiose II (CSF). O tratamento do extracto com o taxol permite a formação de microtúbulos estáveis que podem ser purificados por sedimentação através de uma almofada de glicerol (Figura 1A). Análise de Coomassie de gel confirma que os sedimentos α / β-tubulina, de uma forma dependente do tipo taxol, e representa a principal espécie proteica recuperados nestas preparações (Figura 1B). Níveis mais baixos de outras proteínas também estão presentes no pelete de taxol, mas não em preparações tratadas com a droga nocodazol despolimerização de microtúbulos, indicando que as proteínas na fracção de taxol especificamente associado com microtúbulos (MAPS).
Uma Agilent Bioanalyzer é utilizado para examinar a composição geral de ARN em todos os X. fracções de extrato tropicalis (Figura 1C ). Ambas as espécies de tRNA e rRNA estão presentes no extrato CSF eo microtúbulos contendo taxol pellet, consistente com os resultados anteriores que a tradução ocorre em microtúbulos e fusos em X. extrair laevis ovo 5,21. Um traço da linha de projecção gel revela o sinal de ARNm é acentuadamente menor no microtubos contendo taxol pelota, mais notavelmente na região migrando acima de rRNA 28S, indicando que um subconjunto de mRNAs com cosediment microtúbulos em X. tropicalis. RNA isolado deste modo é adequado para as experiências de RNA-seq, utilizando reagentes comercialmente disponíveis.
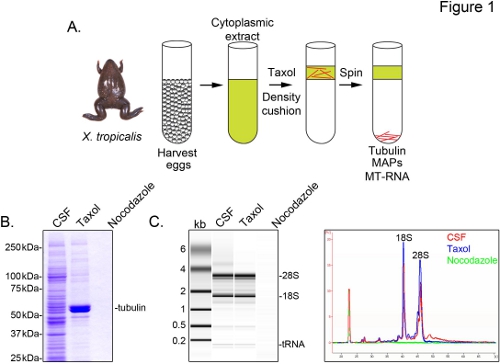
Figura 1. A purificação do MT-RNA para o ARN-seq. (A) Esquema de purificação para isolar o ARN-MT. Os ovos são colhidas a partir fêmea X. trorãs picalis. Após a preparação de um extracto citoplasmático, o taxol é adicionado para induzir a polimerização dos microtúbulos. Os microtúbulos e MT-ARN são purificados por sedimentação através de uma almofada de glicerol. (B) Análise de proteínas isoladas usando o sistema descrito em (A) em gel de Coomassie. Total de CSF em comparação com extracto de proteínas sedimentadas na presença de taxol ou nocodazole. (C) Análise de Bioanalyzer gel de ARN isolado usando o sistema descrito em (A). RNA isolado a partir de CSF em comparação com extrair RNA sedimentadas na presença de taxol ou nocodazole. Tanto a projecção de gel e os traços de linhas são mostradas. Reproduzido com permissão da Sharp, et al., (2011). Clique aqui para ver a imagem ampliada .
Discussão
Neste relatório nós descrevemos um método simples para a produção de extratos de ovos CSF-presos de X. tropicalis, 19, e utilizar este extracto para estudar RNAs microtúbulos associados 7. O procedimento básico para a produção de extratos de ovos CSF-presos de X. tropicalis, é o mesmo que o utilizado para X. laevis com algumas diferenças fundamentais. Um dos aspectos mais difíceis de trabalhar com X. tropicalis sapos é a obtenção de ovos de alta qualidade o suficiente para fazer um extrato com a nucleação de microtúbulos ou atividade de montagem do fuso comparável ao X. extrai laevis ovo. Para alcançar melhores condições de postura de ovos, evitando o deslizamento de meiose II parada do ciclo celular, o intervalo entre as injeções de hormônio para X. tropicalis, é mais curto do que o utilizado para X. laevis, e a temporização com a terceira injecção de hCG para o início da postura de ovos é também muito menor. Com X. laevis o momento da injecção de hCG tele início da postura de ovos é tal que é conveniente e eficiente para os ovos a serem definidos durante a noite em buffer. No entanto, devido ao curto tempo entre a injecção de hCG e a postura de ovos com X. Tropicalis é frequentemente necessário para expressar manualmente os ovos de rãs. Outra diferença significativa entre fazer extrato de ovo dos dois sapos diferentes é o passo dejellying. Com X. laevis os ovos são tão grandes que é fácil de determinar quando o casaco geléia tenha dissolvido por observar de perto como os ovos são espaçados no copo. Como a reação dejellying começa, os ovos começam a se arrumar mais densamente. No entanto, X. tropicalis ovos são muito menores e que pode ser bastante difícil de determinar quando o revestimento de gelatina é dissolvido pela ovo densidade de empacotamento sozinho. Verificou-se que o método mais fiável para determinar quando o revestimento de gelatina é dissolvida é monitorizar a orientação do animal (preto) e vegetais (branco) pólos. Quando todo o vegetal pólos Orient para a parte inferior do recipiente o revestimento geleia foi removida suficiente para prosseguir com o extracto. Finalmente, enquanto X. extracto de ovo laevis podem ser armazenados a temperaturas baixas (4-12 ° C), observou-se que é crítico para manter X. extracto de ovo tropicalis, à temperatura ambiente (20-25 ° C) durante a preparação e manipulações experimentais para preservar a actividade bioquímica. Por causa das diferenças na facilidade de utilização é preferível utilizar X. laevis rãs para a produção de extrato de ovo. No entanto, para as experiências que requerem ou são facilitadas por um organismo com um genoma sequenciado, X. tropicalis é um excelente sistema alternativo.
O método que descrevemos neste relatório utiliza taxol como um agente de estabilização dos microtúbulos para induzir a polimerização dos microtúbulos. Nós escolhemos este método porque o taxol é um agente de estabilização de microtúbulos robusto que facilita o isolamento em larga escala de microtúbulos purificada. O método them que descrevemos poderia provavelmente ser melhorada por meio da comparação das proteínas e RNAs microtúbulos associados, utilizando métodos alternativos de polimerização de microtúbulos. Alternativas poderiam incluir polimerização usando polimerização GTP induzida (técnica clássica), 22 ou usando Ran-GTP como polymerizer microtúbulos para imitar os microtúbulos induzidas pelo conjunto do eixo da cromatina-driven 23. Finalmente, a utilização de núcleos de esperma purificado para induzir a polimerização dos microtúbulos seria o mais próximo imitar aos tipos de microtúbulos, que são nucleados durante a mitose (centrossoma, a cromatina, e cinetocoro mediada). Inconvenientes para estas fontes alternativas de nucleação de microtúbulos são os agentes de nucleação que não são tão facilmente disponíveis como o taxol e eles não nucleada ou estabilizar microtúbulos tão eficientemente como o taxol. Assim, cada um destes métodos seria mais difícil de utilizar para purificações em grande escala. A vantagem de se comparar vários tipos diferentes de nucleadores de microtúbulosé que poderia ser possível identificar as proteínas e / ou RNAs que são específicos para cada via de nucleação de microtúbulos.
O método que descrevemos aqui aproveita extractos citoplasmáticos de anfíbios. No entanto, esta abordagem poderia ser estendida para o uso do sistema de extracto de outros organismos. Extractos mitóticas foram descritos a partir de células de cultura de tecidos humanos sincronizados 24 que fielmente recapitular muitos aspectos da montagem de microtúbulos. Nós utilizamos com sucesso estes extractos identificar ARN associada a microtúbulos a partir de células HeLa 5. Similar esquemas de purificação de microtúbulos têm sido descritas por muitos organismos diferentes 25,26, embora os RNAs dos microtúbulos associados ainda não foram examinados. A abordagem aqui descrita pode ser usada com qualquer organismo que pode produzir um extracto citoplasmático concentrada capaz de nucleação microtúbulos.
Finalmente, embora a abordagem que deescriba discute aqui a purificação de microtúbulos e proteínas e RNAs associados, esta abordagem pode ser generalizada a outras estruturas subcelulares. Enquanto mRNAs mais localizadas não tenham sido identificados utilizando os métodos bioquímicos recentes avanços em tecnologias de sequenciação de DNA e RNA que esta abordagem seja um método atraente para identificar ARN localizadas. Nesta abordagem, qualquer estrutura subcelular ou sub-embrião de interesse pode ser isolado ou purificado. Em seguida, as proteínas e RNAs associados podem ser identificados em grande escala do genoma. RNAs pode então ser comparado com o teor de ANR das células totais ou embrião para identificar ARN localizadas enriquecidos. Esta abordagem poderia ser usada com os ovos inteiros (animal e vegetal separação, semelhante à abordagem que identificou os primeiros RNAs localizadas em Xenopus 27), actina RNAs associados, RNAs ER-associados, RNAs mitocôndrias associadas, ou a qualquer estrutura subcelular que pode ser purificado com RNAs associados intactas. Baseadonosso trabalho em RNA associada aos microtúbulos podemos prever que este seria um excelente método para descobrir novas proteínas que funcionam em um determinado local. Além disso, a identificação da localização e extensão de todos os RNAs localizadas irá fornecer insights sobre como as células e embriões usar localização mRNA para controlar a expressão de genes.
Divulgações
Não há conflitos de interesse declarados.
Materiais
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Xenopus tropicalis | NASCO | LM00823MX | |
| human Chorionic Gonadotropin | Sigma-Aldrich | CG10 | |
| HEPES | Sigma-Aldrich | H4034 | |
| EDTA | Sigma-Aldrich | E5134 | |
| NaCl | Sigma-Aldrich | S3014 | |
| KCl | Sigma-Aldrich | P9541 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| CaCl2 | Sigma-Aldrich | C8106 | |
| sucrose | Sigma-Aldrich | S0389 | |
| NaOH | Sigma-Aldrich | S5881 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Leupeptin | Sigma-Aldrich | L9783 | |
| Pepstatin | Sigma-Aldrich | P5318 | |
| Chymostatin | Sigma-Aldrich | C7268 | |
| Cytochalasin D | Sigma-Aldrich | C8273 | |
| Gelatin, porcine skin | Sigma-Aldrich | G1890 | |
| PIPES | Sigma-Aldrich | P6757 | |
| Taxol | Sigma-Aldrich | T7191 | |
| Nocodazole | Sigma-Aldrich | M1404 | |
| Trizol | Invitrogen | 15596-026 | |
| L-Cysteine, free base | USB Corporation | 14030 | |
| Cichlid Lake Salt | Seachem | 47894 | |
| Marine salt | Seachem | SC7111 | |
| NaHCO3 | Sigma-Aldrich | S6014 | |
| EQUIPMENT | |||
| 1 ml syringes | BD Biosciences | 309659 | |
| 18 gauge needles | BD Biosciences | 305195 | |
| 30 gauge needles | BD Biosciences | 305106 | |
| Rubbermaid Plastic bucket | Amazon | 6306 | |
| Beckman Polyallomer 2 x ½ inch Ultracentrifuge tubes | Beckman | 326819 | |
| 15 ml round-bottomed glass centrifuge tubes | Fisher Scientific | 45500-15 | |
| Rubber adapter sleeves for 15 ml tubes | Kimble-Chase | 45550-15 | |
| 5 ¾ inch glass Pasteur pipettes | Fisher Scientific | 13-678-20A | |
| 14 ml polypropylene round-bottom tube | BD Biosciences | 352059 | |
| Sorvall HB-6 rotor | Thermo Scientific | 11860 | |
| Sorvall RC-6 centrifuge | Thermo Scientific | 46910 | |
Referências
- Martin, K. C., Ephrussi, A. mRNA localization: gene expression in the spatial dimension. Cell. 136, 719-730 (2009).
- Besse, F., Ephrussi, A. Translational control of localized mRNAs: restricting protein synthesis in space and time. Nat. Rev. Mol. Cell Biol. 9, 971-980 (2008).
- Lawrence, J. B., Singer, R. H. Intracellular localization of messenger RNAs for cytoskeletal proteins. Cell. 45, 407-415 (1986).
- Mingle, L. A., et al. Localization of all seven messenger RNAs for the actin-polymerization nucleator Arp2/3 complex in the protrusions of fibroblasts. J. Cell Sci. 118, 2425-2433 (2005).
- Blower, M. D., Feric, E., Weis, K., Heald, R. Genome-wide analysis demonstrates conserved localization of messenger RNAs to mitotic microtubules. J. Cell Biol. 179, 1365-1373 (2007).
- Eliscovich, C., Peset, I., Vernos, I., Mendez, R. Spindle-localized CPE-mediated translation controls meiotic chromosome segregation. Nat. Cell Biol. 10, 858-865 (2008).
- Sharp, J. A., Plant, J. J., Ohsumi, T. K., Borowsky, M., Blower, M. D. Functional analysis of the microtubule-interacting transcriptome. Mol. Biol Cell. 22, 4312-4323 (2011).
- Lecuyer, E., et al. Global analysis of mRNA localization reveals a prominent role in organizing cellular architecture and function. Cell. 131, 174-187 (2007).
- Mili, S., Moissoglu, K., Macara, I. G. Genome-wide screen reveals APC-associated RNAs enriched in cell protrusions. Nature. 453, 115-119 (2008).
- Holt, C. E., Bullock, S. L. Subcellular mRNA localization in animal cells and why it matters. Science. 326, 1212-1216 (2009).
- Desai, A., Murray, A., Mitchison, T. J., Walczak, C. E. The use of Xenopus egg extracts to study mitotic spindle assembly and function in vitro. Methods Cell Biol. 61, 385-412 (1999).
- Gache, V., Waridel, P., Luche, S., Shevchenko, A., Popov, A. V. Purification and mass spectrometry identification of microtubule-binding proteins from Xenopus egg extracts. Methods Mol. Med. 137, 29-43 (2007).
- Gache, V., et al. Xenopus meiotic microtubule-associated interactome. PLoS One. 5, e9248 (2010).
- Cross, M. K., Powers, M. Obtaining eggs from Xenopus laevis females. J. Vis. Exp. (18), e890 (2008).
- Cross, M. K., Powers, M. Preparation and fractionation of Xenopus laevis egg extracts. J. Vis. Exp. (18), e891 (2008).
- Cross, M., Powers, M. In vitro nuclear assembly using fractionated Xenopus egg extracts. J. Vis. Exp. (19), e908 (2008).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends Genet. 27, 507-515 (2011).
- Hellsten, U. The genome of the Western clawed frog Xenopus tropicalis. Science. 328, 633-636 (2010).
- Brown, K. S. Xenopus tropicalis egg extracts provide insight into scaling of the mitotic spindle. J. Cell Biol. 176, 765-770 (2007).
- Godfrey, E. W., Sanders, G. E. Effect of water hardness on oocyte quality and embryo development in the African clawed frog (Xenopus laevis). Comp. Med. 54, 170-175 (2004).
- Groisman, I., et al. CPEB, maskin, and cyclin B1 mRNA at the mitotic apparatus: implications for local translational control of cell division. Cell. 103, 435-447 (2000).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38, 29-34 (2006).
- Kalab, P., Pu, R. T., Dasso, M. The ran GTPase regulates mitotic spindle assembly. Curr. Biol. 9, 481-484 (1999).
- Gaglio, T., Saredi, A., Compton, D. A. NuMA is required for the organization of microtubules into aster-like mitotic arrays. J. Cell Biol. 131, 693-708 (1995).
- Hughes, J. R., et al. A microtubule interactome: complexes with roles in cell cycle and mitosis. PLoS Biol. 6, e98 (2008).
- Suprenant, K. A., Tempero, L. B., Hammer, L. E. Association of ribosomes with in vitro assembled microtubules. Cell Motil. Cytoskeleton. 14, 401-415 (1002).
- Rebagliati, M. R., Weeks, D. L., Harvey, R. P., Melton, D. A. Identification and cloning of localized maternal RNAs from Xenopus eggs. Cell. 42, 769-777 (1985).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados