Method Article
Produzione di
In questo articolo
Riepilogo
Descriviamo la raccolta di non fecondato Xenopus tropicalis Uova e la produzione di un estratto uovo II-arrestato meiosi. Questo estratto uovo può essere usata per purificare microtubuli e RNA associata ai microtubuli.
Abstract
Molti organismi localizzano mRNA a specifiche destinazioni subcellulari per l'espressione del gene di controllo spazialmente e temporalmente. Recenti studi hanno dimostrato che la maggioranza del trascrittoma è localizzato in una posizione non casuale nelle cellule ed embrioni. Un approccio per identificare mRNA localizzate è purificare biochimicamente una struttura cellulare di interesse e per identificare tutte le trascrizioni associati. Utilizzando le tecnologie high-throughput sequencing sviluppati di recente, è ora semplice per identificare tutti gli RNA associati a una struttura subcellulare. Per facilitare l'identificazione trascrizione è necessario lavorare con un organismo con un genoma completamente sequenziato. Un sistema interessante per la purificazione biochimica delle strutture cellulari sono estratti di uova prodotte dalla rana Xenopus laevis. Tuttavia, X. laevis attualmente non ha un genoma completamente sequenziato, che ostacola l'identificazione trascrizione. In questo articolo si descrive un metodoper la produzione di estratti di uova da una rana correlato, X. tropicalis, che ha un genoma completamente sequenziato. Noi forniamo i dettagli per la polimerizzazione dei microtubuli, purificazione e isolamento trascrizione. Mentre questo articolo viene descritto un metodo specifico per l'identificazione di trascritti associata ai microtubuli, riteniamo che possa essere facilmente applicato ad altre strutture subcellulari e fornirà un potente metodo per l'identificazione di RNA localizzate.
Introduzione
Controllo spaziale e temporale dell'espressione genica è importante per tutte le cellule, ed è particolarmente importante per il controllo dei primi embrionali picchiettare 1. Controllo spaziale di espressione genica è ottenuta attraverso la localizzazione attiva di mRNA per le destinazioni specifiche all'interno di cellule o embrioni. In molti tipi di cellule molto grandi, (ad es ovociti, embrioni e neuroni) localizzazione mRNA viene utilizzato per limitare l'espressione della proteina al sito di azione della proteina codificata. Dal momento che un mRNA localizzata può catalizzare molti cicli di produzione di proteine è più efficiente di localizzare un mRNA che di localizzare molecole proteiche individuali. MRNA localizzati sono in genere translationally repressi fino a raggiungere la loro destinazione, che serve a limitare ulteriormente la localizzazione della proteina codificata 2. Oltre ai numerosi casi ben documentati di localizzazione di RNA per controllare patterning embrionale, diversi studi hanno documentato mRNA che sono localizzatial sito di azione della proteina codificata. Esempi importanti includono localizzazione della β-actina e Arp2 3/3 4 mRNA per il bordo di fibroblasti motili e localizzazione degli mRNA per molti regolatori mitotici a meiotica e mitotica mandrini 5-7.
Molti degli esempi classici di mRNA localizzati sono stati identificati attraverso schermi genetici per le mutazioni effetto materno e successivamente sono stati determinati a codificare RNA localizzate. Tuttavia, recenti studi di genome-wide hanno cominciato a fornire una conoscenza più ampia nel campo di applicazione RNA localizzate. Un recente nella schermata di ibridazione in situ in embrioni di Drosophila ha dimostrato che circa il 70% di tutti gli mRNA hanno una localizzazione specifica, tra cui molte nuove destinazioni 8. Purificazione di pseudopodi da fibroblasti di topo ha identificato un gruppo eterogeneo di mRNA localizzato 9. Lavoro da nostro gruppo utilizzando purificazione biochimica dei microtubuli da meiotica Xenopus uovo estrae identificate centinaia di mRNA che copurify con il mandrino 5,7. Il nostro lavoro ha dimostrato che la maggioranza dei microtubuli localizzate mRNA codificano proteine che funzionano nel controllo della mitosi, supportando l'idea che mRNA sono localizzati al sito di azione della proteina codificata. Inoltre, la capacità di rilevare mRNA arricchimento in una frazione subcellulare da purificazione biochimica evidenzia la potenza di questo approccio per la identificazione di mRNA localizzate.
RNA più localizzate utilizzano trasporto attivo sul citoscheletro, sia actina o microtubuli, per raggiungere il trasporto verso la destinazione finale 10. Per ottenere una migliore comprensione della portata e tipi di RNA che sono localizzati a destinazioni specifiche utilizzando un approccio biochimico è necessario disporre di un sistema in vitro che possono ricapitolare processi citoscheletriche. Uno dei sistemi di primo ministro per lo studio della biologia del citoscheletro è estratti di uova prodotteda uova non fecondate dalla rana Xenopus laevis. X. estratti di uova laevis sono stati utilizzati per decenni per studiare una vasta gamma di processi citoscheletro e hanno contribuito molto alla nostra comprensione dei meccanismi e le molecole che controllano assemblaggio del citoscheletro e la dinamica 11. Inoltre, X. estratti uovo laevis sono suscettibili di purificazioni larga scala di microtubuli e proteine associate 12,13 e ci sono metodi ben progettato per la produzione di vari tipi di estratti uovo 14-16. Tuttavia, per gli studi genomici ci sono molti inconvenienti all'utilizzo di X. laevis come sistema modello.
Per decenni rane Xenopus laevis sono stati un potente sistema per lo studio della biologia dello sviluppo e delle cellule, a causa delle grandi dimensioni dell'oocita e robusto sviluppo esterno 17. Inoltre, lo sviluppo di sistemi di estratto di uova che possono ricapitolare molti processe cellulares in una provetta ha reso questa rana un modello sperimentale potente. Tuttavia, Xenopus laevis è stata ostacolata dalla mancanza di una sequenza completa del genoma, che è stato rallentato dalla natura allotetraploide del contrasto genome.In, una specie simile, Xenopus tropicalis, ha un genoma diploide che è stato sequenziato nel 2010 18. Mentre X. tropicalis non è sperimentalmente trattabile come X. laevis 17 la disponibilità di un genoma sequenziato rende un sistema modello attraente per eseguire analisi a livello del genoma.
In questo rapporto si descrive un metodo per rendere meiosi II-, citostatici estratti fattore-arrestati (CSF) da X. tropicalis 19. Descriviamo poi un metodo semplice per purificare microtubuli e RNA associati di questo estratto. Gli RNA possono poi essere convertiti in librerie suscettibili di sequenziamento utilizzando tecnologie di sequenziamento recentemente sviluppati ad alto rendimento. Una volta che le librerievengono sequenziati possono essere allineati al genoma della rana per identificare specifici mRNA che si arricchiscono nel campione rispetto al microtubulo estratto totale. Questo fornisce un potente metodo per rilevare microtubuli mirata localizzazione mRNA su un genoma scala. Oltre ad essere in grado di rilevare mRNA localizzate, l'uso di sequenziamento ad alta produttività e un genoma sequenziato offrono la possibilità di scoprire nuovi trascritti che non sono attualmente presenti nelle annotazioni database pubblico.
Protocollo
1. Generazione di X. tropicalis Uova
Tutti Xenopus tropicalis rane sono ordinate da NASCO. Le nostre rane sono alloggiati in un habitat acquatici sistema di ricircolo dell'acqua mantenuta a 27 ° C. Ci sono molte opzioni per i sistemi di acqua per la cura di X. tropicalis. Qualche buona informazione generale su questa specie di rana si possono trovare sui siti web dei laboratori di Harland e Grainger ( http://tropicalis.berkeley.edu/home/ , http://www.faculty.virginia.edu/xtropicalis / ). Le nostre rane sono mantenuti in tankwater costituito da (0.4 g Ciclid Lago di sali, 0,6 g di sale marino, 0.625 g di NaHCO3 per litro d'acqua, pH 7.0) 20. Ciò si traduce in una ricetta conduttività di ~ 1800 mS, che è una salinità elevata per X. tropicalis. Tuttavia, abbiamo scoperto che le nostre rane prosperano in this ambiente e la qualità degli ovociti è migliorata. Ricette tankwater alternativi si trovano sopra le risorse elencate per il generale X. cura tropicalis.
- Rane vengono iniettati con gonadotropina corionica umana (hCG) per tre giorni consecutivi per stimolare la deposizione delle uova: in primo luogo, preparare due concentrazioni di soluzione di hCG. Risospendere 10.000 U di polvere liofilizzata hCG in 10 ml sterile, deionizzata H 2 O per una concentrazione finale di 1.000 U / ml. Poi, diluire 1 ml di soluzione di hCG 1.000 U / ml in 9 ml di H 2 O per una concentrazione finale di 100 U / ml. Conservare entrambe le soluzioni a 4 ° C.
- Il giorno 1, preparare 4-6 rane per la deposizione delle uova, iniettando con hCG tra le 2:00-03:00. Iniettare ciascuna rana nella dorsale linfa cieco, vicino alla cloaca con 0,2 ml di soluzione di hCG 100 U / ml. Avere le rane velocemente durante le successive due iniezioni sarà ridurre al minimo la quantità di rifiuti presenti rana durante la deposizione delle uova, ma è facoltativo.
- Il giorno 2, iniettare le stesse ranecon 0,2 ml U / ml soluzione 100 hCG tra 2:00-03:00.
- Il giorno 3, iniettare le stesse rane con 0,2 ml di soluzione di hCG 1.000 U / ml, tra le 7:00-10:00. Impostare le rane a deporre le uova: riempire un secchio di plastica 6 quarti con tankwater fresco, aggiungere le rane e posto al buio a 25 ° C. Dopo questa iniezione, deposizione delle uova inizierà dopo 4 ore e sarà completata entro 7 ore. Le rane devono deporre le uova in un ambiente che è mantenuto a un minimo di 25 ° C.
- Fai estrarre soluzioni e disporre di attrezzature pronte immediatamente prima di raccogliere le uova.
20X MMR: HEPES 100 mM, pH 7.8, 2 mM EDTA pH 7.8; 2 M NaCl, 40 mM KCl, 20 mM MgCl2, 40 mM CaCl 2. Autoclave e conservare a temperatura ambiente. Preparare 1 L di 1X MMR appena prima di estrarre preparazione.
XB 10X: 100 HEPES, pH7.7; 10 mM MgCl2, 1 mM CaCl 2, 1 M KCl; 500 mM saccarosio. Autoclave e conservare a 4 ° C. Preparare 1 l di 1x XB just prima di estrarre preparazione. Soluzione Dejelly: preparare 250 ml di soluzione di cisteina 3% in H 2 O deionizzata e pH a 7,8-8,0 con NaOH 10 N. Preparare appena prima estrarre preparazione.
CSF-XB: prendere 200 ml di 1X XB e aggiungere 2 ml di 0,5 M dell'EGTA pH 7.7 e 200 ml 1 M MgCl2. Preparare appena prima estrarre preparazione.
CSF-XB +: prendere 50 ml di CSF-XB e aggiungere 50 ml di LPC (10 mg / ml di ogni stock di leupeptina, Pepstatin e Chymostatin in DMSO). Aggiungere 50 microlitri Citocalasina D (10 mg / ml in DMSO). Preparare appena prima estrarre preparazione.
Preparare una soluzione di gelatina 0,2% in acqua deionizzata H 2 O, forno a microonde per sciogliere e filtrare sterilizzare. Conservare a temperatura ambiente.
Prenotazione 2 Beckman 2 x ½ tubi ultracentrifuga pollici.
Preparare due provette da 15 ml a fondo rotondo in vetro con 0,5 ml di H 2 O in ciascuna per attutire l'ultracentrifuga tubo.
Fai Sfaccettate in vetro pipette Pasteur. Far scattare la fine off del 5 ¾ pipette di vetro di pollice per esporre un'ampia apertura, ed esporre alla fiamma per lisciare il nuovo puntale a vista.
- Preparare un bicchiere di vetro da 500 ml per memorizzare le uova agitando una soluzione di gelatina 0,2% circa per rivestire le pareti del becher. Eliminare la soluzione di gelatina da bicchiere dopo l'uso.
- Raccogliere le uova dal secchio di plastica utilizzato per la posa di 6-7 ore dopo la terza iniezione al giorno 3. Se lo si desidera, premere delicatamente ogni rana una volta per ottenere tutte le uova restanti. Lavare le uova una volta con tankwater fresco e trasferimento in bicchiere da 500 ml in vetro rivestito con soluzione di gelatina 0,2%.
2. Preparazione di estratto da X. tropicalis Uova
Tutte le fasi di preparazione dell'estratto possono essere effettuate a temperatura ambiente di circa 25 ° C. Durante i lavaggi, è importante mantenere l'uovos sommersa sotto liquido in modo che rimangano bagnati. Esposizione all'aria può provocare le uova per sfuggire arresto del ciclo cellulare o lisi.
- Decantare il più tankwater possibile riservandosi abbastanza liquido per mantenere le uova bagnato. Inclinare il bicchiere contenente uova di lato e aggiungere ~ 300 ml 1X MMR lentamente alla parete del bicchiere, in modo che l'agitazione fisica delle uova è minimizzato. Lasciate che depositano le uova, poi decantare il surnatante contenente detriti. X. uova tropicalis sono filata in questa fase, in modo in cui le uova attivati avviene dopo dejellying. Ripetere per un totale di tre lavaggi 1X MMR.
- Dejelly le uova. Decantare il più MMR possibile e aggiungere metà della soluzione dejelly. Swirl continuamente per circa 5 min. Sciogliendo cappotti gelatina sarà visibile nel surnatante dopo un paio di minuti. Decantare ed aggiungere la soluzione dejelly rimanente. Continuare a girare continuamente fino a quando le uova imballare molto strettamente e tutto orientare con il loro polo vegetale (il polocon pigmento bianco) verso il fondo del piatto. Decantare rapidamente fuori come soluzione molto dejelly possibile. Una volta che le uova sono dejellied sono molto sensibili alle manipolazioni meccaniche.
- Aggiungere con cautela XB alle uova. Nel primo lavaggio XB, togliere le uova che sono sfuggiti CSF arresto rimuovendo lisati, gonfi, uova bianche, e pseudocleavage. Activated X. uova tropicalis tendono a stabilirsi in alto al centro, in modo da utilizzare una pipetta di plastica trasferimento a tirare questi fuori. Rimuovere anche pezzi di pelle e rifiuti rana. Lavare le uova per un totale di tre volte con ~ 300 ml di soluzione 1X XB, rimestando delicatamente le uova tra un lavaggio e permettendo loro di stabilirsi sul fondo del bicchiere. Come prima, decantare quanto più di ogni soluzione di lavaggio possibile, mantenendo le uova bagnato.
- Lavare le uova due volte con CSF-XB e decantare.
- Aggiungi CSF-XB + alle uova. Utilizzando un incendio lucidato gelatina-trattato pipetta Pasteur, uova a tubi ultra-centrifuga di trasferimento con CSF-XB +, facendo attenzione a non esporre l'eGGS nell'aria. Posto all'interno delle provette da centrifuga 15 ml di vetro con il cuscino d'acqua.
- Spin uova in una centrifuga clinica a 200 xg per 1 minuto, aumentare la velocità a 800 xg e girare per 30 sec.
- Utilizzare un aspiratore per rimuovere il più possibile dal tampone uova. Dovrebbero essere quasi asciutti in cima. Spostare rapidamente le uova in una Sorvall RC-6 centrifuga dotata di un rotore HB-6 (o equivalente) e spin 17.000 xg per 15 min a 20 ° C.
- Rimuovere lo strato citoplasmatico giallo tra pigmento e strati lipidici usando un ago di calibro 18 attaccato ad una siringa da 1 ml. Forare il lato del tubo e tirare il corpo della siringa lentamente per ottenere lo strato estratto citoplasmatico. Evitare di granuli di pigmento, per quanto possibile.
- Trasferimento citoplasma al nuovo tubo ultracentrifugazione. È normale che l'estratto di apparire leggermente nuvoloso in questa fase. Posto all'interno del tubo di vetro da 15 ml per centrifuga con cuscino d'acqua. Gira ancora 17.000 xg per 10 min a 20 ° C. Ripetere extraction con 18 gauge.
- Trasferire citoplasma di una provetta da microcentrifuga 1,5. Stimare il volume estratto e diluire Citocalasina D e LPC 1:1.000 in estratto. Mescolare bene con una punta di pipetta 1 ml, pipettare su e giù molte volte senza introduzione di bolle d'aria. Una resa tipica di una colonia rana sano è di circa 300-500 ml di estratto / rana. Per conservare attività massima, è necessario immagazzinare l'estratto ed eseguire manipolazioni sperimentali a temperatura ambiente (20-25 ° C).
3. Purificazione Taxol stabilizzato microtubuli da X. tropicalis estratto
- Aggiungere a una Taxol 100-200 microlitri aliquota dell'estratto ad una concentrazione finale di 10 pM e incubare a temperatura ambiente per 30 min. Per reazioni di controllo, trattare un volume equivalente di estratto con la microtubuli destablilzing Nocodazole farmaco (10 mM). Riserva 100 pl di estratto non trattato per l'analisi.
- Diluire il farmaco-trattatoestrarre con 10 volumi BRB-80 (80 mm TUBI pH 6.8, 1 mM MgCl2, 1 mM EGTA) + 30% di glicerolo. Assemblare 14 tubi in polipropilene a fondo rotondo ml contenenti 10 ml di BRB-80 + 60% cuscino glicerolo. Utilizzando un'ampia foro punta di pipetta, la reazione strato estratto farmaco-trattato delicatamente sulla sommità della BRB-80 + 60% glicerolo cuscino. Centrifugare per 10 minuti a 17.000 xg a 20 ° C in una centrifuga Sorvall RC-6 centrifuga dotata di un rotore HB-6 (o equivalente) e adattatori per provette.
- Aspirare il surnatante contenente unsedimented materiale estratto, e lavare l'interfaccia due volte con acqua deionizzata H 2 O. Aspirare il volume del cuscino restante lentamente, facendo attenzione a non disturbare il pellet simile a gel contenente microtubuli, proteine associate ai microtubuli, e RNA associata ai microtubuli nel campione Taxol-trattato. Il campione Nocodazole-trattato non contiene materiale visibile. Risospendere il pellet in 1 ml TRIzol e procedere con le istruzioni del produttore per isolare l'RNA. UntrEstratto eated (fino a 100 microlitri) può essere direttamente risospeso in 1 ml TRIzol.
- Ora ci sono i kit disponibili in commercio per la preparazione di librerie di trascrittoma adatti per l'RNA-Seq. Questi possono essere acquistati attraverso http://www.illumina.com/ e http://www.454.com/ .
Risultati
Per identificare X. trascrizioni tropicalis associati microtubuli, prepariamo un estratto citosolico da uova non fecondate arrestati in metafase della meiosi II (CSF). Trattamento di questo estratto con taxolo permette la formazione di microtubuli stabili che possono essere purificati mediante sedimentazione attraverso un cuscino di glicerolo (Figura 1A). Coomassie analisi gel conferma che i sedimenti α / β-tubulina in maniera taxolo-dipendente, e rappresenta le principali specie proteiche recuperati in queste preparazioni (Figura 1B). Bassi livelli di altre proteine sono presenti anche nella taxolo pellet, ma non in preparazioni trattate con farmaco depolimerizzazione dei microtubuli nocodazole, indicando che le proteine della frazione taxolo specificamente associato con i microtubuli (MAP).
Un Agilent Bioanalyzer viene utilizzato per esaminare composizione generale RNA in tutto X. tropicalis estratto frazioni (Figura 1C ). Sia rRNA e tRNA specie sono presenti in estratto di CSF e il microtubulo contenente taxolo pellet, in linea con i risultati precedenti che di traduzione si verifica sui microtubuli e mandrini in X. laevis uovo estratto di 5,21. Una traccia linea della proiezione gel rivela il segnale di mRNA è marcatamente inferiore nel microtubuli contenente taxolo pellet, in particolare nella regione di migrazione sopra 28S rRNA, indicando che un sottoinsieme di mRNA cosediment con microtubuli in X. tropicalis. RNA isolato in questo modo è adatto per esperimenti di RNA-seq utilizzano reagenti disponibili in commercio.
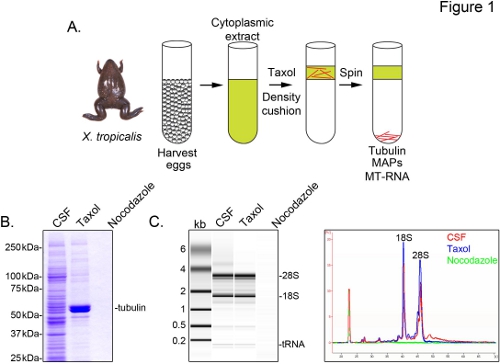
Figura 1. Purificazione di MT-RNA per la RNA-Seq. (A) schema di purificazione per isolare MT-RNA. Le uova vengono raccolte da femmina X. trorane picalis. Dopo la preparazione di un estratto citoplasmatico, taxolo viene aggiunto per indurre polimerizzazione dei microtubuli. Microtubuli e MT-RNA sono purificati mediante sedimentazione attraverso un cuscino di glicerolo. (B) Analisi Coomassie del gel di proteine isolate utilizzando lo schema descritto in (A). CSF totale estratto rispetto alle proteine sedimentate in presenza di taxolo o nocodazole. (C) Bioanalizzatore analisi del gel di RNA isolato utilizzando lo schema descritto in (A). RNA isolato da CSF estratto rispetto a RNA sedimentato in presenza di taxolo o nocodazole. Sia la proiezione gel e le tracce di linea sono mostrati. Ristampato con il permesso di Sharp, et al., (2011). Clicca qui per vedere l'immagine ingrandita .
Discussione
In questo rapporto abbiamo descritto un metodo semplice per la produzione di estratti di uova CSF-arrestati da X. tropicalis 19 e utilizzare questo estratto per studiare RNA associata ai microtubuli 7. La procedura di base per la produzione di estratti di uova CSF-arrestati da X. tropicalis è lo stesso utilizzato per X. laevis con alcune differenze chiave. Uno degli aspetti più difficili da lavorare con X. tropicalis rane è ottenere abbastanza uova di alta qualità per fare un estratto con la nucleazione dei microtubuli o attività di assemblaggio del mandrino paragonabile a X. laevis uovo estrae. Per ottenere i migliori condizioni di posa delle uova, evitando lo scivolamento dalla meiosi II arresto del ciclo cellulare, l'intervallo tra le iniezioni di ormoni per X. tropicalis è più breve di quello utilizzato per X. laevis, e la temporizzazione della terza iniezione hCG per l'inizio di deposizione è anche molto più breve. Con X. laevis la temporizzazione da quella di hCG per tha inizio ovodeposizione è tale che è conveniente ed efficiente per le uova stabilite durante la notte in tampone. Tuttavia, a causa del tempo breve tra di hCG e deposizione delle uova con X. tropicalis è spesso necessario per esprimere manualmente le uova da rane. Un'altra differenza significativa tra fare estratto uovo da due diverse rane è il passo dejellying. Con X. laevis le uova sono così grandi che è facile stabilire quando il cappotto gelatina è sciolto osservando come da vicino le uova sono distanziati nel bicchiere. Poiché la reazione dejellying inizio, le uova iniziano a confezionare più densamente. Tuttavia, X. uova tropicalis sono molto più piccoli e può essere molto difficile determinare quando il cappotto gelatina si è sciolta da uovo imballaggio densità solo. Abbiamo trovato che il metodo più affidabile per determinare quando il cappotto gelatina è dissolta è monitorare l'orientamento dell'animale (nero) e vegetali (bianco) poli. Quando tutto il vegetale poli orient verso il fondo del beaker del mantello gelatina è stato rimosso abbastanza per procedere con l'estratto. Infine, mentre X. estratto uovo laevis può essere conservato a temperature fresche (4-12 ° C), abbiamo osservato che è fondamentale mantenere X. estratto uovo tropicalis a temperatura ambiente (20-25 ° C) durante la preparazione e manipolazioni sperimentali preservare attività biochimica. A causa delle differenze nella facilità di uso preferiamo usare X. laevis rane per la produzione di estratto uovo. Tuttavia, per gli esperimenti che richiedono o sono facilitate da un organismo con un genoma sequenziato, X. tropicalis è un ottimo sistema alternativo.
Il metodo che abbiamo descritto in questo rapporto utilizza taxolo come agente microtubuli stabilizzante per indurre la polimerizzazione dei microtubuli. Abbiamo scelto questo metodo perché taxolo è un agente stabilizzante i microtubuli robusto che facilita l'isolamento su larga scala di microtubuli purificati. Il metodo Tha abbiamo descritto potrebbe probabilmente essere migliorato mettendo a confronto le proteine e RNA associati a microtubuli che utilizzano metodi di polimerizzazione dei microtubuli alternativi. Alternative potrebbero includere polimerizzazione con GTP-indotta polimerizzazione (tecnica classica), 22 o utilizzando Ran-GTP come una polimerizzazione dei microtubuli di imitare i microtubuli indotte dal gruppo del mandrino cromatina guidato 23. Infine, l'uso di sperma nuclei purificato per indurre polimerizzazione dei microtubuli sarebbe il più vicino mimica ai tipi di microtubuli che vengono nucleati durante la mitosi (cntrosoma, cromatina, e cinetocoro mediata). Inconvenienti di queste fonti alternative di nucleazione dei microtubuli sono che gli agenti di nucleazione non sono così facilmente disponibili come taxolo e non lo fanno nucleazione o stabilizzano i microtubuli nel modo più efficiente taxolo. Pertanto, ciascuno di questi metodi sarebbe più difficile da usare per purificazioni larga scala. Il vantaggio di confrontando più diversi tipi di nucleizzatori microtubuliè che potrebbe essere possibile identificare le proteine e / o RNA che sono specifici per ciascun percorso di microtubuli nucleazione.
Il metodo che abbiamo descritto qui sfrutta estratti citoplasmatici di anfibi. Tuttavia, questo approccio potrebbe essere esteso per l'utilizzo del sistema estratto da altri microrganismi. Estratti mitotiche sono state descritte da sincronizzati colture cellulari umane 24 che fedelmente ricapitolare molti aspetti della formazione dei microtubuli. Abbiamo utilizzato con successo per identificare questi estratti RNA associata ai microtubuli da cellule HeLa 5. Schemi di purificazione microtubuli simili sono stati descritti per molti organismi differenti 25,26, anche se non sono stati esaminati i microtubuli RNA associati. L'approccio qui descritto potrebbe essere utilizzato con qualsiasi organismo che può produrre un estratto citoplasmatico concentrato capace di nucleazione microtubuli.
Infine, anche se l'approccio che describa qui discute la purificazione dei microtubuli e proteine e RNA associati, questo approccio potrebbe essere generalizzato ad altre strutture subcellulari. Mentre la maggior parte localizzate mRNA non sono stati identificati con metodi biochimici i recenti progressi nelle tecnologie di sequenziamento del DNA e RNA rendono questo approccio un metodo interessante per identificare RNA localizzate. In questo approccio ogni struttura subcellulare o sub-embrione di interesse potrebbe essere isolata o purificata. Poi le proteine e RNA associati possono essere identificati su vasta scala genoma. RNA possono quindi essere confrontati con il contenuto della cella RNA totale o embrione di identificare RNAs localizzate arricchiti. Questo approccio potrebbe essere utilizzato con le uova intere (di origine animale e vegetale separazione, simile al metodo che ha individuato i primi RNA localizzate in Xenopus 27), actina associati RNA, RNA ER-associate, RNA mitocondri associati, o di qualsiasi struttura subcellulare che possono essere purificato con RNA associati intatti. Sulla baseil nostro lavoro sul associata ai microtubuli RNA Prevediamo che questo sarebbe un ottimo metodo per scoprire nuove proteine che funzionano in un determinato luogo. Inoltre, l'identificazione della posizione e l'estensione di tutti gli RNA localizzate fornirà indizi su come le cellule e gli embrioni di utilizzare la localizzazione mRNA per controllare l'espressione genica.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Materiali
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Xenopus tropicalis | NASCO | LM00823MX | |
| human Chorionic Gonadotropin | Sigma-Aldrich | CG10 | |
| HEPES | Sigma-Aldrich | H4034 | |
| EDTA | Sigma-Aldrich | E5134 | |
| NaCl | Sigma-Aldrich | S3014 | |
| KCl | Sigma-Aldrich | P9541 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| CaCl2 | Sigma-Aldrich | C8106 | |
| sucrose | Sigma-Aldrich | S0389 | |
| NaOH | Sigma-Aldrich | S5881 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Leupeptin | Sigma-Aldrich | L9783 | |
| Pepstatin | Sigma-Aldrich | P5318 | |
| Chymostatin | Sigma-Aldrich | C7268 | |
| Cytochalasin D | Sigma-Aldrich | C8273 | |
| Gelatin, porcine skin | Sigma-Aldrich | G1890 | |
| PIPES | Sigma-Aldrich | P6757 | |
| Taxol | Sigma-Aldrich | T7191 | |
| Nocodazole | Sigma-Aldrich | M1404 | |
| Trizol | Invitrogen | 15596-026 | |
| L-Cysteine, free base | USB Corporation | 14030 | |
| Cichlid Lake Salt | Seachem | 47894 | |
| Marine salt | Seachem | SC7111 | |
| NaHCO3 | Sigma-Aldrich | S6014 | |
| EQUIPMENT | |||
| 1 ml syringes | BD Biosciences | 309659 | |
| 18 gauge needles | BD Biosciences | 305195 | |
| 30 gauge needles | BD Biosciences | 305106 | |
| Rubbermaid Plastic bucket | Amazon | 6306 | |
| Beckman Polyallomer 2 x ½ inch Ultracentrifuge tubes | Beckman | 326819 | |
| 15 ml round-bottomed glass centrifuge tubes | Fisher Scientific | 45500-15 | |
| Rubber adapter sleeves for 15 ml tubes | Kimble-Chase | 45550-15 | |
| 5 ¾ inch glass Pasteur pipettes | Fisher Scientific | 13-678-20A | |
| 14 ml polypropylene round-bottom tube | BD Biosciences | 352059 | |
| Sorvall HB-6 rotor | Thermo Scientific | 11860 | |
| Sorvall RC-6 centrifuge | Thermo Scientific | 46910 | |
Riferimenti
- Martin, K. C., Ephrussi, A. mRNA localization: gene expression in the spatial dimension. Cell. 136, 719-730 (2009).
- Besse, F., Ephrussi, A. Translational control of localized mRNAs: restricting protein synthesis in space and time. Nat. Rev. Mol. Cell Biol. 9, 971-980 (2008).
- Lawrence, J. B., Singer, R. H. Intracellular localization of messenger RNAs for cytoskeletal proteins. Cell. 45, 407-415 (1986).
- Mingle, L. A., et al. Localization of all seven messenger RNAs for the actin-polymerization nucleator Arp2/3 complex in the protrusions of fibroblasts. J. Cell Sci. 118, 2425-2433 (2005).
- Blower, M. D., Feric, E., Weis, K., Heald, R. Genome-wide analysis demonstrates conserved localization of messenger RNAs to mitotic microtubules. J. Cell Biol. 179, 1365-1373 (2007).
- Eliscovich, C., Peset, I., Vernos, I., Mendez, R. Spindle-localized CPE-mediated translation controls meiotic chromosome segregation. Nat. Cell Biol. 10, 858-865 (2008).
- Sharp, J. A., Plant, J. J., Ohsumi, T. K., Borowsky, M., Blower, M. D. Functional analysis of the microtubule-interacting transcriptome. Mol. Biol Cell. 22, 4312-4323 (2011).
- Lecuyer, E., et al. Global analysis of mRNA localization reveals a prominent role in organizing cellular architecture and function. Cell. 131, 174-187 (2007).
- Mili, S., Moissoglu, K., Macara, I. G. Genome-wide screen reveals APC-associated RNAs enriched in cell protrusions. Nature. 453, 115-119 (2008).
- Holt, C. E., Bullock, S. L. Subcellular mRNA localization in animal cells and why it matters. Science. 326, 1212-1216 (2009).
- Desai, A., Murray, A., Mitchison, T. J., Walczak, C. E. The use of Xenopus egg extracts to study mitotic spindle assembly and function in vitro. Methods Cell Biol. 61, 385-412 (1999).
- Gache, V., Waridel, P., Luche, S., Shevchenko, A., Popov, A. V. Purification and mass spectrometry identification of microtubule-binding proteins from Xenopus egg extracts. Methods Mol. Med. 137, 29-43 (2007).
- Gache, V., et al. Xenopus meiotic microtubule-associated interactome. PLoS One. 5, e9248 (2010).
- Cross, M. K., Powers, M. Obtaining eggs from Xenopus laevis females. J. Vis. Exp. (18), e890 (2008).
- Cross, M. K., Powers, M. Preparation and fractionation of Xenopus laevis egg extracts. J. Vis. Exp. (18), e891 (2008).
- Cross, M., Powers, M. In vitro nuclear assembly using fractionated Xenopus egg extracts. J. Vis. Exp. (19), e908 (2008).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends Genet. 27, 507-515 (2011).
- Hellsten, U. The genome of the Western clawed frog Xenopus tropicalis. Science. 328, 633-636 (2010).
- Brown, K. S. Xenopus tropicalis egg extracts provide insight into scaling of the mitotic spindle. J. Cell Biol. 176, 765-770 (2007).
- Godfrey, E. W., Sanders, G. E. Effect of water hardness on oocyte quality and embryo development in the African clawed frog (Xenopus laevis). Comp. Med. 54, 170-175 (2004).
- Groisman, I., et al. CPEB, maskin, and cyclin B1 mRNA at the mitotic apparatus: implications for local translational control of cell division. Cell. 103, 435-447 (2000).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38, 29-34 (2006).
- Kalab, P., Pu, R. T., Dasso, M. The ran GTPase regulates mitotic spindle assembly. Curr. Biol. 9, 481-484 (1999).
- Gaglio, T., Saredi, A., Compton, D. A. NuMA is required for the organization of microtubules into aster-like mitotic arrays. J. Cell Biol. 131, 693-708 (1995).
- Hughes, J. R., et al. A microtubule interactome: complexes with roles in cell cycle and mitosis. PLoS Biol. 6, e98 (2008).
- Suprenant, K. A., Tempero, L. B., Hammer, L. E. Association of ribosomes with in vitro assembled microtubules. Cell Motil. Cytoskeleton. 14, 401-415 (1002).
- Rebagliati, M. R., Weeks, D. L., Harvey, R. P., Melton, D. A. Identification and cloning of localized maternal RNAs from Xenopus eggs. Cell. 42, 769-777 (1985).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon