Method Article
Identificação baseada em Microarray de Individual HERV Loci Expressão: Aplicação a descoberta de biomarcadores em câncer de próstata
Neste Artigo
Resumo
Retrovírus humanos endógenos (HERV), que ocupam a 8% do genoma humano, manter reduzidas capacidades de codificação, mas a centenas de milhares de repetições terminais longas (LTRs). Um costume microarray Affymetrix foi desenhado para identificar indivíduo expressão HERV lugar e foi usado em tecidos de câncer de próstata como uma prova de conceito para futuros estudos clínicos.
Resumo
O antígeno prostático específico (PSA) é o principal biomarcador diagnóstico para câncer de próstata em uso clínico, mas que carece de especificidade e sensibilidade, principalmente nos valores de dosagem baixa 1. 'Como usar PSA "continua a ser um assunto atual, tanto para o diagnóstico como uma zona cinzenta que corresponde a uma concentração no soro de 2,5-10 ng / ml, que não permite uma diferenciação clara a ser feita entre o câncer e noncancer 2 ou para acompanhamento do paciente -up como a análise do PSA pós-operatório parâmetros cinéticos podem representar desafios consideráveis para a sua aplicação prática 3,4. Como alternativa, não-codificadoras RNAs (ncRNAs) estão emergindo como moléculas-chave no câncer humano, com o potencial de servir como novos marcadores de doença, por exemplo, no câncer de próstata PCA3 5,6 e revelar aspectos descaracterizados da biologia do tumor. Além disso, os dados do projeto ENCODE publicado em 2012 mostrou que os diferentes tipos de RNA cobrem cerca de 62% do genome. Parece também que a quantidade de motivos reguladores da transcrição é de pelo menos 4,5 vezes maior do que o correspondente aos exões de codificação de proteínas. Assim, repetições terminais longas (LTRs) do retrovírus endógenos humanos (HERV) constituem uma ampla gama de putativa / candidatos sequências reguladoras da transcrição, como seja a sua função principal no retrovírus infecciosos. HERVs, que estão espalhados por todo o genoma humano, provenientes de infecções ancestrais e independentes dentro da linha germinal, seguido por processos de propagação de copiar e colar e levando a Multicópia famílias que ocupam 8% do genoma humano (note que abrangem exons 2% do nosso genoma ). Alguns loci HERV ainda expressar proteínas que têm sido associados com diversas patologias, incluindo câncer 7-10. Nós projetamos um microarray de alta densidade, em formato Affymetrix, com o objetivo de caracterizar de forma otimizada indivíduo expressão HERV loci, a fim de entender melhor se eles podem ser ativos, se eles dirigem ncRNA transcrição ou modulate codificação expressão do gene. Esta ferramenta tem sido aplicado no campo do cancro da próstata (Figura 1).
Introdução
Retrovírus endógenos humanos (também chamados de HERVs) estão espalhados por todo o nosso genoma. Eles se originam de infecções ancestrais e independentes dentro da linha germinal, seguido por processos de propagação de copiar e colar e levando a famílias multicópia. Hoje em dia, já não são mais infecciosos mas ocupam 8% do genoma humano, como um ponto de comparação, abrangem os exões 2% do genoma humano. Os dados do projeto ENCODE publicado em 2012 mostrou que os diferentes tipos de RNA cobrir cerca de 62% do genoma, incluindo um terço em regiões intergênicas. Além disso, verifica-se que a quantidade de motivos reguladores da transcrição é de pelo menos 4,5 vezes maior do que o correspondente aos exões de codificação de proteínas. HERV repetições terminais longas (LTR) representar uma ampla gama de potenciais elementos reguladores da transcrição, como seja a sua função habitual em retrovírus infecciosos. Historicamente, além de alguns loci expressa na placenta ou testículo, acreditava-se geralmente que HERV são silenciosos devido ao epregulação igenetic. Por isso, criamos um microarray de alta densidade, em formato Affymetrix, com o objetivo de caracterizar de forma otimizada indivíduo expressão HERV loci, a fim de entender melhor se eles estão ativos, se eles dirigem lncRNA transcrição ou modular a expressão do gene de codificação. Esta ferramenta apelidada HERV-V2 GeneChip integra 23.583 probesets HERV e pode discriminar 5.573 elementos HERV distintas compostas de LTRs individuais, bem como provírus completos e parciais (Figura 2).
Diagnóstico, Avaliação e plano:
O diagnóstico do câncer de próstata é baseada na dosagem do antígeno prostático específico (PSA) biomarcador em laboratório de análises clínicas, um exame de toque retal para avaliar alteração morfológica da próstata e, finalmente, as biópsias da próstata observados pelo patologista. A falta de especificidade e sensibilidade suficientes entre biomarcadores de cancro convencionais, tais como PSA para o cancro da próstata, tem sido amplamente reconhecida afTER várias décadas de implicações clínicas 1. Inicialmente, o PSA foi proposto para o diagnóstico e tratamento de adenocarcinoma da próstata 11. Foi este último proposto para o rastreio do cancro e acompanhar o desenvolvimento da doença 12. No entanto, ainda há uma pergunta que é feita regularmente: 'como usar PSA. (I) A zona cinzenta que corresponde a uma concentração no soro de 2,5-10 ng / ml não permite uma clara diferença a ser feita entre o câncer e noncancer 2, (ii) dois grandes estudos matricular centenas de milhares de pessoas na Europa e EUA não conseguiram chegar a uma conclusão clara sobre a utilidade da triagem em termos de mortalidade específica da doença 13,14, (iii) a análise dos parâmetros cinéticos de PSA pós-operatório, como a depuração PSA, velocidade do PSA e tempo de duplicação, embora simples na teoria, podem representar desafios consideráveis em 3,4 aplicação prática. Podemos esperar que, nos próximos anos, app biomarcadorcações apoiará uma escolha clínica entre a espera vigilante e mais ou tratamentos menos agressivos, dependendo do fenótipo tumoral. Em relação ao diagnóstico proferida pelo patologista, um primeiro fator limitante vem de um 20% de falso diagnóstico negativo dentro de biópsias da próstata (muitos tipos de câncer são perdidas por amostragem). Uma segunda preocupação lida com a necessidade de um procedimento de biópsia adicional na sequência de um negativo, o qual pode apresentar efeitos adversos.
A prostatectomia radical é atualmente um dos tratamentos-padrão para câncer de próstata. Propõe-se em pacientes saudáveis, com idade 45-65 anos, especialmente no caso de padrões agressivos (Gleason 7 a 10), tumor multifocal ou tumor palpável. Ele agora é feito em nosso departamento de usar a cirurgia assistida robótico. Por causa da crescente evidência de que os marcadores moleculares terá grande importância nos próximos anos, decidimos propor a todos os nossos pacientes a possibilidade de participar de um programa para a próstatabancos de tecidos. Mais precisamente, a expansão dos programas de pesquisa molecular de câncer de próstata resultou em uma exigência crescente de acesso a alta qualidade tecidos tumorais frescas de espécimes de prostatectomia. Esta investigação, em especial as abordagens genômicas, exigiu grandes amostras de alta qualidade de DNA / RNA. Tumoral e tecidos adjacentes "não tumorais 'do mesmo paciente são necessários. Recomendações para manipulação e processamento de prostatectomia radical são projetados para preservar características patológicas que determinam estágio e status da margem e, portanto, potencial novo tratamento e prognóstico. Qualquer método de amostragem de tecido fresco, por conseguinte, não deve comprometer as avaliações patológicas subsequentes, a fim de ser aceitável para o diagnóstico. Dissecção macroscópica da próstata é difícil e uma grande atenção deve ser dada aos tecidos de margem e invasão capsular: qualquer dissecção para a banca de próstata deve ser sempre realizada por um uropathologist treinados de acordo com um acordoprotocolo d. O comitê de ética da Faculdade de Medicina e do Conselho de Medicina do Estado concordou com essas investigações e consentimento informado foi obtido de todos os pacientes incluídos na banca tecidos da próstata.
Protocolo
1. Cirurgia
Uma vez removido pelo cirurgião, manter a próstata em gelo até à sua tomada em carga dos por um patologista.
2. Tratamento de Próstata Tecidos
- Para respeitar o atraso de isquemia perioperatória, transferir espécimes de prostatectomia radical no gelo para o laboratório por pessoal dedicado dentro de 30 minutos após a ablação cirúrgica. O atraso de congelação deve ser menor do que 20-30 minutos (Figura 3A).
- Pesar e manchar a próstata de acordo com o protocolo habitual (por exemplo, verde, no lado direito, preto no lado esquerdo, ver Figuras 3B e 3C).
- Realizar uma grande secção transversal da glândula na face posterior (Figura 3D). Oriente a próstata e colocá-lo no lado anterior. Realizar uma grande secção transversal da glândula no lado posterior com um bisturi estéril.
- Dissecar pedaços de tecido na zona de transição,nas zonas periféricas da esquerda e da direita, deixando as margens intactas (Figura 3E).
- Coloque os núcleos de tecido em um tubo Eppendorf, encaixe de congelamento e armazenamento em nitrogênio líquido (Figura 3F). Se você não está fazendo biobanco proceder diretamente com o passo 2.7.
- Realizar bancário próstata apenas se o comprimento total do cancro em biópsias é superior a 10 mm. Use um fio de sutura para fechar a próstata e para evitar a distorção da glândula e interrupção mínima da margem cirúrgica (Figura 3G). Em seguida, fixar o espécime de prostatectomia radical com formalina e incorporar em parafina de acordo com o procedimento habitual para análise histológica.
- Montar núcleos de tecido congelado verticalmente em cima de um pequeno monte de outubro e fazer cortes em criostato. Faça um primeiro single de congelação 5 mm e manchá-la com azul de toluidina.
- Executar um exame histológico rápido para analisar a natureza do tecido (isto é. Benigna ou maligna). Para tumoraltecidos, estimar a quantidade de células tumorais e selecionar apenas os núcleos com mais de 80% das células tumorais.
- Em seguida, realizar um novo single de congelação 5 mm e manchá-la com hematoxilina, eosina e Safran. Em seguida, corte 15 seções x 30 mm e coloque-o em um tubo Eppendorf livre de RNAse.
- De uma última 5 um corte de congelação de hematoxilina, eosina e Safran e mancha-lo para controlar a quantidade de células tumorais no final do procedimento.
- Colocar o tubo de Eppendorf em gelo seco e enviar a amostra para o laboratório de biologia molecular.
3. Extração de RNA, purificação e controle de qualidade
- Homogeneização. Realizar a homogeneização na presença de 1 ml de tecido mg Trizol/100 até o tecido é completamente dissolvido em solução. Adicionar solução Trizol gradualmente e proceder com cuidado sobre o gelo usando um moedor de mão. Depois de homogeneizada, a solução divida em alíquotas para tubos Eppendorf e deixar em Trizol à temperatura ambiente durantede cinco minutos.
- A separação de fases. Adicionar 300 mL de clorofórmio (ou 150 ul BCP/1.5 ml de Trizol). Vortex 15 segundos, em seguida, deixar à temperatura ambiente durante 2-3 min. Centrifugar a 12000 xg durante 15 min a 2-8 ° C.
- Precipitação de ARN. Transferir cuidadosamente a fase aquosa superior para um novo tubo. Adicionar 750 mL de isopropanol. Incubar à temperatura ambiente por 10 min (agitar por inversão). Incubar 2 horas a -19 ° C/-31 ° C. Centrifugar as amostras a 12.000 xg durante 30 min a 2-8 ° C.
- Lavagem RNA e suspensão. Após a centrifugação, remove-se o sobrenadante. Wash RNA pellet com 1 ml de 80% EtOH (reverso suavemente os tubos). Centrifugar as amostras a 7500 xg durante 10 min a 2-8 ° C. Remover o sobrenadante utilizando P1000 e P10. Permitir EtOH restante se secar ao ar durante 2-3 min. Adicionar 100 de água livre de RNase-pi, os tubos de transferência a 70 ° C num bloco de aquecimento e deixar repousar durante 2-3 min para dissolver o sedimento. Em seguida, coloque no gelo. Guarde-o em -19 armazenamento ° C/-31 ° C para o curto prazo e -80 ° C para armazenamento a longo prazo.
- Purificação de RNA. Purifica-se o ARN, utilizando o kit RNeasy Mini (Qiagen). Resumidamente, começar adicionando 350 mL de tampão RLT a amostra de RNA de 100 mL e misture bem, em seguida, siga o procedimento Qiagen. Finalmente, ter um pi 3 (de 50 ul) alíquota do produto purificado para os controlos de qualidade (passo 3.6). ARN da loja em -19 ° C/-31 ° C durante um período curto ou a -80 ° C durante um armazenamento a longo prazo.
- ARN QC (Figura 4A). Verificar a qualidade do RNA e a integridade do RNA usando um Bioanalyzer (Agilent) e um Nanodrop (Thermo), de acordo com as instruções do fabricante. O ARN Integridade Número (NIR) é usado para avaliar a qualidade do RNA. Em particular, o sucesso na detecção de 18S e 28S picos é altamente recomendável utilizar as amostras em outras etapas.
4. WT-ovação RNA Amplificação
Recomendações para realizar as etapas de amplificação utilizando o WT-Ovkit amplificação ção em condições ideais:
- Correr não menos que oito amostras de amplificação de cada vez para assegurar a precisão de pipetagem. Em seguida, são responsáveis por um volume de resíduos quando a preparação de misturas mestres que exigem uma divisão do kit em três lotes de oito reações.
- Sempre mantenha os reagentes descongeladas e tubos de reacção em gelo salvo ordem em contrário.
- Use apenas uma nova solução de etanol 80% para a purificação.
- Não pare em qualquer fase do protocolo.
- Dilui-se o RNA total para obter uma concentração de 25 ng / ul. Processe 2 uL da amostra diluída.
- Prepare o poli-A RNA espiga no controle de solução através de uma diluição em série do ARN poli-A da com poli-A de controlo de tampão de diluição (Affymetrix), para atingir uma diluição de 1:25.000.
Passo 1: First Strand cDNA Synthesis 4,3-4,8. Os reagentes mencionados são referidos pelo fornecedor da seguinte maneira: A1 (Strand Primeiro Primer Mix), A2 (FiStrand rst Tampão Mix), A3 (Primeira Mix Enzyme Strand).
- Descongelar A1 e A2, à temperatura ambiente. Misture usando um vortex-mixer por 2 segundos e rotação por 2 seg. Em seguida, colocar rapidamente em gelo. Coloque A3 no gelo.
- Coloque 2 ul de ARN total (50 ng) em um tubo de PCR de 0,2 ml, e adicionar 2 mL de A1 (volume final: 4 ul). Cap e girar o tubo por 2 seg.
- Incubar a 65 ° C durante 5 min, em seguida, colocar o tubo em gelo.
- Preparar a primeira cadeia de cDNA como se segue Master Mix (dada por uma única reacção). Misturar por pipetagem e girar brevemente a Master Mix. Imediatamente, colocar no gelo.
Reagente Volume Strand primeiro buffer Mix (A2) 5 ul Poly-A Controle de RNA (1:25.000) 0,5 ul Primeira mistura de enzima Strand (A3) 0,5 ul - Adicionar 6 & #181; l do Master Mix ao / contendo Primer tubo RNA. Misture sacudindo o tubo, rotação por 2 segundos e rapidamente colocar no gelo (volume final: 10 mL).
- Incubar a 4 ° C durante 1 min, depois 25 ° C durante 10 min, em seguida 42 ° C durante 10 minutos e depois 70 ° C durante 15 min. Mantenha-se fresco a 4 ° C. Retire o tubo de reacção do termociclador, girar rapidamente e manter em gelo. Continue imediatamente com o passo Strand cDNA Synthesis Segundo.
Passo 2: Em segundo lugar Strand cDNA Synthesis 4,8-4,12. Os reagentes mencionados são referidos pelo fornecedor da seguinte forma: B1 (Segunda Strand Buffer de Mix) e B2 (Segunda Strand Mix Enzima).
- Girar B2 e B3 por 2 segundos e rapidamente colocar no gelo. Descongele B1 à temperatura ambiente. Misture usando um vortex-mixer por 2 segundos, rotação por 2 segundos e rapidamente colocar no gelo.
- Prepara-se uma segunda cadeia de Master Mix da seguinte forma (determinado por uma única reacção). Misturar por pipetagem e girar o brie Master Mixvoar. Imediatamente, colocar no gelo.
Reagente Volume Strand segundo buffer Mix (B1) 9,75 mL Segundo Strand Mix Enzyme (B2) 0,25 mL - Adicionam-se 10 ul da mistura de mestre para cada tubo de reacção First Strand. Misture pipetando 3x, rotação por 2 segundos e colocar no gelo (volume final: 20 mL).
- Incubar a 4 ° C durante 1 min, depois 25 ° C durante 10 min, em seguida 50 ° C durante 30 min, e em seguida 70 ° C durante 5 min. Mantenha-se fresco a 4 ° C. Retire o tubo de reacção do termociclador, girar rapidamente e manter em gelo. Continue com o passo imediatamente posterior à Segunda Strand Enhancement.
Passo 3: Pós-Segunda Strand Enhancement 4,13-4,15. Os reagentes mencionados são referidos pelo fornecedor da seguinte forma: B1 (Segunda Strand Buffer de Mix), B3 (Reação Enhancement Mix Enzima).
- Prepara-se uma mistura padrão através da combinação de B1 e B3 mistura da seguinte forma (determinado por uma única reacção). Misturar por pipetagem e girar brevemente a Master Mix. Imediatamente, colocar no gelo.
Reagente Volume Strand segundo buffer Mix (B1) 1,9 mL Reacção Aperfeiçoamento mistura enzimática (B3) 0,1 mL - Adicione 2 mL da Mix Master para cada tubo de reacção Segundo Strand. Misture pipetando 3x, rotação por 2 segundos e colocar no gelo (volume final: 22 mL).
- Incubar a 4 ° C durante 1 min, depois 37 ° C durante 15 min, e depois 80 ° C durante 20 min. Mantenha-se fresco a 4 ° C. Retire o tubo de reacção do termociclador, girar rapidamente e colocar no gelo. Continue imediatamente com o passo SPIA Amplification.
Passo 4: cDNA Single (sscDNA) síntese por procedimento SPIA de 4,16 -4.19. Os reagentes mencionados são referidos pelo fornecedor da seguinte forma: C1 (SPIA Primer Mix), C2 (SPIA Tampão Mix), C3 (SPIA Mix Enzima).
- Descongelar o C1 e C2, à temperatura ambiente. Misture usando um vortex-mixer, rotação por 2 segundos e rapidamente colocar no gelo. Descongele C3 no gelo. Misturar o conteúdo invertendo delicadamente 5x. Certifique-se de não introduzir bolhas de ar. Em seguida, gire por 2 segundos e colocar no gelo.
- Prepare uma mistura SPIA-Master, representando um volume de 0,5 resíduos, como segue. Misturar por pipetagem e girar brevemente a Master Mix. Imediatamente, colocar no gelo.
Reagente Volume SPIA-Buffer Mix (C2) 5 ul SPIA-Primer Mix (C1) 5 ul SPIA-Enzyme Mix (C3) 10 ul - Adicionar 20 ml da SPIA Master Mix ao avançado Segundo Strand Reaction tube. Misture pipetando 6-8x, rotação e rapidamente colocar no gelo (volume final: 42 mL).
- Incubar a 4 ° C durante 1 min, depois 47 ° C durante 60 minutos e depois 95 ° C durante 5 min. Mantenha-se fresco a 4 ° C. Retire o tubo de termociclador, girar rapidamente e colocar no gelo.
5. sscDNA Purificação e Controle de Qualidade
- sscDNA purificação. Purificar sscDNA utilizando o kit de purificação QIAquik de PCR (Qiagen). Resumidamente, começar por adição de 200 ul de tampão de PB para os 42 ul do produto de ADNc amplificado, misturar e carga sobre a coluna. Depois siga o procedimento Qiagen. Finalmente, ter um pi 3 (de 30 ul) alíquota do produto purificado sscDNA para os controlos de qualidade (passo 5.2).
- sscDNA produzir e verificação de distribuição de tamanho (Figura 4B). Verificar sscDNA rendimento e a distribuição de tamanho utilizando um Bioanalyzer e um Nanodrop, de acordo com as instruções do fabricante. O tamanho de distribuição de cDNA amplificados devem be normalmente compreendido entre 100 e 1500 bases de comprimento, com um pico de cerca de 600 bases.
6. sscDNA Fragmentação
- Prepare a 2 ug de cDNA em 30 ul, ajustando o volume com a água livre de nuclease.
- Prepare 1x Uma Phor-All-tampão mais (OPA), a partir de um tampão 10x OPA PLUS solução.
- Prepare a 0,2 U / mL de ADNase I (diluição de 5 vezes de 1 U / mL de DNase I).
- Prepara-se uma fragmentação Master Mix da seguinte forma (determinado por uma única reacção):
Reagente Volume 10X One-Phor-All Tampão PLUS 3,6 mL ADNase I (0,2 U / ul) 3 mL - Adicionar 6,6 mL de mistura de fragmentação para os 30 ul de sscDNA.
- Rotação e incuba-se a 37 ° C durante 10 min, em seguida, inactivar a DNase I a 95 ° C durante 10 minutos e manter em gelo. Alíquota 1 &# 181; l do cDNA fragmentada para verificação de distribuição de tamanho com base Agilent.
- sscDNA verificação de distribuição de tamanho (Figura 4C). Verificar distribuição de tamanho sscDNA usando um Bioanalyzer (Agilent). A distribuição de tamanho de ADNc fragmentado deve ser geralmente compreendida entre 35 e 200 bases.
7. Rotulagem de Fragmentada sscDNA
- Diluir o DLR-1a 7,5 mM até 5 mM em DEPC-água.
- Prepara-se uma Master Mix rotular como segue (dada por uma única reacção):
Reagente Volume 5x TdT Tampão de reacção 14 mL CoCl2 (25 mM) 14 mL DLR-1a (5 mM) 1 ul Transferase terminal (400 U / ul) 4,4 mL - Adicionar 33.4 ul de mistura a rotulagempara cada amostra de ADNc fragmentado.
- Misturar sacudindo o tubo, girar rapidamente e incubar a 37 ° C durante 60 min, em seguida, manter em gelo.
8. A hibridação com o microarray HERV Chip
- Prewet GeneChip HERV com 200 ul de pré-hibridação Mix (Affymetrix) e incubar a 50 ° C, 60 rpm, durante 10 min.
- Preparar a mistura de hibridação, como se segue (dada por uma única reacção):
Reagente Volume Controlo Oligo B2 (3 nM) 3,3 mL 20x eucariótica Hibridização Controle 10 ul 2x Hibridização Mix 100 ul 99,9% de DMSO 17,7 mL - Adicionar 131 ul de mistura de Hibridização para os 69 pi de cDNA marcado fragmentado e à temperatura ambiente para fazer um vo definitivolume de 200 ul.
- Misture e desnaturar durante 2 min a 95 ° C, em seguida, incuba-se a 50 ° C durante 5 min e centrifuga-se à velocidade máxima durante 5 min.
- Esvazie o GeneChip HERV prewetted e carregar a preparação alvo de 200 mL. Aplicar-spots difíceis nos dois septos.
- A hibridização a 50 ° C, 60 rpm, durante 18 horas.
- Após 18 horas, esvaziar o GeneChip HERV e armazenar a solução de hibridização recolhida a 4 ° C. Preencha a matriz sonda com 250 mL de tampão de lavagem A. Se os chips não são executadas imediatamente para os fluidos, armazenados a 4 ° C.
9. Lavagem e coloração
- Execute os Fluidics na barra de menu GCOS. Na caixa de diálogo Fluidics, selecione a estação de interesse (1 - 4), em seguida, selecione Shutdown_450 para todos os módulos, em seguida, executar. Mergulhe as 3 linhas de aspiração de fluidos em água Milli-Q. Siga as instruções no ecrã LCD.
- Aplicar o programa Prime_450 para todos os módulos. Coloque o tubo de tampão de lavagem A emum frasco contendo 400 ml de tampão de lavagem A, ea outra para o tampão de lavagem B em um frasco contendo 200 ml de tampão de lavagem B. Então, novamente, siga as instruções no ecrã LCD.
- Levante as agulhas e coloque cocktail 600 mL mancha 1 (SAPE Solution Mix) e 600 mL de cocktail mancha 2 (Anticorpo Solution Mix) contendo microtubos em posições # 1 e # 2, e 800 mL conjunto segurando solução tampão na posição # 3 .
- Atribuir o chip direito de cada módulo, selecione o protocolo FS450-004 e executar cada módulo, seguindo as instruções na tela.
10. Exploração
- Aqueça o scanner GS3000. Ele está pronto para fazer a varredura quando a luz ficar verde.
- Aplicar-spots difíceis para os septos para evitar vazamento em seguida, coloque o chip para o carregador automático ou, alternativamente, diretamente para o scanner. Inicie a digitalização.
- Após a digitalização do chip,. Arquivos cel são gerados. Verifique a imagem e alinhar a grade para o local para identificar as células de sonda ( Figuras 4D-F).
11. Análise de Dados
- O controle de qualidade. Consulte o padrão Affymetrix controles para verificar se os chips HERV-V2 cumprir os critérios de CQ. Para este efeito, as seguintes representações podem ser usados: a distribuição log valor de intensidade (gráficos de densidade e parcelas de caixa), o desvio absoluto médio (MAD) versus a mediana intensidade (MAD-Med) parcelas, as parcelas de fundo, o erro padrão sem escala normalizada (nuse) parcelas ea expressão log relativa (RLE) parcelas.
- Normalização. Além disso, o conjunto de dados deve ser explorada para evidenciar os efeitos inesperados de lote e corrigi-los antes da análise estatística. O pré-processamento de dados inclui, assim, uma correcção de fundo (p. ex. Baseado no sinal de linha de base da sonda triptofano), seguido por normalização de RMA e compactação 15.
- A mineração de dados e busca de genes diferenciais expressa. Normalizar os chips e aplicar um clusteri hierárquicaabordagem ng para explorar o conjunto de dados (Figura 6A). Em seguida, faça uma busca por genes diferencialmente expressos (DEG) através de uma análise clássica significativa de microarray (SAM) 16 procedimento seguido por uma taxa de falsas descobertas (FDR) correção de 17. Note-se que estes passos são totalmente integrados em algumas suites de análise de software como Partek GS mas em alternativa pode ser realizada utilizando o software estatístico R 18 com os pacotes do projeto Bioconductor 19. Após a análise estatística, filtrar o conjunto de dados para excluir os probesets para o qual os valores de expressão são menos de 2 6.
- Visualização e interpretação. Interpretar os resultados do microarray HERV-V2 em uma interface dedicada usando bancos de dados de anotação.
Resultados
O valor dos estudos transcriptomic reside principalmente na qualidade do material biológico de partida. Se a extracção de RNA é realizada em condições óptimas, o ARN Integridade Número (NIR) é tipicamente 7 ou superior (figura 4A). A necessidade de se hibridizar 2 ug de cDNA no chip Affymetrix HERV-V2 implica a utilização de um processo de amplificação. Um passo de amplificação bem sucedida leva a uma distribuição em forma de sino (Figura 4B). Em seguida, a fragmentação DNAse1 é executada a fim de homogeneizar a distribuição de tamanho de cerca de 100 nucleótidos de cDNA antes da hibridação (Figura 4C). Depois de hibridação e de varredura (Figura 4D), uma inspeção visual da imagem permite que se verifique se a grade está bem alinhada com os pontos (Figura 4E) e se os controles de hibridização são consistentes (Figura 4F). Este passo é também útil, a fim de excluir microarrays em que bolhas de ar ou erros ocorreu durante o experimento.
Uma vez que as batatas fritas já passaram QC (Figura 5) e após a normalização, a análise estatística de 5 tumor jogo de par e as amostras de RNA de próstata normais do Hospital Lyon-Sud, levou à identificação de 207 probesets HERV com valores de expressão diferenciais (p.val <0,05) (Figura 6A). Para dar suporte a esses registros e obter informações específicas de próstata, 35 amostras jogo de pares adicionais (cólon, ovário, testículos, mama, pulmão e próstata) foram adicionados à análise eo procedimento SAM-FDR (FDR = 20%), eventualmente identificados 44 probesets HERV específico da próstata. Entre elas, as 10 estruturas HERV mais relevantes são descritas (Figura 6B). Estudos clínicos adicionais serão necessários para avaliar os valores de sensibilidade e especificidade destes biomarcadores candidatos.
pload/50713/50713fig1.jpg "width =" 500px "/>
. Figura 1 Esquema do procedimento geral a partir de clínica (1: prostatectomia pelo clínico e a preparação do tecido pelo patologista) ao banco (2-6: a preparação da amostra, a preparação de destino, o processamento do microarray) que conduz à identificação de candidatos a biomarcadores (7: Análise de biocomputação dos microarrays HERV). Ácidos nucleicos derivadas de tecido normal estão representados na laranja; ácidos nucléicos derivados de área tumoral consistem em uma mistura de normal (laranja) e tumor específico (preto) ácidos nucléicos. Clique aqui para ver a imagem ampliada .
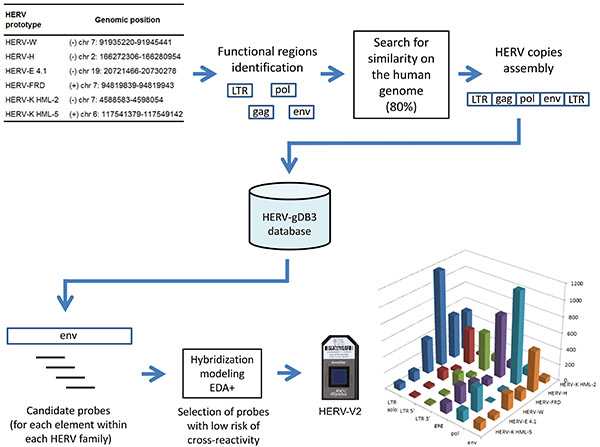
. Figura 2 Concepção e conteúdo do chip HERV-V2: seqüências HERVrecuperado a partir do genoma humano são armazenados numa base de dados denominada HERV-gDB3, então as sondas de candidatos de 25-mer passar através de um processo de modelagem de hibridização específico (EDA +) antes de ser finalmente sintetizado na matriz (os sub-regiões-alvo resultantes estão representadas para cada família). Clique aqui para ver a imagem ampliada .
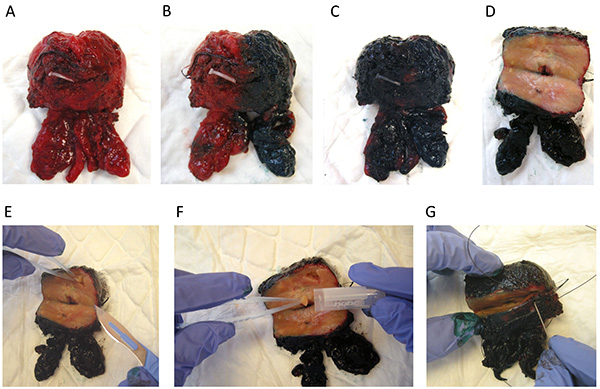
Figura 3. Manipulação da próstata pelo patologista. (A) Fresco espécime prostatectomia radical é transferido para o laboratório. (BC) A próstata é manchada (verde no lado direito, preto no lado esquerdo). (D) grande secção transversal da glândula no lado posterior. (E) Deixando as margens intactas, pieces de tecidos são dissecados a partir de diferentes áreas da glândula da próstata. (F) Os núcleos do tecido são colocados num tubo de Eppendorf. (G) fio de sutura é usado para fechar a próstata e para evitar a distorção da glândula e interrupção mínima da margem cirúrgica. Em seguida, a amostra prostatectomia radical está pronto para a fixação em formol de acordo com o procedimento habitual para análise histológica. Clique aqui para ver a imagem ampliada .
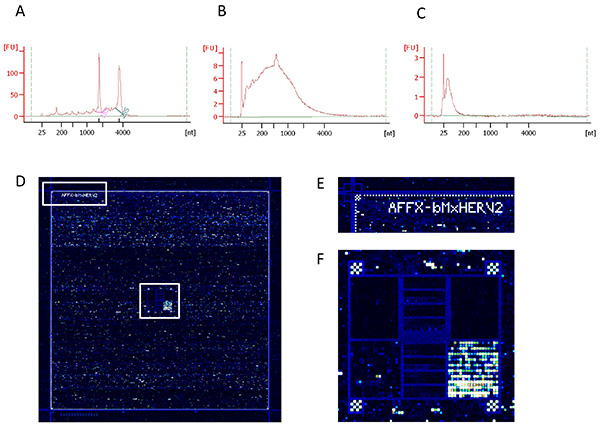
Controlos Figura 4. Qualidade da preparação de ácido nucleico e a eficiência de hibridação. (A) a integridade do RNA, (B) e alvos de cDNA (C) alvos fragmentados utilizados na fase de hibridação amplificada. These três controlos de qualidade foram obtidos com o uso de ARN Bioanalyzer nano chips e o ensaio Eucarioto Nano Serie II. (D) imagem geral da área de HERV-V2 microarray hibridização após a digitalização, (E) alargamento do canto mostrando grade controles de alinhamento superior esquerdo e (F) alargamento da área do centro, mostrando manchas controles de hibridização. Clique aqui para ver a imagem ampliada .
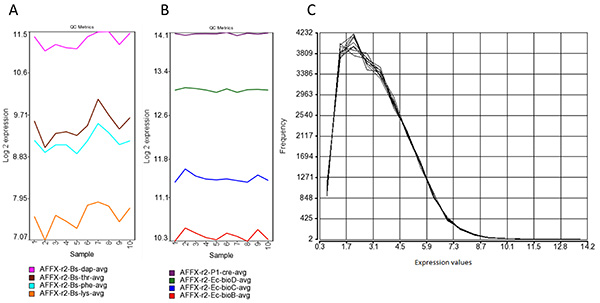
Figura 5. Processamento de sinais. (A) Affymetrix polyA spike-in controles de amplificação. O polyA controla Dap, Thr, Phe e Lys transcrições de B. genes subtilis são cravado na amostra de RNA e servem para avaliar o sucesso global as etapas de preparação do alvo. Intensidade deve ser detectada a diminuir os valores entre estes controlos pico-para assegurar que não havia nenhuma polarização durante a amplificação WT-Ovation entre genes altamente e baixa-expresso. (B) Affymetrix spike-nos controles de hibridização. Estes alvos isolados a partir de E. coli e bacteriófago P1 estarem reforçados antes do procedimento de etiquetagem. Valores crescentes de BIOB, BIOC, BIOD e Cre indicar o sucesso global da hibridização. (C) distribuição da intensidade dos sinais de chips depois RMA normalização. A maioria dos sinais probesets exposição com valores inferiores a 2 6 (background), indicando uma expressão geral restrita principalmente para alguns loci HERV específico. Clique aqui para ver a imagem ampliada .
"width =" ftp_upload/50713/50713fig6.jpg 500px "/>
Análise Figura 6. Dados. (A) análise de agrupamento hierárquico de amostras normais e tumorais. Partitioning agrupamento foi aplicada aos valores de expressão normalizados utilizando um algoritmo de função de distância Euclidiana, agrupando-se em probesets (vermelho) - e para baixo (azul)-regulação entre as amostras normais e tumorais. (B) A seleção dos 10 melhores estruturas HERV identificados como candidato biomarcador de câncer de próstata. Para cada elemento HERV, a família HERV relacionada, as coordenadas genômicas (NCBI 36/hg18) e uma breve descrição da estrutura HERV são dadas. Clique aqui para ver a imagem ampliada .
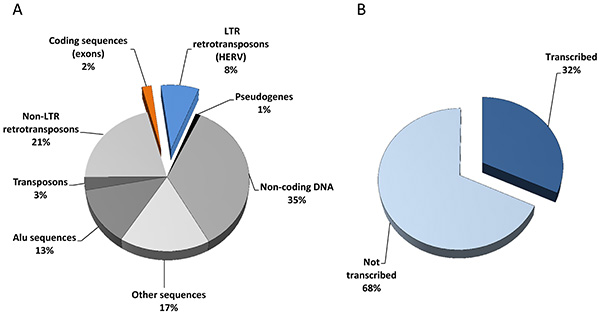
Figura 7. O repertório HERV. (A) O seqüenciamento do genoma humano revelou 25.000 genes codificadores de proteínas (exons, 2%) e uma enorme quantidade de elementos transponíveis, incluindo 200.000 longa-terminal repeat (LTR) retrotransposons (HERV, 8%). (B) A extrapolação a partir do conteúdo de chips HERV-V2 e dados de expressão associados (79 amostras provenientes de oito normal versus tipos de tecidos tumorais) sugerem que um terço do repertório HERV é transcricionalmente ativo. Clique aqui para ver a imagem ampliada .
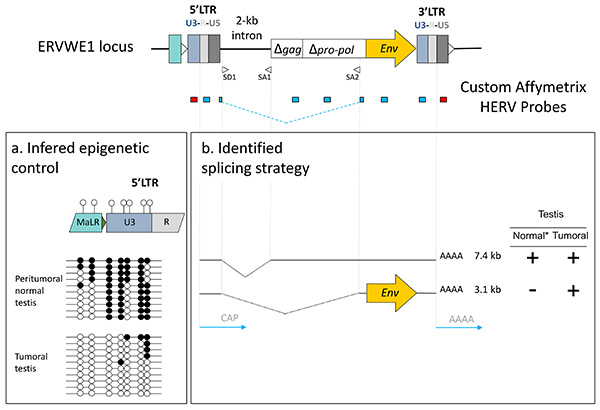
Figura 8. Interpretação funcional de sinais a partir do chip. (A) a identificação Promotor e controle epigenético: U3 sinal negativo (sonda vermelha, 5'LTR) Versus sinal positivo R-U5 (azul sonda, 5'LTR) sugerem transcrição impulsionado-U3, apoiado pelo diferente metilação CpG (círculos pretos sólidos) o conteúdo de U3 em peritumoral normal versus tecidos tumorais. (B) A estratégia de emenda: a putativo envelope de 3,1 kb que codifica para o ARNm expresso exclusivamente no tumor é identificado utilizando junção de processamento SD1/SA2 sobreposição sonda. * Deduzido pela comparação com outros tecidos não-placentários. Clique aqui para ver a imagem ampliada .
Discussão
Ao longo dos últimos 10 anos, a maioria das tentativas de medição expressão HERV usaram técnicas de RT-PCR, quer se concentrar em um locus específico 20-24 ou com base na conservação relativa dos genes pol para avaliar as tendências gerais dentro HERV gêneros 25,26 . Além disso, a amplificação de PCR utilizando os iniciadores altamente degenerados, juntamente com microarrays de baixa densidade destinados a detectar e quantificar a expressão de famílias de HERV 27,28. A fim de traçar a expressão do locus individual dentro de uma família, as abordagens baseadas na amplificação por PCR de regiões conservadas combinada com a clonagem e subsequente sequenciação permitiu elementos distintos transcricionalmente activos das HML-2 29,30 ou HERV-E4.1 31 familiares aos ser identificado. Também terminando por clonagem e sequenciamento de etapas, o genoma repetição técnica de monitoramento de expressão com o objetivo de identificar promotores entre repete identificados HML-2 LTRs solitários humanos específicos ativos 32,33. Desenvolvemos sucessivamente duas gerações de microarrays de alta densidade que se dedicam à análise do transcriptoma HERV, introduzindo metodologias adequadas para o projeto repetido sonda elemento, a fim de minimizar as reações cruzadas entre elementos parálogas dentro de uma família 34,35. O chip HERV-V2 que tem como alvo 2690 provírus distintas e 2883 LTRs de solo do HERV-W, HERV-H, HERV-E 4.1, HERV-FRD, HERV-K HML-2 e HERV-K HML-5 famílias, revelou o expressão de HERV 1718 loci (Figuras 7A e B) em uma ampla gama de tecidos 35, ilustrados no presente documento a identificação de biomarcadores putativos de cancro da próstata. Além disso, o uso de múltiplas probesets num determinado locus é informada sobre a regulação da transcrição. Em primeiro lugar, um sinal negativo U3 em conjunto com um U5 positiva classifica o LTR como um promotor, e sinais negativos, inversamente, U3 e U5 positivos pode reflectir um papel de poliadenilação. Assim, identified 326 LTR do promotor em uma ampla gama de tecidos de 35 e, com base nesta informação dicotómica U3-U5 fornecida pela matriz, foi proposto e confirmado experimentalmente para alguns casos seleccionados tal que a transcrição autónomo foi controlada por um processo de metilação dependentes epigenética 34 (figura 8). Em segundo lugar, a detecção de sinais a partir de por exemplo, LTR, gag e env probesets independentes ou emitida a partir de sondas dirigidas junção de processamento específica é informada sobre a estratégia de splicing proviral, como ilustrado pelo perfil de expressão ERVWE1/Syncytin1 na placenta ou no testículo tumoral 34. Isto indica que o processo de selecção da sonda específica HERV é suficientemente robusta para suportar a identificação da estratégia associada splicing-tecido, de forma tão eficiente como os genes convencionais 36 (Figura 8).
Este método é a primeira tentativa de identificar expressão lócus individualmente HERVutilizando um microarray personalizado de alta densidade com base na tecnologia Affymetrix. As vantagens claramente identificadas no formato de microarranjo para decifrar transcriptoma HERV que consiste de (i) a exploração coordenada de várias famílias de HERV e (ii) a análise simultânea e independente das diferentes regiões para cada locus, por exemplo. U3 e U5 domínios para regiões gag ou env de solo e LTRs provirais e possíveis cruzamentos emendados associados com estruturas provirais, sem qualquer a priori sobre a funcionalidade do elemento HERV. Perspectivas dependem de uma melhoria de anotações nas ferramentas de bioinformática associada à microarray. Isso deve permitir uma para converter sinais de chips em hipóteses biológicas tais como se HERVs ativas evidenciadas dirigir lncRNA transcrição ou modular mais ou menos proximal codificação expressão gênica. De fato, tal hipótese é suportada por estudos recentes que identificaram transcrições ncRNA associados ao câncer de próstata, que incluem componentes de vORF IRAL da família HERV-K retrovírus endógeno ou porções de uma região do promotor LTR viral 37, bem como dois eventos de fusão do gene nomeadamente HERV-K22q11-ETV1 e HERV-K17-ETV 38,39. Tomados em conjunto, esta abordagem transcriptoma todo combinado com a função de LTR e identificações de estratégia de emenda pode ajudar a decifrar o marcador contra os componentes de disparo de expressão HERV em crônica 40,41 e doenças infecciosas 42,43.
Divulgações
Este trabalho foi financiado pela bioMérieux SA, o Hospices Civils de Lyon e da agência pública francesa OSEO (Diagnósticos Avançados para novas abordagens terapêuticas, um programa financiado pelo governo francês dedicado à medicina personalizada). PP, VC, GO, NM, e FM são funcionários da bioMérieux SA. PP, e MN apresentaram pedidos de patente nas conclusões deste trabalho.
Agradecimentos
Agradecemos Cecile Montgiraud, Juliette Gimenez, Magali Jaillard e Bertrand Bonnaud por sua contribuição para o desenvolvimento inicial e otimização do protocolo HERV-V2. Gostaríamos também de agradecer Hader Haidous por sua orientação sobre considerações éticas.
Materiais
| Name | Company | Catalog Number | Comments |
| Trizol | Invitrogen | 15596-026 | |
| RNA poly-A control stock | Affymetrix | 900433 | |
| DNAse 1 | Promega | M6101 | 1,000 U (1 U/µl) |
| Terminal transferase | Roche | 3333574001 | 400 U. Including enzyme and coenzyme (CoCl2). |
| DLR-1a | Affymetrix | 900542 | |
| Hybridization internal controls B2 and 20x Eukaryotic Hybridization Control | Affymetrix | 900454 | |
| GeneChip Hybridization, Wash and staining | Affymetrix | 900720 | Including PreHybridization Mix and 2x Hybridization Mix for 30 reactions |
| 10x One-Phor-All Buffer PLUS | Composition in DEPC-treated water: 100 mM Tris-acetate pH 7.5; 100 mM magnesium acetate; 500 mM potassium acetate. | ||
| RNeasy Mini kit | Qiagen | 74104 | RNA cleanup protocol |
| WT-Ovation RNA amplification system | Nugen | 2210-24 | |
| QIAquik PCR purification kit | Qiagen | 28104 | |
| EQUIPMENT | |||
| Material Name | Company | Catalogue Number | Comments |
| Nanodrop 1000 | Thermo Scientific | ||
| GeneChip Scanner 3000 7G | Affymetrix | GS30007G | Optional: autoloader |
| GeneChip Fluidics Station 450 | Affymetrix | FS450 | |
| GeneChip Hybridization 640 Oven | Affymetrix | 640 | Includes 4 GeneChip Probe array carriers |
| Workstation loaded with GeneChip Operating Software (GCOS) including the GeneChip Scanner 3000 High-Resolution Scanning Patch | |||
| HERV-V2 chip | Affymetrix | Custom array. For microarray availability (for research use only), please contact: François Mallet Laboratoire Commun de Recherche Hospices Civils de Lyon-bioMérieux Medical Diagnostic Discovery Department Centre Hospitalier Lyon Sud, Bâtiment 3F 69495, Pierre Bénite cedex France Phone: 33 (0)4 72 67 87 85 Email: francois.mallet@biomerieux.com | |
| HERV-V2 conception Dedicated database and annotations The construction of a dedicated database, grouping genomic HERV sequences belonging to 6 HERV families, has been achieved by the following procedure: (i) the most complete and representative sequence of each HERV family was selected from the literature and defined as a prototype sequence (Figure 2). (ii) The 6 prototypes were functionally annotated with reference to their LTR (U3/R/U5) and internal parts (gag/pol/env). (iii) RepeatMasker 44 was then applied using these functional sequences as input libraries. A genome-wide search of all related sequences was performed over the human genome on the basis of a minimum 80% homology (NCBI 36/hg18). (iv) Finally, the functional sequences retrieved by this process were assembled into distinct loci on the basis of their genomic location and eventually implemented in a dedicated HERV database. This database, called HERV-gDB3, contains 10,035 individual HERV loci35. Locus-specific probes design Starting from HERV-gDB3, overlapping tracks of 25-mer candidate probes were firstly generated. Each candidate probe was then aligned against the human genome using KASH 45 in order to assess the cross-hybridization potentialities. This latter estimation was performed by a model developed specifically for this purpose and referred to as EDA+. Briefly, the principle of EDA+ is to take into account the instability brought by mismatches and gaps in a 25-mer target/probe hybridization complex. Candidate probes exhibiting low cross-hybridization risks (i.e. a low number of non-specific genomic targets) are selected and lastly assembled into probesets. Custom HERV GeneChip microarray The custom HERV GeneChip integrates 23,583 HERV probesets and can discriminate 5,573 distinct HERV elements, composed of solo LTRs, complete and partial proviruses (Figure 2). The standard Affymetrix control probes for unbiased amplification and hybridization were also included in the microarray. | |||
Referências
- Artibani, W. Landmarks in prostate cancer diagnosis: the biomarkers. BJU Int. 110, Suppl 1. 8-13 (2012).
- Girometti, R., et al. Negative predictive value for cancer in patients with "gray-zone" PSA level and prior negative biopsy: preliminary results with multiparametric 3.0 Tesla MR. J. Magn. Reson. Imaging. 36, 943-950 (2012).
- You, B., et al. Prognostic value of modeled PSA clearance on biochemical relapse free survival after radical prostatectomy. Prostate. 69, 1325-1333 (2009).
- Vickers, A. J., Brewster, S. F. PSA Velocity and Doubling Time in Diagnosis and Prognosis of Prostate Cancer. Br. J. Med. Surg. Urol. 5, 162-168 (2012).
- Bussemakers, M. J., et al. DD3: a new prostate-specific gene, highly overexpressed in prostate cancer. Cancer Res. 59, 5975-5979 (1999).
- Vlaeminck-Guillem, V., Ruffion, A., Andre, J., Devonec, M., Paparel, P. Urinary prostate cancer 3 test: toward the age of reason. Urology. 75, 447-453 (2010).
- Buscher, K., et al. Expression of the human endogenous retrovirus-K transmembrane envelope, Rec and Np9 proteins in melanomas and melanoma cell lines. Melanoma Res. 16, 223-234 (2006).
- Ishida, T., et al. Identification of the HERV-K gag antigen in prostate cancer by SEREX using autologous patient serum and its immunogenicity. Cancer Immun. 8, 15(2008).
- Sauter, M., et al. Human endogenous retrovirus K10: expression of Gag protein and detection of antibodies in patients with seminomas. J. Virol. 69, 414-421 (1995).
- Pérot, P., Bolze, P. A., Mallet, F. Viruses: Essential Agents of Life. Witzany, G. , Springer-Verlag. 325-361 (2012).
- Stamey, T. A., Kabalin, J. N. Prostate specific antigen in the diagnosis and treatment of adenocarcinoma of the prostate. I. Untreated patients. Untreated patients. J. Urol. 141, 1070-1075 (1989).
- Catalona, W. J., et al. Measurement of prostate-specific antigen in serum as a screening test for prostate cancer. New Engl. J. Med. 324, 1156-1161 (1991).
- Schroder, F. H. Prostate cancer around the world. An overview. Urol. Oncol. 28, 663-667 (2010).
- Klotz, L. Active surveillance for favorable-risk prostate cancer: background, patient selection, triggers for intervention, and outcomes. Curr. Urol. Rep. 13, 153-159 (2012).
- Irizarry, R. A., et al. Exploration, normalization, and summaries of high density oligonucleotide array probe level data. Biostatistics. 4, 249-264 (2003).
- Tusher, V. G., Tibshirani, R., Chu, G. Significance analysis of microarrays applied to the ionizing radiation response. Proc. Natl. Acad. Sci. U.S.A. 98, 5116-5121 (2001).
- Storey, J. D., Tibshirani, R. Statistical significance for genomewide studies. Proc. Natl. Acad. Sci. U.S.A. 100, 9440-9445 (2003).
- Team, R. D. C. R: A language and environment for statistical computing. , (2008).
- Gentleman, R. C., et al. Bioconductor: open software development for computational biology and bioinformatics. Genome Biol. 5, R80(2004).
- de Parseval, N., Lazar, V., Casella, J. F., Benit, L., Heidmann, T. Survey of human genes of retroviral origin: identification and transcriptome of the genes with coding capacity for complete envelope proteins. J. Virol. 77, 10414-10422 (2003).
- Wang-Johanning, F., et al. Detecting the expression of human endogenous retrovirus E envelope transcripts in human prostate adenocarcinoma. Cancer. 98, 187-197 (2003).
- Smallwood, A., et al. Temporal regulation of the expression of syncytin (HERV-W), maternally imprinted PEG10, and SGCE in human placenta. Biol. Reprod. 69, 286-293 (2003).
- Okahara, G., et al. Expression analyses of human endogenous retroviruses (HERVs): tissue-specific and developmental stage-dependent expression of HERVs. Genomics. 84, 982-990 (2004).
- Buscher, K., et al. Expression of human endogenous retrovirus K in melanomas and melanoma cell lines. Cancer Res. 65, 4172-4180 (2005).
- Forsman, A., et al. Development of broadly targeted human endogenous gammaretroviral pol-based real time PCRs Quantitation of RNA expression in human tissues. J. Virol. Methods. 129, 16-30 (2005).
- Muradrasoli, S., Forsman, A., Hu, L., Blikstad, V., Blomberg, J. Development of real-time PCRs for detection and quantitation of human MMTV-like (HML) sequences HML expression in human tissues. J. Virol. Methods. 136, 83-92 (2006).
- Seifarth, W., et al. Comprehensive analysis of human endogenous retrovirus transcriptional activity in human tissues with a retrovirus-specific microarray. J. Virol. 79, 341-352 (2005).
- Pichon, J. P., Bonnaud, B., Mallet, F. Quantitative multiplex degenerate PCR for human endogenous retrovirus expression profiling. Nat. Protoc. 1, 2831-2838 (2006).
- Flockerzi, A., et al. Expression pattern analysis of transcribed HERV sequences is complicated by ex vivo recombination. Retrovirology. 4, 39(2007).
- Flockerzi, A., et al. Expression patterns of transcribed human endogenous retrovirus HERV-K(HML-2) loci in human tissues and the need for a HERV Transcriptome Project. BMC Genomics. 9, 354(2008).
- Gosenca, D., et al. HERV-E-Mediated Modulation of PLA2G4A Transcription in Urothelial Carcinoma. PLoS ONE. 7, e49341(2012).
- Buzdin, A., Kovalskaya-Alexandrova, E., Gogvadze, E., Sverdlov, E. GREM, a technique for genome-wide isolation and quantitative analysis of promoter active repeats. Nucleic Acids Res. 34, e67(2006).
- Buzdin, A., Kovalskaya-Alexandrova, E., Gogvadze, E., Sverdlov, E. At least 50% of human-specific HERV-K (HML-2) long terminal repeats serve in vivo as active promoters for host nonrepetitive DNA transcription. J. Virol. 80, 10752-10762 (2006).
- Gimenez, J., et al. Custom human endogenous retroviruses dedicated microarray identifies self-induced HERV-W family elements reactivated in testicular cancer upon methylation control. Nucleic Acids Res. 38, 2229-2246 (2010).
- Pérot, P., et al. Microarray-based sketches of the HERV transcriptome landscape. PLoS ONE. 7, e40194(2012).
- Fehlbaum, P., Guihal, C., Bracco, L., Cochet, O. A microarray configuration to quantify expression levels and relative abundance of splice variants. Nucleic Acids Res. 33, e47(2005).
- Prensner, J. R., et al. Transcriptome sequencing across a prostate cancer cohort identifies PCAT-1, an unannotated lincRNA implicated in disease progression. Nat. Biotechnol. 29, 742-749 (2011).
- Tomlins, S. A., et al. Distinct classes of chromosomal rearrangements create oncogenic ETS gene fusions in prostate cancer. Nature. 448, 595-599 (2007).
- Hermans, K. G., et al. Truncated ETV1, fused to novel tissue-specific genes, and full-length ETV1 in prostate cancer. Cancer Res. 68, 7541-7549 (2008).
- Brodziak, A., et al. The role of human endogenous retroviruses in the pathogenesis of autoimmune diseases. Med. Sci. Monit. 18, RA80-RA88 (2012).
- Mullins, C. S., Linnebacher, M. Human endogenous retroviruses and cancer: Causality and therapeutic possibilities. World J. Gastroenterol. 18, 6027-6035 (2012).
- Young, G. R., et al. Resurrection of endogenous retroviruses in antibody-deficient mice. Nature. 491, 774-778 (2012).
- Vander Kuyl, A. C. HIV infection and HERV expression: a review. Retrovirology. 9, 6(2012).
- Smit, A. F. A., Hubley, R. RepeatMasker Open-3.0. , (1996).
- Navarro, G., Raffinot, M. Flexible Pattern Matching in Strings: Practical On-Line Search Algorithms for Texts and Biological Sequences. , Cambridge University Press. (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados