É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Medir a estabilidade da proteína em Viver embriões Zebrafish Usando Fluorescência Decay Após Fotoconversão (FDAP)
Neste Artigo
Resumo
Os níveis de proteína em células e tecidos são muitas vezes fortemente regulada pelo equilíbrio da produção de proteínas e de apuramento. Usando Fluorescência Decay Após Fotoconversão (FDAP), a cinética de apuramento de proteínas pode ser medida experimentalmente in vivo.
Resumo
A estabilidade da proteína influencia muitos aspectos da biologia, e medir a cinética limpeza de proteínas pode fornecer importantes insights sobre sistemas biológicos. Em experiências FDAP, o apuramento das proteínas nos organismos vivos podem ser medidos. Uma proteína de interesse é marcado com uma proteína fluorescente photoconvertible, expressa in vivo e photoconverted, e a diminuição do sinal photoconverted ao longo do tempo é monitorizada. Os dados são então equipado com um modelo de depuração adequada para determinar a meia-vida da proteína. Importante, a cinética de depuração de populações de proteína em diferentes compartimentos do organismo pode ser examinado separadamente aplicando máscaras compartimentais. Esta abordagem tem sido utilizada para determinar os intra- e extracelulares meias-vidas de proteínas de sinalização segregadas durante o desenvolvimento do peixe-zebra. Aqui, descrevemos um protocolo para experimentos FDAP em embriões de peixe-zebra. Deve ser possível usar FDAP para determinar a cinética de depuração dequalquer proteína de qualquer organismo taggable opticamente acessíveis.
Introdução
Os níveis de proteínas em células e organismos são determinados através das suas taxas de produção e de depuração. Proteína de semi-vida pode variar de minutos a dias 1-4. Em muitos sistemas biológicos, a estabilização ou limpeza de proteínas-chave tem efeitos importantes sobre a atividade celular. Modulação da estabilidade da proteína intracelular é necessário para a progressão do ciclo celular 5,6, sinalização de desenvolvimento 7-9, apoptose 10, e a função normal e manutenção de neurónios 11,12. A estabilidade da proteína extracelular afecta a distribuição e disponibilidade de proteínas segregadas, tais como morfogénios 13,14, dentro de um tecido.
Ao longo das últimas décadas, a estabilidade da proteína foi avaliado principalmente em cultura de células usando radioativo pulso de rotulagem ou experiências de perseguição cicloheximida 15. Nesses experimentos pulse-chase, as células são transitoriamente ou expostos a um "pulso" de amino radioativoprecursores de ácidos que são incorporados em proteínas sintetizadas de novo, ou eles são expostos a ciclo-heximida, a qual inibe a síntese proteica. As células em cultura são então recolhidos em diferentes pontos de tempo, e, ou imunoprecipitação seguido por autorradiografia (em experiências de pulse-chase radioactivos) ou imunodetecção (em experiências de cicloheximida) é utilizada para quantificar a depuração de proteína ao longo do tempo.
Ensaios de estabilidade proteína convencionais têm várias deficiências. Primeiro, as proteínas nestes ensaios frequentemente não são expressos nos seus ambientes endógenos, mas sim em cultura de tecidos e por vezes em células a partir de espécies diferentes. Para proteínas cuja estabilidade é dependente do contexto, esta abordagem é problemática. Em segundo lugar, não é possível seguir a depuração de proteínas em células individuais ou organismos ao longo do tempo, e os dados obtidos nestes ensaios reflecte uma média de diferentes populações de células em diferentes pontos de tempo. Como as células individuais podem ter começadocom diferentes quantidades de proteína, pode ter retomado o marcador radioactivo ou cycloheximide em momentos diferentes, ou podem ter diferentes cinética de depuração, tais dados agregados pode ser enganador. Finalmente, no caso das experiências de perseguição cicloheximida, adição do inibidor da síntese de proteína pode ter efeitos fisiológicos não desejadas que podem alterar a estabilidade da proteína 16-18 artificialmente. Estas deficiências podem ser evitadas através do uso de fluorescência Decay Após Fotoconversão (FDAP), uma técnica que utiliza proteínas photoconvertible para medir folga proteína dinamicamente nos organismos vivos 19-25 (ver discussão para limitações da técnica FDAP).
Photoconvertible proteínas são proteínas fluorescentes cuja excitação e emissão propriedades mudam após exposição a determinados comprimentos de onda de luz 26. Uma variante comumente usado é Dendra2, uma proteína photoconvertible "verde-to-vermelho", que tem inicialmente excitação de emissão e propriedades semelhantes às proteínas fluorescentes verdes, mas após a exposição à radiação UV luz- "fotoconversão" -sua excitação / emissão de propriedades tornam-se semelhantes às das proteínas fluorescentes vermelhas 23,27. Importante, nova proteína produzida após fotoconversão não têm as mesmas propriedades de excitação / emissão como a proteína photoconverted, permitindo a dissociação da produção de depuração e em cima fotoconversão e observação de apenas um conjunto de proteínas photoconverted. Marcação de proteínas de interesse com proteínas photoconvertible, portanto, fornece uma maneira conveniente de pulso de rótulo proteínas intactas, organismos vivos opticamente acessíveis.
Em ensaios FDAP (Figura 1A), uma proteína de interesse é marcado com uma proteína photoconvertible e expressa num organismo vivo (Figura 1B). A proteína de fusão é photoconverted, e a diminuição do sinal de photoconverted ao longo do tempo é monitorizado por fluorescenmicroscopia ce (Figura 1C). Os dados são então montados com um modelo apropriado para determinar a meia-vida da proteína de fusão (Figura 1D).
O ensaio aqui descrito FDAP foi desenhado para determinar as semividas extracelulares de proteínas de sinalização segregadas em embriões de peixe-zebra durante a embriogénese precoce 19. No entanto, esta abordagem pode ser adaptada para qualquer organismo modelo transparente que tolera imagens ao vivo, e pode ser utilizado para monitorar a depuração de qualquer proteína intracelular ou extracelular taggable. As variações da técnica aqui descrita não tem sido realizada em células em cultura e 20,23 Drosophila 22 e 21 embriões de ratinho.
Protocolo
1. Gerando um Photoconvertible construção de fusão e injetando dechorionated embriões Zebrafish
- Gerar uma construção funcional contendo a proteína de interesse fundida com uma proteína photoconvertible verde-a-vermelho (ver discussão), em seguida, utilizar a transcrição in vitro para gerar ARNm que codifica tapado a proteína de fusão como em Muller et al., 2012 19.
- Use pronase para remover os córions de cerca de 30 embriões de peixe zebra na fase de uma célula. Alternativamente, dechorionate manualmente embriões usando uma pinça 28.
Nota: Os embriões devem ser dechorionated para imagens subseqüente. Se desejado, os embriões podem ser injectados através do córion dechorionated e depois, imediatamente antes da imagiologia.- Faça uma solução 5 mg / ml estoque de pronase de Streptomyces griseus em meio embrião de peixe-zebra padrão de 19. Rocha suavemente a solução à TA durante 10 min para permitir a protease para dissolver. Alíquota de 2 ml em tubos de microcentrífuga e congelamento a -20 ° C.
- Transferência de embriões nos estágios de uma célula para um vidro 5 cm de diâmetro ou revestidos por placa de Petri de plástico de agarose contendo ~ 8 mL de meio de embrião. Adicionar 2 ml de solução de estoque descongelado pronase para o prato e incubar à temperatura ambiente durante 5 a 10 min.
- Evite expor embriões para o ar ou plástico, como o contato com qualquer um fará com que embriões dechorionated à ruptura. Encha um copo de vidro de 200 ml com meio embrião. Transferir os embriões para o béquer através da inclinação da placa de Petri enquanto submergindo-o no meio.
- Depois de os embriões terem assentado no fundo do copo, derramar a maior parte do meio de embrião, depois verter meio fresco embrião para o copo. A roda leve do meio de verter para o copo faz com embriões a perder seus córions enfraquecidos.
- Repita o passo 1.2.4.
- Transferir os embriões dechorionated a uma injeção prato revestido de agarose 29 usando uma pipeta Pasteur de vidro com uma ponta flambado. Chamejantea ponta da pipeta impede bordas irregulares a partir de embriões mutilações.
- Co-injetar o mRNA e a 3 kDa Alexa488-dextrano conjugado 29,30 (Figura 1B; veja a discussão para sugeriu mRNA e quantidades Alexa488-dextrano). Injectar directamente no centro da célula (não a gema) para assegurar uma distribuição de ARNm e corante fluorescente uma vez começa a clivagem.
Nota: O sinal Alexa488 vai ser utilizado durante a análise dos dados para gerar máscaras compartimentados de modo a distinguir entre a fluorescência intracelular e extracelular. - Transferência de embriões injectados a uma revestidos por 1-2% de agarose poço de uma placa de seis poços de plástico cheio com meio de embrião. Incubar no escuro a 28 ° C até embriões ter atingido o estágio final de esfera 31 (cerca de 5 horas após a fertilização). Verifique embriões, cada um para 2 horas sob um microscópio estereoscópico e remover todos os detritos gerados por embriões que já morreram.
2. Montagem embriões Zebrafish para Photoconversion e imagem em um microscópio invertido confocal
- Use uma lupa para identificar 1-5 embriões saudáveis, e usar uma pipeta Pasteur de vidro com uma ponta flambado para removê-los do prato.
- Suavemente ejectar os embriões para um tubo de microcentrífuga contendo ~ 1 ml de 1% derretido baixo ponto de fusão de agarose na forma de embrião 1x Danieau (ver Materiais Lista) (Figura 2A).
Nota: Agarose deve ter uma temperatura entre 40 e 42 ° C; temperaturas mais elevadas podem danificar os embriões. - Desenhar os embriões para a pipeta, juntamente com alguns agarose. Gentilmente ejetar o agarose e embriões para a tampa de vidro de um prato fundo de vidro (Figura 2B). Assegure-se que a espessura do vidro de cobertura é compatível com o objectivo no microscópio confocal.
- Re-utilizar a pipeta de vidro, se desejar. Para limpar a agarose residual para fora da pipeta e evitar o entupimento, rapidamente médio pipeta embrião cima e para baixo.Coloque um tubo de 15 ml cheio com ~ 5 ml de meio ao lado do embrião estereomicroscópio para esta finalidade.
- Use uma sonda de metal para posicionar os embriões para que o polo animal (blastoderm) enfrenta a tampa de vidro. Trabalhe rapidamente desde a agarose vai solidificar em 20-30 seg. Use a lupa para monitorar as posições dos embriões e reajustar se necessário até que a agarose endurece.
- Repita os passos 2,1-2,4 até que o número desejado de embriões tiver sido montado.
Nota: Em uma experiência típica, quatro gotas de agarose contendo quatro ou cinco embriões cada um vai caber facilmente na tampa de vidro. Cerca de 16 embriões pode ser trabalhada durante um único ensaio ideal (Figura 2C). - Quando a agarose solidificou, encha o prato com fundo de vidro com meio embrião de 1x Danieau.
3. Photoconverting e medindo a diminuição do sinal Photoconverted
Um objectivo 25X ou 40X de água ié adequado para o tamanho e índice de refracção de embriões de peixes-zebra. É melhor utilizar óleo de imersão com o mesmo índice de refracção de água em vez de água real, uma vez que a água irá evaporar-se durante o decurso da experiência de cinco horas. Assegure-se que o óleo de imersão é concebido para ser usado com um objectivo de água (não de petróleo).
- Coloque uma grande gota de óleo de imersão sobre o objetivo de garantir que a película de óleo entre o objetivo e tampa de vidro não vai quebrar como palco move-se para diferentes posições do embrião durante a geração de imagens. Firmemente colocar o prato com fundo de vidro para o palco para que o prato não vai mudar quando o estágio de movimentos. Se possível, utilizar uma fase aquecida a 28 ° C, a temperatura óptima para o desenvolvimento do peixe-zebra.
- Definir a posição de cada embrião no pacote de software do microscópio confocal. Ajuste o z-depth para cada embrião, e tentar alvejar a menos no mesmo plano em cada embrião.
Nota: Cerca de 30 mm a partir do pólo animal é um good profundidade desde a esta profundidade da camada envolvente do embrião pode ser evitada, a área de imagem é maximizada, e dispersão de luz é mínima. Uma fatia óptico único, com uma espessura de 3,3 mm ~ fornece dados suficientes; não há necessidade de adquirir uma pilha de z (ver item 5). - Recolhe dois sinais ao longo do experimento: o sinal "verde" do Alexa488-dextrano conjugado-que irá ser utilizado durante a análise de dados para isolar extracelular e intracelular de fluorescência e o sinal "vermelho" a partir da proteína de fusão após o que é photoconverted.
- Excitar Alexa488 usando um laser de 488 nm e a fluorescência emitida a recolher entre ~ 500 e 540 nm. Nota: Depois de fotoconversão, muitos verde-to-vermelho proteínas photoconvertible (eg, Dendra2) pode ser animado com um laser de 543 nm e emitem fluorescência entre ~ 550 e 650 nm. Ajuste conforme necessário, com base na proteína photoconvertible usado.
- Adquirir "pré-fotoconversão"Imagens, e configurar o software do microscópio confocal a imagem cada uma das posições previamente definidos (a partir do passo 3.2), com as condições de imagem apropriados a cada 10 ou 20 minutos para um curso de tempo de cinco horas (ver seção 5 e Discussão).
- Para photoconvert a proteína de fusão, mudar para uma objectiva 10X e expor os grupos de embriões a luz UV proveniente de uma lâmpada de arco de mercúrio com um filtro de ~ 300-400 nm de excitação com a intensidade de 100% durante 2 minutos. Mudar o foco ao longo do eixo z para promover fotoconversão uniforme (ver item 5). Certifique-se que o óleo de imersão não escorrer para a objetiva de 10x durante fotoconversão.
Nota: O deslocamento do foco durante fotoconversão pode ser automatizada, a fim de evitar a variabilidade entre os experimentadores. - Volte para o objectivo 25X ou 40X imediatamente após fotoconversão. Certifique-se de que as posições previamente definidos a partir do passo 3.2 ainda são precisos. Se o prato deslocado durante photoconversion, re-definir as posições dos embriões.
- Inicie o programa criado no passo 3.4 e permitir imaging para continuar por 5 horas. Registar o tempo decorrido entre o início e fotoconversão de formação de imagens para cada embrião.
- Verifique ocasionalmente no experimento. Monitorar o nível de meio de Danieau e adicionar mais, se necessário. Reinicie o software se estagnou.
- A fim de determinar os valores da fluorescência de fundo que serão utilizadas durante a análise dos dados para estimar a assíntota de um modelo exponencial decrescente, incluem alguns embriões que foram injectados com Alexa488-dextrano, mas não ARNm no experimento. Para determinar o ruído do instrumento, a qual também irá ser utilizado durante a análise de dados subsequente, adquirir uma imagem na ausência de uma amostra.
4. Analisando os dados usando PyFDAP
- Inspecione visualmente o tempo de conjuntos de dados de curso de cada embrião. Descartar conjuntos de dados a partir de embriões que morreram durante imaging, que mudou significativamente, que têm níveis muito baixos de sinal photoconverted, ou que contêm regiões de células que se parecem incomum e pararam de se mover e dividindo (típico de embriões feridos ou doentes).
Nota: Ocasionalmente, bolhas no óleo de imersão ou outros artefatos aparece em uma ou duas imagens em um conjunto de dados de outra forma utilizável. Anote todas as imagens que contêm artefatos; eles serão descartados mais tarde, e os restantes pontos de tempo de tal conjunto de dados ainda podem ser analisados. - Use a PyFDAP pacote de software baseado em Python para analisar os dados FDAP. PyFDAP calcula meias-vidas por determinação da intensidade média de fluorescência vermelha intracelular e extracelular em cada imagem e ajuste dos dados com uma função exponencialmente decrescente 56 (Figura 3).
- Baixe PyFDAP (veja Lista de Materiais).
- Uso PyFDAP para separar sinal intracelular e extracelular photoconverted (Figura 3A, B). NósÊ O sinal Alexa488, que é estritamente intracelular, para criar uma máscara de intracelular. Aplique esta máscara para a imagem do canal vermelho correspondente para evitar pixels intracelulares de ser considerado para o cálculo da intensidade média extracelular. Para medir a intensidade intracelular média, inverter a máscara.
- Em PyFDAP, exibir as imagens mascarados gerados na etapa 4.2.2. Inspecione visualmente essas imagens e descartar conjuntos de dados em que as máscaras não distinguir com precisão intracelular do espaço extracelular (este deve ser rara, note que as membranas celulares estão incluídas nas imagens em que o espaço extracelular tem sido mascarado, mas eles poderiam ser removidos através da alteração do limiar algoritmo ou através da introdução de uma máscara de membrana (por exemplo, utilizando uma membrana-PCP)). Também descartar quaisquer imagens individuais contendo artefatos (por exemplo, bolhas no óleo de imersão) identificados na etapa 4.1.
- Use PyFDAP para calcular médias intensidades de fluorescência intra e extracelulares para each imagem. PyFDAP calcula estas médias pela soma das intensidades de pixels que caem fora da máscara e dividindo pelo número total de pixels somados.
- Coloque os dados de fluorescência (Figura 3C), com a seguinte função exponencial:
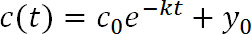
onde t é o tempo de pós-fotoconversão, c (t) é a intensidade de um dado valor de t, c 0 é a intensidade em t = 0, k é a constante de velocidade de eliminação, e y é 0 a assíntota que se aproxima a função como fluorescência diminui (Figura 1D). y 0 pode ser restringida com base nas medições em 19 passo 3.8. - Use PyFDAP para calcular a proteína extracelular e intracelular de semi-vida (τ) a partir das constantes de velocidade de depuração (k) utilizando a seguinte relação:
5. Experimentos controlo para avaliar Fotodegradação, inadvertida Fotoconversão e Fotoconversão Uniformidade
- Avaliando fotodegradação
Nota: A fotodegradação pode causar uma diminuição na intensidade de fluorescência artefacto que reflecte as propriedades de branqueamento de proteína fluorescente para além da eliminação da proteína de interesse.- Para avaliar a possível fotodegradação, execute uma série de experimentos FDAP com intervalos de 10 min entre imagem e um segundo conjunto, com 20 minutos de intervalo entre as imagens (Figura 4). Analisar os dados de ambos os conjuntos de experimentos usando PyFDAP conforme descrito na Seção 4.
- Compare as meias-vidas dos experimentos de 10 e 20 min de intervalo. Meia-vida de 20 min de intervalo experimentos indicam fotodegradação significativa. Se as semividas de ambas as experiências são idênticos, fotobleaching não é uma preocupação significativa.
- Alternativamente, avaliar a fotodegradação através da aquisição de uma série de ~ 30 imagens imediatamente após fotoconversão. Uma diminuição significativa na intensidade de fluorescência indica fotodegradação significativa.
- Se fotodegradação é detectado, usar o poder do laser inferior, diminuir o tempo de criação de imagens, ou considerar o uso de uma proteína photoconvertible mais fotoestável 32.
- Avaliando fotoconversão inadvertida.
Nota: pode ser Dendra2 photoconverted usando iluminação de 488 nm 27. Quando excitante Alexa488 com o laser de 488 nm, tal como descrito no passo 3.3.1, fotoconversão inadvertido e, por conseguinte, um aumento artefactual na semi-vida aparente da proteína de interesse é possível. No entanto, nós e os outros 33 descobriram que 488 nm iluminação é um método ineficiente de fotoconversão em embriões de peixe-zebra.- Use a experiência de controlo descrito na etapa 5.1.1 para detectar fotoconversão inadvertida. Compare as meias-vidas dos experimentos de 10 e 20 min de intervalo. Shorter meias-vidas de 20 min de intervalo experimentos indicam fotoconversão inadvertida significativo. Se as meias-vidas de ambas as experiências são idênticos, fotoconversão inadvertida não é uma preocupação significativa.
- Se fotoconversão inadvertida for detectado, use uma menor potência do laser de 488 nm e os tempos de imagem mais curtos para evitar inadvertidamente photoconverting Dendra2.
- Avaliando uniformidade fotoconversão.
Nota: Se fotoconversão está inclinado para o polo animal do embrião, a diminuição na fluorescência será influenciada pela difusão da proteína ou do movimento das células em planos mais profundas (Figura 5A).- Para determinar se fotoconversão é uniforme, expressar uma proteína secretada photoconvertible (para as experiências com as proteínas de fusão extracelulares) ou uma proteína citoplasmática photoconvertible (para as experiências com as proteínas de fusão intracelulares). Photoconvert como de costume, tgalinha adquirir uma pilha z engloba a maior parte da blastoderme a cada 20 min durante 80 min.
- Se fotoconversão é tendencioso em direção ao pólo animal, a intensidade de fluorescência em planos mais profundos irá aumentar ao longo do tempo devido à difusão ou movimento celular (Figura 5B). Se não uniforme fotoconversão é detectado, o foco mais profundo nos embriões durante fotoconversão.
Resultados
FDAP tem sido usado para determinar a meia-vida de proteínas de sinalização extracelular em embriões de peixe-zebra 19. Uma destas proteínas, estrabismo, induz a expressão de genes durante o desenvolvimento embrionário mesendodermal 34. Estrabismo-Dendra2 activa a expressão de genes mesendodermal em níveis semelhantes aos estrabismo não marcado, como demonstrado por qRT-PCR e hibridização in situ em 19 ensaios. Os embriões foram co-injectados com Alexa488-dextrano e...
Discussão
O sucesso de uma experiência FDAP baseia-se na geração de uma proteína de fusão photoconvertible funcional. Marcação de uma proteína pode afectar a sua actividade biológica e / ou propriedades biofísicas, incluindo a sua localização, a solubilidade, a estabilidade e 36-41. Ser preparados para testar a actividade de várias construções de fusão diferentes, a fim de encontrar um que seja activo. Verificou-se que a mudança da posição da proteína photoconvertible em relação à proteína de in...
Divulgações
Os autores não têm conflitos de divulgar.
Agradecimentos
Os autores gostariam de agradecer Jeffrey Farrell, James Gagnon, e Jennifer Bergmann para comentários sobre o manuscrito. KWR foi apoiado pelo Programa de Bolsas de Pós-Graduação National Science Foundation Research durante o desenvolvimento do ensaio FDAP. Este trabalho foi financiado por doações do NIH para AFS e por doações da Fundação Alemã de Pesquisa (Emmy Noether Programa), a Sociedade Max Planck, e do Programa Human Frontier Science (Prémio Carreira de Desenvolvimento) para PM.
Materiais
| Name | Company | Catalog Number | Comments |
| PyFDAP (download from the following website: http://people.tuebingen.mpg.de/mueller-lab) | Install and operate using the instructions provided on the PyFDAP website; PyFDAP is compatible with Linux, Mac, and Windows operating systems. | ||
| mMessage mMachine Sp6 Transcription Kit | Life Technologies | AM1340 | To generate capped mRNA for injection into embryos |
| Alexa488-dextran conjugate, 3 kDa | Life Technologies | D34682 | Co-inject with mRNA to create intracellular and extracellular masks |
| 6-well plastic dish | BD Falcon | Incubate embryos in agarose-coated wells until ready for mounting | |
| Embryo medium | 250 mg/L Instant Ocean salt, 1 mg/L methylene blue in reverse osmosis water adjusted to pH 7 with NaHCO3 | ||
| Protease from Streptomyces griseus | Sigma | P5147 | Make a 5 mg/ml stock and use at 1 mg/ml to dechorionate embryos at the one-cell stage |
| 5 cm diameter glass Petri dish | For embryo dechorionation | ||
| 200 ml glass beaker | For embryo dechorionation | ||
| Microinjection apparatus | For injection of mRNA and dye into embryos at the one-cell stage | ||
| Stereomicroscope | For injecting and mounting embryos | ||
| 1x Danieau's medium | Dilute low melting point agarose and perform imaging in this medium; recipe: 0.2 mm filtered solution of 58 mM NaCl, 0.7 mM KCl, 0.4 mM MgSO4, 0.3 mM CaCl2, 5 mM HEPES pH 7.2 | ||
| UltraPure low melting point agarose | Invitrogen | 16520-100 | For mounting embryos; use at a concentration of 1% in Danieau's medium: add 200 mg to 20 ml Danieau's medium, microwave until dissolved, then aliquot 1 ml into microcentrifuge tubes; aliquots can be stored at 4 °C, re-melted at 70 °C, and cooled to 40–42 °C when ready to use |
| Glass Pasteur pipette | Kimble Chase (via Fisher) | 63A53WT | For mounting embryos; flame the tip to prevent jagged edges from injuring embryos |
| Metal probe | For positioning embryos during mounting | ||
| Glass bottom dishes | MatTek | P35G-1.5-14-C | Use the appropriate cover glass thickness for your objective; part number listed here is for cover glass No. 1.5 |
| 15 ml tube filled with ~5 ml embryo medium | BD Falcon | For rinsing residual agarose from the Pasteur pipette | |
| Inverted laser scanning confocal microscope | A mercury arc lamp, 488 nm laser, 543 nm laser, and the appropriate filter sets are required | ||
| Heated stage | To maintain embryos at the optimal temperature of 28 °C during the experiment | ||
| Confocal software capable of time-lapse imaging | Must be able to define multiple positions and automatically image them at defined intervals | ||
| 25X or 40X water objective | Objective for imaging | ||
| 10X air objective | Objective for photoconversion | ||
| Immersion oil | Immersion oil with the same refractive index as water |
Referências
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Boisvert, F. M., et al. A Quantitative Spatial Proteomics Analysis of Proteome Turnover in Human Cells. Molecular & Cellular Proteomics. 11 (3), 011429 (2012).
- Belle, A., Tanay, A., Bitincka, L., Shamir, R., O'Shea, E. K. Quantification of protein half-lives in the budding yeast proteome. Proceedings of the National Academy of Sciences of the United States of America. 103 (35), 13004-13009 (2006).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764-768 (2011).
- Parry, D. H., O'Farrell, P. H. The schedule of destruction of three mitotic cyclins can dictate the timing of events during exit from mitosis. Current Biology. 11 (9), 671-683 (2001).
- Holloway, S. L., Glotzer, M., King, R. W., Murray, A. W. Anaphase is initiated by proteolysis rather than by the inactivation of maturation-promoting factor. Cell. 73 (7), 1393-1402 (1993).
- Dharmasiri, N., Estelle, M. Auxin signaling and regulated protein degradation. Trends Plant Sci. 9 (6), 302-308 (2004).
- MacDonald, B. T., Tamai, K., He, X. Wnt/beta-catenin signaling: components, mechanisms, and diseases. Developmental Cell. 17 (1), 9-26 (2009).
- Chen, X., et al. Processing and turnover of the Hedgehog protein in the endoplasmic reticulum. The Journal of Cell Biology. 192 (5), 825-838 (2011).
- Elmore, S. Apoptosis: A Review of Programmed Cell Death. Toxicologic Pathology. 35 (4), 495-516 (2007).
- Yi, J. J., Ehlers, M. D. Emerging Roles for Ubiquitin and Protein Degradation in Neuronal Function. Pharmacological Reviews. 59 (1), 14-39 (2007).
- Rubinsztein, D. C. The roles of intracellular protein-degradation pathways in neurodegeneration. Nature. 443 (7113), 780-786 (2006).
- Müller, P., Schier, A. F. Extracellular Movement of Signaling Molecules. Developmental Cell. 21 (1), 145-158 (2011).
- Eldar, A., Rosin, D., Shilo, B. -. Z., Barkai, N. Self-enhanced ligand degradation underlies robustness of morphogen gradients. Developmental Cell. 5 (4), 635-646 (2003).
- Zhou, P. Determining protein half-lives. Methods In Molecular Biology. 284, 67-77 (2004).
- Woodside, K. H. Effects of cycloheximide on protein degradation and gluconeogenesis in the perfused rat liver. Biochim Biophys Acta. 421 (1), 70-79 (1976).
- Schimke, R. T., Doyle, D. Control of enzyme levels in animal tissues. Annual Review of Biochemistry. 39, 929-976 (1970).
- Kenney, F. T. Turnover of rat liver tyrosine transaminase: stabilization after inhibition of protein synthesis. Science. 156 (3774), 525-528 (1967).
- Müller, P., et al. Differential Diffusivity of Nodal and Lefty Underlies a Reaction-Diffusion Patterning System. Science. 336 (6082), 721-724 (2012).
- Kiuchi, T., Nagai, T., Ohashi, K., Mizuno, K. Measurements of spatiotemporal changes in G-actin concentration reveal its effect on stimulus-induced actin assembly and lamellipodium extension. The Journal of Cell Biology. 193 (2), 365-380 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biology. 13 (2), 17-123 (2011).
- Drocco, J. A., Grimm, O., Tank, D. W., Wieschaus, E. Measurement and Perturbation of Morphogen Lifetime: Effects on Gradient Shape. Biophys J. 101 (8), 1807-1815 (2011).
- Zhang, L., et al. Method for real-time monitoring of protein degradation at the single cell level. BioTechniques. 42 (4), 446-450 (2007).
- Miyawaki, A. Proteins on the move: insights gained from fluorescent protein technologies. Nat Rev Mol Cell Biol. 12 (10), 636-668 (2011).
- Pantazis, P., Supatto, W. Advances in whole-embryo imaging: a quantitative transition is underway. Nat Rev Mol Cell Biol. 15 (5), 327-339 (2014).
- Lukyanov, K. A., Chudakov, D. M., Lukyanov, S., Verkhusha, V. V. Innovation: Photoactivatable fluorescent proteins. Nat Rev Mol Cell Biol. 6 (11), 885-891 (2005).
- Gurskaya, N. G., et al. Engineering of a monomeric green-to-red photoactivatable fluorescent protein induced by blue light. Nature Biotechnology. 24 (4), 461-465 (2006).
- Zou, J., Wei, X. Transplantation of GFP-expressing Blastomeres for Live Imaging of Retinal and Brain Development in Chimeric Zebrafish Embryos. Journal of Visualized Experiments. (41), (2010).
- Yuan, S., Sun, Z. Microinjection of mRNA and Morpholino Antisense Oligonucleotides in Zebrafish Embryos. Journal of Visualized Experiments. (27), (2009).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of Zebrafish Embryos to Analyze Gene Function. Journal of Visualized Experiments. (25), (2009).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Developmental Dynamics. 203 (3), (1995).
- McKinney, S. A., Murphy, C. S., Hazelwood, K. L., Davidson, M. W., Looger, L. L. A bright and photostable photoconvertible fluorescent protein. Nature Methods. 6 (2), 131-133 (2009).
- Dempsey, W. P., Qin, H., Pantazis, P. In Vivo Cell Tracking Using PhOTO Zebrafish. Methods in Molecular Biology. 1148, 217-228 (2014).
- Schier, A. F. Nodal Morphogens). Cold Spring Harbor Perspectives in Biology. 1 (5), a003459-a003459 (2009).
- Otsu, N. A threshold selection method from gray-level histograms. IEEE Trans Syst Man Cybern. 9 (1), 62-66 (1979).
- Pédelacq, J. -. D., Cabantous, S., Tran, T., Terwilliger, T. C., Waldo, G. S. Engineering and characterization of a superfolder green fluorescent protein. Nature Biotechnology. 24 (1), 79-88 (2005).
- Swulius, M. T., Jensen, G. J. The Helical MreB Cytoskeleton in Escherichia coli MC1000/pLE7 Is an Artifact of the N-Terminal Yellow Fluorescent Protein Tag. Journal of Bacteriology. 194 (23), 6382-6386 (2012).
- Landgraf, D., Okumus, B., Chien, P., Baker, T. A., Paulsson, J. Segregation of molecules at cell division reveals native protein localization. Nature Methods. 9 (5), 480-482 (2012).
- Stadler, C., et al. Immunofluorescence and fluorescent-protein tagging show high correlation for protein localization in mammalian cells. Nature Methods. 10 (4), 315-323 (2013).
- Quattrocchio, F. M., Spelt, C., Koes, R. Transgenes and protein localization: myths and legends. Trends Plant Sci. 18 (9), 473-476 (2013).
- Morimoto, Y. V., Kojima, S., Namba, K., Minamino, T. M153R Mutation in a pH-Sensitive Green Fluorescent Protein Stabilizes Its Fusion Proteins. PLoS ONE. 6 (5), e19598 (2011).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2 (12), 905-909 (2005).
- Waters, J. C. Accuracy and precision in quantitative fluorescence microscopy. The Journal of Cell Biology. 185 (7), 1135-1148 (2009).
- Moll, U. M., Petrenko, O. The MDM2-p53 interaction. Mol Cancer Res. 1 (14), 1001-1008 (2003).
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 24 (1), 142-153 (2014).
- Bedell, V. M., et al. In vivo genome editing using a high-efficiency TALEN system. Nature. 491 (7422), 114-118 (2012).
- Hruscha, A., et al. Efficient CRISPR/Cas9 genome editing with low off-target effects in zebrafish. Development. 140 (24), 4982-2987 (2013).
- Hwang, W. Y., et al. Heritable and Precise Zebrafish Genome Editing Using a CRISPR-Cas System. PLoS ONE. 8 (7), 68708 (2013).
- Hwang, W. Y., et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology. 31 (3), 227-229 (2013).
- Zu, Y., et al. TALEN-mediated precise genome modification by homologous recombination in zebrafish. Nature Methods. 10 (4), 329-331 (2013).
- Keller, P. J., Schmidt, A. D., Wittbrodt, J., Stelzer, E. H. K. Reconstruction of Zebrafish Early Embryonic Development by Scanned Light Sheet Microscopy. Science. 322 (5904), 1064-1069 (2008).
- Blanchet, M. H., et al. Cripto Localizes Nodal at the Limiting Membrane of Early Endosomes. Science Signaling. 1 (45), ra13-ra13 (2008).
- Jullien, J., Gurdon, J. Morphogen gradient interpretation by a regulated trafficking step during ligand-receptor transduction. Genes & Development. 19 (22), 2682-2694 (2005).
- Incardona, J. P., et al. Receptor-mediated endocytosis of soluble and membrane-tethered Sonic hedgehog by Patched-1. Proceedings of the National Academy of Sciences of the United States of America. 97 (22), 12044-12049 (2000).
- Scholpp, S., Brand, M. Endocytosis Controls Spreading and Effective Signaling Range of Fgf8 Protein. Current Biology. 14 (20), 1834-1841 (2004).
- Bläßle, A., Müller, P. . PyFDAP: Automated analysis of Fluorescence Decay After Photoconversion (FDAP) experiments. Bioinformatics. In Press, (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados