JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
광 변환 한 후 형광 붕괴를 사용하여 생활 Zebrafish의 태아의 단백질 안정성을 측정 (FDAP)
요약
세포와 조직에서 단백질 수준은 종종 단단히 단백질 생산 및 통관의 균형에 의해 조절된다. 광 (FDAP) 후 형광 감쇄를 사용하면, 단백질의 클리어런스 동력학 실험적 생체 내에서 측정 할 수있다.
초록
단백질 안정성은 생물학의 여러 측면에 영향을 미친다, 단백질의 클리어런스 동역학을 측정하는 중요한 생물학적 시스템에 대한 통찰력을 제공 할 수있다. FDAP 실험에서 생체 내에서 단백질의 클리어런스를 측정 할 수있다. 관심있는 단백질은 형광 단백질 photoconvertible 태그로 생체 내에서 발현시키고 photoconverted하고 시간의 photoconverted 신호의 감소를 모니터한다. 데이터는 단백질의 반감기를 결정하는 적절한 클리어런스 모델 붙인다. 중요하게는, 다른 생물체의 구획 단백질 집단의 동력학 클리어런스는 구획되지 마스크를 적용함으로써 별도로 검사 할 수있다. 이 접근법은 제브라 개발 중에 분비 신호 전달 단백질의 세포 외 및 세포 내 반감기를 결정하는 데 사용되었다. 여기서 우리는 제브라 피쉬 배아에서 FDAP 실험을위한 프로토콜을 설명합니다. 이 간극의 역학을 결정할 FDAP을 사용하는 것이 가능해야광학적으로 접근 유기체의 모든 taggable 단백질.
서문
세포와 생물의 단백질의 수준은 생산 및 통관 자신의 속도에 의해 결정됩니다. 단백질의 반감기는 분에서 일 1-4까지를 범위로 할 수 있습니다. 많은 생물학적 시스템에서 키 단백질의 안정화 또는 통관 세포 활성에 중요한 영향을 미친다. 세포 내 단백질 안정성의 조절은 세포주기 진행 5,6- 발달 시그널링 7-9, 아폽토시스 (10), 정상 기능의 유지 뉴런 11,12 요구된다. 세포 외 단백질의 안정성은 조직 내 분포 및 morphogens 13, 14 등의 분비 단백질의 가용성에 영향을 미친다.
지난 몇 년 동안, 단백질의 안정성은 주로 방사성 펄스 라벨 또는 사이클로 헥 체이스 실험 (15)를 사용하여 세포 배양에서 평가되고있다. 이러한 펄스 추적 실험에서, 세포 중 방사능 일시적 아미노 "펄스"에 노출 될산 전구체 새롭게 합성 된 단백질에 통합하거나 단백질 합성을 저해 사이클로 헥시에 노출되어있다. 배양 된 세포를 상이한 시점에서 수집하고, 어느 (방사성 펄스 추적 실험에서) 오토 라디오이어서 면역 또는 (사이클로 헥 실험에서) 웨스턴 블로 팅은 시간이 지남에 따라 단백질의 클리어런스를 정량화하는데 사용된다.
기존의 단백질 안정성 분석은 몇 가지 단점을 가지고있다. 우선, 단백질의 분석 방법은 종종 그의 내인성 환경에서 발현되지 않고, 다른 종으로부터의 세포 조직 배양에서 때로는. 그 안정성 문맥 의존적 인 단백질이 방법은 문제가있다. 둘째, 시간이 지남에 따라 각각의 세포 또는 생물체 단백질 클리어런스를 수행하는 것이 불가능하고, 상기 분석으로부터의 데이터는 상이한 시점에서 상이한 세포의 집단의 평균을 반영한다. 각각의 세포는 시작 할 수 있기 때문에단백질의 다른 양, 서로 다른 시간에 방사성 라벨 또는 사이클로 헥를 촬영했거나 다른 통관 반응 속도 등의 데이터를 집계 오해의 소지가있을 수있을 수 있습니다. 마지막으로, 사이클로 헥 체이스 실험의 경우, 단백질 합성 억제제의 첨가는 인위적으로 단백질의 안정성을 바꿀 수 16-18 의도하지 않은 생리 학적 효과를 가질 수있다. 이러한 단점은, 광 (FDAP) 후 photoconvertible 단백질이 생물에게 19-25 생활에 동적으로 단백질의 간격을 측정하기 위해 활용하는 기술을 형광 붕괴를 사용하여 방지 할 수 있습니다 (FDAP 기술의 한계에 대한 토론 참조).
Photoconvertible 단백질은 그 여기 및 방출 특성 빛 (26)의 특정 파장에 노출 된 후 변경 형광 단백질이다. 하나는 일반적으로 사용되는 변형 Dendra2, 처음에 전을 가진 "녹색 - 투 - 빨간색"photoconvertible 단백질이다표창장 및 배출 녹색 형광 단백질 유사한 특성,하지만 UV 라이트 - "광"에 노출 된 후 여기를 왔나은 / 방출 특성은 적색 형광 단백질 (23,27)와 유사하게된다. 중요한 것은, 광 변환 후 생성 된 새로운 단백질은 photoconverted 단백질 만 풀의 광에 따라 생산 및 통관의 디커플링과 관찰을 허용, photoconverted 단백질과 동일한 여기 / 방출 특성이 없습니다. photoconvertible 단백질과 관심의 단백질에 태그를 지정하는 것은 따라서 펄스 레이블하는 그대로, 광학적으로 접근 생물체의 단백질을 수있는 편리한 방법을 제공합니다.
FDAP 분석법 (도 1a)에있어서, 목적 단백질은 단백질 photoconvertible 태그로되어 생체 (도 1b)로 표현. 융합 단백질 photoconverted되며, 시간이 지남에 photoconverted 신호의 감소는 fluorescen에 의해 모니터링CE 현미경 (그림 1C). 데이터는 융합 단백질 (도 1D)의 반감기를 측정하기 위하여 적절한 모델이 장착된다.
여기에 설명 FDAP 분석은 19 초 동안 배아 제브라 피쉬 배아 분비 신호 전달 단백질의 세포 외 반감기를 결정하도록 고안되었다. 그러나, 이러한 접근 방식은 라이브 영상을 견뎌, 어떤 taggable 세포 내 또는 세포 외 단백질의 간격을 모니터링하는 데 이용 될 수있는 투명한 모델 생물에 적용 할 수있다. 여기에 설명 된 기술의 변화는 배양 세포 20,23 초파리 (22)와 마우스 (21) 배아에서 수행되었다.
프로토콜
1. Photoconvertible 퓨전 구축 및 삽입하기 Dechorionated Zebrafish의 배아를 생성
- 다음 뮐러 등., 2012 (19)에서와 같이 융합 단백질을 코딩하는 캡핑 된 mRNA를 생성하는 시험 관내 전사를 사용하여, 녹색 대 적색 photoconvertible 단백질 (설명 참조)에 융합 된 관심있는 단백질을 함유하는 기능성 구조체를 생성한다.
- 하나의 세포 단계에서 약 30 제브라 피쉬 배아에서 chorions을 제거하는 프로 나제를 사용합니다. 또한, 수동 집게 (28)를 사용하여 배아를 dechorionate.
주 : 태아의 후속 이미징 dechorionated해야합니다. 원하는 경우, 배아는 촬상 직전에, 나중에 융모막 통해 주입하고 dechorionated 수있다.- 표준 제브라 피쉬 배아 매체 (19)의 스트렙토 마이 세스 (Streptomyces) griseus의에서 프로 나제의 5 ㎎ / ㎖ 원액을 확인합니다. 10 분은 단백질 분해 효소가 분해 할 수 있도록하기 위해 부드럽게 실온에서 솔루션을 바위. 나누어지는 2m-20 ° C에서의 microcentrifuge 튜브에 L과 동결.
- 배아 ~ 8 ml의 배지를 함유 5cm 직경 유리 또는 코팅 된 플라스틱 아가 페트리 접시로 이동 한 세포 단계 배아. 접시에 해동 프로 나제 원액 2 ㎖를 추가하고 5 ~ 10 분 동안 실온에서 품어.
- dechorionated 배아가 파열 될 것 중 하나와 접촉으로, 공기 또는 플라스틱으로 배아를 노출시키지 마십시오. 배아 매체와 200ml의 유리 비커를 입력합니다. 매체에 침수 동안 배양 접시를 기울여 비커에 배아를 전송합니다.
- 배아는 비커의 바닥에 정착 한 후, 다음 비커에 신선한 배아 매체를 부어, 배아 매체의 대부분을 부어. 비커에 쏟아져 매체의 가벼운 소용돌이는 배아가 약화 chorions을 잃게됩니다.
- 단계를 반복 1.2.4.
- 골조 팁 유리 파스퇴르 피펫을 사용하여 아가로 오스 코팅 분사 요리 (29)에 dechorionated 배아를 전송합니다. 불타는피펫 팁은 부상 배아에서 가장자리의 계단 현상을 방지 할 수 있습니다.
- (그림 1B, 제안 mRNA와 Alexa488 - 덱스 트란 양의 토론 참조) mRNA와 3 kDa의 Alexa488 - 덱스 트란 결합체 (29, 30)을 공동-주입. 분열이 시작되면도의 mRNA의 분포와 형광 염료를 보장하기 위해 셀 (안 노른자)의 중심에 직접 주입한다.
주 : Alexa488 신호는 세포 내 및 세포 외 형광을 구별하기 위해 구획되지 마스크를 생성하기 위해 데이터 분석에서 사용된다. - 배아 매체 가득 여섯 아니라 플라스틱 접시의 1 ~ 2 % 아가로 오스 코팅 잘로 전송 주입 배아. 배아 늦게 구 단계 31 (약 5 시간 포스트 수정을)에 도달 할 때까지 28 ° C에서 어둠 속에서 품어. 실체 현미경을 이용하여 배아마다 한 2 시간을 확인하고 사망 한 배아에 의해 생성 된 이물질을 제거합니다.
2. P에 대한 Zebrafish의 배아를 장착거꾸로 공 초점 현미경에 hotoconversion 및 이미지
- 1-5 배아 건강을 식별하고, 접시에서 제거 골조 팁 유리 파스퇴르 피펫을 사용하여 실체 현미경을 사용한다.
- 부드럽게 1X Danieau 배아의 매체에 용해 1 % 저 융점 아가로 오스 ~ 1㎖를 함유 마이크로 원심 분리 튜브로 배아를 토출 (도 2a) (자재 목록 참조).
참고 : 아가로 오스 40, 42 ° C 사이의 온도를 가져야한다; 높은 온도는 배아가 손상 될 수 있습니다. - 일부 아가와 함께 다시 피펫으로 배아를 그립니다. 조심스럽게 유리 바닥 접시 (그림 2B)의 커버 유리 위에 아가로 오스와 배아를 꺼냅니다. 커버 유리의 두께가 공 초점 현미경의 목표와 호환되는지 확인하십시오.
- 원하는 경우 유리 피펫을 다시 사용합니다. 피펫에서 잔류 아가로 오스를 청소하고 위아래로 빠르게 막힘, 피펫 배아 매체를 방지합니다.다음이 목적을위한 실체 현미경으로 배아 매체의 ~ 5 ml로 가득 15 ML 튜브를 놓습니다.
- 동물 극 (배반)는 커버 유리를 향하도록 위치를 배아 금속 프로브를 사용한다. 아가로 오스 20 ~ 30 초에서 응고하기 때문에 신속하게 작업 할 수 있습니다. 배아 '의 위치를 모니터링하고 아가로 오스 견고 때까지 필요한 재조정 실체 현미경을 사용합니다.
- 배아의 원하는 번호가 장착 될 때까지 반복 2.1-2.4 단계를 반복합니다.
참고 : 일반적인 실험에서, 네 다섯 배아 각을 포함하는 4 개의 아가로 오스 방울은 커버 유리에 쉽게 맞습니다. 약 16 배아는 하나의 이상적인 실험 (그림 2C) 동안 이미지를 만들 수 있습니다. - 아가로 오스가 응고되면, 1 배 Danieau의 배아 매체와 유리 바닥 접시를 채운다.
3. Photoconverting 및 Photoconverted 신호의 감소를 측정
25X 또는 40X 물 목표 내가크기와 제브라 피쉬 배아의 굴절률에 적합한이야. 물 여섯 시간 실험의 과정 동안 증발하기 때문에 그것은 수중보다는 실제 물과 같은 굴절률 액침 오일을 사용하는 것이 최선이다. 침지 기름이 물 (석유가 아닌) 목적으로 사용할 수 있도록 설계되어 있는지 확인합니다.
- 목적과 커버 유리 사이의 오일 필름 이미징 동안 다른 배아 위치로 무대 이동으로 중단되지 않도록하기 위해 목표에 몰입 오일의 큰 하락을 놓습니다. 요리 할 때 단계 이동을 이동하지 않도록 확실하게 무대에 유리 바닥 접시를 놓습니다. 가능하면 28 ° C, 제브라 피쉬의 개발을위한 최적의 온도에서 가열 단계를 사용합니다.
- 공 초점 현미경의 소프트웨어 패키지의 각 배아의 위치를 정의합니다. 각각의 배아 용 Z-깊이를 조정하고, 각각의 배아 대략 동일 평면을 대상으로 시도한다.
참고 : 약 30 μm의 동물 극에서이 이동입니다배아의 포락 층을 회피 할 수있는이 깊이에서 보낸 외경 깊이 이미징 영역이 극대화되고, 광 산란이 최소이다. ~ 3.3 ㎛의 두께로 슬라이스 한 광은 충분한 데이터를 제공한다; Z-스택 (섹션 5 참조)을 취득 할 필요가 없다. - 실험 기간 동안 두 개의 신호를 수집 : Alexa488 - 덱스 트란에서 "녹색"신호를 공액-있는이 photoconverted 후 세포 외 및 세포 내 형광 및 융합 단백질에서 "빨간색"신호를 분리하기 위해 데이터 분석시 사용됩니다.
- 488 nm의 레이저를 사용하여 Alexa488을 자극하고, ~ 500 및 540 nm의 사이에 방출되는 형광을 수집합니다. 참고 : 광 변환 후, 많은 녹색 - 투 - 붉은 photoconvertible 단백질 (예를 들어, Dendra2)는 543 nm의 레이저로 흥분하고 ~ 550 및 650 nm의 사이의 형광을 방출 할 수있다. 사용 photoconvertible 단백질에 따라 필요한 조정합니다.
- 취득 "전 광"이미지 및 이미지에 적절한 이미징 조건 (단계 3.2) 이전에 정의 된 각각의 위치를 공 초점 현미경의 소프트웨어를 구성하는 다섯 시간 시간 코스마다 10 또는 20 분 (5 장을보고 토론).
- 융합 단백질을 photoconvert하려면, 2 분 동안 100 %의 출력에서 ~ 300-400 nm의 여기 필터와 수은 아크 램프로부터 UV 광에 배아기를 10X 대물 렌즈로 전환 노출. . 균일 한 광을 (5 장 참조) 홍보 z 축에있는 z 포커스를 이동 침지 오일 광 동안 10 배 목표에 떨어지지 않도록하십시오.
주 : 광 중 포커스의 이동은 실험자 간의 변동을 방지하기 위해 자동화 될 수있다. - 바로 광 변환 한 후 다시 25 배 또는 40 배 목표로 전환합니다. 단계 3.2에서 이전에 정의 된 위치가 여전히 정확한지 확인하십시오. 요리 photoconv 동안 이동하는 경우ersion, 태아의 위치를 다시-정의합니다.
- 3.4 단계에서 만든 프로그램을 시작하고 영상 5 시간 동안 계속 할 수 있습니다. 광 및 각각의 배아에 대한 영상의 시작 사이에 경과 된 시간을합니다.
- 때때로 실험에서 확인합니다. Danieau의 매체의 수준을 모니터링하고 필요하면 더 추가 할 수 있습니다. 이 멈춘 경우 소프트웨어를 다시 시작합니다.
- 지수 감소 모델의 점근선을 추정 데이터 분석에서 사용되는 배경 형광 값을 결정하기 위해, 실험 Alexa488 덱스 트란 아니라 mRNA를 주입 한 배아 일부를 포함한다. 후속 데이터 분석에서 사용되는 기기 노이즈를 측정하기 위해, 샘플의 부재하에 화상을 취득.
4. PyFDAP를 사용하여 데이터를 분석
- 시각 각 배아 시간 코스 데이터 세트를 검사한다. IMAG 중 사망 한 배아에서 데이터 세트를 폐기photoconverted 신호의 매우 낮은 수준, 또는 그 특이한 모양과 이동 (부상 또는 병 배아의 일반) 분할 중지 세포의 영역을 포함하는 상당히 이동 ING,,.
참고 : 때때로, 침지 오일에 기포 나 다른 유물 그렇지 않으면 사용할 수있는 데이터 세트에 하나 또는 두 개의 이미지에 표시됩니다. 아티팩트를 포함하는 모든 이미지를 참고; 나중에는 폐기 될 것이며, 이러한 데이터 세트로부터 남은 시점 여전히 분석 될 수있다. - FDAP 데이터를 분석하는 파이썬 기반의 소프트웨어 패키지 PyFDAP을 사용합니다. PyFDAP 각 화상의 평균 세포 내 및 세포 외 적색 형광 강도를 결정하고, 지수 감소 함수 (56) (도 3)로 데이터를 끼워 맞춤으로써 반감기를 계산한다.
- PyFDAP 다운로드 (참조 재료 목록).
- 사용 PyFDAP 세포 내와 세포 외 photoconverted 신호 (그림 3A를, B)로 구분합니다. 우리세포 내 마스크를 만들기 위해 엄격 세포이다 Alexa488 신호를, 예. 평균 세포 외 농도를 계산할 때 고려되는 세포 내 픽셀을 방지하기 위해 해당 빨강 채널 이미지에이 마스크를 적용합니다. 평균 세포 내 농도를 측정하기 위해, 마스크를 반전.
- PyFDAP, 단계 4.2.2에서 생성 된 마스크 화상을 표시. 시각적으로 이러한 이미지를 검사하고 마스크 정확하게 세포 외 공간에서 세포 내 (이 드문되어야 구별하지 않은 데이터 세트를 폐기, 즉 세포막은 세포 외 공간 마스킹되어있는 이미지에 포함되어 참고하지만 이들은 임계 값을 변경함으로써 제거 될 수 알고리즘 또는 멤브레인 마스크를 도입하여 (예를 들어, 사용하는 막 - CFP)). 또한 단계 4.1에서 확인 된 유물 (예, 침지 기름에 거품)를 포함하는 단일 이미지를 버린다.
- EAC 평균 세포 외 및 세포 내 형광 강도를 계산하는 PyFDAP를 사용하여시간 이미지. PyFDAP 합산은 픽셀들의 총 개수에 의해 마스크 및 분할 벗어나는 픽셀의 휘도를 합산하여 이들의 평균을 계산한다.
- 다음과 같은 지수 함수와 형광 데이터 (그림 3C)를 장착 :
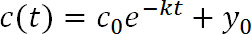
t 시간 후의 광, C (t) t의 주어진 값에서의 강도는이고, C 0 t = 0에서의 강도이고, k는 제거율 상수이며 Y 0 함수가 형광으로 접근 점근선 감소한다 (도 1D). Y는 0 3.8 19 단계에서 측정에 기초하여 제한 될 수있다. - 다음의 관계를 이용하여 제거율 상수 (K)에서 세포 외 및 세포 내 단백질 반감기 (τ)을 계산하기 위해 사용 PyFDAP :

5. 제어 실험 광표백, 부주의 광 및 광 균일 성을 평가하기 위해
- 평가 광표백
주 : 광표백 관심 단백질의 간극 이외에 형광 단백질의 표백 특성을 반영 형광 강도에 인공적 저하 될 수있다.- 가능한 광표백을 평가 이미징 및 영상 사이에 20 분 간격 (그림 4)와 두 번째 세트 사이에 10 분 간격으로 FDAP 실험 한 세트를 수행합니다. 4 절에 설명 된 실험을 이용 PyFDAP 세트 모두로부터의 데이터를 분석한다.
- 도 10 및 20 분 간격 실험에서 반감기 비교. 긴 반감기가 20 분 간격 실험에서 상당한 광표백을 나타냅니다. 두 실험에서 반감기가 동일한 경우, PHOTobleaching는 큰 문제가되지 않습니다.
- 또한, 즉시 광 후 ~ 30 일련의 이미지를 획득하여 photobleaching에 평가합니다. 형광 강도의 유의 한 감소는 상당한 광표백을 나타냅니다.
- 광표백가 검출되었을 경우, 낮은 파워의 레이저를 사용하여 촬상 시간을 단축하거나, 더욱 광 안정성 photoconvertible 단백질 (32)을 사용하는 것을 고려.
- 실수로 광을 평가.
참고 : Dendra2는 488 nm의 조명 (27)를 사용하여 photoconverted 수 있습니다. 488 nm의 레이저로 Alexa488이 흥미 관심 단백질의 겉보기 반감기의 단계 3.3.1, 부주의 광 따라서 인공적 증가하는 경우에 기재된 바와 같이 가능하다. 그러나, 우리와 다른 사람 (33)는 488 nm의 조명은 제브라 피쉬 배아의 광 변환의 비효율적 인 방법이다 것을 발견했다.- 부주의 한 광을 검출하는 단계 5.1.1에서 설명 대조 실험을 사용하여. 도 10 및 20 분 간격 실험에서 반감기 비교. 20 분 간격 실험에서 짧은 반감기는 중요한 실수로 광을 나타냅니다. 두 실험에서 반감기가 동일한 경우, 부주의 한 광은 큰 문제가되지 않습니다.
- 부주의 한 광이 검출되면, Dendra2 photoconverting 실수를 방지하기 위해 낮은 488 nm의 레이저 출력과 짧은 촬상 시간을 사용한다.
- 광 균일 성을 평가.
주 : 광이 동물의 배아 극 치우쳐있는 경우, 형광의 감소가 깊은 평면 (도 5a)로 단백질 또는 세포의 확산 이동에 의해 영향을 받는다.- 광이 균일 여부를 확인하려면, (세포 융합 단백질과 실험) 분비 photoconvertible 단백질 또는 (세포 융합 단백질과 실험) 세포질 photoconvertible 단백질을 발현. 평소와 같이 Photoconvert, t암탉은 80 분마다 20 분 배반 엽의 대부분을 포괄 Z-스택을 획득.
- 광은 동물 극 치우쳐 있으면 깊이 평면에서 형광 강도 때문에 확산 또는 세포 이동 (도 5b)에 시간에 따라 증가 할 것이다. 불균일 한 광이 검출되면, 광 동안 배아을 깊은 초점.
결과
FDAP 지브라 피쉬 19 배아 세포 외 신호 전달 단백질의 반감기를 결정하는 데 사용되었다. 이들 단백질 중 하나 퀸트가 배아 발생 동안 34 mesendodermal 유전자의 발현을 유도한다. 곁눈질-Dendra2가 QRT-PCR에 의해 현장 하이브리드 분석 (19)에서 입증 된 바와 같이, 태그가 곁눈질 비슷한 수준에서 mesendodermal 유전자의 발현을 활성화합니다. 배아 Alexa488 덱스 트란 및 mRNA...
토론
FDAP 실험의 성공은 기능 photoconvertible 융합 단백질의 생성에 의존한다. 단백질에 태그를 자사의 생물학적 활성 및 / 또는 현지화, 용해도, 안정성 36-41을 포함한 생물 물리학 적 특성에 영향을 미칠 수 있습니다. 활성 하나를 찾기 위해 여러 가지 융합 구조의 활동을 테스트 할 준비를하십시오. 발명자들은 관심 단백질 photoconvertible 단백질의 위치를 변경하거나 긴 링커를 사용하여 (예를 ?...
공개
저자는 공개 충돌이 전혀 없습니다.
감사의 말
저자는 원고에 대한 의견에 대한 제프리 파렐, 제임스 가뇽, 제니퍼 버그만에게 감사의 말씀을 전합니다. KWR는 FDAP 분석의 개발 기간 동안 국립 과학 재단 (National Science Foundation) 대학원 연구 활동 프로그램에 의해 지원되었다. 이 작품은 AFS에 NIH로부터 보조금과 독일 연구 재단 (에미 뇌터 프로그램), 막스 플랑크 사회, 오후에 인간 프론티어 과학 프로그램 (경력 개발 상)에서 보조금에 의해 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| PyFDAP (download from the following website: http://people.tuebingen.mpg.de/mueller-lab) | Install and operate using the instructions provided on the PyFDAP website; PyFDAP is compatible with Linux, Mac, and Windows operating systems. | ||
| mMessage mMachine Sp6 Transcription Kit | Life Technologies | AM1340 | To generate capped mRNA for injection into embryos |
| Alexa488-dextran conjugate, 3 kDa | Life Technologies | D34682 | Co-inject with mRNA to create intracellular and extracellular masks |
| 6-well plastic dish | BD Falcon | Incubate embryos in agarose-coated wells until ready for mounting | |
| Embryo medium | 250 mg/L Instant Ocean salt, 1 mg/L methylene blue in reverse osmosis water adjusted to pH 7 with NaHCO3 | ||
| Protease from Streptomyces griseus | Sigma | P5147 | Make a 5 mg/ml stock and use at 1 mg/ml to dechorionate embryos at the one-cell stage |
| 5 cm diameter glass Petri dish | For embryo dechorionation | ||
| 200 ml glass beaker | For embryo dechorionation | ||
| Microinjection apparatus | For injection of mRNA and dye into embryos at the one-cell stage | ||
| Stereomicroscope | For injecting and mounting embryos | ||
| 1x Danieau's medium | Dilute low melting point agarose and perform imaging in this medium; recipe: 0.2 mm filtered solution of 58 mM NaCl, 0.7 mM KCl, 0.4 mM MgSO4, 0.3 mM CaCl2, 5 mM HEPES pH 7.2 | ||
| UltraPure low melting point agarose | Invitrogen | 16520-100 | For mounting embryos; use at a concentration of 1% in Danieau's medium: add 200 mg to 20 ml Danieau's medium, microwave until dissolved, then aliquot 1 ml into microcentrifuge tubes; aliquots can be stored at 4 °C, re-melted at 70 °C, and cooled to 40–42 °C when ready to use |
| Glass Pasteur pipette | Kimble Chase (via Fisher) | 63A53WT | For mounting embryos; flame the tip to prevent jagged edges from injuring embryos |
| Metal probe | For positioning embryos during mounting | ||
| Glass bottom dishes | MatTek | P35G-1.5-14-C | Use the appropriate cover glass thickness for your objective; part number listed here is for cover glass No. 1.5 |
| 15 ml tube filled with ~5 ml embryo medium | BD Falcon | For rinsing residual agarose from the Pasteur pipette | |
| Inverted laser scanning confocal microscope | A mercury arc lamp, 488 nm laser, 543 nm laser, and the appropriate filter sets are required | ||
| Heated stage | To maintain embryos at the optimal temperature of 28 °C during the experiment | ||
| Confocal software capable of time-lapse imaging | Must be able to define multiple positions and automatically image them at defined intervals | ||
| 25X or 40X water objective | Objective for imaging | ||
| 10X air objective | Objective for photoconversion | ||
| Immersion oil | Immersion oil with the same refractive index as water |
참고문헌
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Boisvert, F. M., et al. A Quantitative Spatial Proteomics Analysis of Proteome Turnover in Human Cells. Molecular & Cellular Proteomics. 11 (3), 011429 (2012).
- Belle, A., Tanay, A., Bitincka, L., Shamir, R., O'Shea, E. K. Quantification of protein half-lives in the budding yeast proteome. Proceedings of the National Academy of Sciences of the United States of America. 103 (35), 13004-13009 (2006).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764-768 (2011).
- Parry, D. H., O'Farrell, P. H. The schedule of destruction of three mitotic cyclins can dictate the timing of events during exit from mitosis. Current Biology. 11 (9), 671-683 (2001).
- Holloway, S. L., Glotzer, M., King, R. W., Murray, A. W. Anaphase is initiated by proteolysis rather than by the inactivation of maturation-promoting factor. Cell. 73 (7), 1393-1402 (1993).
- Dharmasiri, N., Estelle, M. Auxin signaling and regulated protein degradation. Trends Plant Sci. 9 (6), 302-308 (2004).
- MacDonald, B. T., Tamai, K., He, X. Wnt/beta-catenin signaling: components, mechanisms, and diseases. Developmental Cell. 17 (1), 9-26 (2009).
- Chen, X., et al. Processing and turnover of the Hedgehog protein in the endoplasmic reticulum. The Journal of Cell Biology. 192 (5), 825-838 (2011).
- Elmore, S. Apoptosis: A Review of Programmed Cell Death. Toxicologic Pathology. 35 (4), 495-516 (2007).
- Yi, J. J., Ehlers, M. D. Emerging Roles for Ubiquitin and Protein Degradation in Neuronal Function. Pharmacological Reviews. 59 (1), 14-39 (2007).
- Rubinsztein, D. C. The roles of intracellular protein-degradation pathways in neurodegeneration. Nature. 443 (7113), 780-786 (2006).
- Müller, P., Schier, A. F. Extracellular Movement of Signaling Molecules. Developmental Cell. 21 (1), 145-158 (2011).
- Eldar, A., Rosin, D., Shilo, B. -. Z., Barkai, N. Self-enhanced ligand degradation underlies robustness of morphogen gradients. Developmental Cell. 5 (4), 635-646 (2003).
- Zhou, P. Determining protein half-lives. Methods In Molecular Biology. 284, 67-77 (2004).
- Woodside, K. H. Effects of cycloheximide on protein degradation and gluconeogenesis in the perfused rat liver. Biochim Biophys Acta. 421 (1), 70-79 (1976).
- Schimke, R. T., Doyle, D. Control of enzyme levels in animal tissues. Annual Review of Biochemistry. 39, 929-976 (1970).
- Kenney, F. T. Turnover of rat liver tyrosine transaminase: stabilization after inhibition of protein synthesis. Science. 156 (3774), 525-528 (1967).
- Müller, P., et al. Differential Diffusivity of Nodal and Lefty Underlies a Reaction-Diffusion Patterning System. Science. 336 (6082), 721-724 (2012).
- Kiuchi, T., Nagai, T., Ohashi, K., Mizuno, K. Measurements of spatiotemporal changes in G-actin concentration reveal its effect on stimulus-induced actin assembly and lamellipodium extension. The Journal of Cell Biology. 193 (2), 365-380 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biology. 13 (2), 17-123 (2011).
- Drocco, J. A., Grimm, O., Tank, D. W., Wieschaus, E. Measurement and Perturbation of Morphogen Lifetime: Effects on Gradient Shape. Biophys J. 101 (8), 1807-1815 (2011).
- Zhang, L., et al. Method for real-time monitoring of protein degradation at the single cell level. BioTechniques. 42 (4), 446-450 (2007).
- Miyawaki, A. Proteins on the move: insights gained from fluorescent protein technologies. Nat Rev Mol Cell Biol. 12 (10), 636-668 (2011).
- Pantazis, P., Supatto, W. Advances in whole-embryo imaging: a quantitative transition is underway. Nat Rev Mol Cell Biol. 15 (5), 327-339 (2014).
- Lukyanov, K. A., Chudakov, D. M., Lukyanov, S., Verkhusha, V. V. Innovation: Photoactivatable fluorescent proteins. Nat Rev Mol Cell Biol. 6 (11), 885-891 (2005).
- Gurskaya, N. G., et al. Engineering of a monomeric green-to-red photoactivatable fluorescent protein induced by blue light. Nature Biotechnology. 24 (4), 461-465 (2006).
- Zou, J., Wei, X. Transplantation of GFP-expressing Blastomeres for Live Imaging of Retinal and Brain Development in Chimeric Zebrafish Embryos. Journal of Visualized Experiments. (41), (2010).
- Yuan, S., Sun, Z. Microinjection of mRNA and Morpholino Antisense Oligonucleotides in Zebrafish Embryos. Journal of Visualized Experiments. (27), (2009).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of Zebrafish Embryos to Analyze Gene Function. Journal of Visualized Experiments. (25), (2009).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Developmental Dynamics. 203 (3), (1995).
- McKinney, S. A., Murphy, C. S., Hazelwood, K. L., Davidson, M. W., Looger, L. L. A bright and photostable photoconvertible fluorescent protein. Nature Methods. 6 (2), 131-133 (2009).
- Dempsey, W. P., Qin, H., Pantazis, P. In Vivo Cell Tracking Using PhOTO Zebrafish. Methods in Molecular Biology. 1148, 217-228 (2014).
- Schier, A. F. Nodal Morphogens). Cold Spring Harbor Perspectives in Biology. 1 (5), a003459-a003459 (2009).
- Otsu, N. A threshold selection method from gray-level histograms. IEEE Trans Syst Man Cybern. 9 (1), 62-66 (1979).
- Pédelacq, J. -. D., Cabantous, S., Tran, T., Terwilliger, T. C., Waldo, G. S. Engineering and characterization of a superfolder green fluorescent protein. Nature Biotechnology. 24 (1), 79-88 (2005).
- Swulius, M. T., Jensen, G. J. The Helical MreB Cytoskeleton in Escherichia coli MC1000/pLE7 Is an Artifact of the N-Terminal Yellow Fluorescent Protein Tag. Journal of Bacteriology. 194 (23), 6382-6386 (2012).
- Landgraf, D., Okumus, B., Chien, P., Baker, T. A., Paulsson, J. Segregation of molecules at cell division reveals native protein localization. Nature Methods. 9 (5), 480-482 (2012).
- Stadler, C., et al. Immunofluorescence and fluorescent-protein tagging show high correlation for protein localization in mammalian cells. Nature Methods. 10 (4), 315-323 (2013).
- Quattrocchio, F. M., Spelt, C., Koes, R. Transgenes and protein localization: myths and legends. Trends Plant Sci. 18 (9), 473-476 (2013).
- Morimoto, Y. V., Kojima, S., Namba, K., Minamino, T. M153R Mutation in a pH-Sensitive Green Fluorescent Protein Stabilizes Its Fusion Proteins. PLoS ONE. 6 (5), e19598 (2011).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2 (12), 905-909 (2005).
- Waters, J. C. Accuracy and precision in quantitative fluorescence microscopy. The Journal of Cell Biology. 185 (7), 1135-1148 (2009).
- Moll, U. M., Petrenko, O. The MDM2-p53 interaction. Mol Cancer Res. 1 (14), 1001-1008 (2003).
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 24 (1), 142-153 (2014).
- Bedell, V. M., et al. In vivo genome editing using a high-efficiency TALEN system. Nature. 491 (7422), 114-118 (2012).
- Hruscha, A., et al. Efficient CRISPR/Cas9 genome editing with low off-target effects in zebrafish. Development. 140 (24), 4982-2987 (2013).
- Hwang, W. Y., et al. Heritable and Precise Zebrafish Genome Editing Using a CRISPR-Cas System. PLoS ONE. 8 (7), 68708 (2013).
- Hwang, W. Y., et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology. 31 (3), 227-229 (2013).
- Zu, Y., et al. TALEN-mediated precise genome modification by homologous recombination in zebrafish. Nature Methods. 10 (4), 329-331 (2013).
- Keller, P. J., Schmidt, A. D., Wittbrodt, J., Stelzer, E. H. K. Reconstruction of Zebrafish Early Embryonic Development by Scanned Light Sheet Microscopy. Science. 322 (5904), 1064-1069 (2008).
- Blanchet, M. H., et al. Cripto Localizes Nodal at the Limiting Membrane of Early Endosomes. Science Signaling. 1 (45), ra13-ra13 (2008).
- Jullien, J., Gurdon, J. Morphogen gradient interpretation by a regulated trafficking step during ligand-receptor transduction. Genes & Development. 19 (22), 2682-2694 (2005).
- Incardona, J. P., et al. Receptor-mediated endocytosis of soluble and membrane-tethered Sonic hedgehog by Patched-1. Proceedings of the National Academy of Sciences of the United States of America. 97 (22), 12044-12049 (2000).
- Scholpp, S., Brand, M. Endocytosis Controls Spreading and Effective Signaling Range of Fgf8 Protein. Current Biology. 14 (20), 1834-1841 (2004).
- Bläßle, A., Müller, P. . PyFDAP: Automated analysis of Fluorescence Decay After Photoconversion (FDAP) experiments. Bioinformatics. In Press, (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유