Method Article
Transmissão de digitalização em grande escala Electron Microscopy (Nanotomy) de saudável e Ofendidos Zebrafish Cérebro
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Large-scale 2D electron microscopy (EM), or nanotomy, is the tissue-wide application of nanoscale resolution EM. Here we describe a universal method for nanotomy applied to investigate the zebrafish larval brain in health and upon non-invasive brain injury.
Resumo
microscopia em grande escala de elétrons 2D (EM), ou nanotomy, é a aplicação de todo o tecido da microscopia eletrônica de resolução nanoescala. Outros e que anteriormente aplicada EM larga escala para ilhotas pancreáticas humanas da pele, cultura de tecidos e toda larvas do peixe 1-7. Aqui nós descrevemos um método universalmente aplicável para EM digitalização escala tecido para detecção imparcial de recursos sub-celulares e moleculares. Nanotomy foi aplicado para investigar o saudável e um cérebro de peixe zebra neurodegenerativa. Nosso método é baseado em protocolos de preparação de amostra padronizados EM: fixação com glutaraldeído e ósmio, seguido por incorporação de resina de epóxi, ultrafinos de corte e montagem de ultrafinos-seções sobre grades de um furo, seguido de pós coloração com uranyl e chumbo. Em larga escala de imagens 2D em mosaico são adquiridos usando uma varredura EM conectada a um gerador de varredura ampla área externa usando EM transmissão de varredura (STEM). Grande escala imagens EM são tipicamente ~ 5 - 50 g de pixels itamanho n, e melhor visualizado usando arquivos HTML com zoom, que pode ser aberto em qualquer navegador web, semelhante a mapas HTML geográficas on-line. Este método pode ser aplicado ao tecido (ser humano), as secções transversais de animais inteiros, bem como cultura de tecidos 1-5. Aqui, os cérebros de peixe-zebra foram analisados em um modelo de ablação neuronal não-invasiva. Visualiza-se dentro de um único conjunto de dados de tecidos, celular e alterações subcelulares que pode ser quantificada em vários tipos de células incluindo neurónios, macrófagos e microglia do cérebro. Além disso, nanotomy facilita a correlação de EM com microscopia de luz (CLEM) 8 no mesmo tecido, como grandes áreas de superfície trabalhada anteriormente usando microscopia de fluorescência, pode ser subsequentemente submetida a grande área de EM, resultando na nano-anatomia (nanotomy) de tecidos. Ao todo, nanotomy permite a detecção imparcial de recursos a nível EM de forma quantificável todo o tecido.
Introdução
desenvolvimentos técnicos recentes têm melhorado a versatilidade, aplicabilidade e natureza quantitativa da EM, levando a um renascimento da análise ultra-estrutural. Os avanços incluem 3D EM, em larga escala 2D EM e métodos aperfeiçoados e reagentes para microscopia de luz e microscopia eletrônica correlata (CLEM) para comparar outros modos de análise microscópica diretamente para o nível EM 8-10. Em larga escala de 2D EM é particularmente adequado para quantificar ou identificar (romance) características da doença de patologia humana, estudo de modelos animais para modelos de doença e de cultura de tecidos. Devido à pequena tipicamente campo de visão que é difícil de correlacionar as modificações em alta ampliação com uma escala larga de tecido, bem como para analisar quantitativamente unbiasedly e características ultra-estruturais.
Para a análise patológica de tecido humano ou a avaliação da patologia em modelos animais, hematoxilina e eosina (H & E) colorido seções de formaldeído parafina fixo incorporado (FFPE) tissue é o padrão. Além de simples H & E coloração imunomarcação também é realizada para identificar anomalias patológicas. Se tais tecidos pode ser analisada ao nível EM, tipos específicos de células, e as alterações intracelulares podem ser identificados. A natureza imparcial de EM em grande escala permite encontrar características inesperadas e novos da doença. Por larga escala áreas em up a milímetros quadrados pode ser visualizado. Nós e outros anteriormente nanotomy de rato ilhotas pancreáticas 4, cultura de células 2, cérebro do rato 3, pele e mucosa 7 e toda larvas do peixe 1,5 (www.nanotomy.org) aplicada. Os peixes-zebra são altamente adequados para imagiologia in vivo, em particular para visualizar os tipos de células que são de difícil acesso, em tecidos de mamíferos, incluindo as células cerebrais imunológicas 11. Aqui o procedimento nanotomy é descrito em pormenor, aplicadas a secções coronais de cabeças de peixe-zebra submetidos à ablação condicional neuronal por conversão de metronidazol por neuronalnitro-redutase expressa (www.nanotomy.org) 5,12-14. Todos os dados brutos são apresentados como arquivos HTML com zoom, visualização molecular para mudanças de escala do tecido. A apresentação dos dados brutos permite análises imparciais sobre os conjuntos de dados de outros ângulos de especialistas em todo o mundo.
Protocolo
Todos os experimentos com larvas do peixe foram aprovados pelo Comitê da Universidade de Groningen Experimentação Animal de acordo com as diretrizes nacionais e da UE.
Preparação 1. Amostra
- Fixação and Embedding
- Fixação
- Anestesiar as larvas de peixes em tricaina (0,003%) adicionado ao (NaCl a 5 mM, KCl 0,17 mM, CaCl2 0,33, MgSO4 0,33 mM, HEPES 10 mM, pH 7,6) E3 água do peixe-zebra e teste de profundidade de anestesia picando suavemente com uma ponta de pipeta 200 uL sob microscópio de dissecção até que o movimento não é mais detectado (Figura 1A).
- Quando o processamento de amostras para o EM correlativo depois de adquirir em dados de imagem in vivo utilizando imagiologia confocal ou dois fotões em 1,8% de agarose como descrito, 5,15 correcção larvas em agarose utilizando 4% de PFA em PBS contendo Triton-X-100 (0,05%).
- Corte larvas de agarose e processo paraEM grande escala 5.
- Fix larvas em fresco de paraformaldeído 4% em PBS contendo Triton-X-100 (0,05%) durante 2 horas à temperatura ambiente em tubos de plástico.
Nota: As células cultivadas em cultura (in vitro) pode ser directamente fixado em glutaraldeído, tal como descrito no passo seguinte (1.1.1.5). - Remoção da solução e adiciona-se 100 uL de 0,5% de PFA, glutaraldeído a 2% (GA), e cacodilato de sódio 0,1 M (pH 7,4). Armazenar durante a noite a 4 ° C. Lavar as amostras duas vezes em cacodilato de sódio 0,1 M.
- Cortar cabeças de larvas rostralmente ao hindbrain para facilitar a penetração de ósmio. Use seringas finas / fórceps.
- Sufixo larvas em 1% de tetróxido de ósmio (OsO4) /1.5% ferrocianeto de potássio (K 4 [Fe (CN) 6]) em cacodilato de sódio 0,1 M em gelo durante 2 horas e lavar 3 x 5 min em banho-H2O destilada
- Desidratar em etanol por 20 min incubações em concentrações crescentes de etanol (50%, 70%, 3 vezes 100%).
- Incorporação
- Prepare resina epóxi de acordo com receitas padrão usados em laboratórios de EM.
Nota: Nós usamos resina epoxi como se segue: Mistura de 19,8 g de éter glicídico 100, 9,6 g de anidrido do ácido 2-dodecenilsuccínico e 11,4 g de anidrido methylnadic utilizando um agitador magnético. Adicionar 0,5 g de DMP-30 (tris- (dimetilaminometil) fenol) e misturar novamente. - Incubar durante a noite em larvas de resina epóxi 50% em etanol (v / v) durante a rotação (RT).
- Substitua diluído epoxy-resina com resina pura. Incubar durante 30 min, adicionar resina fresca novamente e incubar durante mais 30 min. Novamente actualizar a resina e, em seguida incubar 3 horas à temperatura ambiente, seguido de 15 min a 58 ° C e mais 1 h à TA sob pressão reduzida (200 mbar).
- Orientar as cabeças em silício plana moldes de incorporação disponíveis comercialmente sob um microscópio de dissecação com uma agulha ou com o palito anterior virado para o lado do molde ou conforme necessário (Figura 1B).
- Polimerizar res epóxino dia para o outro a 58 ° C. Se resina epóxi é ainda muito mole permitir que outro dia para polimerizar e endurecer a 58 ° C. Teste rigidez através da aplicação de pressão usando uma pinça. Se isso deixa facilmente um recuo permitir que outro dia para polimerizar e endurecer a 58 ° C. Quando amostra é completamente endurecido avançar para desbastar e cortar.
- Corte o excesso de resina das stub usando lâminas de barbear (Figura 1B).
- Prepare resina epóxi de acordo com receitas padrão usados em laboratórios de EM.
- Fixação
- Seccionamento, contrastando, e Montagem
- seccionamento
- bloco de resina epóxi seção usando um ultramicrotome.
- Usar uma faca de vidro ou um histoknife diamante para cortar secções de cortes semifinos (500 nm) para o azul de toluidina fucsina / básico (Figura 1D) coloração para detectar o posicionamento direito.
- Transferência de secções semi-finas em lâminas microscópicas por pegá-los com uma pipeta Pasteur de vidro cuja ponta foi fechado pelo derretimento em uma chama. Seco sobre um prato quente uté a água terminar. Mancha durante 10 segundos com 1% de azul de toluidina em água em uma placa quente e após enxaguamento com água, a mancha durante 10 segundos com 0,05% fucsina básica em 1% de tetraborato de sódio. Examinar com um microscópio de luz normal em 10X a 40X.
- Quando o local apropriado / orientação dentro do cérebro é alcançado, continuar o corte do bloco de resina epóxi com uma faca de diamante para cortar cortes ultrafinos (~ 70 nm; Figura 1E)
- Use estruturas anatômicas facilmente identificáveis e em torno do cérebro, incluindo poços olfativos, olhos, limites matéria cinzenta-brancas, para identificar a região de interesse durante ultrafinos de corte e para ajustar o ângulo de corte quando a amostra é inclinada.
- Seção de montagem no único slot grades L2 x 1 cobre (Figura 1F), para permitir a aquisição ininterrupta por barras da grade. Use Formvar grades revestidas para apoiar as seções.
- contrastando
- cortes ultrafinos mancha na grades durante 2 min em 2% de acetato de uranilo em metanol seguido por citrato de chumbo de Reynolds durante mais 2 min 16.
- seccionamento
2. Aquisição de Imagem
- Large Scale STEM
Nota: Nós usamos EM digitalização com um gerador de varredura externa capaz de adquirir vários grandes campos de visão em alta resolução 3,5,7,17 usando a detecção de STEM. Tipicamente, uma imagem gerada desta forma é equivalente aos campos de visão de ~ 100 imagens Microscópio Electrónico de Transmissão (TEM), reduzindo significativamente a quantidade de costura.- Montagem da amostra no microscópio
- Coloque grade com seção no suporte de amostras grade do múltiplo e transferir para a câmara do SEM.
- Alinhamento do Detector STEM
- Coloque a grade com a amostra sob o feixe e movê-lo para que ele será aprox. 5 mm a partir do poste de lente. Adquirir uma imagem a 20 kV usandoo detector na lente.
- Concentre-se na borda da rede e ajustar a distância de trabalho para 4,0 mm.
- Mova o suporte de amostra para uma posição segura e trazer o detector STEM.
- O centro do furo do detector STEM na imagem de campo girando os ajustes parafusos enquanto os de imagem na velocidade máxima com o detector na lente.
- Cuidadosamente trazer de volta o suporte da amostra que só vai caber entre a lente do pólo e do detector.
- Adquirir uma imagem com o detector na lente ter a certeza de estar na área de seção.
- Mudar para STEM detecção (meio de ganho e de todos os quadrantes definidos em "menos") e adquirir uma imagem e selecione a área a ser digitalizada.
- Levante voltagem de aceleração de 21 kV e esperar para a estabilização do feixe (imagem estável novamente) e, em seguida, ir para 29 kV em passos de 2 kV.
- Obtenção de imagens
- Pré-irradiar a amostra (para evitar alterações de brilho causadas pelo e-beam) por zoompara fora de modo que a área total a ser digitalizado encaixa janela de imagem.
- Mudar a abertura 120 um (para manter a gama dinâmica adequada os ganhos do detector têm de ser reduzida uma etapa).
- De-foco para a imagem está desfocada.
- Faça a área digitalizada tão apertada quanto possível usando a opção de digitalização área reduzida.
- Defina a velocidade de verificação de tal modo que um quadro é digitalizada em aprox. 1-2 seg.
Nota: Dependendo do tamanho e do tecido isso normalmente leva ½ h (100 x 100 um FOV) de até 3 h (1000 x 500 mm). - Aumentar em pelo menos 100X e digitalizar uma pequena área por 10 seg.
Nota: Quando esta área não alterar o brilho em comparação ao seu redor, pré-irradiação é suficiente. - Trazer de volta a abertura 30 mm (e STEM ganho em média)
- Focar a amostra.
- Alinhar abertura usando o wobbler (em ~ 40,000X ampliação).
- Enquanto em foco selecionar o mais leve / área de recurso, defina a velocidade de verificação de tal forma que detalhessão visíveis.
- No software microscópio ajustar brilho e contraste, observando cuidadosamente o histograma para manter todos os pixels na faixa dinâmica. Faça o mesmo para os mais escuros área / recursos.
- Volte para a área brilhante e verifique novamente. Seja no lado seguro para que haja algum espaço em ambos os lados do histograma.
- Zoom out para que a área total a ser digitalizado se encaixa a janela de imagem, e iniciar o grande programa de aquisição de área.
- Use a opção de assistente para configurar um mosaico ao selecionar uma área da tela. Use tamanho do pixel 2-5 nm, dependendo do que detalhes são necessários. Use tempo de permanência 3 mS para STEM.
- Selecione a opção "autofocus na telha anterior", use o autofocus grande software de aquisição de escala, com configurações da empresa padrão, a caixa de seleção "verificar a configuração antes da execução" e definir onde salvar os dados.
- Pressione "otimizar" para verificar as configurações de microscópio.
Nota: Agora o tempo necessário será shpróprio. Isso pode ser de até 72 horas por 1 x 0,5 mm áreas. - Na janela tipo "resolução máxima telha" 20.000 telhas para individuais não será maior do que 20k x 20k, evitar efeitos de borda arredondada. Pressione o botão "executar".
- Quando perguntado para verificar a focagem e Brightness / Contrast (B / C), desligue gerador de varredura externa e cuidadosamente ajustar o foco e astigmatismo no software microscópio. Não altere B / C.
Nota: A fase de podem ser movidos e ampliação alterado, mas a ampliação deve ser ajustado de volta para o tamanho do pixel desejado. - Ligue gerador de varredura externa novamente e pressione "continuar".
- Montagem da amostra no microscópio
Análise 3. Dados
- Abra o programa visualizador de arquivos EM grande escala e abra o arquivo "info mosaico". Isto irá abrir todos os tif-arquivos azulejos. Esta preferência é realizado em outra estação de trabalho a não obstruir novas aquisições.
- Escolha a opção 'aupara costurar toda mosaico (parâmetros modo de sobrepor-se a metade, costurando limiar de 0,90, redução de ruído automática. Em caso critérios de costura não pode ser cumprida, zoom in e colocar manualmente azulejo na posição e clique em "Continuar como está").
- Exportar como um arquivo HTML, ou, alternativamente, como um único TIF.
Note que para TIF exportar um downscaling pode ser necessário. Incluir o tamanho final do pixel no nome do arquivo que permite medições mais tarde. - Execute as anotações no arquivo TIF abrindo-o em um programa de edição de imagem. Em paralelo, abra o arquivo html com zoom em um navegador da web para observação resolução ideal.
Resultados
Em larga escala EM conjunto de dados de cortes coronais da cabeça de larvas de peixe-zebra de 1 semana de idade mostra muitos tecidos e características celulares na imagem em escala de um único grande (www.nanotomy.org).
Nanotomy de seções do cérebro de controle revela características típicas ultra-estruturais de tecido neural do cérebro anterior rostral incluindo pacotes olfativos fibra, núcleos neuronais e neuronais sub-compartimentos incluindo sinapses (Figura 2A, www.nanotomy.org). tipos de células na cabeça que podem ser distinguidos incluem condrócitos com grandes vacúolos no maxilar inferior, mielina osmiophilic das células nervosas olfativas, células endoteliais de uma pequena embarcação, estruturas incluem a meninge (a contrapartida peixe-zebra das meninges, o encasing camada membranoso o cérebro de mamíferos). estruturas subcelulares incluem o glicocálice da epiderme, morfologias diferentes e focos nucleares em cel diferente l tipos e organelas, incluindo aparelho de Golgi, retículo endoplasmático (ER), mitocôndrias e vesículas sinápticas e densidades pós-sinápticas.
Inesperadamente os núcleos de células do epitélio do ventrículo são muito electrões denso comparado com outras células dispersas mais lateralmente no cérebro. Além disso, estes núcleos ventriculares mostram focos escuro que estão ausentes em outras células no cérebro e aparecem diferentes em tamanho e estrutura da heterocromatina, que se encontra nos núcleos da microglia fagocítica (Figura 2). Células que revestem o ventrículo no desenvolvimento do peixe-zebra incluem células gliais principalmente radiais e progenitores neuronais, o que pode refletir um estado de cromatina alterada do que as células mais diferenciadas. Como as células-tronco no tecido são geralmente bastante raro, nanotomy correlacionar rotulagem de tecido para marcadores de células-tronco com escala tecido EM poderia, portanto, ser usados para identificar características ultra-estruturais de células-tronco.
ve_content "fo: manter-together.within-page =" 1 "> grande escala EM (www.nanotomy.org) de uma secção coronal em uma larva de peixe-zebra submetidos à ablação neuronal revela muitos tecidos específicos, e as características moleculares e celulares que não são encontrados em animais de controlo. recursos celulares incluem microglia fagocítica e vacúolos o tamanho das células, provavelmente representando células que morrem (Figura 2B, www.nanotomy.org). eM estudos anteriores identificaram microglia em vertebrados 18,19. Aspectos característicos da microglia incluem núcleos alongados , aglomerados de cromatina irregular ao lado do envelope nuclear, aparelho de Golgi proeminente, polirribossomos livres, retículo endoplasmático granular (ER) com cisternas longa e estreita, relativamente escuro citoplasma / denso, e numerosas inclusões, tais como phagosomes, gotículas lipídicas e lisossomos. Todos estes características, atribuídas a microglia em tecidos de mamíferos, foram também encontradas nestas células em cérebros de peixe-zebra (Figura 2B, www.nanotomy.org). 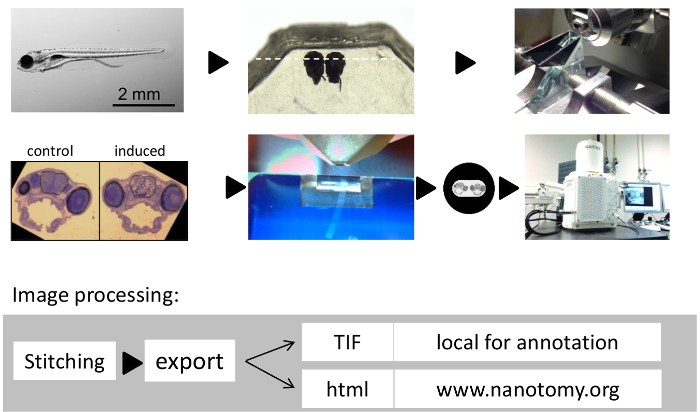
Figura 1: Fluxo de trabalho de tecido em larga escala Electron Microscopy em Brains Zebrafish. (A) 7 dias de idade, larva de peixe-zebra. (B) cabeças de larvas Zebrafish lado a lado embutidos em resina epóxi. Faca (C) Vidro para semi -thin seccionamento e orientando a amostra. Seção Representante cortes semifinos toluidina azul manchado mostrando dois (D) side-by-side incorporado larvas do peixe. (E) diamante faca para cortar ultrafinos seções (70 nm). (F) imagem editada em (D) da seção do cérebro de larvas de peixe-zebra inteira na grade de um buraco para indicar o posicionamento. (G) sistema de microscopia usado para grande escala 2D eM neste estudo. (H) Fluxo de processamento de imagem. por favor clique aquipara ver uma versão maior desta figura.
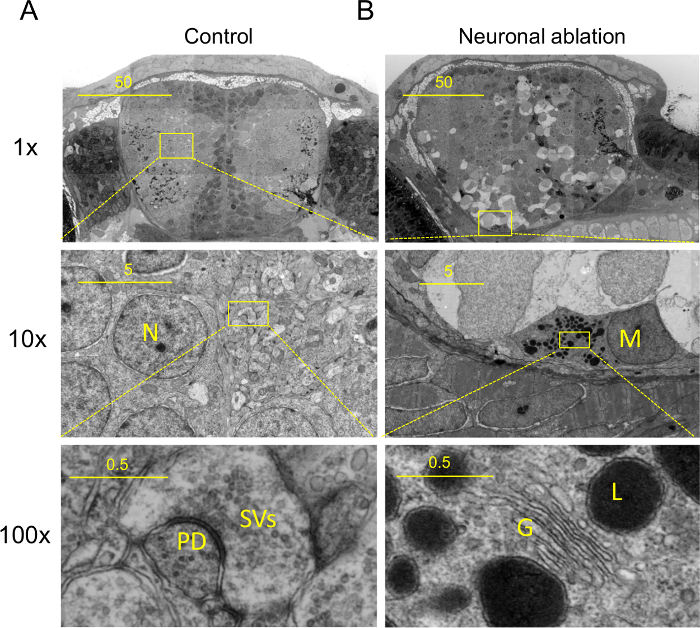
. Figura 2: Nanotomy Resultados:. A partir de Macromoléculas ao tecido (A) Nanotomy de cérebros de 7 dias de controle após a fertilização e (b) tratados (ablação neuronal; Figura 1), mostrando características específicas ao neuronal ablated degenerativa do cérebro (B) Estes incluem fagocíticas microglia, células escuras submetidos a morte celular e aparência esponjosa de tecido neural ((A) e (B)). Painéis superiores (adaptado de 5): 10X visão ampliada da região indicada em painéis de média. painéis Média: vista 10X ampliada da região indicada nos painéis inferiores. (A, painel do meio) Alto ampliação de neurópilo mostrando sinapse em (A, painel inferior), vesículas sinápticas (SVS) edensidade pós-sináptica (PD). (B, painel médio) Alto ampliação de microglia amebóides ou possivelmente um macrófago (painel do meio, M) mostrando características típicas amebóides microgliais (painel inferior), incluindo aparelho de Golgi proeminente (G), inclusões incluindo vacúolos lisossomais (L). Barras de escala: 50 mm (superior), 5 m (meio) e 0,5 m (fundo). Este valor foi modificado a partir de 5. Por favor clique aqui para ver uma versão maior desta figura , ou visite o on-line de dados completo (nanotomy.org).
Discussão
EM permite a análise de contexto celular com imagens de alta resolução das macromoléculas em contexto biológico. No entanto, isso normalmente limita o campo de visão. Maior escala 3D EM é particularmente adequado para a conectividade mapeamento neuronal através da criação de resolução nanoescala 3 reconstruções tridimensionais, exigindo processamento de dados complexos 10. Em contraste, 2D larga escala EM requer apenas uma única seção e costura dos dados de imagem e avaliação dos dados é possível por qualquer pessoa com acesso a um navegador de Internet. Nós e outros anteriormente utilizado EM grande escala para analisar tecidos e animais inteiros. Quanto mais se aproxima, TEM e costura à base de SEM tem suas próprias vantagens. Aqui, a transmissão de varredura EM (STEM) foi usada que permite a geração de um grande campo de visão em alta resolução. Normalmente, uma imagem STEM é equivalente aos campos de visão de cerca de 100 imagens de TEM, reduzindo significativamente a quantidade de costura quando imagiologia grande fields de vista em alta resolução. Se mais de alta resolução é necessária, pode ser vantajoso MET. HASTE tem a vantagem sobre TEM que as amostras não contrastou pode ser utilizado com um bom contraste ultra-6.
Além disso, o método descrito aqui pode ser simplesmente ajustadas para utilização com uma detecção electrónica dispersa de volta (DEB) em secções montadas em lâminas de Si, o alargamento do uso de vários sistemas de microscópio. Tecidos HTML-zoomable arquivos EM são muito útil para a quantificação, a partilha de dados que podem não ter sido analisados ao seu conteúdo completo, combinando LM e os dados de EM (Clem) 8, fins de apresentação na investigação científica, para analisar os dados do paciente, e para a educação. Alternativamente, em EM grande escala num SEM, mas não de uma ETM, bolachas de sílica pode ser usado, o qual tem duas vantagens principais: BSD pode ser utilizado, o que é mais, geralmente disponíveis no microscópios electrónicos de varrimento de detecção STEM. Em segundo lugar, a montagem de grandes secções (> 1 mm 2 áreas dejuros) é simples. Montagem sobre grelhas de fenda simples é trabalhoso e tecnicamente desafiador. Comparação detalhada entre TEM, SEM e STEM é detalhado em outros lugares 6. Uma desvantagem de imagiologia BSD é que, em comparação com caule, as imagens têm um aumento de ruído. Isto pode em parte ser compensada pelo aumento do tempo de permanência do pixel, resultando em tempos de aquisição mais longos.
Embora para preparação de amostras de processamento relativamente padrão EM (fixação, incorporação e de corte) é necessária 5-7, é tecnicamente desafiador para cortar grandes cortes ultrafinos completamente desprovido de artefatos. As seções são muito frágeis, facilmente quebrar, dobrar ou são destruídas durante o exame, o que normalmente leva várias horas por conjunto de dados. No entanto, devido à partilha on-line dos dados brutos imparciais, deve tornar-se possível comparar mais facilmente com dados publicados, e usar conjunto de dados publicados no domínio aberto como controlos. Actualmente, alguns dispositivos EM capaz de Laanálise rge escala estão em uso e, portanto, a acessibilidade a esta técnica é um pouco limitada, embora a maioria dos centros de imagem acolher os esforços de colaboração.
Para as secções de grande escala o tecido tem de ser correctamente fixada em toda a amostra. É por isso uma mistura do jejum, mas moderada fixador PFA é utilizado em combinação com a lenta mas forte fixador GA. Corte e pegar grandes seções sem artefatos é difícil. Trabalhando com wafers em combinação com BSD é mais fácil em comparação com a coleta seções em grades único furo. Heavy metal post coloração é mais crítica em relação a EM clássica. Desde a seção inteira é imaginada cada artefato será visível. usuários TEM normalmente têm dificuldade em mudar para um SEM, por causa da diferença na operação microscópio.
Quantificação e partilha de dados - Quantificar características subcelulares é difícil em imagens EM individuais. A possibilidade de zoom in e out de grande escalaimagens permite prontamente a identificação das células de interesse, que pode ser seguido por meio de medições em nano-escala no interior das células. Estes conjuntos de dados indicam que tipos específicos de células podem ser rapidamente identificados e quantificados de forma imparcial nestas secções de tecido de grande porte, com base em características detectáveis em diferentes escalas. Por exemplo microglia podem ser identificados com base na sua morfologia e citoplasma denso. Posteriormente, após zoom em células individuais, características subcelulares e moleculares em nanoescala dessas células pode ser medida dentro do mesmo conjunto de dados, como mostramos anteriormente para a largura ER dentro de uma larga escala de tecido EM cultura conjunto de dados 2. Uma vantagem adicional é que nanotomy hospedagem de conjuntos de dados de grande escala on-line irá permitir que outros para inspecionar os dados, talvez para outros recursos, e tirar suas conclusões sobre a nova hipótese.
CLEM - Além de facilitar EM quantitativo, EM grande escala torna mais fácil correlacionar microsco luzrotulagem pic EM nível 8. No presente exemplo, mostra-se a presença da microglia fagocíticas num modelo de ablação do peixe-zebra. Uma questão importante em neurociência é o que as funções e as contribuições individuais são de microglia e potenciais outras fontes de fagocíticas e células do sistema imunológico. Estudos EM preliminares mostraram características subcelulares distintas de microglia na doença 18. Infelizmente, é difícil rotular selectivamente microglia em particular numa configuração patológica, uma vez que exibem grande sobreposição com outras células do sistema imunológico na expressão genética, a morfologia e função. Portanto, não é claro se a microglia e quando ao nível ultra-estrutural diferente a partir de células imunes de outras fontes, incluindo derivados de monócitos macrófagos infiltrantes. Entender se existem diferenças ultra-estruturais entre estas células irá proporcionar um ponto de partida para a análise das diferenças funcionais. Combinando marcadores transgênicos ou expressão selectiva e CLEM permite que detexão de características ultra-estruturais seletiva para populações específicas.
Diagnóstico & apresentação e educação - Ao visualizar nanoescala a microescala dentro de um único conjunto de dados de dados EM é muito facilitada a um público amplo. Com o aumento das possibilidades e ferramentas para a EM-larga escala correlativa e prevemos um renascimento do EM em pesquisa básica e médica. O nosso método aqui representado é aplicado a um modelo de lesão de cérebro de peixe zebra 5, mas foi usado no tecido humano 7, cérebro de rato 3, num modelo de rato de diabetes 4 e em cultura de células de 2, e também pode ser utilizado em combinação com uma ETM abordagem com base na 1 que mostra a versatilidade deste método. O operador microscópio não é mais a gravação altamente selecionada e imagens, portanto, tendenciosas, mas todos os inúmeros recursos ultra-estruturais são registrados e aberto para análise em todo o mundo.
Divulgações
Os autores não têm nada para revelar.
Agradecimentos
Maior parte do trabalho foi realizada no UMCG Microscopia e Imaging Center (UMIC), que é patrocinado pela NWO 175-010-2009-023 e ZonMW 91.111.006; STW "Microscopia Vale 12718" para BNGG. Este trabalho foi patrocinado por uma concessão ZonMW Veni, uma bolsa de integração na carreira Marie Curie (economia morrendo neurônios) e uma bolsa de Alzheimer Nederland para TJvH
Materiais
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| low melting point agarose | VWR | 444152G | |
| tricaine | Sigma | E10521 | |
| triton-X-100 | Sigma | X100 | |
| glutaraldehyde | Polysciences | 1909 | |
| sodium cacodylate | Sigma | C0250 | |
| osmium tetroxide | Electron Microscopy Sciences | 19114 | |
| potassium ferrocyanide (K4[Fe(CN)]6) | Merck | 4984 | |
| ethanol | VWR | 20821.365 | |
| uranyl acetate | Merck | 8473 | |
| sodium tetraborate | Merck | 1063808 | |
| toluidene blue | Merck | 1273 | |
| basic fuchsin | BDH | 340324 | |
| lead citrate | BDH | discontinued | |
| EPON | |||
| 2-dodecenylsuccinicacid anhydride | Serva | 20755 | |
| methylnadic anhydride | Serva | 29452 | |
| glycid ether 100 | Serva | 21045 | |
| DMP-30 | Polysciences | 553 | |
| standard flat embedding mold | Electron Microscopy Sciences | 70901 | |
| diamond knife | Diatome Inc. | ||
| copper grids | Electron Microscopy Sciences | ||
| double sided carbon tape | Electron Microscopy Sciences | ||
| scanning EM Zeiss Supra55 | Zeiss | ||
| ultramicrotome Leica EM UC7 | Leica | ||
| atlas external scan generator | Fibics |
Referências
- Faas, F. G., et al. Virtual nanoscopy: generation of ultra-large high resolution electron microscopy maps. J Cell Biol. 198, 457-469 (2012).
- Kuipers, J., et al. FLIPPER, a combinatorial probe for correlated live imaging and electron microscopy, allows identification and quantitative analysis of various cells and organelles. Cell Tissue Res. 360, 61-70 (2015).
- Kuwajima, M., Mendenhall, J. M., Lindsey, L. F., Harris, K. M. Automated transmission-mode scanning electron microscopy (tSEM) for large volume analysis at nanoscale resolution. PLoS One. 8, e59573(2013).
- Ravelli, R. B., et al. Destruction of tissue, cells and organelles in type 1 diabetic rats presented at macromolecular resolution. Sci Rep. 3, 1804(2013).
- van Ham, T. J., et al. Intravital correlated microscopy reveals differential macrophage and microglial dynamics during resolution of neuroinflammation. Dis Model Mech. 7, 857-869 (2014).
- Kuipers, J., de Boer, P., Giepmans, B. N. Scanning EM of non-heavy metal stained biosamples: Large-field of view, high contrast and highly efficient immunolabeling. Exp Cell Res. 15477, 202-207 (2015).
- Sokol, E., et al. Large-Scale Electron Microscopy Maps of Patient Skin and Mucosa Provide Insight into Pathogenesis of Blistering Diseases. J Invest Dermatol. 135, 1763-1770 (2015).
- de Boer, P., Hoogenboom, J. P., Giepmans, B. N. Correlated light and electron microscopy: Ultrastructure lights up! Nat Methods. Nat Methods. 12, 503-513 (2015).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12, 51-54 (2015).
- Helmstaedter, M., et al. Connectomic reconstruction of the inner plexiform layer in the mouse retina. Nature. 500, 168-174 (2013).
- Oosterhof, N., Boddeke, E., van Ham, T. J. Immune cell dynamics in the CNS: Learning from the zebrafish. Glia. 63, 719-735 (2015).
- van Ham, T. J., Kokel, D., Peterson, R. T. Apoptotic cells are cleared by directional migration and elmo1- dependent macrophage engulfment. Curr Biol. 22, 830-836 (2012).
- Davison, J. M., et al. Transactivation from Gal4-VP16 transgenic insertions for tissue-specific cell labeling and ablation in zebrafish. Dev Biol. 304, 811-824 (2007).
- Distel, M., Wullimann, M. F., Koster, R. W. Optimized Gal4 genetics for permanent gene expression mapping in zebrafish. Proc Natl Acad Sci U S A. 106, 13365-13370 (2009).
- van Ham, T. J., Mapes, J., Kokel, D., Peterson, R. T. Live imaging of apoptotic cells in zebrafish. FASEB J. 24, 4336-4342 (2010).
- Reynolds, E. S. The use of lead citrate at high pH as an electron-opaque stain in electron microscopy. J Cell Biol. 17, 208-212 (1963).
- Kuwajima, M., Mendenhall, J. M., Harris, K. M. Large-volume reconstruction of brain tissue from high-resolution serial section images acquired by SEM-based scanning transmission electron microscopy. Methods Mol Biol. 950, 253-273 (2013).
- Stensaas, L. J., Reichert, W. H. Round and amoeboid microglial cells in the neonatal rabbit brain. Z Zellforsch Mikrosk Anat. 119, 147-163 (1971).
- Mori, S., Leblond, C. P. Identification of microglia in light and electron microscopy. J Comp Neurol. 135, 57-80 (1969).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados