Method Article
Discriminação e Caracterização de Populações Heterocelulares Usando Técnicas de Imagem Quantitativa
Neste Artigo
Resumo
Desenvolvemos um novo protocolo para estudar a dinâmica da população heterocelular em resposta a perturbações. Este manuscrito descreve uma plataforma baseada em imagens que produz conjuntos de dados quantitativos para a caracterização simultânea de múltiplos fenótipos celulares de populações heterocelulares de maneira robusta.
Resumo
Os processos celulares são complexos e resultam da interação entre vários tipos de células e seu ambiente. As técnicas existentes de biologia celular muitas vezes não permitem uma interpretação precisa dessa interação. Usando uma abordagem quantitativa baseada em imagens, apresentamos um protocolo de alto conteúdo para caracterizar as respostas fenotípicas dinâmicas ( ou seja , mudanças de morfologia, proliferação, apoptose) de populações de células heterogêneas para mudanças nos estímulos ambientais. Destacamos nossa capacidade de distinguir entre tipos de células baseados em intensidade de fluorescência ou características de morfologia inerentes dependendo da aplicação. Esta plataforma permite uma caracterização mais abrangente da resposta da subpopulação a perturbações ao tempo mais curto, quantidades menores de reagentes e menor probabilidade de erro do que os ensaios tradicionais de biologia celular. No entanto, em alguns casos, as populações de células podem ser difíceis de identificar e quantificar com base em células complexasCaracterísticas importantes e exigirá resolução de problemas adicionais; Destacamos algumas dessas circunstâncias no protocolo. Demonstramos esta aplicação usando a resposta ao fármaco em um modelo de câncer; No entanto, pode ser facilmente aplicado de forma mais ampla a outros processos fisiológicos. Este protocolo permite identificar subpopulações dentro de um sistema de co-cultura e caracterizar a resposta particular de cada estímulo externo.
Introdução
Os ensaios baseados em células foram um campo de trabalho nas pesquisas básicas e nas configurações de desenvolvimento de medicamentos. No entanto, as limitações desses ensaios padrão tornaram-se cada vez mais evidentes com a discordância entre dados clínicos e in vitro e a falha na maioria dos medicamentos para receber aprovação da FDA. Aqui, apresentamos um novo método para a utilização de imagem quantitativa para análise simultânea de fenótipos heterocelulares em resposta a estímulos ambientais relevantes que ocorrem.
Os ensaios tradicionais baseados em células que são utilizados para medir a viabilidade celular incluem: ensaios de exclusão de azul de tripano, MTT / MTS e coloração de citometria de fluxo de anexina V-FITC. Os ensaios de exclusão de Trypan Blue, embora simples e baratos, requerem um grande número de células, são demoradas, e muitas vezes são influenciadas pela polarização do usuário 1 . Os ensaios MTT e MTS medem indiretamente a viabilidade celular através de medidas da taxa metabólica mitocondrial. No entanto, a atividade metabólicaAs células de células podem ser afetadas por diferentes condições de cultura (como a mídia ou a concentração de oxigênio), o que leva a resultados imprecisos e evita a padronização em tipos de células e condições 2 , 3 . Outra grande desvantagem dessas técnicas é a incapacidade de distinguir entre múltiplos tipos de células - a maioria dos sistemas biológicos são heterocelulares. Embora os métodos de citometria de fluxo tenham a capacidade de distinguir entre múltiplas populações de células, os rótulos das células são necessários, a amostragem dinâmica é desafiadora e, ao usar células aderentes, esta aplicação torna-se demorada e propensa a erros.
Outros fenótipos celulares importantes, incluindo mudanças morfológicas, ocorrem em resposta a estímulos ambientais, mas não são capturados por ensaios tradicionais baseados em células. O perfil dos estados celulares através da caracterização morfológica e das semelhanças de mapeamento em amostras é uma ferramenta poderosa e imparcial com aA fim de fornecer novos conhecimentos em muitos aspectos da pesquisa básica e translacional, incluindo biologia celular básica e descoberta de drogas 4 . Além disso, a morfologia das células tumorais mostrou-se correlacionada com subtipos de tumor 5 e agressividade 6 . Por isso, é de grande interesse estudar essas características celulares e como elas se relacionam com perturbações ambientais específicas. Além disso, pode-se usar diferenças nas características morfológicas para discriminar entre subpopulações em sistemas de co-cultura. A rotulação fluorescente de células tem queda ( isto é, alterando as propriedades das células inerentes, demorado) e, portanto, métodos adicionais para classificar os tipos de células são vantajosos.
A imagem baseada em microscopia é um método alternativo para o perfil de fenótipos celulares de forma multiplexada, quantitativa e robusta. Neste manuscrito, aplicamos nosso pipeline de imagens quantitativas para destacar a evoluçãoDinâmica nítida de populações de células heterogêneas dentro de um tumor. Concentramo-nos na interação entre as células do câncer de pulmão não celular pequeno (NSCLC) e os Fibroblastos associados ao câncer (CAFs), o tipo de célula estromal mais prevalente encontrado nos tumores. CAFs foram implicados na iniciação, progressão e resposta terapêutica do tumor; Portanto, realizar ensaios fenotípicos em células tumorais na ausência de CAFs pode ser enganador 7 , 8 , 9 . Especificamente, avaliamos os efeitos de CAFs em células tumorais em resposta a erlotinib, uma molécula pequena visando o receptor do fator de crescimento epidérmico (EGFR), que é freqüentemente usado no tratamento clínico de NSCLC. Utilizamos uma plataforma de rastreio de alto conteúdo e o software de análise de imagem que o acompanha para avaliação; No entanto, na tentativa de tornar esta metodologia acessível a outros pesquisadores, também desenvolvemos um protocolo downstream comparável usando o software open-source:CellProfiler 10 e CellProfiler Analyst 11 . A maioria dos ensaios de rastreio de alto conteúdo baseados em imagens são analisados com software comercializado específico para um determinado modelo de instrumento. Os resultados são difíceis de replicar em outros laboratórios com diferentes softwares porque os algoritmos subjacentes são muitas vezes proprietários. Utilizando esta tubulação baseada em imagem, foram medidas a proliferação celular, a morte e a morfologia de cada subpopulação de uma cultura heterocelular em resposta ao tratamento medicamentoso utilizando a classificação baseada em fluorescência e morfologia. O protocolo a seguir fornece uma metodologia robusta para testar processos celulares complexos.
Protocolo
1. Cultura celular
NOTA: Aqui as respostas fenotípicas das células NSCLC (H3255) ao agente de segmentação EGFR, o erlotinib, quando co-cultivado com fibroblastos pulmonares (CCD-19Lu GFP), foram investigados. Para o mesmo conjunto de dados, foi realizada a classificação baseada em fluorescência e baseada em morfologia das duas populações de células (H3255 e CCD-19Lu GFP) para exemplificar sua concordância. No entanto, apenas um método de classificação precisa ser usado e deve ser escolhido com base no aplicativo.
- Preparar 500 mL de RPMI-1640 suplementado com 10% de FBS inactivado pelo calor e 1% de penicilina / estreptomicina.
NOTA: O meio de crescimento e os suplementos podem ser substituídos conforme necessário para outros tipos de células. - Células de cultura em placas de 10 cm3 como populações puras em 5% de CO 2 a 37 ° C e passam a proporção apropriada a cada 3-4 dias.
2. Preparação de células
- Para preparar uma suspensão celular, remova cEll media e células de lavagem com 5 mL de salina tamponada com fosfato 1X (PBS).
- Aspirar o PBS, adicionar 1 mL de tripsina a 0,05% aquecida a 37 ° C e incubar durante 5 minutos (ou até que as células não sejam mais aderidas à placa) a 37 ° C.
- Para neutralizar a tripsina, adicione 4 mL de RPMI às placas H3255 e CCD-19Lu GFP e pipeteie as soluções celulares em tubos cônicos de 15 mL.
- Centrifugação de células a 150 xg durante 5 minutos à TA.
- Aspirar o sobrenadante e ressuspender os grânulos celulares em 10 mL de meio RPMI em tubos cônicos para se preparar para a semeadura.
3. Revestimento de células
- Contar as células para padronizar a semeadura.
- Inverta os tubos para misturar as células em solução. Pipetar 10 μL de cada suspensão de células em um tubo de microcentrífuga e adicionar 10 μL de azul de tripano.
- Pipetar 10 μL desta solução em um slide de contagem de células e inserir no contador de células automatizado.
NOTA: Outros métodos de contagem de células ( ie. Hemocitómetro) podem ser utilizados. - Repita a contagem de células pelo menos três vezes, ou até que as contagens obtidas sejam consistentes.
- Células de sementes em uma placa de vários poços
NOTA: O número de placas para semente depende do número de pontos de tempo que serão fotografados. Alternativamente, uma placa pode ser re-imaginada ao longo do tempo se as células forem transduzidas com histona-2B-GFP (ou outras manchas nucleares lentivirais estáveis que proporcionem uma segmentação nuclear adequada).- Semente um total de 1.500 células / poço em 100 μL de RPMI. Prepare três suspensões de células para plaquear três populações de células: H3255, CCD-19Lu GFP e 50% de H3255 + 50% CCD-19Lu GFP. Execute cada um em triplicado com configurações de placas idênticas para cada ponto de tempo.
NOTA: As densidades iniciais de semeadura devem ser otimizadas para cada linha celular e tamanho da placa. - Células de semente em placas de 3 x 96 poços usando uma pipeta multicanal. Incubar células O / N a 37 ° C, 5% de CO 2 .
- Semente um total de 1.500 células / poço em 100 μL de RPMI. Prepare três suspensões de células para plaquear três populações de células: H3255, CCD-19Lu GFP e 50% de H3255 + 50% CCD-19Lu GFP. Execute cada um em triplicado com configurações de placas idênticas para cada ponto de tempo.
4. Dosagem de drogas
- Adicionar DMSO ao pó de erlotinib para fazer uma solução-mãe de erlotinib final de concentração de 10 mM.
NOTA: O medicamento pode ser substituído conforme desejado. - Diluir o fármaco com meio de cultura de células para 2x a concentração final com a concentração final mais alta a 10 μM e diluir em série quatro vezes a uma proporção de 1:10, para um total de cinco concentrações de fármaco e nenhum controle de drogas.
NOTA: se a concentração de DMSO for igual ou superior a 0,1% v / v com a maior dose de fármaco, o controle de drogas não deve conter quantidade equivalente de DMSO para garantir que os efeitos observados observados não sejam devidos a toxicidade de DMSO. - Pipetar 100 μL de solução de fármaco no poço apropriado para uma concentração final de fármaco de 1x diluída em meio.
5. Aquisição de imagens
NOTA: As imagens foram adquiridas nos dias 0, 2 e 3. Dependendo dos tipos de células e dos processos celulares em estudo, oOs pontos de tempo desejados podem ser estudados.
- Mancha células para se preparar para imagens.
- Elimine a solução de corante nas seguintes concentrações finais.
- Para a classificação baseada em fluorescência, prepare mancha nuclear de 5 μg / mL e mancha de células mortas 5 μM em PBS (ver Tabela de Materiais ).
- Para a classificação baseada em morfologia, prepare mancha nuclear de 5 μg / mL, mancha de células mortas 5 μM e mancha celular de 5 μM em PBS (ver Tabela de Materiais ).
- Adicione 20 μL de solução de corante a cada poço. Incubar durante 30 min a 37 ° C protegido da luz.
- Elimine a solução de corante nas seguintes concentrações finais.
- Otimize e adquira imagens.
- Retire a placa do poço da incubadora, limpe a parte inferior da placa com 70% de EtOH e coloque a placa na câmara de imagem.
- Na guia "Configuração", clique no botão '+' em 'Seleção de Canal' para adicionar os canais apropriados paraImagem (por exemplo, luminosos, manchas nucleares, manchas de células mortas, GFP e RFP).
- Clique em "Seleção de layout" e pegue imagens de teste empilhadas em z a cada 2 μm começando a 0 μm e terminando em 20 μm para identificar o plano de foco. Insira esta distância para cada canal em 'Altura'.
- Clique em "Instantâneo" em cada canal para avaliar as intensidades e otimizar os tempos de exposição. Insira os valores mais altos ou mais baixos em "Tempo" conforme necessário.
- No lado direito da tela, destaque os poços apropriados (para serem imaginados) no esquema da placa. No esquema de poços abaixo, destaque os vinte e cinco campos a serem imaginados de forma semelhante.
- Em 'Executar Experimento', identifique a placa em 'Nome da placa' na aba esquerda e clique no botão 'Iniciar' (embaixo) para executar o protocolo de aquisição de imagem com um objetivo 10X para gerar imagens TIFF em escala de cinza nos canais acima mencionados.
NOTA: instância passo a passoAs regras para o protocolo de imagem podem diferir entre os instrumentos, mas os valores dos parâmetros ainda devem ser otimizados. - Repita a imagem nos dias dois e três.
6. Análise de imagem
NOTA: Toda análise de imagem foi realizada usando software proprietário. No entanto, uma vez que esta não é uma análise pública, análises comparáveis também foram projetadas no CellProfiler 2.2 e no CellProfiler Analyst 2.0, com um breve protocolo listado abaixo e um protocolo detalhado fornecido como material suplementar (as imagens de teste já são carregadas em condutas para testar o fluxo de trabalho). As imagens desta experiência foram agrupadas em uma base por poço para que cada poço pudesse ser carregado individualmente. As tubulações CellProfiler abaixo contêm uma expressão regular que analisa as informações de metadados de cada nome de arquivo de imagem e permite que as imagens sejam agrupadas por canal.
- Baixe e instale o software de código aberto: 'CellProfiler9; E 'CellProfiler Analyst'. Aceite todas as opções e complementos padrão durante a instalação.
- Classifique as culturas heterocelulares em subpopulações.
- Abra o software 'CellProfiler', clique em 'Arquivo' | 'Oleoduto de importação' | 'De arquivo' e selecione o arquivo apropriado ("Fluorescence_Classification.cppipe" para classificação baseada em fluorescência ou "Morphology_Classification.cppipe" para classificação baseada em morfologia).
NOTA: Estes arquivos contêm protocolos para o processamento básico de imagem e segmentação de núcleos / células. Devido à mancha Hoechst desigual presente sobre essas células, os núcleos foram segmentados e ampliados e, em seguida, usados para mascarar a imagem para permitir a segmentação de núcleos com intensidades mais baixas.- Selecione a tubagem de classificação baseada em fluorescência para classificar células como H3255 ou CCD-19Lu com base na intensidade de eGFP e para calcular os recursos de morfologia.
NOTA: CCDAs células de 19Lu foram transduzidas com GFP. - Selecione a tubulação de classificação baseada em morfologia para segmentar núcleos e células, e para calcular características de morfologia.
NOTA: É necessária mais análise a jusante em 'CellProfiler Analyst' para classificação. As células mortas são classificadas através da classificação baseada em fluorescência.
- Selecione a tubagem de classificação baseada em fluorescência para classificar células como H3255 ou CCD-19Lu com base na intensidade de eGFP e para calcular os recursos de morfologia.
- Clique em 'Exibir configurações de saída' no canto inferior esquerdo da tela. Selecione a localização dos arquivos de imagem para análise ('Pasta de Entrada Padrão') e o destino para os dados extraídos ('Pasta de Saída Padrão').
- Clique em 'Analisar imagens' para começar a análise. Quando a análise estiver completa, clique em "OK" e vá para a "Pasta de saída padrão" para ver os dados calculados.
NOTA: Exemplo de valores de parâmetros e imagens estão disponíveis em Supplemental File 1-CellProfilerProtocol.pdf . - Para a classificação baseada em morfologia (somente), faça o seguinte.
- Abra o software 'CellProfiler Analyst', selecione o arquivo de propriedades do banco de dados gerado no CellProfiler e clique em 'Abrir'.
- Clique na guia "Classificador" na parte superior esquerda da tela. Para chamar imagens de células aleatórias da experiência, clique em 'Fetch'; As imagens aparecerão na janela não classificada.
- Classifique manualmente as células como positivas (H3255) ou negativas (CCD-19Lu) selecionando e arrastando as células para o compartimento apropriado (consulte o Arquivo Suplementar 2-Pipeline.pdf : Figura B ). Depois de classificar pelo menos 50 células por subpopulação, clique em 'Train Classifier' e, em seguida, clique em 'Verificar progresso'.
NOTA: As células são classificadas através de um algoritmo de aprendizagem de máquina de floresta aleatória supervisionado pelo usuário 12 . Se a precisão não for superior ao limite aprovado (> 90%), talvez seja necessário voltar e otimizar a segmentação celular em 'CellProfiler', ou as células podem não ser ideais para classiFingindo pela morfologia. - Clique em 'Pontuação Todos' para gerar uma tabela com contagem de células para cada subpopulação.
- Abra o software 'CellProfiler', clique em 'Arquivo' | 'Oleoduto de importação' | 'De arquivo' e selecione o arquivo apropriado ("Fluorescence_Classification.cppipe" para classificação baseada em fluorescência ou "Morphology_Classification.cppipe" para classificação baseada em morfologia).
Resultados
Nós geramos um conjunto de imagens consistindo de 25 campos / poço, 54 poços / placa (3 populações de células x 6 concentrações de drogas x 3 repetições), em três placas para um total de 4.050 imagens individuais. Os conjuntos de imagens geradas ao longo do experimento foram analisados usando softwares proprietários (ver tabela de materiais) para extrair várias propriedades quantitativas das células ( ou seja , morfologia, fluorescência) que poderiam então ser usadas para classificar as subpopulações celulares. No entanto, como o software comercial utilizado tem acesso limitado, foram criadas as encanamentos downstream comparáveis no CellProfiler e CellProfiler Analyst.
Classificação Heterocelular em Subpopulações
Os núcleos foram identificados e segmentados com base na mancha do DNA (aqui Hoechst) e populações de células foram classificadas quer com base na fluorescência ou moRphology ( Figura 1 ). Para a classificação baseada em fluorescência, os fibroblastos (CCD-19Lu) foram previamente transduzidos com GFP-lentivírus. Os níveis de intensidade GFP foram medidos para cada núcleo e aqueles que foram calculados acima do limite aceito (com base no sinal de fundo) foram classificados como CCD-19Lu, enquanto aqueles abaixo foram identificados como células tumorais (H3255). Para a classificação baseada em morfologia, as células foram previamente coradas com uma mancha celular não tóxica (veja a tabela de materiais) e isso foi usado para identificar e segmentar o citoplasma. Um algoritmo de aprendizado de máquina foi treinado com ~ 50-100 células de cada população. As características morfológicas foram identificadas que eram significativamente diferentes entre as populações, que foram então usadas para projetar um classificador linear para distinguir as células CCD-19Lu e H3255. Os protocolos de classificação de fluorescência e morfologia foram 97,4% (n = 1403) concordantes na distinção entre a população de duas célulasEm condições não tratadas e 92,5% (n = 916) concordantes em condições tratadas com fármaco (eretinib 1 μM) ( Figura 2 ).
Análises fenotípicas de subpopulações
Além de discriminar entre os tipos de células, buscamos caracterizar as propriedades fenotípicas de cada subpopulação. Os ensaios de multiplexagem economizam tempo e reagentes, aumentam a consistência e fornecem informações adicionais sobre o sistema em estudo. Existem muitas saídas fenotípicas potenciais e deve-se escolhê-las com base nas questões de interesse. Aqui, foram investigadas mudanças na morfologia celular e no status de viabilidade em resposta ao tratamento com erlotinib. Após três dias de tratamento medicamentoso, observou-se uma diminuição da área nuclear e um aumento na área celular das células H3255 ( Figura 3A ). A diferença média na área nuclear entreAs populações tratadas "sem drogas" e "fármaco" foram estatisticamente significativas através de um teste t de dois lados tipo-2 (variação igual) ( p = 7,92 x 10 -16 ). Nós levantamos a hipótese de que esta observação é uma resposta celular ao estresse imposto pelo tratamento medicamentoso.
Também é interessante estudar se um medicamento tem um efeito citotóxico ( ou seja, aumento do número de células mortas ao longo do tempo) ou citostático ( ou seja, diminuição do número de partos celulares ao longo do tempo) em células, pois isso tem um impacto clínico profundo. Por exemplo, um efeito citostático do medicamento induz a prisão de crescimento ainda não elimina as células do tumor, portanto existe o potencial de células cancerosas para reiniciar a proliferação celular uma vez que o medicamento é removido. Os efeitos de drogas muitas vezes podem ser o contexto, a concentração e o tipo de célula dependente. Anteriormente observamos erlotinib provocando uma resposta citotóxica em um tipo de célula, enquanto mostrava acResposta ytostatica em outros 13 .
Os ensaios de viabilidade tradicionais produzem o número relativo de células e, portanto, não discriminam as prisões de crescimento e a morte celular. Aqui, células mortas foram identificadas com base na mancha de iodeto de propídio ( Figura 3B ). Foram observados efeitos citotóxicos e citostáticos de erlotinib em células H3255, com aumento do número de óbitos e diminuição do número de nascimentos após o tratamento com drogas ( Figura 3C ). Vale ressaltar que o número de células mortas cai após o dia 1 provavelmente devido à depuração celular. As células CCD-19Lu não foram afetadas pelo fármaco. Uma vantagem adicional desta plataforma é a geração de dados quantitativos. Por exemplo, em nossa experiência de co-cultura, uma subpopulação inicial de 1.118 (75,8%) células H3255 foi de 2.817 (87,9%) ou 396 (57,2%) após três dias sem ou com erlotinaTratamento ib, respectivamente ( Figura 4 ). Como podemos gerar contagens de células reais em vez de porcentagem relativa (como com os métodos de citometria de fluxo), concluímos que a alteração na composição durante o tratamento de drogas é devido a uma diminuição das células H3255 e não ao aumento do CCD-19Lu. Não vale a pena nada que as taxas de mortalidade possam ser subestimadas devido à depuração celular, que é difícil de avaliar experimentalmente e provavelmente difere através dos tipos de células.
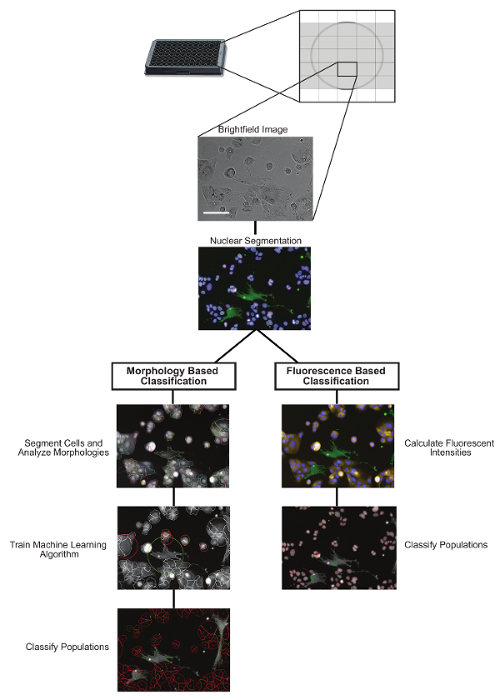
Figura 1: Visão geral do Protocolo de Análise de Imagem. Duas potenciales pipelines de análise de imagem a jusante para classificar populações heterocelulares usando classificação baseada em morfologia ou classificação baseada em fluorescência. Barras de escala = 100 μm. Clique na elaE para ver uma versão maior dessa figura.

Figura 2: Concordância entre Morfologia e Classificação Baseada em Fluorescência. ( A ) Gráfico de Concordância exibindo a sobreposição dos dois protocolos de classificação. As mesmas células foram classificadas como H3255 usando a classificação baseada em morfologia e fluorescência. Os dois protocolos estavam de acordo, com classificação de 97,4% (n = 1403) de células não tratadas e 92,5% (n = 916) de células tratadas com erlotinib (Nota: a área branca é muito pequena para visualizar). ( B ) imagens 10X que descrevem exemplos de boa e fraca concordância entre a classificação baseada em fluorescência e baseada em morfologia. As setas brancas apontam células que foram classificadas de forma inconsistente entre as plataformas. Imagem de entrada: azul - núcleo (Hoechst); Verde - CCD19Lu (GFP). ClassiImagens de ficação: Vermelho - H3255; Verde - CCD-19Lu. Barra de escala = 100 μm. Clique aqui para ver uma versão maior dessa figura.
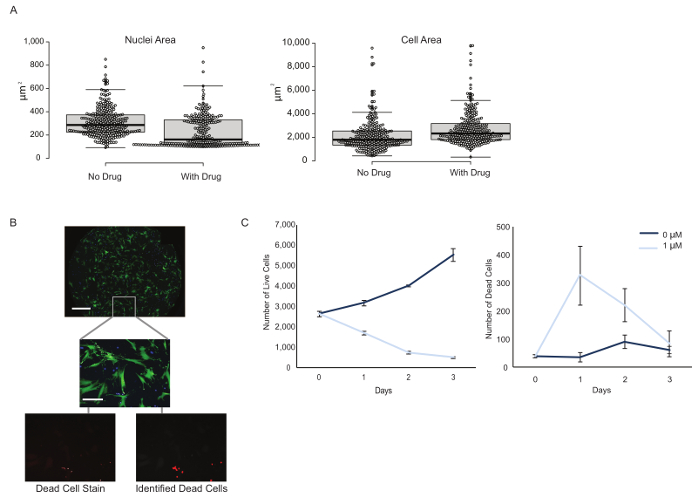
Figura 3: Medições fenotípicas multiplexadas de uma configuração experimental única. ( A ) As características morfológicas, como os núcleos e a área celular, foram calculadas no nível celular único na presença e ausência de fármaco. Nota: As áreas celulares que medem menos de 100 μm 2 foram consideradas detritos e excluídas das análises. O gráfico de caixa representa a mediana com faixas de primeiro e terceiro quartil e barras de erro de intervalo de confiança de 95%. ( B ) As células H3255 (azul) e CCD-19Lu (verde) foram co-cultivadas e as células mortas foram identificadas com base na intensidade do propídioMancha iodida (vermelha) e imagem usando um objetivo 10X. Barra de escala = 1 mm (painel superior); 100 μm (imagens do fundo). ( C ) O número total de células vivas e mortas foi calculado durante três dias com ou sem tratamento medicamentoso, com uma diminuição óbvia no número de células vivas e aumento das células mortas com a adição de erlotinib. As barras de erro representam o erro padrão da média com base em três repetições. Clique aqui para ver uma versão maior dessa figura.
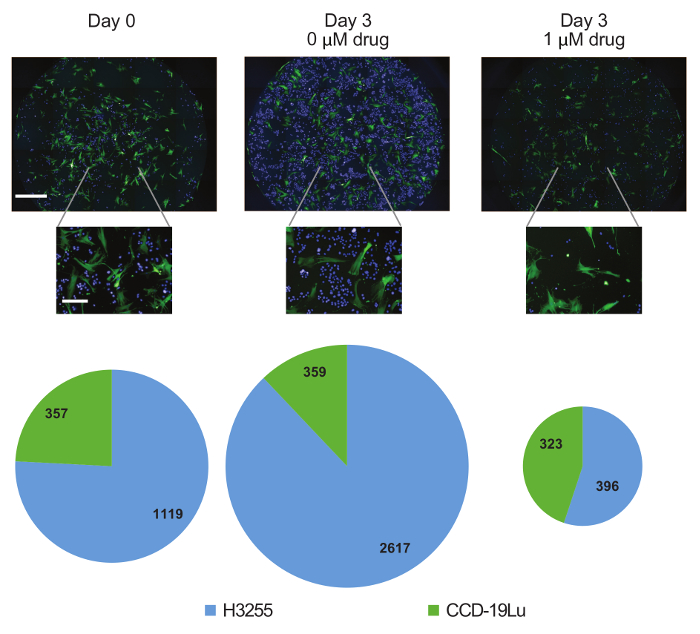
Figura 4: Dinâmica de subpopulação ao longo do tempo. Imagens representativas 10X de poços contendo H3255 (azul) e CCD-19Lu (verde) no dia 0 ou dia 3 com e sem medicamento. As células pertencentes a cada subpopulação foram contadas e os gráficos de torta proporcionais mostram aMudança cíclica na composição da população em amostras. Barras de escala = 1 mm (painéis do meio, shpwn in; eftmost panel), 1 mm (imagem superior), 100 μm (imagens de baixo). Clique aqui para ver uma versão maior dessa figura.
Discussão
The protocol described above improves upon current cell biology assays by providing more comprehensive insights into phenotypic dynamics of multiple cell types in response to environmental perturbations while using reduced reagents and time. A major advantage of this experimental design is the ability to analyze multiple phenotypes with a single setup and generate quantitative data characterizing these phenotypes on a single cell level. One technical advantage to this platform is the ease of initial troubleshooting compared to other assays. Because this method is image-based, one is able to visualize the wells for apparent over/under seeding. It is advisable to have cells in the exponential growth phase for the duration of the experiment and not be limited by nutrient or spatial constraints or confounded by senescence due to scarce seeding. Otherwise, birth and death rates may not be reproducible between experiments. For reference, CellPD is a publicly available program for computation of birth and death rates14. Additionally, one can visualize whether adequate concentrations of dyes were added to each well. Pipetting issues in an individual well could result in missegmentation and skewed data, but can easily be detected with the aforementioned protocol.
Unfortunately, not all cell types may be amenable to this application. It is important to be able to accurately segment the nuclei and cells, therefore analysis of cells that organize in more sphere-like or clumped structures may not be suitable. For some cells, it also may be advantageous to use a cell strainer prior to seeding to ensure initial seeding of single cells. In addition, the linear classifier technique is only applicable to cell types that can be readily distinguished based on morphology features.
For the success of the protocol, it is important to first optimize the imaging conditions, as the validity of downstream analyses is dependent on the quality of the images. While here we performed experiments using a high-content screening platform, image acquisition can also be performed using any fluorescent microscope (although an automated imaging platform is ideal for high-throughput approaches). Test images should be taken prior to each imaging time point to ensure that there are no problems with the microscope or protocol. The signal to noise ratio should be high, especially for the channels that will be used for segmentation (i.e. nuclei, cell stains). Additionally, it is important to image in the optimal plane of focus. If the images are out of focus, segmentation becomes much more difficult and the calculated morphological features will likely be inaccurate. Illumination differences between fields can cause problems with image segmentation as well. Large differences in brightness make the automated selection of threshold values difficult. Additionally, if there are heterogeneous fluorescence intensities between cells, a single threshold value may not sufficiently segment all the cells in an image. In this analysis, these problems were overcome by creating masks around brighter and dimmer cell populations and segmenting each population separately.
While the phenotypes under investigation in this protocol are limited to live, dead, and morphological characterization, they can easily be expanded to investigate other features. For example, functional genetic studies can be added with RNAi, overexpression, or other chemical perturbations.
In this paper, the capabilities of the protocol to measure the response of non-small cell lung cancer cells to erlotinib in the presence and absence of CAFs was demonstrated. However, this is merely one example of the many cell types and microenvironmental parameters that can be tested. We have extended this protocol to be used with other cell types and drug studies, including primary cells isolated from patient tumors13,15.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Este trabalho foi financiado pelo National Cancer Institute (NCI) Subvenções U54CA143798 e U54CA143907 para estabelecer Centros de Ciência Física-Oncologia (PS-OCs) no Dana-Farber Cancer Institute e University of Southern California, respectivamente. SM Mumenthaler recebeu um prêmio PS-OC transnetwork que apoiou alguns desses trabalhos.
Gostaríamos de expressar nossa mais profunda gratidão aos nossos apoiantes filantrópicos, particularmente a família Stephenson, Emmet, Toni e Tessa, por sua doação da plataforma Operetta HCS. Também gostaríamos de agradecer a J. Foo para orientação e os membros da equipe do Centro de Medicina Molecular: D. Agus para orientação clínica e orientação, K. Patsch para discussões significativas com projeto experimental, R. Rawat para ajuda em protocolos de análise de imagem , J. Katz para assistência técnica com a Operetta, e P. Macklin e D. Ruderman para discussões e comentários úteis.
Materiais
| Name | Company | Catalog Number | Comments |
| RPMI-1640 | Corning | 10-040-CV | cell culture medium |
| Fetal Bovine Serum (FBS) | Gemini | 100-106 | medium supplement |
| Penicillin/streptomycin | Gibco | 15-140-122 | medium supplement |
| TC20 | Biorad | 1450102 | cell counter |
| TC20 slides with trypan blue | Biorad | 1450003 | cell counter slides |
| 96-well plates | Corning | 3904 | clear bottom black plates |
| Erlotinib | LC Laboratories | E-4007 | |
| DMSO | VWR | 317275-100ML | solution to resuspend drug |
| Hoechst | Invitrogen | H21491 | nuclear dye |
| Propidium Iodide (PI) | Invitrogen | P1304MP | dead cell stain |
| Cell Tracker Orange CMRA | Life Technologies | C34551 | whole cell stain |
| Operetta high content imaging system | Perkin Elmer | ||
| CellProfiler | Broad Institue | version 2.2.0 (rev 9969f42) | http://cellprofiler.org/releases/ |
| Cell culture incubator | Any cell culture incubator will be suitable - cells were cultured under 37 ºC at 5% CO2. | ||
| 15 mL Falcon conical tubes | Falcon | 14-959-53A | |
| 10 cm2 cell culture plates | TPP | 93040 |
Referências
- Kanemura, Y., et al. Evaluation of in vitro proliferative activity of human fetal neural stem/progenitor cells using indirect measurements of viable cells based on cellular metabolic activity. J Neurosci Res. 69 (6), 869-879 (2002).
- Plumb, J. A., Milroy, R., Kaye, S. B. Effects of the pH dependence of 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-tetrazolium bromide-formazan absorption on chemosensitivity determined by a novel tetrazolium-based assay. Cancer Res. 49 (16), 4435-4440 (1989).
- Hsu, S., et al. Green tea polyphenols induce differentiation and proliferation in epidermal keratinocytes. J Pharmacol Exp Ther. 306 (1), 29-34 (2003).
- Caicedo, J. C., Singh, S., Carpenter, A. E. Applications in image-based profiling of perturbations. Curr Opin Biotechnol. 39, 134-142 (2016).
- Sero, J. E., et al. Cell shape and the microenvironment regulate nuclear translocation of NF-kappaB in breast epithelial and tumor cells. Mol Syst Biol. 11 (3), 790 (2015).
- Chen, J. F., et al. Subclassification of prostate cancer circulating tumor cells by nuclear size reveals very small nuclear circulating tumor cells in patients with visceral metastases. Cancer. 121 (18), 3240-3251 (2015).
- Ohlund, D., Elyada, E., Tuveson, D. Fibroblast heterogeneity in the cancer wound. J Exp Med. 211 (8), 1503-1523 (2014).
- Paraiso, K. H., Smalley, K. S. Fibroblast-mediated drug resistance in cancer. Biochem Pharmacol. 85 (8), 1033-1041 (2013).
- Kalluri, R. The biology and function of fibroblasts in cancer. Nat Rev Cancer. 16 (9), 582-598 (2016).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100 (2006).
- Jones, T. R., et al. CellProfiler Analyst: data exploration and analysis software for complex image-based screens. BMC Bioinformatics. 9, 482 (2008).
- Dao, D., et al. CellProfiler Analyst: interactive data exploration, analysis and classification of large biological image sets. Bioinformatics. 32 (20), 3210-3212 (2016).
- Garvey, C. M., et al. A high-content image-based method for quantitatively studying context-dependent cell population dynamics. Sci Rep. 6, 29752 (2016).
- Juarez, E. F., et al. Quantifying differences in cell line population dynamics using CellPD. BMC Syst Biol. 10 (1), 92 (2016).
- Mumenthaler, S. M., et al. The Impact of Microenvironmental Heterogeneity on the Evolution of Drug Resistance in Cancer Cells. Cancer Inform. 14, 19-31 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados