Method Article
Combinando reticulação química e espectrometria de massa de complexos de proteínas intactas para estudar a arquitetura de montagens de proteínas com multi- subunidades
Neste Artigo
Resumo
A arquitetura de complexos de proteínas é essencial para a sua função. Combinar várias técnicas de espectrometria de massa provou poderosa para estudar seu assembly. Nós fornecemos protocolos para cross-linking química e espectrometria de massa nativa e mostrar como essas técnicas complementares ajudam a elucidar a arquitetura de assemblies multi o subunit da proteína.
Resumo
Proteínas interagem com seus ligantes de forma ativa e dinâmica módulos (assemblies) que realizam várias funções celulares. Portanto, é fundamental para a compreensão dos processos celulares elucidar essas interações. No entanto, muitos complexos de proteína são assemblies dinâmicos e não são acessíveis por técnicas estruturais convencionais. Espectrometria de massa contribui para a investigação estrutural destes conjuntos, e particularmente a combinação de várias técnicas de espectrometria de massa fornece insights valiosos sobre seu arranjo estrutural.
Neste artigo, descrevemos a aplicação e a combinação de duas técnicas complementares de espectrometria de massa, ou seja cross-linking química juntamente com espectrometria de massa e espectrometria de massa nativa. Cross-linking química envolve a ligação covalente de aminoácidos nas proximidades usando reagentes químicos. Após digestão com proteases, reticulado de di-peptídeos são identificados por espectrometria de massa e sites de interações proteína são descobertos. Espectrometria de massa nativa por outro lado é a análise de conjuntos de proteína intacta na fase gasosa de um espectrômetro de massa. Ele revela que stoichiometries de proteína, bem como interações proteína e ligante. Ambas as técnicas, portanto, fornecer informações complementares sobre a estrutura da proteína-ligante assemblies e sua combinação provada poderosa em estudos anteriores.
Introdução
A investigação estrutural de assemblies de proteína tornou-se particularmente importante para a compreensão dos processos celulares. Consequentemente, muitos, técnicas foram desenvolvidos e melhorados em biologia estrutural1. No entanto, essas técnicas às vezes são limitadas na sua aplicação devido ao tamanho, flexibilidade ou heterogeneidade dos complexos proteína sob investigação. Espectrometria de massa pode lidar com esses desafios e, portanto, surgiu como uma poderosa ferramenta na biologia estrutural2,3,4,5. A maior vantagem de espectrometria de massa, no entanto, é a capacidade de identificar inequivocamente proteínas mesmo em misturas complexas e heterogêneas6. Para este fim, as proteínas geralmente são digeridas com endoproteinases e a mistura resultante do peptídeo é separada por cromatografia líquida e eluída diretamente para o espectrômetro de massa. Massas de peptídeo são posteriormente determinadas e íons precursor são selecionados para maior fragmentação dos peptides. Proteínas são então identificadas por busca do peptide e massas de fragmento correspondente contra um banco de dados conhecido. Este procedimento não só permite a identificação de peptídeos/proteínas, mas também suas modificações borne-translational que causar uma mudança em massa de peptídeos o precursor e os íons de fragmento carregando a modificação7. Algumas das muitas técnicas de espectrometria de massa estrutural se baseiam neste princípio4,8. Por exemplo, rotulagem técnicas tais como o hidrogênio deutério troca9,10, química rotulagem estratégias11,12, ou o radical hidroxilo pé impressão13,14 , dar insights sobre a acessibilidade de superfície das proteínas sob certas condições.
Outra técnica é (química) cross-linking envolvendo a ligação covalente de aminoácidos nas proximidades através de seus grupos funcionais. Por isso, reagentes químicos, reagentes UV-ativável ou ácidos aminados são independentes de15,16. Depois do cross-linking, as proteínas geralmente são hidrolisadas com proteases e reticulados di-peptídeos são analisados por espectrometria de massa acoplada a cromatografia líquida. A identificação dos produtos do cross-linking por banco de dados de pesquisa, no entanto, requer o uso de software especializado que concatenar sequências peptídicas de várias proteínas e regiões díspares17,18,19. O uso de cross-linkers químicas tem a vantagem que podem ser empregada para quase todos os complexos de proteína de interesse e não exige a incorporação de aminoácidos UV-ativável, que só pode ser alcançado quando expressar a proteína de interesse em células hospedeiras. Como tal, cross-linking é uma ferramenta versátil e foi empregado com sucesso em muitos estudos estruturais de proteínas grandes módulos (assemblies)20.
Espectrometria de massa de proteína intacta complexos (às vezes chamado de 'nativa' espectrometria de massa), por outro lado, envolve a análise de proteínas intactas e complexos sem hidrólise de proteínas em peptídeos. Ele revela a composição, heterogeneidade, estequiometria, topologia e subunidade interações da proteína complexos21,22. Em combinação com a mobilidade do íon, espectrometria de massa nativa mais permite a determinação da sua conformação23,24. Isso o torna uma ferramenta poderosa para a investigação estrutural de complexos de proteínas que são difíceis de avaliar por técnicas estruturais convencionais. No entanto, espectrometria de massa nativa requer buffers de análise que mantêm interações da proteína não-covalente durante a ionização electrospray. Isto geralmente é conseguido usando amortecedores aquosas, voláteis, tais como acetato de amônio25. Além disso, o instrumento modificações são necessárias para evitar a dissociação durante a transmissão em fase gasosa, o espectrômetro de massa26. Aplicado desta forma, muitos complexos de proteína (grande) foram analisados. Exemplos impressionantes são os estudos de ribossomas intacta27, ATP sintases28ou vírus29.
A combinação de espectrometria de massa nativa e cross-linking provou ser particularmente bem sucedida em estudos anteriores. Por exemplo, os stoichiometries de complexos de acompanhante, incluindo um inesperado dímero de Hsp70, poderiam ser obtidos experimentos de espectrometria de massa dos complexos proteína intacta, enquanto cross-linking química revelou o regime das proteínas na módulos (assemblies)30,31. Em um estudo diferente, foram estudados os efeitos das modificações borne-translational sobre uma intacta ATP sintase complexo. Espectrometria de massa nativa forneceu insights sobre a estabilidade de complexos de proteína na presença ou ausência de fosforilação ou acetilação de32,33. Então, um comparativo do cross-linking estratégia34 revelou mudanças conformacionais da proteína complexa sob diferentes condições.
Aqui, nós fornecemos os protocolos para identificação de proteínas por espectrometria de massa, cross-linking (química) e espectrometria de massa nativa, incluindo a análise dos dados e interpretação (Figura 1). A combinação de resultados complementares obtidas a partir desses métodos com a ajuda de dois complexos de proteína bem caracterizadas é demonstrada35. Nosso protocolo pode ser aplicado a qualquer conjunto de proteína que pode ser purificado em determinados pureza e concentração. A abordagem é em alguns casos limitados pela análise dos dados dos dados, ou seja, o tamanho do banco de dados do cross-linking empregada, que determina a necessária busca espaço e tempo. Além disso, a validação manual de ligações cruzadas identificadas é muitas vezes necessária e reduz ainda mais a saída. Espectrometria de massa nativa é limitada principalmente pela qualidade da amostra, por exemplo, amortecedores e adutos usado durante a purificação e a possibilidade de trocá-los por buffers aquosos e volátil. No entanto, complexos da proteína purificados para análise estrutural geralmente têm a qualidade necessária para análise de sucesso com nossos protocolos.
Protocolo
1. purificação de proteínas complexos
- Prepare a proteína complexa de acordo com protocolos padrão otimizados.
Nota: O protocolo é demonstrado aqui com o complexo RvB1/B2 de Chaetomium thermophilum e o carbamoil fosfato sintetase (CPS) de Escherichia coli. RvB1/B2 e CPS foram purificados como descrito36,,37. Todas as proteínas complexas requer um protocolo individual de purificação. Ajuste o protocolo de acordo. Buffers de amina livre como tampão fosfato salino (PBS) ou ácido 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic (HEPES) devem ser usados para cross-linking química. Substitua o tampão durante a purificação se possível.
Atenção: Não se aplicam quaisquer métodos ou reagentes que perturbam o assembly nativo da proteína complexa.
2. proteína massa baseada em espectrometria de identificação
-
Eletroforese em gel
Nota: Há gel de diferentes sistemas disponíveis e cada laboratório usa sua própria configuração. Ajuste as condições de acordo com o sistema do gel. Use luvas e avental em todo o protocolo como queratina está entre os contaminantes mais comuns na análise de espectrometria de massa.- Aplica 7.5 amortecedor da amostra 4 x µ l e 3 µ l x 10 agente redutor (concentração final 50mm ditiotreitol (DTT)) a 20 µ l da amostra da proteína. Use 10 µM CPS ou 2,5 µM RvB1/B2. Spin para baixo por 1 min em 16.200 × g e calor por 10 min a 70 ° C.
Nota: Quando utilizar um sistema de gel de diferentes ajustar os volumes necessários e a temperatura de aquecimento. - Prepare um gel de gradiente de 4-12% para a electroforese. Diluir o buffer corrente 20 vezes com água e encha a câmara de electroforese. Uso 2-(N-Morpholinos) etanesulfónico ácido (MES) buffer para proteínas de menor peso molecular (2-200 kDa) e 3-(N-Morpholinos) ácido (MOPS) buffer para proteínas maiores (14-200 kDa). Adicione antioxidantes de 0,5 mL para a câmara interna.
Nota: Quando utilizar um sistema de gel de diferentes preparar buffer de execução e o gel de acordo com os protocolos do fabricante. - Carregar um marcador de proteína adequada (pre-manchados, imaculados, diferentes tamanhos moleculares) na primeira cavidade e carregar as amostras da proteína em cavidades do restantes.
- Proteínas separadas por 35 min (MES) ou 50 min (MOPS) a 200 V.
Nota: Ajuste os tempos de eletroforese e tensão para gel de diferentes sistemas. - Para manchar as bandas de proteína, transferir o gel em um caixa de coloração de gel e cubra o gel com uma Coomassie à base de água, solução de coloração. Incube durante a noite e à temperatura ambiente num agitador de gel de horizontal.
- Destain o gel, substituindo a solução de coloração com água. Repita este passo várias vezes (aproximadamente 3 - 5 vezes) até o fundo de gel parece claro.
- Aplica 7.5 amortecedor da amostra 4 x µ l e 3 µ l x 10 agente redutor (concentração final 50mm ditiotreitol (DTT)) a 20 µ l da amostra da proteína. Use 10 µM CPS ou 2,5 µM RvB1/B2. Spin para baixo por 1 min em 16.200 × g e calor por 10 min a 70 ° C.
-
Digestão em-gel
Nota: Para evitar contaminação, use solventes de grau HPLC em todo o protocolo de digestão e para todas as etapas a seguir (ou seja, análise de espectrometria de massa). Prepare todas as soluções antes da utilização e filtrado de água e bicarbonato de amónio.- Corte as bandas de proteínas que são visualizadas por Coomassie azul mancham do gel usando um bisturi. Corte com cuidado as bandas de proteínas em pequenos pedaços de aproximadamente 1 mm × 1 mm. enxaguar o bisturi com água entre bandas de diferentes proteínas. Lave as bandas de gel com água e acetonitrilo (ACN).
- Reduzir as ligações de bissulfeto com TDT, alkylate resíduos de cisteína com iodoacetamide e digerir proteínas com tripsina conforme descrito anteriormente,38; Geralmente, uma relação de enzima: proteína de 01:20 até 1: 100 é utilizada. Use bicarbonato de amónio de 100 mM durante o protocolo de digestão.
- Extrato de peptídeos em duas etapas.
- Primeiro incubar os pedaços de gel com bicarbonato de amónio e ACN e recolher o sobrenadante contendo peptídeos. Em segundo lugar, incube os pedaços de gel com 5% (v/v) de ácido fórmico e do Grupo ACN. Incube por 15 min a cada passo. Combine ambos os sobrenadantes. Seque os peptídeos extraídos por evaporação de solventes em uma centrífuga a vácuo.
Nota: Peptídeos secados podem ser armazenados a-20 º C durante vários meses.
- Primeiro incubar os pedaços de gel com bicarbonato de amónio e ACN e recolher o sobrenadante contendo peptídeos. Em segundo lugar, incube os pedaços de gel com 5% (v/v) de ácido fórmico e do Grupo ACN. Incube por 15 min a cada passo. Combine ambos os sobrenadantes. Seque os peptídeos extraídos por evaporação de solventes em uma centrífuga a vácuo.
-
Espectrometria de massa acoplada a cromatografia líquida
- Dissolva os peptídeos secados em ácido fórmico a 2% ACN/0.1%. Dissolva os peptídeos num banho sonication durante 2-3 min e spin para baixo em uma centrífuga a 16.200 x g durante 30 min. transferência das amostras dentro dos frascos de mostruário.
Nota: Ajuste o volume de acordo com a quantidade de proteína. Para bandas de Coomassie bem manchadas usamos 20 µ l. - Injete 5 µ l da amostra para o sistema de nano-LC-MS/MS utilizando o mostruário. Coloque a mistura de peptídeo em uma pré-coluna fase reversa C18 (C18, 150 µm identificação, 2 cm, tamanho de poros de 5 µm) do desalt e concentrar os peptídeos on-line.
- Use a 0,1% (v/v) de ácido fórmico, como fase móvel A e 80% ACN/0.1% (v/v) de ácido fórmico como mobile fase B. separar os peptídeos em uma coluna analítica de C18 de fase reversa (C18, 75 µm, carteira de identidade, de 50 cm, tamanho de poro de 3 µm) usando um gradiente de 4-80% B (contendo 0,1% de ácido fórmico) em 300 nL / mais de 65 min de min.
Nota: Uma fonte de íon nanospray é usada para transferir eluted peptídeos para o espectrômetro de massa. - Usar condições de MS (típicas): pulverizar a tensão de 1,6 kV; capilar temperatura de 250 ° C; energia de colisão normalizado de 30. Opere o espectrômetro de massa no modo dependente de dados.
- Adquirir os espectros de MS no analisador de massa (por exemplo, orbitrap) (m/z 350−1, 600) com uma resolução de 70.000 e um alvo de controle automático de ganho de 3 x 106. Selecione 20 dos íons mais intensos para a fragmentação do HCD em um alvo de controle automático de ganho de 1 x 105. Dinamicamente excluir selecionado anteriormente íons por 30 s. Exclude isoladamente carregado íons, bem como íons com o estado de carga não reconhecido.
Nota: Calibração interna dos espectros de massa foi realizada usando o bloqueio opção massa39.
- Dissolva os peptídeos secados em ácido fórmico a 2% ACN/0.1%. Dissolva os peptídeos num banho sonication durante 2-3 min e spin para baixo em uma centrífuga a 16.200 x g durante 30 min. transferência das amostras dentro dos frascos de mostruário.
-
Pesquisando o banco de dados
Nota: Há diferentes softwares disponíveis para a pesquisa de banco de dados. MaxQuant40, por exemplo, está disponível gratuitamente.- Converta arquivos RAW para .mgf de arquivos usando uma ferramenta de conversão de pXtract (http://pfind.ict.ac.cn/downloads.html).
- Realizar pesquisa de banco de dados usando parâmetros de pesquisa típico: banco de dados, swissprot; tolerância de massa do peptide, 10 ppm; fragmento de tolerância em massa, Da 0,5; enzima, tripsina; sites de clivagem perdidas, 2; modificações na variáveis, carbamidomethylation (cisteína) e oxidação (metionina).
Nota: Altere os parâmetros de pesquisa de acordo com as configurações experimentais. - Inspecione o resultado da pesquisa de banco de dados. Avaliar o escore de proteína, número de peptídeos identificados, golo de peptídeo e precisão em massa; a cobertura de sequência retorna a cobertura de proteína identificada.
Nota: Cada motor de busca tem seu algoritmo de Pontuação individual. Avalie o sistema de Pontuação pela qualidade dos espectros de massa em tandem observado o peptídeo. Os principais sinais de um bom espectro devem mostrar série de íon (completa) para verificar se o peptídeo identificado. Geralmente, a pontuação de peptídeo é baseada em probabilidade, ou seja, a pontuação de peptídeo é uma medida para como provável a sequência do peptídeo identificados coincide com o espectro obtido. A pontuação de proteína é geralmente derivada de escores do peptide e é usada para classificar uma proteína em uma lista de proteínas identificadas.
3. espectrometria de complexos de proteína intacta (espectrometria de massa nativa)
- Preparação de emissores de ouro-revestido para ionização electrospray
- Use um extrator de micropipeta para preparar nanoflow emissores de capilares de vidro, conforme descrito anteriormente,25,,41. Use os capilares de borosilicato de 0,78 mm de diâmetro interno.
Nota: Alterando os parâmetros para agulha puxando, a forma da ponta e o tamanho podem ser modificados e ajustados para a amostra. Capilares com espessura de diâmetro e parede interna diferente estão disponíveis. - Revestir os capilares de vidro com material condutor (por exemplo, ouro ou paládio); é comum o uso de um aplicador por pulverização catódica, gerando um plasma de ouro. Siga as instruções do fabricante para obter o revestimento de boa qualidade.
Nota: O revestimento deve ser suficiente para obter um pulverizador estável ao aplicar tensões capilares comuns (veja abaixo).
- Use um extrator de micropipeta para preparar nanoflow emissores de capilares de vidro, conforme descrito anteriormente,25,,41. Use os capilares de borosilicato de 0,78 mm de diâmetro interno.
- Preparação da amostra para espectrometria de massa nativa
Nota: Sais, detergentes ou grandes quantidades de glicerol não são compatíveis com ionização electrospray. Portanto, o buffer de purificação é trocado por um buffer de voláteis, aquoso. acetato de amônio 200 mM é comumente usado. Uso tamanho Exclusão centrifugação colunas ou dispositivos de ultrafiltração para troca de amortecedor. Em alguns casos, a estabilidade do complexa ou atividade poderá ser afetada pela troca de amortecedor. Avaliar os espectros de massa cuidadosamente e verificar a actividade do complexo. Adicione cofactores ou aditivos para o buffer de análise, se necessário.- Use colunas de tamanho Exclusão centrifugação para troca rápida de reserva. Remover o buffer de armazenamento por centrifugação a 1.000 g de x e 4 ° C, durante 1 min. descarte o fluxo através de. Lavar três vezes adicionando 500 µ l 200 mM acetato de amónio, seguido por centrifugação. Carrega a 20 µ l da amostra da proteína para a coluna e centrifugar a 1.000 x g e 4 ° C por 4 min.
Nota: A concentração da proteína complexa deve ser de 1-10 µM. O procedimento pode ser repetido se a componentes não-volátil ainda perturbam a análise. - Para concentrar a amostra de proteína e trocar o buffer no mesmo experimento usar filtros centrífugos. Use uma membrana de filtração de poros tamanho 50% menor que o tamanho das proteínas analisadas.
- Transferir a amostra de proteína para o dispositivo de filtragem e adicionar o acetato de amônio 200 mM. Spin para baixo a 15.000 x g e descartar a fluir. Adicionar o acetato de amônio 200 mM e repita a centrifugação. Repita este passo várias vezes. Siga as instruções do fabricante para a velocidade de centrifugação. Realizar a centrifugação a 4 ° C.
Atenção: Proteínas de membrana tendem a precipitar-se sobre a membrana do dispositivo de filtro.
- Transferir a amostra de proteína para o dispositivo de filtragem e adicionar o acetato de amônio 200 mM. Spin para baixo a 15.000 x g e descartar a fluir. Adicionar o acetato de amônio 200 mM e repita a centrifugação. Repita este passo várias vezes. Siga as instruções do fabricante para a velocidade de centrifugação. Realizar a centrifugação a 4 ° C.
- Use colunas de tamanho Exclusão centrifugação para troca rápida de reserva. Remover o buffer de armazenamento por centrifugação a 1.000 g de x e 4 ° C, durante 1 min. descarte o fluxo através de. Lavar três vezes adicionando 500 µ l 200 mM acetato de amónio, seguido por centrifugação. Carrega a 20 µ l da amostra da proteína para a coluna e centrifugar a 1.000 x g e 4 ° C por 4 min.
- Análise de espectrometria de massa de complexos de proteína intacta
Nota: Existem diferentes tipos de espectrómetros de massa de fabricantes diferentes que podem ser modificados por espectrometria de massa nativa, por exemplo, espectrómetros de massa quadrupolo tempo-de-voo (Q-ToF) ou orbitrap espectrómetros de massa. O protocolo descrito abaixo foi realizado num instrumento de Q-ToF.- Coloque um capilar revestido de ouro no porta-capilar e preencher o capilar com 1-4 µ l da amostra de proteína. Abra a ponta da agulha com uma pinça.
- Conectar o titular capilar com a fonte de nano-electrospray e ajustar a posição do capilar. Posicione a ponta do capilar em 0.5-1.5 cm para o orifício do cone. Utilize gás de 80-150 L/h nanoflow para iniciar o spray e ajustar o fluxo de gás para manter um spray estável.
- Ajuste os parâmetros na melodia-página do instrumento Q-ToF. Condições de partida típicas são: tensão capilar, 1,50 kV; tensão de cone, 80 V; Energia de RF lente 1, 80 V; energia de colisão, 20 V; abertura 3, 13,6 V. modificar estes parâmetros para obter espectros de massa boas. Iniciar a aquisição clicando no botão "adquirir" e combinar tantos exames quanto possível para obter um bom espectro de massa.
Nota: Recomendamos a combinação de pelo menos 100 varreduras.
- Espectrometria de massa em tandem de complexos de proteína intacta
- Adquira o espectro de massa como descrito acima (protocolo seção 3.3). Escolha um íon precursor de um complexo de proteínas.
- Mudar do MS em modo de MS/MS no arquivo de aquisição. Definir a seleção de MS/MS para o precursor em massa.
- Inicie a aquisição em baixa energia. Combinar vários exames (aproximadamente 20 exames) para verificar se a seleção do correto precursor massa. Aumenta a energia de colisão até se dissocia do complexo de proteínas. Para obter um bom espectro de massa combinar pelo menos 500 exames.
Nota: Despojados complexos às vezes têm baixa intensidade. Combinando a tantos exames quanto possível pode aumentar a relação sinal-ruído e da resolução.
- Dissociação na solução de complexos de proteína intacta
Nota: Para ganhar a introspecção adicional em interações da proteína dentro dos complexos de proteínas, dissociação em solução pode ser executada.- Prepare a amostra de proteína, como descrito acima (seção 3.2). Adicionar solventes para a amostra de proteína ou alterar o pH para dissociar complexos intactos em sub complexos. Solventes típicos são o metanol, isopropanol e ACN; Altere o pH do acetato de amónio pela adição de ácido acético ou solução de amônia. Aquisição de espectros de massa como descrito acima (seções 3.3 e 3.4).
- Varia a quantidade de solvente ou o intervalo de pH para gerar vários complexos sub. Normalmente, a quantidade de solvente é 5-50% (concentração final), e a faixa de pH típico é de 4-9. Comece com uma baixa concentração de solvente (5%) ou pequenas alterações no pH e adquirir um espectro de massa (veja seção 3.3).
- Aumentar a quantidade de solvente ou alterar o pH em etapas até sub complexos são gerados em solução. Aquisição de espectros de massa para analisar complexos sub.
- Calibrar dados
Nota: Espectros de massa adquiridos são calibrados externamente usando solução de (CsI) de iodeto de césio.- Dissolva 100 mg de CsI em 1 mL de água.
- Adquira um espectro de massa de CsI. Varia a energia de colisão para obter clusters de CsI na mesma gama de m/z como o complexo de proteínas analisadas acima.
Cuidado: CsI rapidamente precipita-se na ponta do emissor e contamina o cone. Adquirir apenas como muitos examina como necessário para obter um espectro suficiente. Remova o emissor da fonte quando terminar. - Fazer um arquivo de calibração utilizando o espectro de massa adquirido e um arquivo de referência do CsI.
- Aplica a calibração de espectros de massa adquiridos.
Cuidado: Calibração de espectros de massa pode ser uma mudança permanente dos dados brutos. Se não calibrado espectros são necessários, faça uma cópia de backup do arquivo.
- Análise e processamento de dados
Nota: Existem muitas ferramentas de software disponível gratuitamente para análise de dados de espectros de massa nativos; por exemplo, Massign42 ou UniDec43. O protocolo abaixo descreve a análise manual dos dados com a ajuda do software do instrumento, bem como o uso de Massign para amostras complexas. Este software é bem adequada para a análise de espectros de massa complexas. Siga as instruções fornecidas on-line para uso do programa (http://massign.chem.ox.ac.uk/).- Para análise de dados, suave espectros ajustando parâmetros de suavização. Espectros de centroide ajustando parâmetros. Calcular massas complexas de dois picos adjacentes da proteína complexa ' envelope pico usando ferramentas de software do instrumento.
Cuidado: Suavização muito intenso pode causar perda de dados (por exemplo, perda de ligantes acoplados). - Para análise com Massign42, gere uma lista de pico para o espectro de massa. Linearizar pontos de dados do espectro e suave. Use as várias ferramentas de software para atribuir complexos da proteína, calcular a composição complexa ou simular envelopes pico complexo.
- Para análise de dados, suave espectros ajustando parâmetros de suavização. Espectros de centroide ajustando parâmetros. Calcular massas complexas de dois picos adjacentes da proteína complexa ' envelope pico usando ferramentas de software do instrumento.
4. química do cross-linking acoplada com espectrometria de massa
Nota: Existem várias estratégias do cross-linking disponível. Aqui, descrevemos o uso do Bis (sulfosuccinimidyl) suberate (BS3), um cross-linker amina-reativo que é comumente usada para estudar as interações da proteína-proteína.
- Dissolva 1,43 mg BS3 na água, 100 µ l prepare uma solução stock de 25 mM.
Nota: Outros reagentes como disuccinimidyl suberate (DSS) não são solúveis em água e geralmente são dissolvidos em dimetilsulfóxido (DMSO). Alguns cross-linkers também estão disponíveis em formas de desvendar-rotulado pesadas. Incorporação dos isótopos estáveis pesados gera pares de pico de di-peptídeos reticulados em espectros de MS, que ajuda na avaliação de dados. Quando usando diferencialmente rotulados cross-linkers, as soluções de ambas as variantes são preparadas e mistura 1:1.
Cuidado: Para evitar a dissociação dos complexos proteína por DMSO, uma solução altamente concentrada é preparada e diluída com água ou buffer antes da reação do cross-linking. - Adicione BS3 para o complexo de proteínas. Use quantidades variadas de BS3 variando de 0,5-5 mM para identificar a concentração ideal cross-linker. Incube as misturas de reação a 25 ° C, durante 1 h em um thermomixer. Usar géis gradiente de 4-12% e executar SDS-PAGE para avaliar resultados do cross-linking (consulte a Figura 2 , por exemplo).
Nota: A concentração ideal de BS3 é atingida quando maiores bandas de peso molecular da proteína, que não são visíveis no controle non-cross-linked, são obtidas durante SDS-PAGE, enquanto única subunidades são ainda visíveis (Figura 2). - Repeti a reação do cross-linking com a concentração ideal de BS3. Incube as misturas de reação a 25 ° C, durante 1 h em um thermomixer.
Nota: Alguns complexos de proteínas não são estáveis à temperatura ambiente. A reação do cross-linking também pode ser executada no gelo; no entanto, o tempo de reação precisa ser ajustada. -
Saciar a reação do cross-linking, adicionando o buffer de amina (por exemplo, 50-100 mM de tampão Tris, pH 7,5, concentração final) ou executar a precipitação do álcool etílico para remover qualquer reagente residual do cross-linking.
- Adicionar água ou buffer para a mistura de reação para atingir um volume final de 200 µ l. adicionar 600 µ l gelada etanol e 20 µ l 3 M de acetato de sódio, pH 5,3. Homogeneizar e incubar a-20 º C por 2 h ou durante a noite.
Nota: Como alternativa, as proteínas podem ser precipitadas a 80 ° C por 30 min ou em nitrogênio líquido. - Spin para baixo a 16.200 x g e 4 ° C por 30 min. Retire com cuidado o sobrenadante.
- Lave o sedimento com 1 mL de etanol a 80% (v/v) gelada. Spin para baixo a 16.200 x g e 4 ° C por 30 min. Retire com cuidado o sobrenadante. Seque a pelota em uma centrífuga a vácuo.
- Adicionar água ou buffer para a mistura de reação para atingir um volume final de 200 µ l. adicionar 600 µ l gelada etanol e 20 µ l 3 M de acetato de sódio, pH 5,3. Homogeneizar e incubar a-20 º C por 2 h ou durante a noite.
- Execute SDS-PAGE das proteínas do reticulado. Corte as bandas de gel e digerir o proteína em gel, como descrito acima (seções 2.1 e 2.2).
Nota: Digestão em solução também pode ser realizada, no entanto, geralmente requer etapas adicionais de separação (por exemplo, cromatografia de exclusão dos peptides digeridos). -
Realize a espectrometria de massa acoplada a cromatografia líquida conforme descrito acima (seção 2.3). Como peptídeos reticulados são geralmente baixa abundante, aplicam-se as seguintes variações para a análise de espectrometria de massa para aumentar a profundidade analítica da amostra reticulada.
- Use gradientes mais tempo durante a separação de cromatografia líquida (por exemplo, 90 min, em vez de 65 min, veja acima).
- Exclua os peptídeos duplamente carregados de fragmentação HCD.
Nota: Duplamente carregados peptídeos são geralmente intra-cross-linked peptídeos ("laço peptídeos" ou "tipo 1" ligações cruzadas). - Quando usando diferencialmente rotulados reagentes do cross-linking, use a opção "escolher pico" durante a análise, ou seja, fragmentação HCD é desencadeada pela presença de pares de pico da diferença de massa definida em espectros de massa.
Nota: O pico "escolher" opção não pode estar disponível em todos os espectrômetro de massa.
- Uso pLink software18 para identificação de di-peptídeos reticulados. Use bancos de dados minimizados para identificação. Parâmetros de busca típicos são: instrumento de espectros, HCD; enzima, tripsina; no máximo. sites de clivagem perdidas, 3; modificações variáveis, oxidação (metionina) e carbamidomethylation (cisteína); cross-linker, BS3; comprimento de peptídeo de min., 4; no máximo. comprimento do peptide, 100; min. massa de peptídeo, 400 Da; no máximo. massa do peptide, 10.000 Da; FDR, 1%.
Nota: Quando utilizar diferencialmente rotulados cross-linkers, o aumento da massa causado pelo vinculador precisa ser configurado. Há também outros softwares usados para identificação de ligações cruzadas, por exemplo, xQuest17, MassMatrix19ou XlinkX15. - Avalie os resultados da pesquisa de banco de dados com a qualidade dos espectros de fragmentação. Espectros aceitáveis das ligações cruzadas devem mostrar série de iões de ambos os peptídeos (pelo menos 4 íons adjacentes) com uma razoável relação sinal-ruído.
Nota: Quando utilizar diferencialmente rotulados reagentes do cross-linking, pares de pico em espectros de MS podem ser usados como um controle de qualidade adicional. - Se necessário, visualize os resultados do cross-linking em redes de interação de proteínas usando ferramentas de software (por exemplo, XVis, XiNET). Uso bar parcelas circulares ou parcelas para visualização das interações da proteína.
Nota: As duas ferramentas de software estão disponíveis gratuitamente em um servidor web. Siga as instruções detalhadas nos respectivos sites (https://xvis.genzentrum.lmu.de/ e http://crosslinkviewer.org/).
Resultados
A análise estrutural de proteínas e os complexos que formam é fundamental para a compreensão de sua função. Espectrometria de massa contribui consideravelmente para a investigação estrutural em que ele pode ser aplicado a quase todos os complexos turísticos, independentemente da dimensão ou heterogeneidade da amostra. Podemos exemplificar o protocolo usando dois complexos de proteína bem caracterizadas; Primeiro, a hetero-dodecamer RvB1/B2 de c. thermophilum e segundo, o CPS de hetero-octameric complexo de e. coli.
Primeiro, nós identificamos os componentes de proteína dos dois complexos. Para isso, as proteínas foram separadas por SDS-PAGE (Figura 2) e gel de bandas foram cortadas do gel. Depois de em-gel a digestão das proteínas, a mistura de peptídeo foi analisada por espectrometria de massa acoplada a cromatografia líquida e massas do peptide e fragmento foram submetidas à pesquisa de banco de dados. Seguindo este fluxo de trabalho, identificamos todas as subunidades de proteína dos dois complexos com alta confiança, ou seja, um número elevado de peptídeos com golo de peptídeo razoável observou-se rendendo a cobertura alta sequência para todas as subunidades de proteína (tabela 1 ).
Então analisamos o intacto RvB1/B2 complexo por espectrometria de massa nativa (Figura 3A). O espectro de massa revelou duas espécies, uma em aproximadamente 8.000 m/z e outra espécie em aproximadamente 11.000-12.000 m/z. As massas calculadas para essas espécies correspondem o hexamérica (RvB1)3(RvB2)3 anel (aproximadamente 310 kDa) e o dodecameric duplo-anel (RvB1)6(RvB2)6 (cerca de 620 kDa). Ambas as séries de pico mostram duas populações; Estas originam uma mistura de subunidades de RvB2 sua marcados e não marcas na complexos. Uma estrutura de cristal para o complexo RvB1/B2 foi obtida anteriormente36 e mostra o arranjo do duplo-anel (Figura 3B). O espectro de massa nativo confirma, portanto, a estequiometria do dodecamer intacto e além disso, revela um complexo estável de sub. Além disso, as populações co existentes são identificadas.
Para identificar sites de interação da proteína no complexo RvB1/B2, nós quimicamente reticulado o complexo purificado com cross-linker BS3. Nós primeiro titulado a quantidade de BS3 durante a reação do cross-linking para determinar a concentração ideal. BS3 é amina específicos e covalentemente cadeias de laterais de lisina de ligações, bem como o N-termini de proteínas. A reação do cross-linking foi seguida por SDS-PAGE (Figura 2A). O complexo de non-cross-linked mostrou subunidades tanto RvB1 e RvB2. Adicionar BS3 a mistura de reacção causada ligação covalente das proteínas resultando em bandas de proteína no peso molecular mais elevado. O gel de SDS mostra que quantidades crescentes de BS3 produzem quantidades mais elevadas de reticulado espécie enquanto subunidades proteicas Cruz não-vinculadas são reduzidas. Em seguida, corte as bandas de proteínas do gel e seguiram o protocolo fornecido acima para identificar sites de interação de proteínas. Um espectro de exemplo de um reticulado di-peptídeo é mostrado (Figura 3). O espectro mostra série y de iões de ambos os peptídeos confirmando essa interação de proteínas. No total, obtivemos 14 interações da proteína, incluindo quatro ligações cruzadas entre subunidades RvB1 e RvB2 e duas ligações cruzadas entre duas cópias do RvB2 (tabela 2). Os resultados de BS3 cross-linking são visualizados em uma rede de interação (Figura 3D) mostrando interações intra moleculares, bem como interações entre diferentes subunidades. Ligações cruzadas intra moleculares sugerem que tanto RvB1 como RvB2 subunidades dobram de uma forma que domínios N - e C-terminal estão em estreita proximidade. Note, que interações intra moleculares não podem ser distinguidas de interacções inter moleculares de subunidade mesma neste caso. Inter moleculares ligações cruzadas entre as duas subunidades também foram observadas. Destas, duas ligações cruzadas inter moleculares entre RvB1 e RvB2 podem ser visualizadas na estrutura validando a abordagem do cross-linking. As outras ligações inter moleculares cruzadas estão localizadas em loops flexíveis que não estejam incluídos na estrutura de cristal. Também identificamos duas ligações cruzadas em RvB2 que contém as mesmas sequências peptídicas. Estas ligações cruzadas sem ambiguidade podem ser classificadas como inter molecular como eles devem ser provenientes de duas cópias da mesma proteína (Figura 3D). Nossos experimentos do cross-linking revelaram sites de interação da proteína dentro do complexo, mas também dentro as subunidades de proteína, fornecendo insights sobre seu arranjo estrutural que também pode ser confirmado pela estrutura existente de cristal (Figura 3B).
O segundo complexo de proteína que estudamos foi CPS. O espectro de massa nativo (Figura 4A) revelou três complexos de proteína entre 6.000 e 12.000 m/z. O maior complexo de 640 kDa corresponde a hetero-octamer intacto. Os complexos menores representam dois sub complexos; o dímero das pequenas e grandes subunidades de CPS (160 kDa) e um hétero-tetrâmero contendo duas cópias de cada subunidade (320 kDa). Estes complexos sub entregam primeiros insights sobre o conjunto de proteínas; ou seja, as grandes e pequenas subunidades estão em contato direto (como revelado por hetero-dímero) e o tetrâmero pode ser um produto de dois dímeros. Para obter mais informações sobre o arranjo estrutural no complexo de CPS intacto, realizamos a espectrometria de massa em tandem (MS/MS) da hetero-octamer e o hétero-tetrâmero. Em ambos os casos, a subunidade pequena dissociada do precursor, sugerindo que a subunidade pequena está localizada na periferia da Assembleia (Figura 4B). Com efeito, a subunidade pequena é periférica no cristal disponível estrutura (Figura 4)44.
Química do cross-linking usando o cross-linker BS3 também foi realizado. A ligação covalente das subunidades CPS usando quantidades crescentes, foi reforçada. Após a digestão das proteínas e análise dos peptides como descrito acima, muitas interações da proteína dentro da subunidade grande e uma ligação transversal na subunidade pequena foram obtidas (tabela 2). Além disso, semelhante do complexo RvB1/B2, encontramos duas ligações cruzadas inter moleculares entre duas cópias de grande subunidade CPS. Estas ligações cruzadas Coloque as duas subunidades grandes frente para o outro em seus lados C-terminal. Em um estudo anterior, combinando a espectrometria de massa estrutural e modelagem computacional35, nós identificamos três interações adicionais na grande subunidade que provavelmente se originam a partir da interface de duas cópias de grande subunidade validado pelo a estrutura de cristal e o modelo obtido (Figura 4 e tabela 2). Essas interações permitem que o arranjo do núcleo CPS complexo constituído por quatro subunidades grandes. No entanto, não há ligações de subunidade inter cruzadas entre subunidades grandes e os pequenos foram observadas. Inspecionando a estrutura de cristal disponível (Figura 4), torna-se aparente que a superfície de interação entre o núcleo tetramérica do complexo, consistindo a subunidade grande e as subunidades periféricas pequenas é muito pequena, o que pode explicar a ausência de interações inter subunidade. Isto é confirmado por espectrometria de massa nativa que mostrou que a subunidade pequena prontamente dissocia-se dos complexos intactos provavelmente devido a uma interface de ligação pequena. Não obstante, interações da proteína no CPS complexo combinado do cross-linking química e espectrometria de massa nativa permitem deduzir seu arranjo estrutural (Figura 4).
Tomados em conjunto, a combinação de espectrometria de massa nativa e química do cross-linking acoplada com espectrometria de massa identificação de peptídeos reticulados, permite a reconstituição do arranjo estrutural dos dois complexos de exemplo. Enquanto o cross-linking química revelou o arranjo das subunidades da proteína, por exemplo, as interações entre RvB1 e RvB2 ou dentro do núcleo tetramérica da CPS, espectrometria de massa nativa entregue stoichiometries de proteína dos complexos intactos e subcomplexes predominantes. No caso de CPS, para os quais não há interações inter moleculares entre as duas subunidades poderiam ser observadas pela química do cross-linking, espectrometria de massa nativa sugere que cada subunidade grande interage com uma subunidade pequena (Figura 4). Espectrometria de massa em tandem propôs a localização periférica de subunidade pequena no complexo e uma pequena interface entre as duas subunidades.
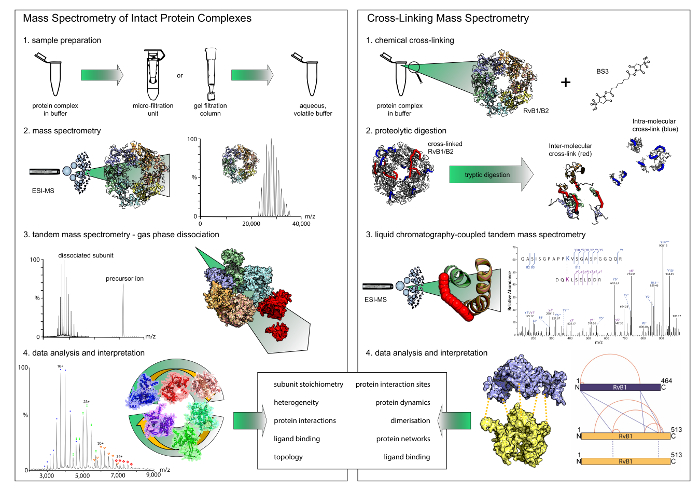
Figura 1: fluxo de trabalho de espectrometria de massa nativa e cross-linking. Ambas as técnicas resultados complementares. Enquanto a espectrometria de massa nativa revela stoichiometries e módulos de interação, cross-linking dá insights sobre os sites de interação da proteína dentro dos complexos. Observe que cross-linking química só revela interações binárias. (A), o primeiro passo na espectrometria de massa nativa é troca de buffer para um buffer de voláteis e aquosa, usando unidades de filtro ou colunas de gel filtração. Espectrometria de massa dos complexos proteína intacta então revela sua estequiometria. Em experimentos de espectrometria de massa em tandem, subunidades periféricas são dissociadas. (B) para cross-linking química, o complexo de proteínas é incubado com um reagente do cross-linking. As proteínas reticuladas são então digeridas em peptídeos, que são posteriormente analisados por espectrometria de massa acoplada a cromatografia líquida. Clique aqui para ver uma versão maior desta figura.
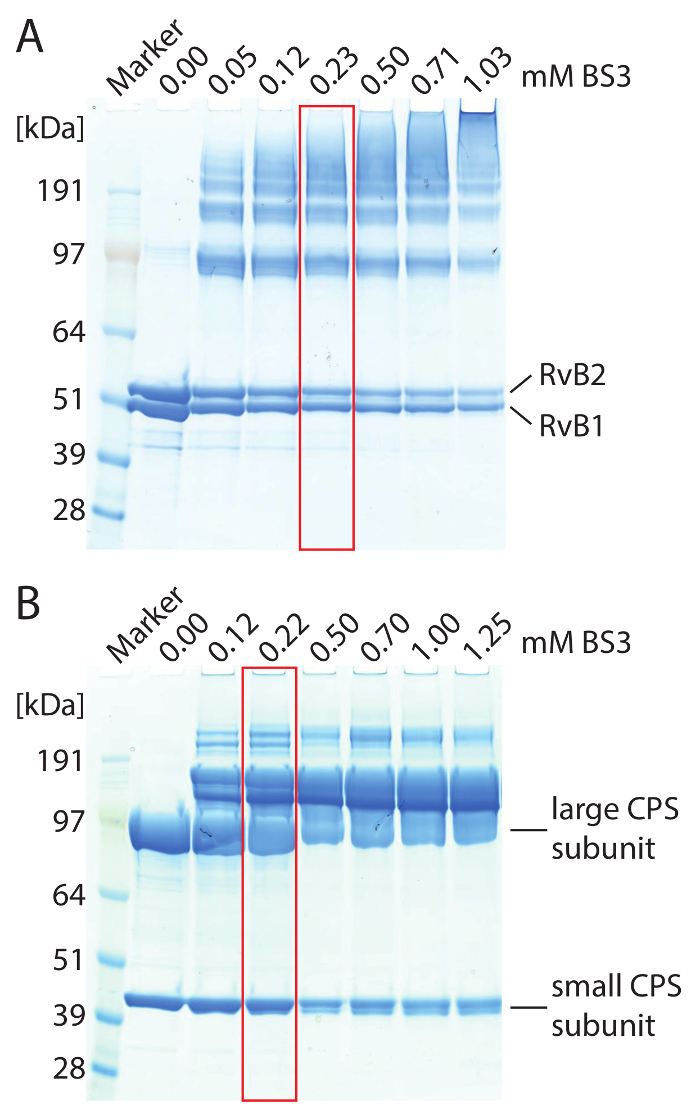
Figura 2: SDS-PAGE de reticulado RvB1/B2 (A) e CPS (B) complexos. (A) 2,5 µM RvB1/B2 foram carregados por gel de raia. A concentração de BS3 era variada. Non-Cruz-lig RvB1/B2 mostra as subunidades de dois proteína em cerca de 50 kDa. Adição de BS3 causou enlace covalente das subunidades da proteína resultando em bandas de proteína no peso molecular mais elevado. A quantidade de espécies reticulados é aumentada com altas concentrações de BS3. Condições óptimas do cross-linking são destaque (vermelho). (B) 10 µM CPS foram carregados por gel de raia. O grande (90 kDa) e pequeno (40 kDa) subunidades CPS são obtidas. Adição de BS3 causou enlace covalente das subunidades da proteína resultando em bandas de proteína no peso molecular mais elevado. Condições óptimas do cross-linking são destaque (vermelho). Clique aqui para ver uma versão maior desta figura.
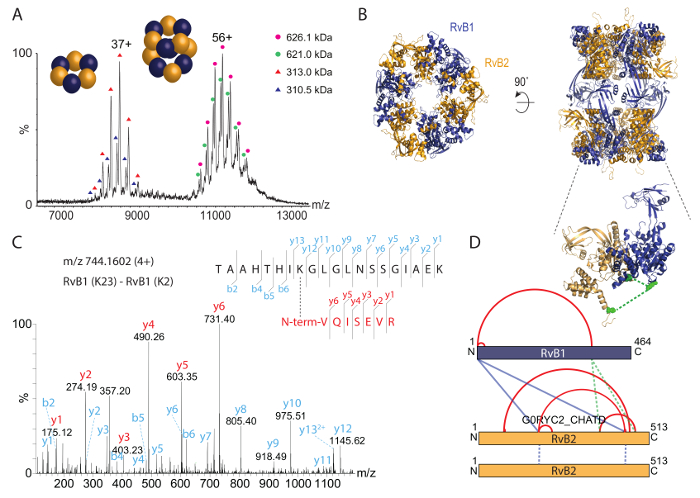
Figura 3: espectrometria de massa nativa e cross-linking química do complexo RvB1/B2. (A), o espectro de massa nativa revela duas espécies de RvB1/B2; o dodecamer intacto (ou seja, de6(RvB2) (RvB1)6) em aproximadamente 11.000 a 12.000 m/z e o hexamérica anel (RvB1)3(RvB2)3 em aproximadamente 8.000 m/z. Ambas as espécies mostram duas populações resultantes de RvB2 sua marcados e não marcados. O espectro foi modificado de35. (B) o cristal estrutura de RvB1/B2 é mostrada (PDB ID 4WVY). Alternância de RvB1 e RvB2 subunidades formam dois anéis hexamérica. (C) espectro de fragmentação de um reticulado di-peptídeo. O N-terminal da RvB1 foi reticulado com K23 de RvB1... série de y-íon foram obtido para ambos os peptídeos (vermelho e ciano). (D) Intra e inter protein interações obtidas no complexo RvB1/B2. Intra-cross-links são mostrados em vermelho, inter-cross-links são mostrados em azul. A inserção mostra duas ligações cruzadas inter moleculares entre subunidades RvB1 e RvB2, que podem ser visualizadas na estrutura de cristal (verde, inserção). Interações que se originam de duas cópias de RvB2 são mostradas como azuis linhas pontilhadas. Clique aqui para ver uma versão maior desta figura.
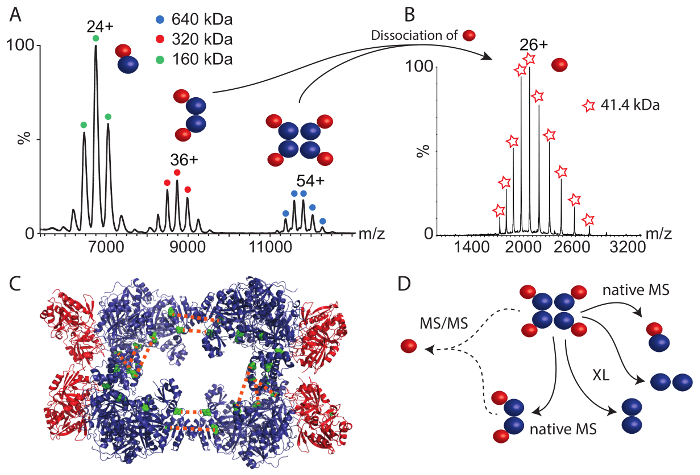
Figura 4: espectrometria de massa nativa e cross-linking química dos CPS. (A), o espectro de massa nativa do CPS mostra três complexos. O hetero-dímero (160 kDa), hétero-tetrâmero (320 kDa) e a hetero-octamer (640 kDa). O espectro foi modificado de35. (B) espectrometria de massa em Tandem da tetramérica e octameric CPS complexo revelou dissociação de pequena subunidade CPS. (C) o cristal estrutura do CPS é mostrada (PDB ID 1BXR). As grandes subunidades formam um núcleo tetramérica e as pequenas subunidades estão localizadas na periferia do complexo. Inter moleculares ligações cruzadas entre duas cópias de grande subunidade são mostradas (verdes). (D) interações das grandes e pequenas subunidades CPS. Espectrometria de massa nativa revelou subcomplexes e sugere uma localização periférica de subunidade pequena. Ligações químicas cruzadas indicam arranjos no núcleo tetramérica da CPS. Clique aqui para ver uma versão maior desta figura.
Tabela 1: resultados de pesquisa de banco de dados. As proteínas foram identificadas através da espectrometria de massa acoplada a cromatografia líquida e banco de dados. Os nomes de proteína, número e descrição, bem como a proteína de adesão em massa são dadas. A pontuação de proteína, número dos espectros observados por proteína e o número de sequências peptídicas observados estão listados. Os cinco peptídeos com maior pontuação de peptídeos de mascote estão listados para cada subunidade de proteína. Clique aqui para baixar este arquivo.
Tabela 2: ligações cruzadas observaram em RvB1/B2 e CPS. As subunidades dos complexos e os resíduos de reticulado são dadas. O tipo de ligação cruzada (intra ou inter molecular) revelou-se de sequências de peptídeo sobreposição ou um anterior estudo35. Clique aqui para baixar este arquivo.
Discussão
Protocolos são fornecidos para análise estrutural de espectrometria de massa base de complexos multi o subunit da proteína. As duas técnicas, descritas no protocolo, principalmente, entregar resultados complementares e são well-suited para obter insights sobre os arranjos estruturais dentro da proteína (-ligante) complexos que são difíceis de estudar por técnicas estruturais convencionais. Espectrometria de massa nativa oferece insights sobre stoichiometries de proteína, bem como interações da proteína através da análise de subcomplexes e módulos de interação estável. Cross-linking, por outro lado, produz informações sobre sites de contato diretos. Dependendo o cross-linker usado, uma certa flexibilidade pode ou deve ser incluída na análise.
Os protocolos fornecidos são em geral fácil de executar e não demorado. O protocolo inteiro pode ser executado dentro de uma semana e pode ser aplicado a quase todos os complexos da proteína, apesar de uma certa quantidade de proteína complexa é necessária para a análise bem sucedida. Preparação da amostra é simples e não requer especificamente os complexos de proteína purificada. No entanto, uma armadilha comum é a contaminação da amostra durante a preparação da amostra para identificação de espectrometria de massa-baseadas da proteína. Estas contaminações na maioria dos casos incluem a queratina que se originam de pó, pele ou cabelo. Portanto, extra tais como usando luvas e jalecos, filtração buffers de aquosas e usando solventes de alta pureza tenha cuidados durante a preparação da amostra para identificação de espectrometria de massa-baseadas da proteína. Outras proteínas contaminantes tais como acompanhantes geralmente são introduzidas durante a purificação de proteínas, por exemplo, quando usando marcas de afinidade. Nestes casos, purificação de proteínas deve ser melhorada, por exemplo, aumentando a etapas de lavagem. Em qualquer caso, as contaminações de proteína na amostra são facilmente identificadas durante a pesquisa de banco de dados, omitindo o filtro de taxonomia (i.e., procurando contra proteínas de todas as espécies). Se apenas alguns peptídeos são observados (ou seja, uma cobertura de baixa proteína poderia ser obtida), apesar de amostra suficiente estiver disponível, talvez seja necessário usar uma protease diferente durante a digestão. Em geral a tripsina produz um número suficiente de peptídeos; no entanto, em alguns casos, tais como proteínas de membrana ou membrana domínios de proteínas, é reduzir o número de sítios de clivagem tryptic e outras enzimas como alvo aminoácidos hidrofóbicos são uma escolha melhor.
Em termos de instrumentação, um instrumento particularmente modificado é necessário para espectrometria de massa nativa que mantém interações não covalentes durante a transferência para a fase gasosa. Vários tipos de instrumento foram introduzidos, incluindo instrumentos Q-ToF e orbitrap. Enquanto espectrómetros de massa Q-ToF modificados estão disponíveis comercialmente para espectrometria de massa nativa desde vários anos, este último só recentemente foram introduzido e na maioria dos casos requerem modificação especializados45. No entanto, a aplicação de instrumentos de alta resolução permitiu estudar a ligação de vários ligantes e sua quantificação46,47 e é promissor para aplicações futuras.
Para identificar di-peptídeos reticulados por espectrometria de massa acoplada a cromatografia líquida, podem ser aplicados os procedimentos padrão, com poucas modificações. No entanto, a pesquisa de banco de dados é o fator limitante como software especializado pode raramente lidam com grandes bases de dados e reduzidos bancos de dados contendo as subunidades de proteína dos complexos são necessários. Estudos recentes usados espectrometria de massa-cleavable cross-linkers visando interações da proteína em toda a célula lysates48,49. O uso de cross-linkers químicos que fragmentam em experimentos de espectrometria de massa em tandem principalmente produz peptídeos lineares (modificados pelo cross-linker), que podem ser identificados por mais fragmentação e banco de dados à procura de peptídeos lineares e isso reduz tempo de pesquisa e espaço de pesquisa computacional. No entanto, para realizar estas experiências, um analisador de massa ion trap ou um espectrômetro de massa híbrido com uma armadilha do íon é necessário. Em geral, como falsos positivos são uma questão importante, espectros de massa de reticulado peptídeos são frequentemente validados manualmente pela qualidade dos seus espectros de fragmento que amplia enormemente o tempo de análise de dados. Desenvolvimento de sistemas robustos de pontuação que podem ser aplicados sem mais etapas de validação são, portanto, possíveis aplicações futuras. Uma maneira de melhorar a análise de dados e reduzir o número de falsos positivos foi a introdução de cálculos da taxa falsa da descoberta e sua aplicação para cross-linking de conjuntos de dados50.
Em geral, as técnicas aqui descritas podem ser complementadas com ainda mais técnicas de espectrometria de massa (por exemplo, rotulagem covalente) para aumentar a saída da análise. Outras modificações e melhoramentos dos protocolos podem ser facilmente implementados. Como tal comparativo do cross-linking34 desvenda mudanças conformacionais na Assembleia de proteína. Novos desenvolvimentos em espectrometria de massa nativa hoje em dia permitem a análise de proteínas de membrana51,52 e suas interações com lipídios28,52,53,54 . Novos desenvolvimentos de espectrómetros de massa de alta resolução para espectrometria de massa nativa ter alargado o aplicativo e vinculação de ligante, por exemplo, vinculação de lipídios, proteínas de membrana, agora podem ser incluídos na análise45, 46. em combinação com abordagens de modelagem computacional, estas técnicas podem entregar modelos estruturais de diferentes resolução55. Se não há estruturas de cristal estão disponíveis para as subunidades de complexas ou única intactas, espectrometria de massa pode entregar primeiros insights sobre a topologia do complexo desconhecido e interações da proteína. Dependendo das técnicas utilizadas e os resultados obtidos, modelos de baixa resolução do complexo desconhecido podem ser obtidos56,57,58. Se homologia modelos ou estruturas de cristal estão disponíveis, as informações estruturais recebidas de espectrometria de massa podem render até quase nativo modelos59.
Em comparação com outras técnicas estruturais, espectrometria de massa tem a vantagem que requer quantidades de amostra baixa, ele pode lidar com amostras heterogêneas e é aplicável a complexos de proteínas de tamanho ilimitado. Além disso, a espectrometria de massa permite a investigação de sistemas dinâmicos da proteína. Populações diferentes da proteína ou proteína complexa que existem em solução geralmente são analisadas em conjunto e, portanto, ao contrário com outras técnicas estruturais que requerem a seleção de determinadas populações, todas as conformações são mantidas durante análise e são tributáveis em um experimento. Abordagens do cross-linking quantitativas foram recentemente introduzidas34,60,,61 e são promissoras para futuros aplicativos descrevendo mudanças conformacionais em condições diferentes.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Agradecemos nossos colegas para debates úteis. Também agradecemos Ilme Schlichting e Karl-Peter Hopfner fornecendo complexos da proteína. Reconhecemos que o financiamento do Ministério Federal da educação e pesquisa (BMBF, ZIK programa, 03Z22HN22), os fundos europeus de Desenvolvimento Regional (Pedro, ZS/2016/04/78115) e o MLU Halle-Wittenberg a C.S. e financiamento do Wellcome Trust (109854/658 Z/15/Z) de A.P.
Materiais
| Name | Company | Catalog Number | Comments |
| Chemicals, Reagents, Consumables | |||
| 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid (HEPES) | Sigma Aldrich | H4034 | buffer |
| Acetic acid | Sigma Aldrich | 695092 | pH |
| Acetonitrile Optima LC/MS | Fisher Chemicals | A955-1 | solvent |
| Amicon Ultra centrifugal devices (different MWCO) | Millipore | i.e. UFC500396 | buffer exchange, concentration |
| Ammonium acetate solution | Sigma Aldrich | A2706 | native MS |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | in-gel digestion |
| Ammonium solution | Sigma Aldrich | 9859 | pH |
| Bis(Sulfosuccinimidyl)suberate-d0 (BS3-d0) | Thermo Scientific | 21590 | cross-linker |
| Bis(Sulfosuccinimidyl)suberate-d4 (BS3-d4) | Thermo Scientific | 21595 | cross-linker |
| Caesium iodide | Sigma Aldrich | 203033 | calibration |
| Calcium chloride | Sigma Aldrich | C5670 | in-gel digestion |
| Capillaries (1.0 OD × 0.78 ID × 100 L mm) | Harvard Apparatus | 30-0038 | native MS |
| Disuccinimidyl suberate (DSS) | Thermo Scientific | 21655 | cross-linker |
| DL-Dithiothreitol | Sigma Aldrich | D5545 | in-gel digestion |
| DMSO (dimethyl sulfoxide) | Sigma Aldrich | D8418 | solvent |
| Ethanol | Fisher Chemicals | BP2818 | solvent |
| Formic acid Optima LC/MS | Fisher Chemicals | A117-50 | solvent supplement |
| Instant Blue Coomassie staining solution | expedeon | ISB1L | staining solution for gel electrophoresis |
| Invitrogen NuPAGE 4-12% Bis-Tris gels (1 mm, 10 well) | life technologies | NP0323BOX | gel electrophoresis |
| Iodoacetamide | Sigma Aldrich | I1149 | In-gel digestion |
| Isopropyl alcohol, HPLC grade | Fisher Chemicals | P750717 | solvent |
| Methanol | Fisher Chemicals | A456-212 | solvent |
| Micro BioSpin 6 columns | BioRad | 732-6222 | buffer exchange |
| NuPAGE Antioxidant | invitrogen | NP0005 | running buffer, gel electrophoresis |
| NuPAGE LDS Sample Buffer (4 ×) | invitrogen | NP0007 | sample loading buffer (non-reducing) for gel electrophoresis |
| NuPAGE MES SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE MOPS SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE Sample Reducing Agent (10 ×) | invitrogen | NP0004 | reducing Agent for gel electrophoresis |
| PBS - phosphate buffered saline | Sigma Aldrich | P4417 | buffer |
| SeeBlue Plus2 Prestained Standard Protein Marker | invitrogen | LC5925 | prestained Protein Marker for gel electrophoresis |
| Sodium acetate | Sigma Aldrich | S2889 | ethanol precipitation |
| Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 252859 | buffer |
| Trypsin | Sigma Aldrich | TRYPSEQ-RO (11418475001) | in-gel digestion |
| Vivaspin centrifugal devices (different MWCO) | Sartorius | i.e. VS0101 | buffer exchange, concentration |
| Water for HPLC | Sigma Aldrich | 270733-2.5L-M | solvent |
| Name | Company | Catalog Number | Comments |
| Instruments | |||
| Centrifuge Heraeus Fresco 21, bench-top centrifuge | Thermo Scientific | 75002425 | |
| Flaming / Brown Micropipette puller Model P-1000 | Sutter Instruments | P-1000 | |
| Gelelectrophoresis chamber Xcell SureLock MiniCell | Invitrogen | EI0001 | |
| Gold Coater Quorum Q150R S | Quorum Technologies Ltd. | Q150RS | |
| Horizontal gel shaker Rotamax 120 T | Heidolph Instruments | 544-41200-00 | |
| Q-TOF Ultima mass spectrometer, MS Vision high mass upgrade | Waters (Micromass) | - | |
| reversed-phase C18 analytical column Acclaim PepMap (C18, 75 mm I.D., 50 cm, 3 mm pore size) | Thermo Scientific | 164570 | |
| reversed-phase C18 pre-column (C18, 150 mm I.D., 2 cm, 5 mm pore size) | Thermo Scientific | 164213 | |
| scalpel | Fisher Scientifc | 10463989 | |
| SpeedVac SPD121P vacuum centrifuge | Thermo Scientific | SPD121DP-230 | |
| Thermomixer C | Eppendorf | 5382000015 | |
| Tweezers AA | Sigma Aldrich | Z680184-1EA | |
| Ultrasonic cleaner USC-TH, sonication bath | VWR | 142-0084 | |
| UltiMate Dionex 3000 nano-LC system, coupled to Q-Exactive plus hybrid mass spectrometer (nano-ESI source) | Thermo Scientific | 0726030+ | |
| Name | Company | Catalog Number | Comments |
| Software, Software Tools, Database search | |||
| Mascot | www.matrixscience.com | ||
| Massign | http://massign.chem.ox.ac.uk | ||
| MassLynx | Micromass | ||
| MassMatrix | Xu, H. J Proteome Res. 9 (2010) | ||
| MaxQuant | Cox, J. Nat. Biotechn. 26 (2008) | ||
| pLink | Yang, B. Nat Methods 9 (2012) | ||
| pXtract conversion tool | http://pfind.ict.ac.cn/downloads.html | ||
| UniDec | Marty, M.T. Anal. Chem. 87 (2015) | ||
| XCalibur | Thermo Scientific | ||
| XiNET | http://crosslinkviewer.org | ||
| XLinkX | Liu, F. Curr Op Struct Biol 35 (2015) | ||
| xQuest | Rinner, O. Nat Methods 5 (2008) | ||
| Xvis | https://xvis.genzentrum.lmu.de |
Referências
- Campbell, I. D. Timeline: the march of structural biology. Nat Rev Mol Cell Biol. 3 (5), 377-381 (2002).
- Liko, I., Allison, T. M., Hopper, J. T., Robinson, C. V. Mass spectrometry guided structural biology. Curr Opin Struct Biol. 40, 136-144 (2016).
- Robinson, C. V. From molecular chaperones to membrane motors: through the lens of a mass spectrometrist. Biochem Soc Trans. 45 (1), 251-260 (2017).
- Lossl, P., van de Waterbeemd, M., Heck, A. J. The diverse and expanding role of mass spectrometry in structural and molecular biology. EMBO J. 35 (24), 2634-2657 (2016).
- Wang, L., Chance, M. R. Protein Footprinting Comes of Age: Mass Spectrometry for Biophysical Structure Assessment. Mol Cell Proteomics. 16 (5), 706-716 (2017).
- Steen, H., Mann, M. The ABC's (and XYZ's) of peptide sequencing. Nat Rev Mol Cell Biol. 5 (9), 699-711 (2004).
- Olsen, J. V., Mann, M. Status of large-scale analysis of post-translational modifications by mass spectrometry. Mol Cell Proteomics. 12 (12), 3444-3452 (2013).
- Schmidt, C., Robinson, C. V. Dynamic protein ligand interactions--insights from MS. FEBS J. 281 (8), 1950-1964 (2014).
- Marcsisin, S. R., Engen, J. R. Hydrogen exchange mass spectrometry: what is it and what can it tell us?. Anal Bioanal Chem. 397 (3), 967-972 (2010).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Leitner, A., Lindner, W. Current chemical tagging strategies for proteome analysis by mass spectrometry. J Chromatogr B Analyt Technol Biomed Life Sci. 813 (1-2), 1-26 (2004).
- Leitner, A., Lindner, W. Chemistry meets proteomics: the use of chemical tagging reactions for MS-based proteomics. Proteomics. 6 (20), 5418-5434 (2006).
- Kiselar, J. G., Chance, M. R. Future directions of structural mass spectrometry using hydroxyl radical footprinting. J Mass Spectrom. 45 (12), 1373-1382 (2010).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chem Rev. 107 (8), 3514-3543 (2007).
- Liu, F., Heck, A. J. Interrogating the architecture of protein assemblies and protein interaction networks by cross-linking mass spectrometry. Curr Opin Struct Biol. 35, 100-108 (2015).
- Rappsilber, J. The beginning of a beautiful friendship: cross-linking/mass spectrometry and modelling of proteins and multi-protein complexes. J Struct Biol. 173 (3), 530-540 (2011).
- Rinner, O., et al. Identification of cross-linked peptides from large sequence databases. Nat Methods. 5 (4), 315-318 (2008).
- Yang, B., et al. Identification of cross-linked peptides from complex samples. Nat Methods. 9 (9), 904-906 (2012).
- Xu, H., Hsu, P. H., Zhang, L., Tsai, M. D., Freitas, M. A. Database search algorithm for identification of intact cross-links in proteins and peptides using tandem mass spectrometry. J Proteome Res. 9 (7), 3384-3393 (2010).
- Leitner, A., Faini, M., Stengel, F., Aebersold, R. Crosslinking and Mass Spectrometry: An Integrated Technology to Understand the Structure and Function of Molecular Machines. Trends Biochem Sci. 41 (1), 20-32 (2016).
- Heck, A. J. Native mass spectrometry: a bridge between interactomics and structural biology. Nat Methods. 5 (11), 927-933 (2008).
- Sharon, M., Robinson, C. V. The role of mass spectrometry in structure elucidation of dynamic protein complexes. Annu Rev Biochem. 76, 167-193 (2007).
- Goth, M., Pagel, K. Ion mobility-mass spectrometry as a tool to investigate protein-ligand interactions. Anal Bioanal Chem. , (2017).
- Uetrecht, C., Rose, R. J., van Duijn, E., Lorenzen, K., Heck, A. J. Ion mobility mass spectrometry of proteins and protein assemblies. Chem Soc Rev. 39 (5), 1633-1655 (2010).
- Hernandez, H., Robinson, C. V. Determining the stoichiometry and interactions of macromolecular assemblies from mass spectrometry. Nat Protoc. 2 (3), 715-726 (2007).
- Sobott, F., Hernandez, H., McCammon, M. G., Tito, M. A., Robinson, C. V. A tandem mass spectrometer for improved transmission and analysis of large macromolecular assemblies. Anal Chem. 74 (6), 1402-1407 (2002).
- Rostom, A. A., et al. Detection and selective dissociation of intact ribosomes in a mass spectrometer. Proc Natl Acad Sci U S A. 97 (10), 5185-5190 (2000).
- Zhou, M., et al. Mass spectrometry of intact V-type ATPases reveals bound lipids and the effects of nucleotide binding. Science. 334 (6054), 380-385 (2011).
- Uetrecht, C., et al. High-resolution mass spectrometry of viral assemblies: molecular composition and stability of dimorphic hepatitis B virus capsids. Proc Natl Acad Sci U S A. 105 (27), 9216-9220 (2008).
- Morgner, N., et al. Hsp70 forms antiparallel dimers stabilized by post-translational modifications to position clients for transfer to Hsp90. Cell Rep. 11 (5), 759-769 (2015).
- Schmidt, C., Beilsten-Edmands, V., Robinson, C. V. The joining of the Hsp90 and Hsp70 chaperone cycles yields transient interactions and stable intermediates: insights from mass spectrometry. Oncotarget. 6 (21), 18276-18281 (2015).
- Schmidt, C., Beilsten-Edmands, V., Mohammed, S., Robinson, C. V. Acetylation and phosphorylation control both local and global stability of the chloroplast F1 ATP synthase. Sci Rep. 7, 44068 (2017).
- Schmidt, C., et al. Comparative cross-linking and mass spectrometry of an intact F-type ATPase suggest a role for phosphorylation. Nat Commun. 4, 1985 (2013).
- Schmidt, C., Robinson, C. V. A comparative cross-linking strategy to probe conformational changes in protein complexes. Nat Protoc. 9 (9), 2224-2236 (2014).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Anal Chem. 89 (3), 1459-1468 (2017).
- Lakomek, K., Stoehr, G., Tosi, A., Schmailzl, M., Hopfner, K. P. Structural basis for dodecameric assembly states and conformational plasticity of the full-length AAA+ ATPases Rvb1 · Rvb2. Structure. 23 (3), 483-495 (2015).
- Mareya, S. M., Raushel, F. M. A molecular wedge for triggering the amidotransferase activity of carbamoyl phosphate synthetase. Biochemistry. 33 (10), 2945-2950 (1994).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Olsen, J. V., et al. Parts per million mass accuracy on an Orbitrap mass spectrometer via lock mass injection into a C-trap. Mol Cell Proteomics. 4 (12), 2010-2021 (2005).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Kirshenbaum, N., Michaelevski, I., Sharon, M. Analyzing large protein complexes by structural mass spectrometry. J Vis Exp. (40), (2010).
- Morgner, N., Robinson, C. V. Massign: an assignment strategy for maximizing information from the mass spectra of heterogeneous protein assemblies. Anal Chem. 84 (6), 2939-2948 (2012).
- Marty, M. T., et al. Bayesian deconvolution of mass and ion mobility spectra: from binary interactions to polydisperse ensembles. Anal Chem. 87 (8), 4370-4376 (2015).
- Thoden, J. B., Wesenberg, G., Raushel, F. M., Holden, H. M. Carbamoyl phosphate synthetase: closure of the B-domain as a result of nucleotide binding. Biochemistry. 38 (8), 2347-2357 (1999).
- Rose, R. J., Damoc, E., Denisov, E., Makarov, A., Heck, A. J. High-sensitivity Orbitrap mass analysis of intact macromolecular assemblies. Nat Methods. 9 (11), 1084-1086 (2012).
- Gault, J., et al. High-resolution mass spectrometry of small molecules bound to membrane proteins. Nat Methods. 13 (4), 333-336 (2016).
- Mehmood, S., et al. Mass spectrometry captures off-target drug binding and provides mechanistic insights into the human metalloprotease ZMPSTE24. Nat Chem. 8 (12), 1152-1158 (2016).
- Kaake, R. M., et al. A new in vivo cross-linking mass spectrometry platform to define protein-protein interactions in living cells. Mol Cell Proteomics. 13 (12), 3533-3543 (2014).
- Liu, F., Rijkers, D. T., Post, H., Heck, A. J. Proteome-wide profiling of protein assemblies by cross-linking mass spectrometry. Nat Methods. 12 (12), 1179-1184 (2015).
- Walzthoeni, T., et al. False discovery rate estimation for cross-linked peptides identified by mass spectrometry. Nat Methods. 9 (9), 901-903 (2012).
- Barrera, N. P., Di Bartolo, N., Booth, P. J., Robinson, C. V. Micelles protect membrane complexes from solution to vacuum. Science. 321 (5886), 243-246 (2008).
- Barrera, N. P., et al. Mass spectrometry of membrane transporters reveals subunit stoichiometry and interactions. Nat Methods. 6 (8), 585-587 (2009).
- Laganowsky, A., et al. Membrane proteins bind lipids selectively to modulate their structure and function. Nature. 510 (7503), 172-175 (2014).
- Marcoux, J., et al. Mass spectrometry reveals synergistic effects of nucleotides, lipids, and drugs binding to a multidrug resistance efflux pump. Proc Natl Acad Sci U S A. 110 (24), 9704-9709 (2013).
- Politis, A., Schmidt, C. Structural characterisation of medically relevant protein assemblies by integrating mass spectrometry with computational modelling. J Proteomics. , (2017).
- Hall, Z., Politis, A., Robinson, C. V. Structural modeling of heteromeric protein complexes from disassembly pathways and ion mobility-mass spectrometry. Structure. 20 (9), 1596-1609 (2012).
- Pukala, T. L., et al. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- Politis, A., et al. Topological models of heteromeric protein assemblies from mass spectrometry: application to the yeast eIF3:eIF5 complex. Chem Biol. 22 (1), 117-128 (2015).
- Politis, A., et al. A mass spectrometry-based hybrid method for structural modeling of protein complexes. Nat Methods. 11 (4), 403-406 (2014).
- Fischer, L., Chen, Z. A., Rappsilber, J. Quantitative cross-linking/mass spectrometry using isotope-labelled cross-linkers. J Proteomics. 88, 120-128 (2013).
- Yu, C., et al. Developing a Multiplexed Quantitative Cross-Linking Mass Spectrometry Platform for Comparative Structural Analysis of Protein Complexes. Anal Chem. 88 (20), 10301-10308 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados