Method Article
Сочетание химических Cross-linking и масс-спектрометрии комплексов интактных белков для изучения архитектуры мульти субъединицы белка сборок
В этой статье
Резюме
Архитектура белковых комплексов имеет важное значение для их функционирования. Сочетание различных масс-спектрометрических методов оказался мощным, чтобы изучить их сборки. Мы предоставляем протоколы для химической сшивки и родной масс-спектрометрии и показать, как эти дополнительные методы помогают уточнить архитектура мульти субъединицы белка сборок.
Аннотация
Белки взаимодействуют с их лигандами формы активного и динамичного сборкам, которые осуществляют различные клеточные функции. Изучение этих взаимодействий Поэтому основополагающее значение для понимания клеточных процессов. Однако многие комплексы протеина являются динамические сборки и не доступны структурными аналогами. Масс-спектрометрия способствует структурной расследование этих Ассамблей, и особенно сочетание различных масс-спектрометрических методов обеспечивает ценную информацию в их структурные договоренности.
В этой статье мы описывают приложение и сочетание двух дополнительных масс-спектрометрических методов, а именно химической сшивки в сочетании с масс-спектрометрии и родной масс-спектрометрии. Химической сшивки включает ковалентная связь аминокислот в непосредственной близости с помощью химических реагентов. После переваривания с протеаз сшитого ди пептиды идентифицируются по масс-спектрометрии и белковых взаимодействий сайты открыты. Родной масс-спектрометрии с другой стороны является анализ сборок интактных белков в газовой фазе масс-спектрометр. Он показывает stoichiometries белка, а также взаимодействия белков и лиганда. Обе методики поэтому доставить дополнительную информацию о структуре белка лиганд сборок и их сочетание, оказался мощным в предыдущих исследованиях.
Введение
Структурные исследования белка сборок стало особенно важное значение для понимания клеточных процессов. Следовательно многие методы разработаны и улучшены в структурной биологии1. Однако эти методы иногда ограничены в их применение из-за размера, гибкости или неоднородность белковых комплексов под следствием. Масс-спектрометрия может справиться с этими проблемами и, таким образом, стала мощным инструментом в структурной биологии2,3,4,5. Самое главное преимущество масс-спектрометрии, однако, является возможность однозначно идентифицировать белки даже в сложной и неоднородной смеси6. С этой целью обычно белки усваиваются с endoproteinases и полученную смесь пептидов разделенных жидкостной хроматографии и непосредственно этого eluted в масс-спектрометр. Пептид массы впоследствии определяются и прекурсоров ионы выбираются для дальнейшей фрагментации пептидов. Белки, затем обозначаются поиска пептида и соответствующий фрагмент масс против известных базы данных. Эта процедура позволяет не только идентификация пептидов/белков, но также их столб-поступательные изменения, которые вызывают массовые сдвиг пептиды прекурсоров и фрагмент ионов, перевозящих модификации7. Некоторые из многих методов в структурных масс-спектрометрии основаны на4,этот принцип8. Например обозначая методы, такие как водорода дейтерия обмен9,10, химические маркировки стратегии11,12, или Гидроксильная группа ногой печати13,14 , дать понимание поверхности доступность белков при определенных условиях.
Другой метод (химической) сшивки с участием ковалентная связь аминокислот в непосредственной близости от через их функциональных групп. Для этого химические реактивы, УФ активируемых реагентов или аминокислоты являются занятых15,16. После сшивки, белки обычно гидролизуется с протеаз и сшитого ди пептиды анализируются жидкостной хроматографии в сочетании масс-спектрометрии. Выявление сшивки продуктов в базе данных поиска, однако, требует использования специализированного программного обеспечения, которое сцепить пептид последовательности различных белков и различных регионах17,18,19. Использование химических сшивок имеет то преимущество, что он может быть использован для почти каждый комплекс протеина интереса и не требует включения УФ активируемых аминокислот, которые может быть достигнуто только в том случае, когда выражение протеина интереса в клетки хозяина. Таким образом сшивки является универсальным инструментом и успешно применяется во многих структурных исследованиях даже большой белок сборки20.
Масс-спектрометрии комплексов интактных белков (иногда называется «родной» масс-спектрометрия), с другой стороны, включает в себя анализ интакт белками и белковых комплексов без гидролиза в пептиды. Это показывает, композиция, неоднородность, стехиометрии, топология и Субблок взаимодействия белковых комплексов21,22. В сочетании с ионной подвижности родной масс-спектрометрии далее позволяет определять их конформацию23,24. Это делает его мощным инструментом для структурной расследования белковых комплексов, которые трудно оценить структурными аналогами. Однако родной масс-спектрометрии требует анализа буферов, которые поддерживают non ковалентные белковых взаимодействий во время электроспрей ионизации. Обычно это достигается с помощью водный, летучие буферов например аммония ацетат25. Кроме того инструмент изменения необходимы для предотвращения диссоциации во время передачи в газовой фазе масс-спектрометр26. Применяется в таким образом, были проанализированы многие комплексы (большой) белок. Впечатляющие примеры являются исследования нетронутыми рибосомы27, АТФ synthases28или29вирусов.
Сочетание родной масс-спектрометрии и cross-linking оказался особенно успешным в предыдущих исследованиях. К примеру, stoichiometries сопровождающий комплексов, включая неожиданные Hsp70 димер, могут быть получены из экспериментов масс-спектрометрии комплексов интактных белков, химической сшивки выявлены механизмы белков в сборки,3031. В различные исследования были изучены последствия столб-поступательные изменения на нетронутыми АТФ-синтазы комплекса. Родной масс-спектрометрии предусмотрено понимание сложных стабильность белков в наличие или отсутствие фосфорилирование или ацетилирования32,33. Сравнительные сшивки стратегии34 затем показали конформационные изменения белкового комплекса в различных условиях.
Здесь мы предоставляем протоколы для идентификации белков, масс-спектрометрия, сшивки (химические) и родной масс-спектрометрии, включая анализ данных и интерпретации (рис. 1). Сочетание взаимодополняющих результатов, полученные из этих методов, с помощью двух хорошо изученных белковых комплексов является продемонстрировали35. Наш протокол может применяться любой Ассамблее белков, которые могут быть очищены в определенные чистоты и концентрации. Подход в некоторых случаях ограничивается анализа данных сшивки данных, т.е., размер базы данных используется, которая определяет требуемый поиск пространства и времени. Кроме того ручной проверки выявленных перекрестных ссылок часто требуется и далее уменьшает выход. Родной масс-спектрометрии ограничивается главным образом образец качества, например, буферов и аддукты используется во время очищения и возможность обменять их на водной и летучих буферов. Однако белковых комплексов, очищенный для структурного анализа обычно имеют качества, необходимые для успешного анализа с нашими протоколы.
протокол
1. Очистка белковых комплексов
- Подготовьте комплекс согласно оптимизированные стандартные протоколы белков.
Примечание: Протокол свидетельствует здесь с RvB1/B2 комплекс от Chaetomium Тенелюбы и carbamoyl фосфат синтазы (CPS) от кишечной палочки. Как описано36,37были очищены RvB1/B2 и CPS. Каждый комплекс белков требует индивидуальной очистки протокол. Соответствующим образом настройте протокол. Амин свободных буферов как фосфат амортизированное saline (PBS) или 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic кислоты (HEPES) должен использоваться для химической сшивки. Если возможно замените буфера во время очистки.
Предупреждение: Не применять любые методы или реагенты, которые мешают родной Ассамблеи комплекс белков.
2. Масса на основе спектрометрии белков идентификации
-
Электрофорез геля
Примечание: Существует различные гель систем и каждая лаборатория использует свои собственные настройки. Настройка условий согласно системе геля. Надевайте перчатки и лаборатории пальто всей протокола как кератины среди наиболее распространенных загрязняющих веществ в масс-спектрометрических анализа.- Применить буфер образца 4 x 7,5 мкл и восстанавливающего агента в мкл 10 x 3 (конечная концентрация 50 мм Дитиотреитол (DTT)) к 20 мкл пример белка. Использование 10 мкм CPS или 2,5 мкм RvB1/B2. Уменьшается на 1 мин на 16200 × g и тепла в течение 10 минут при 70 ° C.
Примечание: Когда с помощью различных гелевая отрегулировать необходимых объемов и температуры нагрева. - Подготовка 4-12% градиента гель для электрофореза. Разбавить идущий буфер 20 раз с водой и заполнить камеры электрофореза. Использование 2-(N-Морфолино) ethanesulfonic кислота (MES) буфер для белков меньший молекулярный вес (2-200 кДа) и 3 - propanesulfonic кислоты (N-Морфолино) (МОПЫ) буфер для крупных белков (14-200 кДа). Добавьте 0,5 мл антиоксидант внутри камеры.
Примечание: Когда с использованием различных гель системы подготовить идущий буфер и гель по данным производителя протоколы. - Загрузить подходящий белка маркера (предварительно окрашенные, безупречная, различных молекулярных размеров) в полости первой и загрузить образцы протеина в оставшиеся пустоты.
- Отдельный белков для 35 мин (MES) или 50 мин (МОПЫ) на 200 V.
Примечание: Отрегулируйте время электрофореза и напряжения для различных гель систем. - Пятно белка полосы, передача гель в гель окраски окно и накрыть гель на водной основе Кумасси, окрашивание раствора. Инкубируйте на ночь и при комнатной температуре на горизонтальной гель шейкер.
- Отмойте гель, заменив окрашивание раствора с водой. Повторите это действие несколько раз (примерно 3 - 5 раз) до тех пор, пока гель фон появляется ясно.
- Применить буфер образца 4 x 7,5 мкл и восстанавливающего агента в мкл 10 x 3 (конечная концентрация 50 мм Дитиотреитол (DTT)) к 20 мкл пример белка. Использование 10 мкм CPS или 2,5 мкм RvB1/B2. Уменьшается на 1 мин на 16200 × g и тепла в течение 10 минут при 70 ° C.
-
В гель пищеварения
Примечание: Чтобы избежать загрязнения, используйте растворители класса ВЭЖХ в протоколе пищеварения и все следующие шаги (то есть, масс-спектрометрических анализ). Подготовка всех решений до использования и фильтрат воды и бикарбонат аммония.- Вырезать полосы белков, которые визуализируются Кумасси синий пятно от геля с помощью скальпеля. Аккуратно вырежьте белка полосы на мелкие кусочки из примерно 1 мм × 1 мм. Промойте скальпеля с водой между диапазонами различных белков. Промойте гель полос с водой и ацетонитриле (АКС).
- Уменьшить Скрепления дисульфида с DTT, алкилата остатков цистеина с iodoacetamide и дайджест белки с трипсином как описано38; обычно используется фермент: белка соотношение 1:20 до 1: 100. Используйте бикарбонат аммония 100 мм в протоколе пищеварения.
- Экстракт пептидов в два этапа.
- Сначала Проинкубируйте гель куски бикарбонат аммония и АКС и собирать пептид содержащих супернатант. Во-вторых Проинкубируйте гель штук с муравьиной кислоты 5% (v/v) и АКС. Инкубируйте 15 мин на каждом шагу. Объединить оба supernatants. Сухие извлеченные пептиды путем выпаривания растворителей в вакуумной центрифуги.
Примечание: Сушеные пептиды могут храниться при температуре-20 ° C в течение нескольких месяцев.
- Сначала Проинкубируйте гель куски бикарбонат аммония и АКС и собирать пептид содержащих супернатант. Во-вторых Проинкубируйте гель штук с муравьиной кислоты 5% (v/v) и АКС. Инкубируйте 15 мин на каждом шагу. Объединить оба supernatants. Сухие извлеченные пептиды путем выпаривания растворителей в вакуумной центрифуги.
-
Жидкостной хроматографии в сочетании масс-спектрометрии
- Растворите сушеные пептидов в 2% ACN/0.1% муравьиной кислоты. Растворяются пептидов в sonication баня для 2-3 мин и спин вниз в центрифуге на 16200 x g для 30 min. передачи образцов в автоматический пробоотборник флаконов.
Примечание: Отрегулируйте громкость по количеству протеина. Для хорошо окрашенные Кумасси полос мы используем 20 мкл. - Придать 5 мкл образца к системе нано LC-MS/MS, используя Автоматический пробоотборник. Загрузки пептидной смесь на Обратная фаза с18 до столбец (C18, 150 мкм и.д., 2 см, размер пор 5 мкм) для опреснения и сконцентрировать пептиды онлайн.
- Используйте Муравьиная кислота 0,1% (v/v), как подвижная фаза A и ACN/0.1% (v/v) 80% муравьиной кислоты как мобильный фазы B. отдельные пептиды обратная фаза C18 аналитической столбца (C18, 75 мкм и.д., 50 см, размер пор 3 мкм) с использованием градиента 4-80% B (содержащие 0,1% муравьиной кислоты) в 300 nL / мин свыше 65 мин.
Примечание: Источник ионов наноспрей используется для передачи eluted пептидов в масс-спектрометр. - (Типичный) MS условия использования: спрей напряжение 1,6 кв; капиллярные температуры 250 ° c; нормализованное столкновений энергия 30. Действуют масс-спектрометр в зависящих от данных режиме.
- Приобрести МС спектры в масс-анализатор (например, Орбитрэп) (m/z 350−1, 600) с разрешением 70000 и автоматическая регулировка усиления управления цели 3 x 10-6. Выберите 20 самых интенсивных ионов для фрагментации HCD автоматическая регулировка усиления управления цель 1 х 105. Динамически исключение ранее выбранных ионов для 30 s. исключить поодиночке предъявлено непризнанных заряд ионов, а также ионы.
Примечание: Внутренняя калибровка массового спектров выполнялось с помощью блокировки массового вариант39.
- Растворите сушеные пептидов в 2% ACN/0.1% муравьиной кислоты. Растворяются пептидов в sonication баня для 2-3 мин и спин вниз в центрифуге на 16200 x g для 30 min. передачи образцов в автоматический пробоотборник флаконов.
-
База данных поиска
Примечание: Существует различное программное обеспечение для базы данных поиска. MaxQuant40, например, свободно доступны.- Конвертировать .raw файлы в .mgf файлы с помощью инструмента преобразование pXtract (http://pfind.ict.ac.cn/downloads.html).
- Выполните поиск в базе данных, используя параметры типичных поиска: база данных, swissprot; пептид массового терпимости, 10 ppm; фрагмент массового терпимости, 0.5 Да; фермент, трипсина; пропущенные расщепления сайтов, 2; переменной модификации, carbamidomethylation (цистеин) и окисления (метионин).
Примечание: Измените параметры поиска по данным экспериментальных настройки. - Проверьте результат поиска базы данных. Оценивать белка оценка, количество выявленных пептиды, пептид баллы и массовых точности; охват последовательности возвращает белка охвата выявленных.
Примечание: Каждая поисковая система имеет свой индивидуальный скоринга алгоритм. Оцените пептида балльной системе по качеству тандем массового спектры наблюдается. Основные сигналы хорошего спектра должен показать серии Ион (полная) для проверки выявленных пептид. Обычно Оценка пептид, основанной на вероятности, т.е., пептид Оценка представляет собой меру как вероятно выявленных пептид последовательность соответствует полученной спектра. Оценка белка обычно является производным от пептида баллы и используется для ранга белка в список выявленных белков.
3. масс-спектрометрии комплексов интактных белков (родной масс-спектрометрия)
- Подготовка золото покрытием излучателей для ионизации электроспрей
- Используйте съемник микропипеткой подготовить nanoflow излучателей от стеклянных капилляров, как описано ранее25,41. Использование боросиликатное капилляров с внутренним диаметром 0,78 мм.
Примечание: Путем изменения параметров для потянув иглу, оконечности форму и размер можно изменить и скорректировать для образца. Доступны с различными внутренний диаметр и толщина стенки капилляров. - Слой стекла капилляров с проводящего материала (например, золото или палладий); Использование распыления нанесения покрытий, генерации золото плазмы является общим. Следуйте инструкциям производителя, чтобы получить хорошее качество покрытия.
Примечание: Покрытие должно быть достаточным, чтобы получить стабильный спрей при применении общих капиллярного напряжения (см. ниже).
- Используйте съемник микропипеткой подготовить nanoflow излучателей от стеклянных капилляров, как описано ранее25,41. Использование боросиликатное капилляров с внутренним диаметром 0,78 мм.
- Пробоподготовка для родной масс-спектрометрия
Примечание: Соли, моющие средства или большим количеством глицерина не совместимы с ионизацией электроспрей. Таким образом очистка буфера обменялись летучих, водный буфера. Ацетат аммония 200 мм обычно используется. Использовать размер исключения спин столбцы или ультрафильтрации устройства для буфера обмена. В некоторых случаях комплекс стабильности или деятельности могут быть затронуты буфера обмена. Тщательно оценить массы спектры и проверить деятельность комплекса. При необходимости добавьте добавок или кофакторов анализа буфер.- Используйте размер исключения спин столбцы для быстрого буфера обмена. Удалить в буфер хранения центрифугированием на 1000 x g и 4 ° C в течение 1 мин отбросить через поток. Вымойте три раза путем добавления 500 мкл 200 мм аммония ацетат, следуют центрифугирования. Загрузите 20 мкл образец протеина на столбец и центрифуги 1000 x g и 4 ° C на 4 мин.
Примечание: Концентрация белкового комплекса должна быть 1-10 мкм. Процедуру можно повторять, если энергонезависимой компоненты по-прежнему мешают анализа. - Концентрат белка образца и обмениваться буфер в том же эксперимента использовать центробежные фильтры. Используйте Фильтрующая мембрана поры размер 50% меньше, чем размер белков проанализирована.
- Передача образец протеина в фильтрации устройства и добавить 200 мм аммония ацетат. Уменьшается на 15 000 x g и отбросить через поток. Добавить 200 мм аммония ацетат и повторите центрифугирования. Повторите это действие несколько раз. Следуйте инструкциям производителя для центрифугирования скорости. Выполните центрифугированием при 4 ° C.
Осторожностью: Мембранные белки, как правило, осадок на мембране фильтра устройства.
- Передача образец протеина в фильтрации устройства и добавить 200 мм аммония ацетат. Уменьшается на 15 000 x g и отбросить через поток. Добавить 200 мм аммония ацетат и повторите центрифугирования. Повторите это действие несколько раз. Следуйте инструкциям производителя для центрифугирования скорости. Выполните центрифугированием при 4 ° C.
- Используйте размер исключения спин столбцы для быстрого буфера обмена. Удалить в буфер хранения центрифугированием на 1000 x g и 4 ° C в течение 1 мин отбросить через поток. Вымойте три раза путем добавления 500 мкл 200 мм аммония ацетат, следуют центрифугирования. Загрузите 20 мкл образец протеина на столбец и центрифуги 1000 x g и 4 ° C на 4 мин.
- Масс-спектрометрических анализ нетронутыми белковых комплексов
Примечание: Существуют различные типы от разных производителей, которые могут быть изменены для родной масс-спектрометрии, например, (Q-ToF) время полета квадрупольного масс-спектрометров или Орбитрэп масс-спектрометров масс-спектрометров. Протокол, в описанный ниже была исполнена на Q-ToF инструменте.- Золото покрытием капилляров в капиллярной держателя и заполнения капилляра с 1-4 мкл образец протеина. Откройте кончик иглы с пинцет.
- Подключиться капиллярная держатель с нано электроспрей источником и отрегулировать положение капилляров. Расположите наконечник капилляр на 0,5-1,5 см на конус отверстия. Используйте 80-150 Л/ч nanoflow газ инициировать спрей и отрегулировать поток газа для поддержания стабильной спрей.
- Отрегулируйте параметры на странице Настройка Q-ToF инструмента. Типичный стартовые условия являются: капиллярный напряжения, 1,50 кв; конус напряжения, 80 V; Объектив 1 РЧ, 80 V; энергию столкновения, 20 V; Диафрагма 3, 13,6 V. изменить эти параметры, чтобы получить хорошее массы спектры. Начать приобретение, нажав кнопку «приобрести» и объединить столько сканирует как можно получить хороший массовых спектр.
Примечание: Мы рекомендуем совместить по меньшей мере 100 сканирований.
- Тандемные масс-спектрометрии комплексов интактных белков
- Приобрести массовых спектр, как описано выше (протокол раздел 3.3). Выберите прекурсоров Ион белкового комплекса.
- Изменить MS в режим МС/МС в файле приобретения. Настроить выбор МС/МС предвестником массы.
- Начните приобретение на энергию низкой столкновения. Объединить несколько сканов (примерно 20 сканов) чтобы проверить выбор правильного прекурсоров массы. Увеличение энергии столкновения до тех пор, пока разъединяет комплекс белков. Для получения хорошей массовых спектр объединить по крайней мере 500 сканирования.
Примечание: Раздели комплексов иногда имеют низкой интенсивности. Сочетая столько сканирует как можно может увеличить соотношение сигнал шум и резолюции.
- В решение диссоциации нетронутыми белковых комплексов
Примечание: Для получения дополнительных понимания взаимодействия протеина внутри белковых комплексов, может выполняться в решение диссоциации.- Подготовьте образец протеина, как описано выше (раздел 3.2). Добавить растворители образец протеина или изменить рН не присоединяться нетронутыми комплексов в суб комплексов. Типичные растворители являются метанол, изопропиловый спирт и АКС; Измените рН Ацетат аммония, добавлением уксусной кислоты или раствора аммиака. Приобрести массового спектры, как описано выше (разделы 3.3 и 3.4).
- Изменять количество растворителя или рН для создания различных суб комплексов. Как правило количество растворителя составляет 5-50% (конечная концентрация) и типичный рН-4-9. Начать с низким содержанием растворителей (5%) или незначительные изменения в pH и приобрести массового спектра (см. раздел 3.3).
- Увеличить количество растворителя или изменить рН ступенчатой пока суб комплексы создаются в растворе. Приобрести массового спектры для анализа суб комплексов.
- Калибровки данных
Примечание: Приобретенных массового спектры калибруются внешне с использованием раствора йодистого (CsI) цезия.- Растворите 100 мг CsI в 1 мл воды.
- Приобрести массовые спектр CsI. Различаются энергию столкновения получить CsI кластеров в том же диапазоне m/z как комплекс белков проанализированы выше.
Предупреждение: CsI быстро осаждается на кончике излучатель и загрязняет конуса. Приобретать только столько сканирует как необходимых для получения достаточной спектра. Удалите из источника после завершения излучатель. - Сделайте файл калибровки, используя приобретенные массовых спектр и CsI справочного файла.
- Применить калибровки для приобретенных массового спектров.
Предупреждение: Калибровка массового спектров может быть постоянного изменения исходных данных. Если необходимы некалиброванных спектры, сделайте резервную копию файла.
- Обработка и анализ данных
Примечание: Существует много инструментов свободно доступного программного обеспечения для анализа данных о родной спектры массы; Например, Massign42 или UniDec43. Протокол ниже описывает анализ данных вручную с помощью инструмента программного обеспечения, а также использование Massign для сложных образцов. Это программное обеспечение хорошо подходит для анализа сложных массового спектров. Следуйте инструкциям онлайн для использования программы (http://massign.chem.ox.ac.uk/).- Для анализа данных гладкие спектры, регулируя параметры сглаживания. Центроид спектры, регулируя параметры. Расчет сложных массы из двух смежных вершин комплекс белков ' пик конверт с помощью инструмента программного обеспечения.
Предупреждение: Слишком интенсивный сглаживания может привести к потере данных (например, потеря связанных лигандов). - Для анализа с Massign42генерировать список пик для массовых спектр. Линеаризации данных точек спектра и гладкой. Используйте различные программные средства для назначения белковых комплексов, вычисления сложного состава или имитировать сложный пик конверты.
- Для анализа данных гладкие спектры, регулируя параметры сглаживания. Центроид спектры, регулируя параметры. Расчет сложных массы из двух смежных вершин комплекс белков ' пик конверт с помощью инструмента программного обеспечения.
4. Химическая, структурообразования зарядовой связью с масс-спектрометрии
Примечание: Есть многочисленные сшивки стратегии. Здесь мы описывают использование бис (sulfosuccinimidyl) suberate (BS3), Амин реактивной крест-компоновщик, который обычно используется для изучения взаимодействия протеин протеина.
- Растворите 1,43 мг BS3 в 100 мкл воды подготовить раствор 25 мм.
Примечание: Другие реагентов как disuccinimidyl suberate (DSS) не растворимы и обычно растворены в диметилсульфоксида (ДМСО). Некоторые сшивок также доступны в тяжелых формах гетерогенны помечены. Включение тяжелых стабильных изотопов генерирует пары пик сшитого ди-пептидов в МС спектры, которая помогает во время оценки данных. При использовании дифференциально помечены сшивок, запасов решения обоих вариантов готовятся и смешанные 1:1.
Предупреждение: Чтобы избежать диссоциации белковых комплексов, ДМСО, высококонцентрированный раствор готовится и разбавленным с водой или буфера до сшивки реакции. - Добавьте BS3 комплекс белков. Использовать различное количество BS3 начиная от 0.5-5 мм для определения оптимального крест-компоновщик концентрации. Инкубируйте реакции смесей при 25 ° C в течение 1 ч в thermomixer. Использование 4-12% градиента гели и выполняют SDS-PAGE для оценки сшивки результаты (см. Рисунок 2 пример).
Примечание: Оптимальная концентрация BS3 достигается, когда выше молекулярный вес белка полосы, которые не видны в элементе управления, связанный с Креста, полученные во время SDS-PAGE пока единого подразделения по-прежнему видны (рис. 2). - Повторите сшивки реакции с оптимальной концентрации BS3. Инкубируйте реакции смесей при 25 ° C в течение 1 ч в thermomixer.
Примечание: Некоторые белковых комплексов не стабильны при комнатной температуре. Сшивки реакции могут также выполняться на льду; Однако время реакции необходимо скорректировать. -
Утолить реакция структурообразования, добавляя Амин буфера (например, 50-100 мм трис-буфер, pH 7.5, конечная концентрация) или выполнить этанола осадков для удаления любых остаточных сшивки реагента.
- Добавить воду или буфера в реакционную смесь для достижения окончательный объем 200 мкл. добавить 600 мкл ледяной этанола и 20 мкл 3 M ацетат натрия, рН 5.3. Тщательно перемешать и инкубировать и при-20 ° C для 2 h или на ночь.
Примечание: в качестве альтернативы, белки можно осаждают при 80 ° C за 30 мин или в жидком азоте. - Спин вниз на 16200 x g и 4 ° C на 30 мин тщательно удалить супернатант.
- Помойте лепешка с 1 мл ледяной 80% (v/v) этанола. Спин вниз на 16200 x g и 4 ° C на 30 мин тщательно удалить супернатант. Сухие гранулы в вакуумной центрифуги.
- Добавить воду или буфера в реакционную смесь для достижения окончательный объем 200 мкл. добавить 600 мкл ледяной этанола и 20 мкл 3 M ацетат натрия, рН 5.3. Тщательно перемешать и инкубировать и при-20 ° C для 2 h или на ночь.
- Выполните SDS-PAGE сшитого белков. Вырезать полосы гель и переваривать белок в гель, как описано выше (разделы 2.1 и 2.2).
Примечание: Пищеварение в решение также может быть выполнена, однако, это обычно требует дополнительных разделения шаги (например, размер гель-проникающей хроматографии переваривается пептидов). -
Выполните жидкостной хроматографии в сочетании масс-спектрометрии, как описано выше (раздел 2.3). Как сшитого пептидов, как правило низкие изобилии, применяются следующие изменения к масс-спектрометрических анализа для увеличения аналитической глубины сшитого образца.
- Используйте градиенты дольше во время разделения жидкостной хроматографии (например, 90 мин вместо 65 мин, см. выше).
- Исключите вдвойне заряженных пептиды из HCD фрагментации.
Примечание: Вдвойне заряженных пептиды, обычно внутри-кросс-связаны пептидов («петля пептиды» или «тип 1» перекрестные ссылки). - При использовании дифференциально помечены сшивки реагентов, используйте параметр «пик комплектации» во время анализа, т.е., HCD фрагментации вызвана наличием пик пар определенных массовой разницей в массовых спектры.
Примечание: «Сбор пик» вариант не может быть доступна на каждом масс-спектрометр.
- Использование палить18 программное обеспечение для идентификации сшитого ди-пептидов. Используйте свернутого баз данных для идентификации. Типичный поиска параметры являются: инструмент спектры, HCD; фермент, трипсина; Макс. пропущенные расщепления сайтов, 3; переменной модификации, окисления (метионин) и carbamidomethylation (цистеин); Кросс-линкера, BS3; Минимальная длина пептид, 4; Макс. Длина пептида, 100; min. пептид массы, 400 да; Макс. пептид массы, 10000 да; РУЗВЕЛЬТ, 1%.
Примечание: При использовании дифференциально помечены сшивок, увеличение массы, вызванные компоновщик будет необходимо настроить. Есть также другие часто используемые программного обеспечения для идентификации перекрестных ссылок, например, xQuest17, MassMatrix19или XlinkX15. - Оцените результаты поиска базы данных по качеству фрагментации спектров. Приемлемыми спектров перекрестные ссылки следует показать серии Ион обоих пептидов (по крайней мере 4 соседних ионов) на разумное соотношение сигнал шум.
Примечание: Когда с использованием дифференциально помечены сшивки реагентов, пик пар в МС спектры может использоваться как дополнительный контроль качества. - При необходимости Визуализируйте сшивки результаты в протеин взаимодействия сетей с помощью программных средств (например, XVis, XiNET). Использование бар участков или круговой участков для визуализации белковых взаимодействий.
Примечание: Оба программные средства свободно доступны на веб-сервере. Подробные инструкции на соответствующих веб-сайтах (https://xvis.genzentrum.lmu.de/ и http://crosslinkviewer.org/).
Результаты
Структурный анализ белков и комплексы, которые они формируют имеет основополагающее значение для понимания их функции. Масс-спектрометрия значительно способствует структурной расследование в том, что он может быть применен к почти каждый комплекс интересов независимо от размера или отведайте неоднородности. Мы иллюстрируют протокол с помощью двух хорошо изученных белковых комплексов; Во-первых, гетеро dodecamer RvB1/B2 от C. Тенелюбы и во-вторых, CPS гетеро octameric комплекс от кишечной палочки.
Во-первых мы определили белковых компонентов двух комплексов. Для этого белки были отделены от SDS-PAGE (рис. 2) и гель полосы были отрезаны от геля. После-гель пищеварения белков пептид смесь был проанализирован жидкостной хроматографии в сочетании масс-спектрометрии и пептида и фрагмент масс были подвергнуты базы данных поиска. После этого рабочий процесс, мы определили всех субблоков протеина двух комплексов с высокой степенью уверенности, т.е., большое количество пептиды с разумной пептид оценки было отмечено, приносит высокий последовательности покрытие для всех субблоков протеина (Таблица 1 ).
Затем мы проанализировали нетронутыми комплекс RvB1/B2 по родной масс-спектрометрии (рис. 3A). Массовые спектр показал два вида, один на примерно 8000 m/z и другой вид на примерно 11000-12000 m/z. Вычисляемые масс для этих видов соответствуют кольцо3 3(RvB2) hexameric (RvB1) (около 310 кДа) и dodecameric двойной кольцо (RvB1)6(RvB2)6 (примерно 620 кДа). Обе серии пик показать две популяции; они происходят из смеси его с тегами и без тегов подразделения RvB2 в комплексах. Кристаллическая структура для RvB1/B2 комплекс ранее было получено36 и показывает расположение двойной-кольца (рис. 3B). Родной массовых спектр поэтому подтверждает стехиометрии нетронутыми dodecamer и Кроме того показывает стабильные подкомплекса. Кроме того определены совместно существующих популяций.
Для идентификации белков взаимодействия сайтов в комплексе RvB1/B2, мы химически сшитого очищенный комплекс с BS3 крест-компоновщик. Мы сначала титруют количество BS3 во время реакции структурообразования для определения оптимальной концентрации. BS3 — Амин конкретных и ковалентно ссылки лизин боковых цепей, а также N-Термини белков. Сшивки реакция последовала SDS-PAGE (рис. 2A). Кросс несвязанном комплекс показали RvB1 и RvB2 подразделения. Добавление реакционной смеси BS3 вызвал ковалентная связь белков, привело полосы белка на более высокой молекулярной массой. Геля SDS показывает, что все большее количество BS3 доходность выше количества видов сшитого хотя кросс несвязанном белковых субъединиц сокращаются. Затем режем белка полосы от геля и затем протокол, представленным выше для выявления сайтов взаимодействия протеина. Пример спектр сшитого ди пептид показано (рис. 3 c). Спектр показывает серия y Ион обоих пептидов, подтверждающие этот протеин взаимодействия. В общей сложности, мы получили 14 белковых взаимодействий, включая четыре перекрестные ссылки между подразделениями, RvB1 и RvB2 и двух поперечных связей между двумя копиями RvB2 (Таблица 2). Результаты от BS3 cross-linking визуализируются в сети взаимодействия (рис. 3D) показаны внутри молекулярных взаимодействий, а также взаимодействия между различными подразделениями. Перекрестные ссылки внутри молекулярная предполагают, что RvB1 и RvB2 субблоков раз в способ, что N - и C-терминал домены находятся в непосредственной близости. Обратите внимание, что внутри молекулярных взаимодействий нельзя отличить от между молекулярных взаимодействий же подразделения в этом случае. Также наблюдались между молекулярной перекрестные ссылки между двумя подразделениями. Из них два между молекулярной перекрестные ссылки между RvB1 и RvB2 могут быть визуализированы в структуре проверка сшивки подход. Другие между молекулярной перекрестные ссылки расположены в гибкой петли, которые не включены в кристаллической структуре. Мы также определили две перекрестные ссылки в RvB2 содержащий же последовательности пептид. Эти перекрестные ссылки, однозначно могут быть классифицированы как между молекулярной, как они должны происходить из двух копий же белка (рис. 3D). Наши сшивки эксперименты показывают протеина взаимодействия сайтов в пределах комплекса, но и внутри белковых субъединиц, обеспечивая понимание их структурный механизм, который также могут быть подтверждены существующей структуры кристалла (рис. 3B).
Второй комплекс белков, который мы изучали был CPS. Родной массовых спектр (рис. 4A) выявили три белковых комплексов между 6000 до 12 000 m/z. Крупнейший комплекс 640 кДа соответствует нетронутыми гетеро octamer. Меньшие комплексы представляют собой две суб комплексов; Димер малых и больших CPS субблоков (160 кДа) и гетеро Тетрамер, содержащий две копии каждого субъединицы (320 кДа). Эти вложенные комплексы доставить первое понимание белка Ассамблеи; то есть, больших и малых субъединиц находятся в непосредственном контакте (как показал гетеро димер) и Тетрамер может быть продуктом двух димеры. Чтобы получить более подробную информацию о структурного механизма в нетронутыми CPS комплекса, мы провели тандемные масс-спектрометрия (МС/МС) гетеро octamer и гетеро Тетрамер. В обоих случаях, небольшой субъединицы в отрыве от прекурсоров, предполагая, что небольшой Субблок расположен в периферии Ассамблеи (Рисунок 4B). Действительно небольшие подразделения периферийных в доступные кристалл структуру (рис. 4 c)44.
Также была исполнена химической сшивки с помощью Кросс-линкера BS3. Использование увеличения сумм, ковалентная связь CPS подразделений был расширен. После переваривания белков и анализа пептидов, как описано выше многие взаимодействий протеина внутри большой субъединицы и один перекрестных ссылок в небольшой подгруппы были получены (Таблица 2). Кроме того аналогичные сложные RvB1/B2, мы нашли два между молекулярной перекрестные ссылки между двумя копиями большой субъединицы CPS. Эти перекрестные ссылки место два крупных подразделений, лицом друг к другу на их сторонах C-терминала. В предыдущем исследовании сочетая структурных масс-спектрометрии и численное моделирование35, мы определили три дополнительные взаимодействия в большой субблок, которые скорее возникают из интерфейса двух копий большой субблок подтверждены Кристаллическая структура и полученная модель (Рисунок 4 c и Таблица 2). Эти взаимодействия позволяют Расположение CPS основной комплекс, состоящий из четырех крупных подразделений. Однако не межучрежденческой подгруппы перекрестные ссылки между малых и больших подразделений были замечены. Изучив имеющиеся кристаллическую структуру (рис. 4 c), становится очевидным, что поверхности взаимодействия между tetrameric ядро комплекса, состоящий из большой субъединицы и периферийных мелких подразделений очень мала, которые могли бы объяснить отсутствие взаимодействия между субъединицы. Это подтверждается родной масс-спектрометрии, который показал, что небольшой Субблок легко отделяет от нетронутыми комплексы, скорее всего, из-за небольшой привязки интерфейса. Тем не менее взаимодействий протеина в CPS комплекса в сочетании с химической сшивки и родной масс-спектрометрия позволяют, выводя их структурного механизма (рис. 4 d).
Взятые вместе, сочетание родной масс-спектрометрии и химической сшивки в сочетании с масс-спектрометрических идентификации сшитого пептидов, позволяет воссоздания механизма структурных комплексов как пример. В то время как химической сшивки показали расположение субблоков протеина, например взаимодействия между RvB1 и RvB2 или внутри tetrameric ядро CPS, родной масс-спектрометрии доставлены белка stoichiometries нетронутыми комплексов и распространены Субкомплексы. В случае CPS, для которого можно наблюдать не между молекулярных взаимодействий между двумя подразделениями по химической сшивки, родной масс-спектрометрии свидетельствует о том, что каждый большой субблок взаимодействует с одной небольшой субъединицы (рис. 4 d). Тандемные масс-спектрометрии предложил периферийное положение малых субъединицы в комплекс и небольшой интерфейс между обоих подразделений.
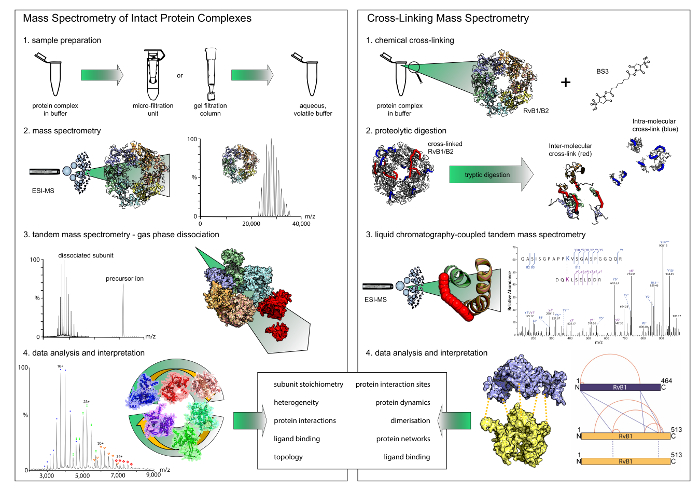
Рисунок 1: процесс родной масс-спектрометрии и cross-linking. Обе методики доставить дополнительные результаты. Хотя родной масс-спектрометрии показывает, stoichiometries и взаимодействия модулей, сшивки дает понимание сайтов взаимодействия протеина внутри комплексы. Обратите внимание, что только химической сшивки показывает двоичные взаимодействий. (A) первый шаг в родной масс-спектрометрия является буфером обмена летучих и водный буфер с помощью фильтра единиц или гель фильтрации столбцов. Масс-спектрометрии комплексов интактных белков затем показывает их стехиометрии. В экспериментах тандемные масс-спектрометрии отделить периферийных подразделений. (B) для химической сшивки, комплекс белков инкубировали с cross-linking реагента. Затем сшитого белки перевариваются в пептидов, которые затем анализируются жидкостной хроматографии в сочетании масс-спектрометрии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
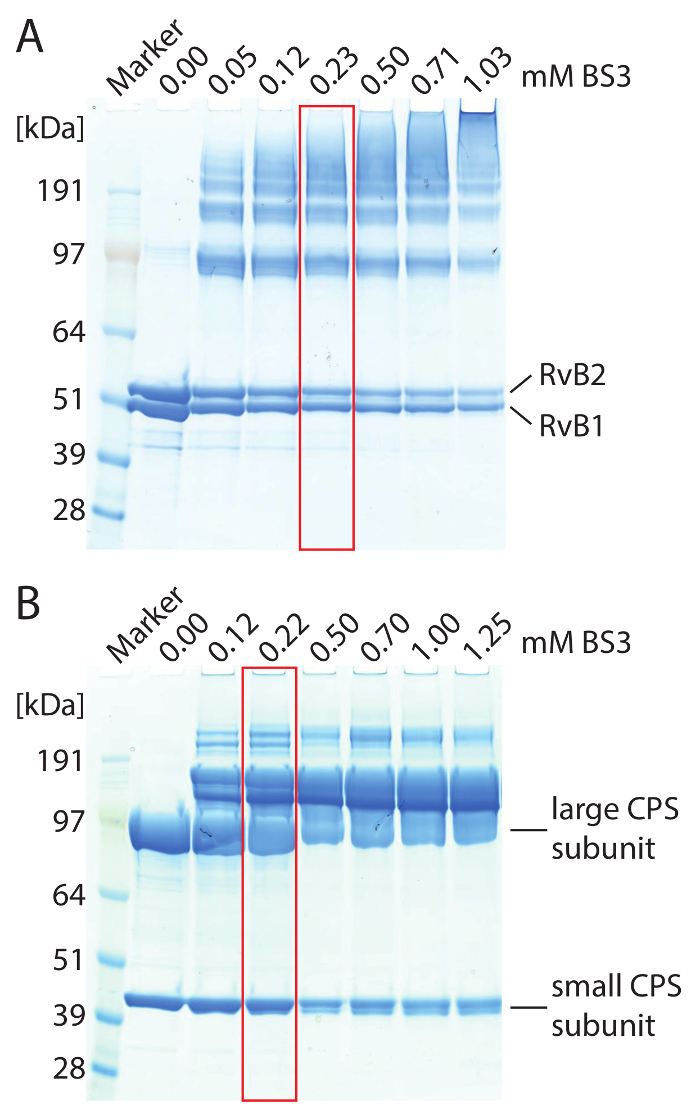
Рисунок 2: SDS-PAGE из сшитого RvB1/B2 (A) и CPS (B) комплексы. (A) 2,5 мкм RvB1/B2 были загружены на гель Лейн. Концентрация BS3 была различной. Non кросс связаны RvB1/B2 показывает двух белковых субъединиц приблизительно 50 kDa. Добавление BS3 вызвал ковалентная связь субблоков протеина, привело полосы белка на более высокой молекулярной массой. С более высокими концентрациями BS3 увеличивается количество сшитых видов. Оптимальные условия сшивки, выделенный (красный). (B) 10 мкм CPS были загружены на гель Лейн. Большой (90 кДа) и малых (40 кДа) получаются CPS субъединиц. Добавление BS3 вызвал ковалентная связь субблоков протеина, привело полосы белка на более высокой молекулярной массой. Оптимальные условия сшивки, выделенный (красный). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
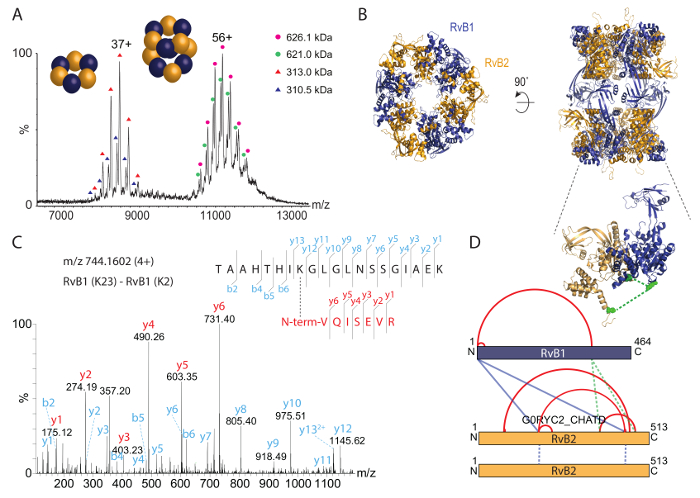
Рисунок 3: родной масс-спектрометрии и химической сшивки RvB1/B2 комплекс. (A) родной массовых спектр показывает два вида RvB1/B2; нетронутыми dodecamer (т.е.,6(RvB2) (RvB1)6) на примерно 11 000 до 12 000 m/z и hexameric кольцо (RvB1)3(RvB2)3 в приблизительно 8000 m/z. Оба вида показывают две популяции, вытекающие из его с тегами и без тегов RvB2. Спектра был изменен с35. (B) кристалл структура RvB1/B2 показана (PDB ID 4WVY). Переменный RvB1 и RvB2 подразделения образуют два кольца hexameric. (C) фрагментация спектр сшитого ди пептид. N-го RvB1 был Пенополиолефины с K23 RvB1. y Ион серии были получены для обоих пептидов (красный и голубой). (D) внутри и между protein взаимодействия полученные в комплексе RvB1/B2. Внутри-кросс-ссылки указаны в красном, inter-cross-links отображаются синим цветом. Вставки показывает два между молекулярной перекрестные ссылки между RvB1 и RvB2 подразделений, которые могут быть визуализированы в кристаллической структуре (зеленый, вставить). Взаимодействия, которые происходят из двух копий RvB2 отображаются как синие пунктирные линии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
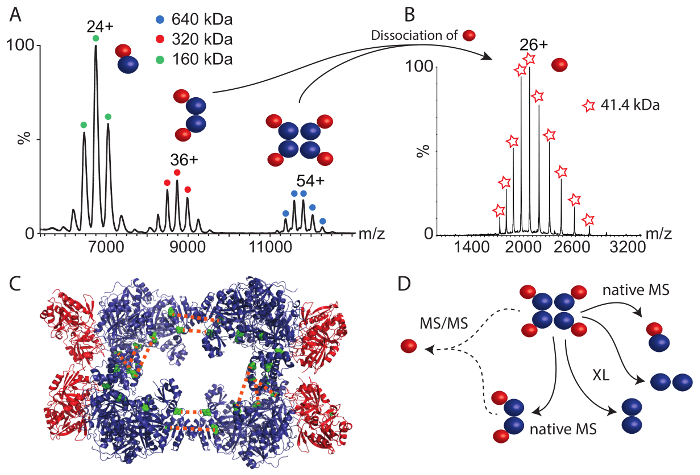
Рисунок 4: родной масс-спектрометрии и химической сшивки CPS. (A) родной массовых спектр CPS показывает три комплексы. Гетеро димер (160 кДа), гетеро Тетрамер (320 кДа) и гетеро octamer (640 кДа). Спектра был изменен с35. (B) тандем масс-спектрометрии, tetrameric и octameric CPS комплекс показал диссоциации небольшой субъединицы CPS. (C) кристалл структура CPS показана (PDB ID 1BXR). Крупные подразделения образуют tetrameric ядро и небольшие подразделения расположены на периферии комплекса. Между молекулярной перекрестные ссылки между двумя копиями большой субъединицы показываются (зеленый). (D) взаимодействия подразделений CPS больших и малых. Родной масс-спектрометрии показали Субкомплексы и предлагает периферийное положение малых субъединицы. Химические перекрестные ссылки указывают меры в tetrameric ядре CPS. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Таблица 1: результаты поиска в базе данных. Белки были определены жидкостной хроматографии в сочетании масс-спектрометрии и базы данных поиска. Имена белка, присоединение номер и описание, а также белка приводятся массовой. Оценка белка, количество наблюдаемых спектров на белок и количество наблюдаемых пептид последовательностей перечислены. Для каждого субъединицы белка перечислены пять пептиды с наивысшими баллами пептиды талисман. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Таблица 2: перекрестные ссылки наблюдается в RvB1/B2 и CPS. Субблоков комплексы и сшитого остатков даны. Типа перекрестных ссылок (внутри - или между молекулярной) было выявлено из перекрывающихся пептид последовательностей или предыдущие исследования35. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Протоколы предназначены для массы на основе спектрометрии структурного анализа многолетних Субблок белковых комплексов. Двух методов, описанных в протоколе, главным образом доставить дополнительные результаты и хорошо подходит, чтобы получить понимание структурных договоренностей в рамках белка (-лиганд) комплексов, которые трудно изучать, структурными аналогами. Родной масс-спектрометрии обеспечивает понимание stoichiometries белка, а также взаимодействия протеина путем анализа Субкомплексы и стабильного взаимодействия модулей. Сшивки, с другой стороны, дает информацию о прямых контактов. В зависимости от крест-компоновщик используется определенную гибкость могут или должны быть включены в анализ.
Предоставленные протоколы являются в целом легко выполнять и не отнимает много времени. Весь протокол может быть выполнен в течение одной недели и может быть применен к почти всех белковых комплексов, хотя, для успешного анализа требуется определенное количество комплекс белков. Подготовка образца проста и не требует специально очищенный протеин комплексов. Однако одной типичной ошибкой является загрязнение образца во время подготовки проб для определения массы на основе спектрометрии белков. Эти загрязнений в большинстве случаев включают кератины, происходящих от пыли, кожи или волос. Поэтому осторожность как носить перчатки и лаборатории пальто, фильтрационные водный буферов и с использованием высокой чистоты растворителей следует принять в ходе подготовки проб для определения массы на основе спектрометрии белков. Другие загрязняющие белков, таких как сопровождающим обычно вводятся во время очистки белков, например, при использовании сходства Теги. В этих случаях следует улучшить очищение протеина, например путем увеличения Стиральная шаги. В любом случае, белковых загрязнений в образце легко определяются в ходе поиска по базе данных, минуя фильтр таксономии (то есть, Поиск против белков от всех видов). Если только несколько пептиды наблюдаются (т.е., низкое содержание белка покрытия могут быть получены), хотя достаточно образец доступен, он может быть необходимо использовать различные протеиназ во время пищеварения. В целом трипсина дает достаточное количество пептиды; Однако в некоторых случаях, таких как мембранных белков или домены мембранных белков, уменьшается количество сайтов tryptic расщепления и другие ферменты, ориентация гидрофобные аминокислоты являются лучшим выбором.
С точки зрения инструментария особенно измененный документ необходим для родной масс-спектрометрии, которая поддерживает non ковалентные взаимодействий во время передачи в газовой фазе. Были введены несколько типов инструментов, включая инструменты Q-ToF и Орбитрэп. В то время как изменение Q-ToF масс-спектрометров коммерчески доступны для родной масс-спектрометрии с нескольких лет, последний только недавно появились и в большинстве случаев требуют специализированных модификации45. Однако применение инструментов с высоким разрешением позволило изучению связывания нескольких лигандов и46,их количественная оценка47 и является перспективным для будущих приложений.
Чтобы определить сшитого ди пептиды, жидкостной хроматографии в сочетании масс-спектрометрии, могут применяться стандартные процедуры с несколькими изменениями. Однако поиск по базе данных является ограничивающим фактором, специализированное программное обеспечение редко могут иметь дело с большими базами данных, а также сокращение базы данных, содержащие субблоков протеина комплексов требуются. Недавние исследования использовали Массовое спектрометрирование горные сшивок ориентации взаимодействий протеина в всю ячейку лизатов48,49. Использование химических сшивок, которые фрагмента в тандеме масс-спектрометрии экспериментов основном дает линейной пептиды (изменена крест-компоновщик), которые могут быть идентифицированы по дальнейшей фрагментации и базы данных поиска линейной пептидов, и это уменьшает время поиска и вычислительной поиск пространства. Однако чтобы выполнить эти эксперименты, требуется масс-анализатор для ионной ловушки или гибридный масс-спектрометр с ионной ловушки. В общем как ложных срабатываний являются важным вопросом, массовые спектры сшитого пептиды часто проверяются вручную по качеству их спектры фрагмент который простирается время анализа данных чрезвычайно. Разработка надежных скоринговых систем, которые могут быть применены без дальнейших шагов проверки являются поэтому потенциальных будущих приложений. Одним из способов для улучшения анализа данных и уменьшить количество ложных срабатываний было введение ложных обнаружения скорость расчетов и их применение для сшивки50наборов данных.
В общем, описанные здесь методы могут быть дополнены далее масс-спектрометрии методов (например, ковалентных маркировки) увеличить вывод из анализа. Другие изменения и улучшения протоколов могут быть легко реализованы. Как таковой сравнительных сшивки34 распутывает конформационные изменения в Ассамблее белка. Дальнейшие события в родной масс-спектрометрии в настоящее время позволяют анализ мембранных белков51,52 и их взаимодействия с липидами28,52,,5354 . Новые разработки с высоким разрешением масс-спектрометров для родной масс-спектрометрии расширили приложение и Связывание лиганда, например, Связывание липидов в мембранных белков, теперь могут быть включены в анализ45, 46. в сочетании с подходами, численное моделирование, эти методы могут доставить структурной модели различной резолюции55. Если не кристаллические структуры доступны для нетронутыми сложных или единого подразделения, масс-спектрометрия может доставить первое понимание взаимодействия протеина и топология неизвестного комплекса. В зависимости от используемых методов и полученных результатов с низким разрешением модели неизвестного комплекса могут быть получены56,,5758. Если доступны кристаллические структуры или модели гомологии, структурной информации, полученной от масс-спектрометрии может принести даже почти родной модели59.
По сравнению с другими структурные методы, масс-спектрометрия имеет то преимущество, что она требует низкой выборки сумм, он может справиться с разнородными образцами и применима к белковых комплексов неограниченного размера. Кроме того масс-спектрометрии позволяет исследование систем динамических белка. Различных популяциях белка или комплекс белков, которые существуют в растворе обычно анализируются вместе и поэтому, в отличие от с другими структурными методами, которые требуют выбора определенных групп населения, все конформации поддерживаются во время анализ и являются оценимый в одном эксперименте. Недавно появившаяся34,,6061 и количественных сшивки подходы являются многообещающими для будущих приложений, описывающих конформационные изменения в различных условиях.
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Мы благодарим наших коллег за полезные дискуссии. Мы также благодарим Ilme Шлихтинг и Карл-Петер Hopfner за предоставление белковых комплексов. Мы признаем, что финансирование от федерального министерства для образования и исследования (BMBF, Зик программы, 03Z22HN22), европейские региональные фонды развития (EFRE, ZS/2016/04/78115) и MLU Галле-Виттенберг C.S. и финансирование от Уэллком траст (109854/658 Z/15/Z) чтобы а.п.
Материалы
| Name | Company | Catalog Number | Comments |
| Chemicals, Reagents, Consumables | |||
| 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid (HEPES) | Sigma Aldrich | H4034 | buffer |
| Acetic acid | Sigma Aldrich | 695092 | pH |
| Acetonitrile Optima LC/MS | Fisher Chemicals | A955-1 | solvent |
| Amicon Ultra centrifugal devices (different MWCO) | Millipore | i.e. UFC500396 | buffer exchange, concentration |
| Ammonium acetate solution | Sigma Aldrich | A2706 | native MS |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | in-gel digestion |
| Ammonium solution | Sigma Aldrich | 9859 | pH |
| Bis(Sulfosuccinimidyl)suberate-d0 (BS3-d0) | Thermo Scientific | 21590 | cross-linker |
| Bis(Sulfosuccinimidyl)suberate-d4 (BS3-d4) | Thermo Scientific | 21595 | cross-linker |
| Caesium iodide | Sigma Aldrich | 203033 | calibration |
| Calcium chloride | Sigma Aldrich | C5670 | in-gel digestion |
| Capillaries (1.0 OD × 0.78 ID × 100 L mm) | Harvard Apparatus | 30-0038 | native MS |
| Disuccinimidyl suberate (DSS) | Thermo Scientific | 21655 | cross-linker |
| DL-Dithiothreitol | Sigma Aldrich | D5545 | in-gel digestion |
| DMSO (dimethyl sulfoxide) | Sigma Aldrich | D8418 | solvent |
| Ethanol | Fisher Chemicals | BP2818 | solvent |
| Formic acid Optima LC/MS | Fisher Chemicals | A117-50 | solvent supplement |
| Instant Blue Coomassie staining solution | expedeon | ISB1L | staining solution for gel electrophoresis |
| Invitrogen NuPAGE 4-12% Bis-Tris gels (1 mm, 10 well) | life technologies | NP0323BOX | gel electrophoresis |
| Iodoacetamide | Sigma Aldrich | I1149 | In-gel digestion |
| Isopropyl alcohol, HPLC grade | Fisher Chemicals | P750717 | solvent |
| Methanol | Fisher Chemicals | A456-212 | solvent |
| Micro BioSpin 6 columns | BioRad | 732-6222 | buffer exchange |
| NuPAGE Antioxidant | invitrogen | NP0005 | running buffer, gel electrophoresis |
| NuPAGE LDS Sample Buffer (4 ×) | invitrogen | NP0007 | sample loading buffer (non-reducing) for gel electrophoresis |
| NuPAGE MES SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE MOPS SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE Sample Reducing Agent (10 ×) | invitrogen | NP0004 | reducing Agent for gel electrophoresis |
| PBS - phosphate buffered saline | Sigma Aldrich | P4417 | buffer |
| SeeBlue Plus2 Prestained Standard Protein Marker | invitrogen | LC5925 | prestained Protein Marker for gel electrophoresis |
| Sodium acetate | Sigma Aldrich | S2889 | ethanol precipitation |
| Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 252859 | buffer |
| Trypsin | Sigma Aldrich | TRYPSEQ-RO (11418475001) | in-gel digestion |
| Vivaspin centrifugal devices (different MWCO) | Sartorius | i.e. VS0101 | buffer exchange, concentration |
| Water for HPLC | Sigma Aldrich | 270733-2.5L-M | solvent |
| Name | Company | Catalog Number | Comments |
| Instruments | |||
| Centrifuge Heraeus Fresco 21, bench-top centrifuge | Thermo Scientific | 75002425 | |
| Flaming / Brown Micropipette puller Model P-1000 | Sutter Instruments | P-1000 | |
| Gelelectrophoresis chamber Xcell SureLock MiniCell | Invitrogen | EI0001 | |
| Gold Coater Quorum Q150R S | Quorum Technologies Ltd. | Q150RS | |
| Horizontal gel shaker Rotamax 120 T | Heidolph Instruments | 544-41200-00 | |
| Q-TOF Ultima mass spectrometer, MS Vision high mass upgrade | Waters (Micromass) | - | |
| reversed-phase C18 analytical column Acclaim PepMap (C18, 75 mm I.D., 50 cm, 3 mm pore size) | Thermo Scientific | 164570 | |
| reversed-phase C18 pre-column (C18, 150 mm I.D., 2 cm, 5 mm pore size) | Thermo Scientific | 164213 | |
| scalpel | Fisher Scientifc | 10463989 | |
| SpeedVac SPD121P vacuum centrifuge | Thermo Scientific | SPD121DP-230 | |
| Thermomixer C | Eppendorf | 5382000015 | |
| Tweezers AA | Sigma Aldrich | Z680184-1EA | |
| Ultrasonic cleaner USC-TH, sonication bath | VWR | 142-0084 | |
| UltiMate Dionex 3000 nano-LC system, coupled to Q-Exactive plus hybrid mass spectrometer (nano-ESI source) | Thermo Scientific | 0726030+ | |
| Name | Company | Catalog Number | Comments |
| Software, Software Tools, Database search | |||
| Mascot | www.matrixscience.com | ||
| Massign | http://massign.chem.ox.ac.uk | ||
| MassLynx | Micromass | ||
| MassMatrix | Xu, H. J Proteome Res. 9 (2010) | ||
| MaxQuant | Cox, J. Nat. Biotechn. 26 (2008) | ||
| pLink | Yang, B. Nat Methods 9 (2012) | ||
| pXtract conversion tool | http://pfind.ict.ac.cn/downloads.html | ||
| UniDec | Marty, M.T. Anal. Chem. 87 (2015) | ||
| XCalibur | Thermo Scientific | ||
| XiNET | http://crosslinkviewer.org | ||
| XLinkX | Liu, F. Curr Op Struct Biol 35 (2015) | ||
| xQuest | Rinner, O. Nat Methods 5 (2008) | ||
| Xvis | https://xvis.genzentrum.lmu.de |
Ссылки
- Campbell, I. D. Timeline: the march of structural biology. Nat Rev Mol Cell Biol. 3 (5), 377-381 (2002).
- Liko, I., Allison, T. M., Hopper, J. T., Robinson, C. V. Mass spectrometry guided structural biology. Curr Opin Struct Biol. 40, 136-144 (2016).
- Robinson, C. V. From molecular chaperones to membrane motors: through the lens of a mass spectrometrist. Biochem Soc Trans. 45 (1), 251-260 (2017).
- Lossl, P., van de Waterbeemd, M., Heck, A. J. The diverse and expanding role of mass spectrometry in structural and molecular biology. EMBO J. 35 (24), 2634-2657 (2016).
- Wang, L., Chance, M. R. Protein Footprinting Comes of Age: Mass Spectrometry for Biophysical Structure Assessment. Mol Cell Proteomics. 16 (5), 706-716 (2017).
- Steen, H., Mann, M. The ABC's (and XYZ's) of peptide sequencing. Nat Rev Mol Cell Biol. 5 (9), 699-711 (2004).
- Olsen, J. V., Mann, M. Status of large-scale analysis of post-translational modifications by mass spectrometry. Mol Cell Proteomics. 12 (12), 3444-3452 (2013).
- Schmidt, C., Robinson, C. V. Dynamic protein ligand interactions--insights from MS. FEBS J. 281 (8), 1950-1964 (2014).
- Marcsisin, S. R., Engen, J. R. Hydrogen exchange mass spectrometry: what is it and what can it tell us?. Anal Bioanal Chem. 397 (3), 967-972 (2010).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Leitner, A., Lindner, W. Current chemical tagging strategies for proteome analysis by mass spectrometry. J Chromatogr B Analyt Technol Biomed Life Sci. 813 (1-2), 1-26 (2004).
- Leitner, A., Lindner, W. Chemistry meets proteomics: the use of chemical tagging reactions for MS-based proteomics. Proteomics. 6 (20), 5418-5434 (2006).
- Kiselar, J. G., Chance, M. R. Future directions of structural mass spectrometry using hydroxyl radical footprinting. J Mass Spectrom. 45 (12), 1373-1382 (2010).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chem Rev. 107 (8), 3514-3543 (2007).
- Liu, F., Heck, A. J. Interrogating the architecture of protein assemblies and protein interaction networks by cross-linking mass spectrometry. Curr Opin Struct Biol. 35, 100-108 (2015).
- Rappsilber, J. The beginning of a beautiful friendship: cross-linking/mass spectrometry and modelling of proteins and multi-protein complexes. J Struct Biol. 173 (3), 530-540 (2011).
- Rinner, O., et al. Identification of cross-linked peptides from large sequence databases. Nat Methods. 5 (4), 315-318 (2008).
- Yang, B., et al. Identification of cross-linked peptides from complex samples. Nat Methods. 9 (9), 904-906 (2012).
- Xu, H., Hsu, P. H., Zhang, L., Tsai, M. D., Freitas, M. A. Database search algorithm for identification of intact cross-links in proteins and peptides using tandem mass spectrometry. J Proteome Res. 9 (7), 3384-3393 (2010).
- Leitner, A., Faini, M., Stengel, F., Aebersold, R. Crosslinking and Mass Spectrometry: An Integrated Technology to Understand the Structure and Function of Molecular Machines. Trends Biochem Sci. 41 (1), 20-32 (2016).
- Heck, A. J. Native mass spectrometry: a bridge between interactomics and structural biology. Nat Methods. 5 (11), 927-933 (2008).
- Sharon, M., Robinson, C. V. The role of mass spectrometry in structure elucidation of dynamic protein complexes. Annu Rev Biochem. 76, 167-193 (2007).
- Goth, M., Pagel, K. Ion mobility-mass spectrometry as a tool to investigate protein-ligand interactions. Anal Bioanal Chem. , (2017).
- Uetrecht, C., Rose, R. J., van Duijn, E., Lorenzen, K., Heck, A. J. Ion mobility mass spectrometry of proteins and protein assemblies. Chem Soc Rev. 39 (5), 1633-1655 (2010).
- Hernandez, H., Robinson, C. V. Determining the stoichiometry and interactions of macromolecular assemblies from mass spectrometry. Nat Protoc. 2 (3), 715-726 (2007).
- Sobott, F., Hernandez, H., McCammon, M. G., Tito, M. A., Robinson, C. V. A tandem mass spectrometer for improved transmission and analysis of large macromolecular assemblies. Anal Chem. 74 (6), 1402-1407 (2002).
- Rostom, A. A., et al. Detection and selective dissociation of intact ribosomes in a mass spectrometer. Proc Natl Acad Sci U S A. 97 (10), 5185-5190 (2000).
- Zhou, M., et al. Mass spectrometry of intact V-type ATPases reveals bound lipids and the effects of nucleotide binding. Science. 334 (6054), 380-385 (2011).
- Uetrecht, C., et al. High-resolution mass spectrometry of viral assemblies: molecular composition and stability of dimorphic hepatitis B virus capsids. Proc Natl Acad Sci U S A. 105 (27), 9216-9220 (2008).
- Morgner, N., et al. Hsp70 forms antiparallel dimers stabilized by post-translational modifications to position clients for transfer to Hsp90. Cell Rep. 11 (5), 759-769 (2015).
- Schmidt, C., Beilsten-Edmands, V., Robinson, C. V. The joining of the Hsp90 and Hsp70 chaperone cycles yields transient interactions and stable intermediates: insights from mass spectrometry. Oncotarget. 6 (21), 18276-18281 (2015).
- Schmidt, C., Beilsten-Edmands, V., Mohammed, S., Robinson, C. V. Acetylation and phosphorylation control both local and global stability of the chloroplast F1 ATP synthase. Sci Rep. 7, 44068 (2017).
- Schmidt, C., et al. Comparative cross-linking and mass spectrometry of an intact F-type ATPase suggest a role for phosphorylation. Nat Commun. 4, 1985 (2013).
- Schmidt, C., Robinson, C. V. A comparative cross-linking strategy to probe conformational changes in protein complexes. Nat Protoc. 9 (9), 2224-2236 (2014).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Anal Chem. 89 (3), 1459-1468 (2017).
- Lakomek, K., Stoehr, G., Tosi, A., Schmailzl, M., Hopfner, K. P. Structural basis for dodecameric assembly states and conformational plasticity of the full-length AAA+ ATPases Rvb1 · Rvb2. Structure. 23 (3), 483-495 (2015).
- Mareya, S. M., Raushel, F. M. A molecular wedge for triggering the amidotransferase activity of carbamoyl phosphate synthetase. Biochemistry. 33 (10), 2945-2950 (1994).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Olsen, J. V., et al. Parts per million mass accuracy on an Orbitrap mass spectrometer via lock mass injection into a C-trap. Mol Cell Proteomics. 4 (12), 2010-2021 (2005).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Kirshenbaum, N., Michaelevski, I., Sharon, M. Analyzing large protein complexes by structural mass spectrometry. J Vis Exp. (40), (2010).
- Morgner, N., Robinson, C. V. Massign: an assignment strategy for maximizing information from the mass spectra of heterogeneous protein assemblies. Anal Chem. 84 (6), 2939-2948 (2012).
- Marty, M. T., et al. Bayesian deconvolution of mass and ion mobility spectra: from binary interactions to polydisperse ensembles. Anal Chem. 87 (8), 4370-4376 (2015).
- Thoden, J. B., Wesenberg, G., Raushel, F. M., Holden, H. M. Carbamoyl phosphate synthetase: closure of the B-domain as a result of nucleotide binding. Biochemistry. 38 (8), 2347-2357 (1999).
- Rose, R. J., Damoc, E., Denisov, E., Makarov, A., Heck, A. J. High-sensitivity Orbitrap mass analysis of intact macromolecular assemblies. Nat Methods. 9 (11), 1084-1086 (2012).
- Gault, J., et al. High-resolution mass spectrometry of small molecules bound to membrane proteins. Nat Methods. 13 (4), 333-336 (2016).
- Mehmood, S., et al. Mass spectrometry captures off-target drug binding and provides mechanistic insights into the human metalloprotease ZMPSTE24. Nat Chem. 8 (12), 1152-1158 (2016).
- Kaake, R. M., et al. A new in vivo cross-linking mass spectrometry platform to define protein-protein interactions in living cells. Mol Cell Proteomics. 13 (12), 3533-3543 (2014).
- Liu, F., Rijkers, D. T., Post, H., Heck, A. J. Proteome-wide profiling of protein assemblies by cross-linking mass spectrometry. Nat Methods. 12 (12), 1179-1184 (2015).
- Walzthoeni, T., et al. False discovery rate estimation for cross-linked peptides identified by mass spectrometry. Nat Methods. 9 (9), 901-903 (2012).
- Barrera, N. P., Di Bartolo, N., Booth, P. J., Robinson, C. V. Micelles protect membrane complexes from solution to vacuum. Science. 321 (5886), 243-246 (2008).
- Barrera, N. P., et al. Mass spectrometry of membrane transporters reveals subunit stoichiometry and interactions. Nat Methods. 6 (8), 585-587 (2009).
- Laganowsky, A., et al. Membrane proteins bind lipids selectively to modulate their structure and function. Nature. 510 (7503), 172-175 (2014).
- Marcoux, J., et al. Mass spectrometry reveals synergistic effects of nucleotides, lipids, and drugs binding to a multidrug resistance efflux pump. Proc Natl Acad Sci U S A. 110 (24), 9704-9709 (2013).
- Politis, A., Schmidt, C. Structural characterisation of medically relevant protein assemblies by integrating mass spectrometry with computational modelling. J Proteomics. , (2017).
- Hall, Z., Politis, A., Robinson, C. V. Structural modeling of heteromeric protein complexes from disassembly pathways and ion mobility-mass spectrometry. Structure. 20 (9), 1596-1609 (2012).
- Pukala, T. L., et al. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- Politis, A., et al. Topological models of heteromeric protein assemblies from mass spectrometry: application to the yeast eIF3:eIF5 complex. Chem Biol. 22 (1), 117-128 (2015).
- Politis, A., et al. A mass spectrometry-based hybrid method for structural modeling of protein complexes. Nat Methods. 11 (4), 403-406 (2014).
- Fischer, L., Chen, Z. A., Rappsilber, J. Quantitative cross-linking/mass spectrometry using isotope-labelled cross-linkers. J Proteomics. 88, 120-128 (2013).
- Yu, C., et al. Developing a Multiplexed Quantitative Cross-Linking Mass Spectrometry Platform for Comparative Structural Analysis of Protein Complexes. Anal Chem. 88 (20), 10301-10308 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены