É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Avaliação da Interface sináptica de células T humano primário do sangue periférico e tecido linfoide
Neste Artigo
Resumo
O protocolo descreve uma técnica para estudar a capacidade das células T policlonais humanas de primário para formar sinápticas interfaces usando bilayers do lipid planar. Usamos esta técnica para mostrar a capacidade de formação de sinapse diferencial de células T humanas primárias derivado dos gânglios linfáticos e sangue periférico.
Resumo
A atual compreensão da dinâmica e características estruturais das interfaces sinápticas de células T tem sido largamente determinadas com o uso de vidro apoiados bilayers planares e in vitro-T células derivadas de clones ou linhas1,2 ,3,4. Como estes resultados aplicam-se para as células humanas primárias T isoladas do sangue ou linfoides tecidos não é conhecido, em parte devido a dificuldades na obtenção de um número suficiente de células para análise5. Aqui abordamos isto através do desenvolvimento de uma técnica de slides de fluxo multicanal para construir bilayers do lipid planar contendo moléculas de aderência e ativando a explorar. A baixa altura dos slides fluxo promove a sedimentação celular rápida para sincronizar o acessório de celular: BICAMADA, permitindo aos pesquisadores estudar a dinâmica da formação sináptica interface e a cinética da liberação de grânulos. Podemos aplicar esta abordagem para analisar a interface sináptica de sómente 104 105 primária criopreservado T células isoladas de linfonodos (LN) e sangue periférico (PB). Os resultados revelam que a técnica de bicamada lipídica planar romance permite o estudo das propriedades biofísicas de primárias células T humanas, derivadas do sangue e tecidos no contexto da saúde e na doença.
Introdução
Conhecimento científico das características estruturais de sinapses imunes de células T e sua ligação com a atividade funcional das células T foi gerado principalmente a partir do estudo de linhagens celulares e clones derivado do PB. A que ponto esses achados se relacionam com primárias células T obtidos de sangue ou tecidos linfoides humanos permanece obscuro, como as interfaces sinápticas de células T que residem nos tecidos linfoides e outros não foram analisadas até agora. Importante, dados emergentes sugerem que o residente em tecidos e órgãos linfoides-derivado de células T podem ter diferenças significativas em seu fenótipo e atividade funcional em comparação com aqueles em PB6,7. Isto solidificou ainda mais a necessidade de compreender melhor as características da interface sináptica de células T na primárias células T humanas.
Para este fim, nós desenvolvemos uma abordagem de escala mini novela explorar bilayers do lipid construídos em slides de fluxo multicanal, permitindo-na realizar a imagem de interfaces de T-celular/BICAMADA com menos de 105 primário T células isoladas de humano PB e LN. Esta nova técnica permite o estudo das propriedades biofísicas de primário T-célula sinápticas interfaces humanas a fim de melhor modelo e perceber na vivo interações célula-célula.
Protocolo
Este estudo foi realizado em conformidade com a declaração de Helsinque. Foi obtido consentimento escrito de todos os participantes, e amostras de sangue e linfonodo foram adquiridas com a aprovação do Conselho de revisão institucional da Universidade da Pensilvânia (IRB #809316, IRB # 815056). Todos os seres humanos eram adultos. Amostras de sangue de cordão foram gentilmente cedidas pelo trabalho e entrega do departamento de obstetrícia & ginecologia na Universidade de Thomas Jefferson. Todas as amostras foram identificadas eliminação.
1. isolamento de CD4+ T células para uma análise de imagem
-
Descongele uma alíquota de 1 mL contendo 107 congelado sangue periférico células mononucleares (PBMC) ou células mononucleares nó de linfa (LNMCs) de amostras coletadas. Em uma capa de estéril, adicione as células descongeladas para 9 mL de RPMI suplementado com penicilina/estreptomicina e glutamina.
- Centrifugate as células durante 10 minutos a 300 x g a 4 ° C, aspirar o sobrenadante e ressuspender as células em 5 mL de RPMI complementado contendo 10% FBS (médio completo). Incubar as células durante a noite em uma incubadora de CO2 a 37 ° C.
- No dia seguinte, purificar CD4+ T células por Fima negativa classificação usando um kit comercialmente disponível de acordo com as instruções do fabricante.
- Para medir o número de CD4 recém purificada+ T células, misturar 5 µ l de suspensão de células com igual volume de uma solução de azul de Tripan. Carregar um hemocytometer com uma célula-trypan azul mistura e contar as células vivas dentro de 5 seções do hemocytometer.
- Tirar a média do número de células e determinar o número de células na suspensão de célula original: o número de células/1 mL = a contagem média x 2 x 104. Se o número total de células isoladas é muito pequeno, use as células como é sem contar.
- Centrifugar as células em 300 x g durante 10 minutos e ressuspendê-los em um buffer de ensaio (20 mM HEPES, pH 7,4, 137 mM NaCl, 2mm Na2HPO4, 5mm D-glicose, 5 mM KCl, 1 mM MgCl2, 2mm CaCl2e 1% de albumina de soro humano) em 105 < /C13 > células/50 µ l ou menos e manter as células a 4 ° C (para 1 a 2 h) até que esteja pronto para usar nos experimentos.
- Elimine todos os resíduos biológicos de acordo com as orientações institucionais relevantes.
- Se desejar como uma população de células de controle, preparar células CD8 T ativadas do sangue do cordão PBMC, coloque 107 células em 5 mL de meio completo em um frasco de cultura T25 coberto com uma mistura de anticorpos anti-CD3 e anti-CD28 em 10 µ g/mL e 1 µ g/mL , respectivamente.
- No dia seguinte, retire as células de sangue de cordão ativado o balão, lavá-los 1x com fresco médio de completar e expandir as células na presença de IL-2 recombinante (100 U/mL) por 2 semanas.
- Purificar o sangue do cordão umbilical CD8+ T células classificando Fima negativa usando o kit comercialmente disponível de acordo com as instruções do fabricante. Contar as células e trocar a mídia para o buffer de ensaio conforme descrito nas etapas 1.3-1.6 para LN e CD8 PB+ T células.
2. componentes para a preparação de Bilayers do Lipid Planar
- Preparar 3 tipo de lipossomas conforme descrito em outro lugar5: (a) 0,4 milímetros DOPC (1,2-dioleoyl -sn- glicero-3-fosfocolina) lipossomas, lipossomas DOPC (b) 0,4 milímetros contendo 33 mol % cães-NTA (1,2-dioleoyl -sn- glicero - 3-[(N-(5- amino-1-carboxypentyl) ácido iminodiacetic) Succinil] (sal de amónio)) lipídios, (c) 0,4 mM DOPC lipossomas contendo 4 mol % Biotinyl-Cap-PE (1,2-dioleoyl -sn- glicero-3-phosphoethanolamine - N-(cap biotinyl) (sal de sódio)).
-
Prepare-se 5% solução de caseína como descrito anteriormente,5.
- Dissolver 5 g de caseína em pó em 100 mL de água ultrapura e adicionar 350 µ l de hidróxido de sódio 10 M. Misture tudo em um agitador magnético regular a uma velocidade lenta de acordo com a escala disponível, à temperatura ambiente por 2 h e então durante a noite a 4 ° C. Ajustar o pH para 7,3 e se a solução por 2 h a 100.000 x g a 4 ° C. Filtrar o sobrenadante com um filtro de 0,22 µm estéril e armazenar a solução em alíquotas a-80 ° C.
Cuidado: solução de hidróxido de sódio pode causar queimaduras químicas e provocar cegueira permanente em caso de contacto com os olhos. Usar luvas de borracha, roupas de segurança e proteção para os olhos ao manusear este produto químico ou suas soluções.
- Dissolver 5 g de caseína em pó em 100 mL de água ultrapura e adicionar 350 µ l de hidróxido de sódio 10 M. Misture tudo em um agitador magnético regular a uma velocidade lenta de acordo com a escala disponível, à temperatura ambiente por 2 h e então durante a noite a 4 ° C. Ajustar o pH para 7,3 e se a solução por 2 h a 100.000 x g a 4 ° C. Filtrar o sobrenadante com um filtro de 0,22 µm estéril e armazenar a solução em alíquotas a-80 ° C.
-
Anticorpo anti-CD3 de rótulo com biotina para produzir moléculas de anticorpos mono-bionylated com uma abordagem descrito anteriormente8.
- Prepare uma solução de biotina-PEO4-NHS em dimetilsulfóxido (DMSO) em 0,1 mg/mL. Adicione 3,7 µ l da solução de biotina-PEO4-NHS a 1 mg de anticorpo em 0,5 mL de tampão fosfato salino (PBS) contendo bicarbonato de sódio de 100 mM.
- Incube a mistura por 2 h à temperatura ambiente. Prepare uma solução de éster de Alexa Fluor 488 NHS em 10 mg/mL em DMSO. Adicione a solução de éster de Alexa Fluor 488 NHS ao anticorpo marcado pela biotina-PEO4-NHS em um excesso molar 10 vezes.
- Incube a mistura por 1h à temperatura ambiente com agitação lenta num agitador magnético regular. Separe o corante não acoplado usando cromatografia de exclusão de tamanho.
- Determinar a concentração de anticorpo através da medição da densidade óptica da solução de anticorpo em 280 nm (280). Medir a densidade óptica dos anticorpos etiquetados em 577 nm (577).
- Determine a proporção de corante-para-anticorpo através da seguinte equação:
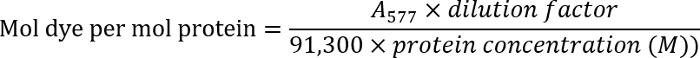
Nota: Encontrar detalhes adicionais no protocolo do fabricante.
-
Expressa uma proteína recombinante de ICAM-1 solúvel em um sistema de expressão de Drosophila conforme descrito anteriormente,3,4,9,10.
- Clone, com codificação de cDNA, o ectodomínio da ICAM-1 em Drosophila expressão vetorial PGTO/V5-His com um promotor inducible metalotioneína para produzir uma proteína recombinante anexada com seus6 marca na extremidade C-terminal.
- Co transfect S2 células com o plasmídeo contendo resultante de ICAM-1 e um vetor de expressão G418. Selecione transfectants estável usando a mídia de Drosophila Schneider suplementada com 10% de soro fetal bezerro (FCS) e 0,5 mg/mL de G418 por 3 semanas. Expandir as células em meio livre de soro inseto e induzir a expressão de proteínas com 0,5 mM de CuSO4 para 3-d.
- Concentre-se no x 10 sobrenadante de cultura e Dialize contra PBS através de um concentrador de fluxo tangencial, como descrito anteriormente,11.
- Aplicar a sobrenadante de cultura concentrada para uma coluna contendo Sepharose com anticorpo monoclonal anti-ICAM-1 covalentemente imobilizado e eluir a ICAM-1 acoplado com um tampão glicina de 50 mM, pH 3.0. Neutralize imediatamente a proteína eluted de ICAM-1 com um tampão Tris de 2 M, pH 8.0.
- Dialize o material eluted contra PBS, pH 8.0 e adicionar o material dializado para um coluna contendo Ni-NTA agarose. Eluir a ICAM-1 solúvel com imidazol 200 mM, pH 8.0. Dialize o material eluted contra um tampão PBS, pH 8.0.
- Rotule o ICAM-1 purificada com éster Cy5 NHS de acordo com as instruções do fabricante.
Nota: A melhor relação de tintura-para-proteína final é 1:1.
- Fab de produzir fragmentos de um anticorpo anti-CD107a por digestão de papaína e purificar o Fab fragmentos por cromatografia de troca iónica, conforme descrito anteriormente3. Rótulo do Fab fragmentos com éster de Alexa Fluor 568 NHS de acordo com as instruções do fabricante.
3. formação de vidro com suporte Planar Lipid Bilayers
- Prepare uma solução fresca piranha ácida misturando 140 mL de ácido sulfúrico concentrado e 60 mL de peróxido de hidrogênio 30%. Lave as lamelas de vidro para os slides de fluxo embebendo-los na solução de ácido piranha de 30 min. espera a lamela de vidro com a pinça de polipropileno tipo tesoura.
Cuidado: Solução de Piranha é um oxidante extremamente forte. Lembre-se de utilizar óculos ou óculos de proteção ou escudo facial juntamente com luvas de borracha grossa em todos os momentos quando manusear a solução. Só trabalho com solução piranha sob uma coifa. Evite o aquecimento do transporte e sacudi-la a qualquer momento durante a utilização, como pode explodir. Recolha os resíduos de piranha em um frasco de vidro com um buraco com chumbo. Entrar em contato com o Comitê de segurança institucional sobre a utilização adequada de resíduos. -
Lavar as lamínulas lavadas 7 x com água ultrapura, transferindo-os sequencialmente em Béquer contendo água doce. Conjunto as lamelas molhadas lado deixar o restante da água rolar fora o vidro limpo, deixando o vidro seco.
- Como alternativa, use uma ponta de pipeta ligada a uma bomba de vácuo para remover as gotículas de água restante cuidadosamente as lamelas.
- No bairro estéreis, realize diluições de vários lipídios para produzir a mistura de lipossomas para fazer bilayers. Em primeiro lugar, combine 37 µ l de lipossomas DOPC e 3 µ l de lipossomas Biotinyl-Cap-PE. Em segundo lugar, misture 14 µ l de lipossomas DOPC e 15 µ l de lipossomas de cães-NTA. Em terceiro lugar, adicione 1 µ l da mistura primeira a 29 µ l do mix segundo para fabricar a mistura final em lipossomas.
-
No bairro estéril, configurar um espaço de trabalho com lamelas secas por perto. µ L de 2 alíquota da mistura final em lipossomas (ver passo 3.3) precisamente no centro de uma calha autoadesivo. Imediatamente e muito justamente alinhar uma lamínula limpa e seca com o slide e baixe suavemente a lamela no lado pegajoso do slide.
- Se preparar mais de um slide, trabalha em um slide em um momento desde que a mistura de lipossoma evapora rapidamente. Vire o slide e usar o anel exterior de fórceps de polipropileno tipo tesoura para aplicar uma pressão suave ao contato periférico da lamela com o slide, certificando-se que o deslizamento é firmemente preso à slide para impedir fugas.
Nota: Não pressione os canais do slide para evitar quebrar ou rachar a lamela. - Vire o slide novamente e marque a posição da bicamada formada, que se parece com uma queda entre a lamínula e o slide de canal, por desenho 4 pontos com um marcador permanente em torno da bicamada no lado externo do slide da assembleia.
- Se preparar mais de um slide, trabalha em um slide em um momento desde que a mistura de lipossoma evapora rapidamente. Vire o slide e usar o anel exterior de fórceps de polipropileno tipo tesoura para aplicar uma pressão suave ao contato periférico da lamela com o slide, certificando-se que o deslizamento é firmemente preso à slide para impedir fugas.
- Antes da primeira injeção de um líquido dentro do canal, designar uma porta do canal como a porta de entrada e o outro como a porta de saída e manter esta designação por todo o experimento.
- Para evitar a formação de bolhas, insira a extremidade da ponta da pipeta diretamente na porta de entrada do canal. Lentamente, encha os canais do slide com 50 µ l de quente (temperatura de quarto pelo menos) buffer de ensaio (ver passo 1.5 para a composição do tampão).
- Prepare uma solução de cloreto de nickel(II) de 0,5 M. Descongelar uma alíquota de 2 mL da solução de caseína em banho-maria a 37 ° C por 30 min e completá-lo com uma solução de cloreto de níquel em uma concentração final de 200 µM.
- Lave os bilayers primeiro injetando 100 µ l da solução de caseína na porta de entrada do canal e removendo imediatamente 100 µ l de fora da porta de saída no slide por pipetagem. Bloquear os bilayers com a mesma solução por injeção de 100 µ l da solução de caseína na porta de entrada de cada canal e incubando o slide para 45 min à temperatura ambiente.
- Descongele alíquotas de Cy5-ICAM-1-His6 e streptavidin proteínas. Combine as proteínas no buffer de ensaio para a concentração final de 2 µ g/mL cada. Centrifugue a solução por 30 min em 20.000 x g e 4 ° C para remover quaisquer agregados.
- Remova o resto da solução de bloqueio da porta de saída do canal de slide por pipetagem. Injete 100 µ l da solução contendo o ICAM-1 e streptavidin na porta de entrada.
- Incube o slide para 45 min à temperatura ambiente. Remova qualquer excesso de solução da proteína da porta de saída. Lave a BICAMADA 2 x primeiro injetando 100 µ l de buffer de ensaio na porta de entrada do canal e imediatamente removendo 100 µ l de fora da porta de saída.
- Dilua um anticorpo anti-CD3 Alexa Fluor-488-com rótulo com o buffer de ensaio para uma concentração final de 2 µ g/mL. Injetar 100 µ l da solução de anticorpo na porta de entrada do slide e incube-lo por 45 min à temperatura ambiente. Remova qualquer excesso de solução da proteína da porta de saída. Lave a BICAMADA 2 x com 100 µ l de buffer de ensaio como na etapa 3.11.
4. imagem latente da interação com a BICAMADA Planar células T
-
Pré-aqueça o palco e o objectivo de um microscópio de fluorescência (TIRF) de reflexão interna total ou confocal, até que a temperatura está incubada a 37 ° C. Configure o slide com o bilayer(s) no palco aquecido. Mover o palco para uma posição apropriada de acordo com as marcas de tinta e focar a BICAMADA empregando a fluorescência de moléculas com rótulo Cy ICAM-1.
- Use um objectivo X 61 para o microscópio confocal, ou um objectivo X 100 para o microscópio TIRF, com configurações de filtro apropriado.
-
Para grânulo versão de imagem por microscopia TIRF, adicionar Alexa Fluor-568-rótulo anti-CD107a fragmentos de anticorpos Fab à suspensão de células em uma concentração final de 4 µ g/mL antes de injetar as células para as portas de entrada.
- Ressuspender o CD4 preparado+ T células isoladas de LN ou PB ou cabo de sangue e injetar 50 µ l de suspensão de célula para a porta de entrada do canal slide contendo a BICAMADA.
- Escolha o número desejado de campos e gravar imagens de cada campo 1 x a cada 2 min para 30 min após a injeção.
- Explorar o campo brilhante, refletidas luz fluorescentes canais e (Alexa 488 e Cy5) do microscópio confocal para adquirir as imagens. Use o modo TIRF para fluorescência Alexa-Fluor-488 e Alexa-Fluor-568 e widefield para fluorescência Cy5, bem como imagens de campo claro, no microscópio TIRF.
5. análise de imagens
- Analise as imagens adquiridas usando o software apropriado. Observe a morfologia celular na imagens de luz transmitidas e excluir agrupado e visivelmente danificada ou pilhas apoptotic da análise. Inclua na análise somente as células que interagir produtivamente com a superfície de BICAMADA (ou seja, células acumulando Alexa-Fluor-488 fluorescência (anticorpos anti-CD3) na interface).
- Determine o tamanho da área de adesão celular durante 20 min após o início da interação célula-BICAMADA.
Nota: A área de adesão é a área escura desenvolvida na interface celular-BICAMADA sobre as imagens de microscopia (IRM) de reflexão de interferência. - Observe qualquer acumulação de fluorescência Cy5-ICAM-1 e uma formação da junção do anel por moléculas de Cy-ICAM-1 segregadas na interface celular-BICAMADA. Se as moléculas de ICAM-1 acumuladas formaram uma junção do anel de adesão pelo menos duas imagens consecutivas, designe tais células como células desenvolvendo um periférico supramolecular ativando cluster (pSMAC)12.
- Avalie a liberação de grânulo medindo a intensidade de fluorescência de Alexa-Fluor-568 na interface de células T-BICAMADA sobre a fluorescência de fundo fora da área de contato nas proximidades para a célula. Designe as células com um rácio de Alexa-Fluor-568 sinal-para-plano de fundo de pelo menos 1.3 como células degranulating.
Resultados
Primeiro, comparamos a estrutura da interface sináptica formada por ativadas CD8 cabo-hemoderivados+ T células expostas a bilayers do lipid construíram ou fluxo em grande escala tradicional em sistemas de células (veja a Tabela de materiais para obter detalhes)1 ,2,3,4 ou em slides de fluxo multicanal. Os bilayers continham fluor...
Discussão
A nova técnica descrita aqui utiliza reagentes semelhantes necessários para construir bilayers planares no fluxo convencional célula5 e pode ser aplicada com sucesso para executar a imagem da principal célula T humana – BICAMADA interfaces3,4 ,15. A técnica oferece uma redução significativa no uso do moléculas fluorescentes e necessita de 10 – 20 x menos células T em comparação com um flux...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi financiado pela subvenção NIH R01AI118694 para Michael R. Betts, que inclui o prêmio sub 566950 para Yuri Sykulev. Agradecemos a Sidney Kimmel Cancer Center Bioimaging recurso compartilhado pelo excelente apoio.
Materiais
| Name | Company | Catalog Number | Comments |
| CD4 T cell isolation kit, human | Miltenyl Biotec | 130-096-533 | |
| CD8 T cells Isolation Kit, human | Miltenyl Biotec | 130-096-495 | |
| DOPC | Avanti Polar Lipids | 850375C | |
| DOGS NTA | Avanti Polar Lipids | 790528C | |
| Biotinyl Cap PE | Avanti Polar Lipids | 870273C | |
| Human Serum Albumin | Octapharma USA | NDC 68982-643-01 | |
| sticky-Slide VI 0.4 | ibidi | 80608 | |
| Coverslips for sticky-Slides | ibidi | 10812 | |
| Bioptech FCS2 Chamber | Bioptech | 060319-2-03 | |
| anti-CD3 antibody | Thermo Fisher Scientific | 16-0037-81 | OKT3 clone, hybridoma cells are available from ATCC |
| anti- CD28 antibody | Genetex | GTX14664 | 9.3 clone |
| Casein | Sigma | C5890 | |
| Biotin-PEO4-NHS | Thermo Fisher Scientific | 21329 | |
| DMSO | Sigma | D2650-5 | |
| Alexa Fluor 488 protein labeling kit with column for labeled protein purification | Thermo Fisher Scientific | A10235 | |
| Alexa Fluor 568 protein labeling kit with column for labeled protein purification | Thermo Fisher Scientific | A10238 | |
| Amersham Cy5 NHS Ester | GE Life Science | PA15101 | |
| pMT/V5-His A, B, C Drosophila Expression Vectors | Thermo Fisher Scientific | V412020 | |
| pcopneo, G418 Drosophila expression vector for positive selection | ATCC | 37409 | |
| Serum free Drosophial media Insect-XPRESS | Lonza | 12-730Q | |
| Hybridoma YN1/1.7.4 | ATCC | CRL1878 | The hybridoma secrets antibody against ICAM-1. |
| Cyanogen bromide-activated-Sepharose 4B | Sigma-Aldrich | C9142 | Utilized for preparation of Sepharose with covelently bound anti-ICAM antibody. |
| MasterFlex tangential flow concentrator | Cole-Parmer | 77601-60 7592-40 | Used for ICAM-1 containing supernatant concentration and dialysis of ICAM-1 containing supernant |
| Centramate Lab Tangential Flow Systems | Pall Laboratory | FS002K10 OS010T12 FS005K10 | Used for ICAM-1 containing supernatant concentration and dialysis of ICAM-1 containing supernant |
| Ni-NTA Agarose | QIAGEN | 30210 | |
| Dialysis tubing | Spectra/Por | 131384 | |
| Papain from papaya latex | Sigma | P3125 | |
| mouse anti-human antibody against CD107a | BD Bioscences | 555798 | Clone H4A3 |
| Ansell Natural Blue Gloves | Fisher Scientific | 19-014-539 | |
| Nalgene Polypropylene Scissor-Type Forceps | Thermo Fisher Scientific | 6320-0010 | |
| Streptavidin | ProZyme | SA10 | |
| Confocal microscope | Nikon | Nikon TiE inverted microscope equipped with PFS for long-term image stability control, 60X oil objectives, 4 lasers with excitation lines at 405, 458, 488, 514, 561, and 640 nm, 2 GaAsP detectors and 2 high sensitivity PMTs, DIC transmitted light, Programmable X,Y,Z stage for multiple positions and stitching of large areas, time lapse functions, Tokai-Hit temperature and CO2-controlled chamber for live imaging, and anti-vibration isolation table | |
| TIRF microscope | Andor | Andor Revolution XD system equipped with Nikon TIRF-E illuminator, Lasers with 405,488,561 and 640 lines, DIC transmitted light, Yokogawa CSU-X1 spinning disk head for confocal imaging, 100/1.49 NA objective, Andor iXon X3 EM-CCD camera, objective heater, and a piezoelectric motorized stage with Perfect Focus System (PFS) | |
| MetaMorph Premier Image Analysis Software | Molecular devices |
Referências
- Grakoui, A., et al. The immunological synapse: a molecular machine controlling T cell activation. Science. 285, 221-227 (1999).
- Somersalo, K., et al. Cytotoxic T lymphocytes form an antigen-independent ring junction. Journal of Clinical Investigation. 113, 49-57 (2004).
- Beal, A. M., et al. Protein kinase C theta regulates stability of the peripheral adhesion ring junction and contributes to the sensitivity of target cell lysis by CTL. The Journal of Immunology. 181, 4815-4824 (2008).
- Beal, A. M., et al. Kinetics of early T cell receptor signaling regulate the pathway of lytic granule delivery to the secretory domain. Immunity. 31, 632-642 (2009).
- Dustin, M. L., Starr, T., Varma, R., Thomas, V. K. Supported planar bilayers for study of the immunological synapse. Current Protocols in Immunology. , (2007).
- Reuter, M. A., et al. HIV-Specific CD8(+) T Cells Exhibit Reduced and Differentially Regulated Cytolytic Activity in Lymphoid Tissue. Cell Reports. 21, 3458-3470 (2017).
- Buggert, M., et al. Limited immune surveillance in lymphoid tissue by cytolytic CD4+ T cells during health and HIV disease. PLoS Pathogens. 14, e1006973 (2018).
- Carrasco, Y. R., Fleire, S. J., Cameron, T., Dustin, M. L., Batista, F. D. LFA-1/ICAM-1 interaction lowers the threshold of B cell activation by facilitating B cell adhesion and synapse formation. Immunity. 20, 589-599 (2004).
- Anikeeva, N., et al. Distinct role of lymphocyte function-associated antigen-1 in mediating effective cytolytic activity by cytotoxic T lymphocytes. Proceedings of the National Academy of Sciences of the United States of America. 102, 6437-6442 (2005).
- Steblyanko, M., Anikeeva, N., Campbell, K. S., Keen, J. H., Sykulev, Y. Integrins Influence the Size and Dynamics of Signaling Microclusters in a Pyk2-dependent Manner. The Journal of Biological Chemistry. 290, 11833-11842 (2015).
- Anikeeva, N., Lebedeva, T., Sumaroka, M., Kalams, S. A., Sykulev, Y. Soluble HIV-specific T-cell receptor: expression, purification and analysis of the specificity. Journal of Immunological Methods. 277, 75-86 (2003).
- Monks, C., Freiberg, B., Kupfer, H., Sciaky, N., Kupfer, A. Three-dimensional segregation of supramolecular activation clusters in T cells. Nature. 395, 82-86 (1998).
- Riddell, S. R., Greenberg, P. D. The use of anti-CD3 and anti-CD28 monoclonal antibodies to clone and expand human antigen-specific T cells. Journal of Immunological Methods. 128, 189-201 (1990).
- Lin, S. J., Yu, J. C., Cheng, P. J., Hsiao, S. S., Kuo, M. L. Effect of interleukin-15 on anti-CD3/anti-CD28 induced apoptosis of umbilical cord blood CD4+ T cells. European Journal of Haematology. 71, 425-432 (2003).
- Anikeeva, N., Sykulev, Y. Mechanisms controlling granule-mediated cytolytic activity of cytotoxic T lymphocytes. Immunologic Research. 51, 183-194 (2011).
- Huppa, J. B., et al. TCR-peptide-MHC interactions in situ show accelerated kinetics and increased affinity. Nature. 463, 963-967 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados