Method Article
Testes anticorpos anti-RDL e Anti-mglutr1 em seções cerebrais de abelhas usando CRISPR-Cas9
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Apresentado aqui é um protocolo para usar o sistema CRISPR-Cas9 para reduzir a produção de uma proteína no cérebro de abelha adulta para testar a especificidade do anticorpo.
Resumo
Cluster Regularmente Interspaced Short Palindromic Repeats (CRISPR)/CRISPR-associated protein 9 (Cas9) é uma técnica de edição de genes amplamente utilizada em estudos de função genética. Usamos este método neste estudo para verificar a especificidade dos anticorpos desenvolvidos contra o inseto GABAA subunidade receptor Resistance to Dieldrin (RDL) e um receptor glutamato metabotropic mGlutR1 (mGluRA). Os anticorpos foram gerados em coelhos contra os peptídeos conjugados específicos das moscas frutíferas (Drosophila melanogaster) bem como para abelhas(Apis mellifera). Usamos esses anticorpos em seções cerebrais de abelhas para estudar a distribuição dos receptores em cérebros de abelhas. Os anticorpos foram purificados contra o peptídeo e testados com imunocoagulação e o método clássico de preadsorção com conjugados peptídeos para mostrar que os anticorpos são específicos para os conjugados correspondentes peptídeos contra os quais foram levantados. Aqui desenvolvemos a técnica CRISPR-Cas9 para testar a redução de metas proteicas no cérebro 48 h após injeção CRISPR-Cas9 com RNAs guia projetado para o receptor correspondente. O método CRISPR-Cas9 também pode ser usado em análises comportamentais nas abelhas adultas quando um ou múltiplos genes precisam ser modificados.
Introdução
O sistema CRISPR/Cas9 recentemente descoberto é uma ferramenta poderosa que tem sido usada para alterar o DNA genômico em vários sistemas e organismos de modelos. Acelerou a pesquisa biomédica e grandes avanços tecnológicos, tornando a modificação do genoma mais eficiente e robusta do que os métodos anteriores1. Nativo da bactéria S. pyógenes, o sistema conta com uma endonuclocaçãa cas9, cuja atividade leva a quebras duplas encalhadas (DSBs) no DNA, e um Guia RNA (gRNA) que direciona a proteína Cas9 para um local específico, dependente de sequência2. Pausas duplas-encalhadas geradas pelo CRISPR/Cas9 podem ser reparadas através de uma junção final não homologous (NHEJ), um processo propenso a erros que pode levar a mudanças de quadros ou reparo direto de homologia quando um modelo de doador está presente. O gRNA em si consiste em um CrispR RNA (crRNA) específico para alvos e um crRNA transativador universal (tracrRNA) que pode ser quimicamente sintetizado e entregue com nuclease Cas9 purificado como um complexo ribonucleoprotein (RNP)2,3. A rotulagem fluorescente do gRNA ou nuclease Cas9 pode permitir a detecção e visualização intracelular de componentes moleculares através de microscopia fluorescente4.
Em nosso trabalho atual, aproveitamos o sistema CRISPR-Cas9 para reduzir os níveis de proteína em cérebros adultos de abelhas. Estudamos o receptor glutamato metabotropic (mGluR) e anticorpos receptores anti-mGlutR1 e os anticorpos de subunidade RDL e anti-RDL do receptor GABAA. Desenvolvemos um método simples para reduzir a quantidade de proteína no cérebro da abelha adulta e a usamos para conduzir testes adicionais dos anticorpos desenvolvidos contra as proteínas correspondentes. O monitoramento da fluorescência do CRISPR-Cas9 nos permitiu estimar as áreas e células envolvidas na redução da proteína.
Usando este método, também caracterizamos os anticorpos anti-mGlutR1 que foram feitos em coelhos contra o peptídeo conjugado. O genoma da abelha codifica um AmGluRA altamente conservado (chamado mGlutR1 de acordo com a nomenclatura NCBI) receptor glutamato metabotropic5. O gene honeybee mGlutR1 tem quatro variantes de splice previstas de acordo com o banco de dados NCBI. Foi relatado que está expresso no sistema nervoso central (SNC) tanto de fase de abelhas pupa quanto adulta e está envolvido na formação de memória de longo prazo5. Anticorpos desenvolvidos contra mGlutR1 podem ser uma ferramenta essencial para estudar o sistema glutamatergico no processo de aprendizagem e memória em abelhas.
Em nossos estudos, também caracterizamos anticorpos anti-RDL desenvolvidos em coelhos imunizados com peptídeos conjugados da subunidade receptorapifera RDL. O gene Honeybee Rdl, AmRdl (XM_006565102.3, banco de dados NCBI), tem 14 variantes de emenda previstas. Um fragmento parcialmente clonado foi relatado no banco de dados da NCBI AF094822.1. A função receptora RDL e sua fisiologia são bem estudadas em insetos6,7,8, incluindo abelhas9,10,11. Anticorpos desenvolvidos contra o anti-RDL podem ser uma ferramenta essencial para estudar o sistema GABAergic no processo de aprendizagem e memória em abelhas.
Um estudo anterior sobre o papel dos receptores de octopamina e tirasmina usou RNAi injetado no cérebro com um teste subsequente da quantidade de proteína pela mancha ocidental12,13. No entanto, a RNAi tem algumas limitações significativas. Há apenas uma janela de curto tempo após a injeção RNAi dentro da qual ocorre uma redução da proteína13. CRISPR-Cas9 foi usado muito recentemente em embriões de abelhas para excluir ou modificar genes em todo o animal14,15,16. Reportamos o uso de CRISPR-Cas9 para reduzir a quantidade de proteína na abelha adulta. Desenvolvemos essa abordagem para abelhas devido à capacidade de acopla-la a estudos comportamentais de aprendizagem e memória em condições laboratoriais controladas17.
No presente trabalho, desenvolvemos anticorpos contra dois receptores e os testamos nas seções cerebrais de abelhas adultas depois que a proteína foi reduzida pela injeção CRISPR-Cas9. Ao mesmo tempo, estabelecemos um projeto experimental que permite o uso do método para experimentos comportamentais.
Protocolo
O protocolo descrito aqui segue as diretrizes de cuidados com animais da Universidade Estadual do Arizona.
1. Isolamento total de proteínas de cérebros de Apis mellifera
NOTA: Use apis mellifera New World Carniolan forasteiros de idade desconhecida para este experimento.
- Coloque uma tela de malha de alumínio sobre a entrada da colmeia para capturar abelhas forager17. Capture cada abelha em um frasco com um pequeno buraco em cada tampa. Coloque os frascos contendo as abelhas no gelo para baixar a temperatura corporal e imobilizá-las. Deixe as abelhas no gelo por não mais do que 3 min.
- Proteja as abelhas imobilizadas em detentores de metais previamente preparados. Certifique-se de que os suportes metálicos sejam construídos para que a abelha possa ser fixada com pequenos pedaços de fita adesiva, mas ainda tenha seu tórax traseiro, asas e cabeça expostas.
ATENÇÃO: Certifique-se de que as abelhas estejam totalmente imobilizadas antes de tentar colocá-las nos titulares. - Alimente as abelhas com uma solução de sacarose de 1 M usando uma seringa de 5 mL até que elas não estejam mais com fome. Coloque as abelhas seguras em uma caixa com uma toalha de papel molhado para garantir um ambiente úmido.
- Disseque o cérebro da abelha rapidamente cortando a cabeça com a tesoura Barraquer Iris (ver Tabela de Materiais)e use a tesoura para abrir a cabeça da frente.
- Corte o cérebro da cápsula da cabeça, pegue o cérebro comfórceps #5 e coloque-o em 100 μL de tampão de lise frio (4-8 °C). O buffer de lise consiste em 120 mM Tris-HCl, 2% sulfato dodecyl de sódio (SDS), 5% glicerol, 0,2 mM dithiothreitol, 1% Triton X-100 e 1-5 μg/mL dos inibidores protease PMSF (fenilmesulfofifofialfluoride), aprotina, benzamidina (pH 6,8) a 4 C °. Homogeneize o cérebro na solução de lise, transformando-se em um pilão por cerca de 2 min.
- Centrífuga a amostra em 12.000 x g por 20 min. Aspirar 90 μL do supernatante e descartar a pelota.
- Tome 1 μL do supernatante para quantificar a proteína total usando um fluorímetro. A concentração aproximada da proteína total foi entre 2-3 mg/mL por abelha. Tome 10 μL do supernatant e adicione 10 μL do buffer de lise e 10 μL de um buffer de 6x Laemmli18. Gire brevemente e ferva por 3 min, depois esfrie no gelo. Gire por 1 min a 10.000 x g para remover todos os detritos. Um décimo de cérebro de abelha contém aproximadamente 25 ng de proteína total.
2. Western Blotting19
- Faça 30 mL de 10% de gel de corrida contendo 12,15 mL de água destilada ultrapura, 7,5 mL de 1,5 M Tris-HCl (pH 8.8), 0,3 mL de 10% SDS, 10 mL de 30% solução de acrilamida de acrilamida-bis, 0,15 mL de 10% de persulfato de amônio (APS) e 20 μL de tetramemetetilenodiamina (TEMED) ).
- Molde o gel entre duas placas de vidro separadas por espaçadores.
- Faça um gel de empilhamento de 20 mL contendo 12,1 mL de água destilada ultrapura, 5,0 mL de 0,5 M Tris-HCl (pH 6.8), 0,2 mL de 10% SDS, 2,6 mL de acrilamida acrilal-bis, 0,1 mL de 10% APS e 20 μL de TEMED.
- Quando o gel em execução se solidificar, despeje um gel de empilhamento. Adicione cuidadosamente o separador de plástico para lançar a faixa de carregamento, evitando bolhas. Espere 15-30 min, até que o gel seja solidificado.
- Comece a carregar o gel usando 5 μL de padrões proteicos. Carregue 20 μL da mistura de lysato a partir da etapa 1,8 por faixa, correspondendo a ~1/15 de um cérebro de abelha ou ~16 ng de proteína total por pista. Execute as amostras por 3,5-4 h no total a 16 mA no gel de empilhamento e 32 mA no gel de corrida. Pare quando o tinéme deixar o gel.
- Transfira as proteínas para as membranas de nitrocelulose em tampão de transferência (25 mM Tris-HCl, 192 mM glicina, 15% metanol) a 0,45 mA por 1h 30 min a 4 °C.
- Para avaliar a eficiência da transferência de proteína após o SDS-PAGE antes da imunolotamento, adicione a solução de coloração Ponceau S (adicione 0,1 g de Ponceau S e 5 mL de ácido ástico à água a um volume final de 100 mL). Armazene a 4 °C por 1 min e enxágue rapidamente com água destilada.
- Rotular cada pista com uma caneta esferográfica, corte a membrana contendo duas faixas com homogeneeidade cerebral e uma faixa com marcador de proteína e coloque cada uma em uma caixa de incubação de manchas ocidentais. Lave 3x por 5 min cada em soro-fato tampão de fosfato (PBS) contendo 0,1% Tween 20 (PBS-Tw).
- Bloqueie a membrana com 10% de NGS (1 mL de soro normal de cabra para 10 mL de PBS-Tw) em uma caixa de incubação de manchas ocidentais por 1h. Faça uma diluição anti-mGlutR1 (5 μL de anticorpo em 10 mL de 10% NGS). Faça diluições anti-RDL1 e anti-RDL2 (5 μL de anticorpo em 10 mL de 10% NGS cada). Substitua a solução de bloqueio em cada caixa pelos anticorpos diluídos e deixe durante a noite (16-24 h) a 4 °C.
- Lave a membrana 3x por 5 min cada em PBS-Tw. Incubar a membrana com anticorpos secundários conjugados anti-coelho IgG HRP às 1:10.000 em 10% NGS PBS-Tw por 2 h à temperatura ambiente (RT). Lave membranas 3x em PBS-Tw, depois 1x em PBS.
- Detecte as bandas usando substrato hrp quimiluminescente ocidental. Misture dois substratos 1:1 (v/v) na RT, coloque todas as membranas na mesma caixa e cubra-as com a mistura de substrato por 2 minutos (em uma sala escura com luz vermelha) na RT. Prossiga para a detecção de proteínas usando um filme de autordiografia com vários tempos de exposição. Geralmente um anticorpo é testado em uma membrana contendo a mesma diluição do cérebro homogeneizado em duas ou três pistas e uma faixa com o marcador de peso.
3. Procedimentos imunocitoquímicos
- Para dissecar cérebros de abelhas para imunocitoquímica, imobilize as abelhas no gelo por 30 s. Depois que as abelhas forem imobilizadas, decapitar a abelha com tesoura e colocar a cabeça em uma solução de 4% paraformaldeído na PBS. Trabalhe o capô da fumaça.
ATENÇÃO: O corpo deve ser cuidadosamente descartado porque o abdômen ainda pode picar após a decapitação. - Cuidadosamente, mas remova rapidamente as antenas, acessa os olhos e corte todo o exoesqueleto superior com tesouras Barraquer Iris. Deixe as cabeças sentarem-se na solução fixa por 10 minutos. Remova o resto do exoesqueleto da cabeça e corte todas as traqueadas restantes.
- Coloque cada cérebro em um tubo de microcentrífuga de 1,5 mL contendo pelo menos 1 mL de solução paraformaldeído de 4% e deixe durante a noite em 4-8 °C.
- Faça uma solução de 7,6% de agarose misturando 3,8 g de agarose e 50 mL de água destilada em um frasco de Erlenmeyer. Micro-ondas a solução até que a agarose liquifica.
NOTA: Um pequeno pedaço de papel de tecido pode ser colocado na abertura do frasco, a fim de evitar que a solução de agarose transborde durante o aquecimento. - Coloque os cérebros fixos de abelhas (3-4 cérebros) em uma placa de Petri de 35 mm e remova o excesso fixado com papel de tecido. Despeje a solução de agarose líquida sobre o cérebro. Oriente os cérebros na agarose para que os lóbulos da antena estejam de frente para cima. Deixe a agarose esfriar e solidificar.
- Depois que a agarose se solidificou, cortar blocos de agarose cada um contendo um cérebro.
- Para secção vibratome, prepare uma placa de 24 poços com cada poço contendo uma cesta com uma malha hidrofóbica na parte inferior. Encha cada poço com 600 μL de PBS.
- Corte cada bloco em seções transversais de 70 μm usando a máquina vibratome e coloque as seções na cesta contendo PBS.
NOTA: Certifique-se de que seções do mesmo cérebro sejam colocadas na mesma cesta. - Lave as seções cerebrais 6x por 10 min cada com PBS-TX para garantir que nenhum fixante permaneça nas seções. Coloque a placa multiwell em um agitador orbital e lave os cérebros a 210 rpm. Antes de cada lavagem certifique-se de substituir a solução PBS-TX por solução PBS-TX fresca. Bloqueie com 1% de soro de burro normal durante a última lavagem.
- Para testar o anticorpo primário anti-mGlutR1, prepare uma diluição de 1:112 de anticorpos anti-mGlutR1 adicionando 9 mL de PBS-TX a 80 μL de anticorpos anti-mGlutR1 em um tubo de centrífuga de 15 mL. Vórtice o tubo brevemente para misturar completamente. A diluição de anticorpos foi determinada em experimentos preliminares.
- Para testar o anticorpo primário anti-RDL, prepare uma diluição de 1:100 de anticorpos anti-RDL adicionando 30 μL de peptídeo anti-RDL 1, 30 μL de peptídeo anti-RDL 2 e 6 mL de PBS-TX em um tubo centrífuga de 15 mL. Vórtice o tubo brevemente para misturar completamente. A diluição de anticorpos foi determinada em experimentos preliminares.
- Adicione 800 μL de solução de anticorpos a cada poço na placa. Cubra a placa multiwell e enrole-a em papel alumínio para evitar a degradação da exposição à luz. Coloque a placa envolta em papel alumínio em um shaker orbital e agite a 210 rpm por 2h. Em seguida, deixe durante a noite na RT sem tremer.
- Depois que as seções cerebrais forem deixadas durante a noite, lave com PBS-TX por 10 min. Repita a etapa de lavagem 6x.
- Prepare os anticorpos secundários (anti-coelho do burro) fazendo uma diluição de 1:225 de anticorpos secundários adicionando 40 μL de anticorpos secundários a 9 mL de PBS-TX.
- Adicione 800 μL da diluição secundária do anticorpo a cada poço. Cubra a placa e enrole-a em papel alumínio. Coloque a placa envolta em papel alumínio no shaker orbital e agite a 210 rpm por 2h. Em seguida, deixe-o durante a noite na RT.
- Lave as seções cerebrais 3x por 10 min cada com PBS-TX e 3x com solução PBS regular.
- Para incorporar as seções nos slides, prepare a solução de incorporação de mídia/glicerol de montagem modificada a partir de Rodriguez et al.20. Adicione 5 g do meio de montagem em PBS de 20 mL e mexa por 16 h com um agitador magnético. Adicione 10 mL de glicerol e mexa por mais 16 h com um agitador magnético. Centrífuga por 15 min a 4.000 x g, pegue o supernatante líquido homogêneo e a alíquota em 1 mL tubos. Mantenha-se em -20 °C
- Incorpore as seções nos slides com uma gota do meio de montagem preparado na etapa 3.17, certificando-se de que cada slide contenha seções de um cérebro.
- Controle de preatopção de imunocoloração com peptídeos conjugados
- Para peptídeos anti-RDL e conjugados, incubar a diluição de trabalho de anticorpos anti-RDL com o peptídeo correspondente conjugado durante a noite na RT no shaker: condição 1) peptídeo de 500 μg conjugado com Keyhole Limpet Hemocyanin (KLH) via glutaraldeído; condição 2) sem conjugação.
- Centrífuga cada mistura por 10 min a 10.000 x g e coletar o supernatant de ambas as condições (passo 3.19.1).
- Incubar as seções seriais do cérebro de abelha sem cada supernatante e processar com os anticorpos secundários descritos acima. Seções de série são seções que se seguem durante os procedimentos de secção vibratome, de modo que a mesma parte do cérebro será exposta a controles positivos e negativos.
- Para o peptídeo anti-mGlutR1 e conjugado, incuba a diluição de trabalho de anticorpos anti-mGlutR1 na RT com o peptídeo conjugado KLH contendo o peptídeo de10-4 M (condição 1) e sem qualquer conjugado (condição 2).
- Centrífuga cada mistura por 10 min a 10.000 x g e coletar o supernatante de ambos os controles.
- Incubar as seções seriais do cérebro de abelha com supernatante de ambas as condições e processo com os anticorpos secundários (passos 3.14-3.15).
- Incorpore seções de ambas as condições para o slide usando mídia de incorporação (passo 3.17).
4. Teste de RDL e mGlutR1 Protein Expression por Imunocytochemistry (seção 3) no Cérebro de Abelha após injeção do sistema CRISPR-Cas9 correspondente
- Projete os guias usando uma ferramenta de design CRISPR-Cas9 online21 usando as sequências de DNA genômica de AmRdl (XM_006565102.3) e sequências de mGlutR1 (XM_006566244.3) (Tabela 1). Ordem como Cas9 crRNA e Cas-9 trarRNA com o corante fluorescente ATTO550 no final de 5'. Peça o Cas9 Nuclease V3 (Ver Tabela de Materiais).
- Prepare os componentes para o sistema CRISPR-Cas 9 fazendo um estoque de 100 μM de cada guia crRNA e tracrRNA usando água sem nuca. Aliquot e loja a -20 °C. Prepare a concentração de trabalho da solução Cas-9 adicionando 2,5 μL de Cas 9 nuclease V3 (10 mg/mL) a 47,5 μL de buffer livre de nucleotídeo para obter uma concentração final de 0,5 μg/μL. Faça gRNA e RNP para injeção.
- Prepare a formação complexa do gRNA para cada RNA guia separadamente (guideRNA:tracrNAATTO550)
- Rotular um tubo de ensaio com o nome do gRNA, adicionar 92 μL de buffer livre de nucleotídeo, 4 μL de 100 μM CRISPR-Cas9 tracrRNA- 5'ATTO550 e 4 μL da solução de crRNA guia. Misture suavemente e gire.
NOTA: Exemplo de rotulagem de tubos gRNA para RDL: GRDL1, GRDL2, GRDL3 (correspondente às guias RDL RNA na Tabela 1). Exemplo de rotulagem de tubos gRNA para mGlutR1: GMGL1, GMGL2, GMGL3(Tabela 1). - Aqueça a solução a 95 °C por 5 min para criar gRNA. Esfrie a mistura na RT por 10 min.
- Rotular um tubo de ensaio com o nome do gRNA, adicionar 92 μL de buffer livre de nucleotídeo, 4 μL de 100 μM CRISPR-Cas9 tracrRNA- 5'ATTO550 e 4 μL da solução de crRNA guia. Misture suavemente e gire.
- Prepare a formação complexa RNP (gRNA: S.p Cas9Nuclease), misturas de entrega e controle.
- Rotular um tubo de ensaio com o nome do RNP, adicionar 6 μL de solução gRNA e 6 μL de 0,5 μg/μL S.p Cas9 Nuclease V3. Misture suavemente e incuba as soluções a 37 °C por 10 min antes da injeção.
NOTA: Exemplo de etiquetas de tubo RNP: RRDL1, RRDL2, RRDL3. - Faça um RNPRDLmix. Para preparar a mistura final usada para injeções, adicione 4 μL de cada RNP de RDL juntos. Mistura RNP/RDL = 4 μL RRDL1 + 4 μL RRDL + 4 μL RRDL3.
- Faça um RNPmGlutR1mix. Misture 4 μL de cada RNP de mGlutR1 juntos para preparar a mistura final usada para injeções (RNPmGlutR1mix) = 4 μL RMG1 + 4 μL RMG2 + 4 μL RMG3.
NOTA: Exemplo de etiquetas de tubo RNP: RMG1, RMG2, RMG3. - Faça uma solução noguideRNA de controle misturando 4 μL de trarRNA, 92 μL de tampão e 4 μL de água em vez de guideRNA (ver seção 4.3). Misture 6 μL desta solução noguideRNA e 6 μL de 0,5 μg/μL Cas9 Nuclease V3 (passo 4.4.1) para produzir a solução de injeção de controle.
- Rotular um tubo de ensaio com o nome do RNP, adicionar 6 μL de solução gRNA e 6 μL de 0,5 μg/μL S.p Cas9 Nuclease V3. Misture suavemente e incuba as soluções a 37 °C por 10 min antes da injeção.
5. Procedimento de injeção
- Imobilize cada abelha como descrito na etapa 1.3 e alimente-as como na etapa 1.4. Em seguida, prepare dois conjuntos de duas caixas de madeira para soltar as abelhas após a injeção: uma caixa branca (condição experimental) e uma caixa preta (controle). Cada caixa deve conter um pente pequeno e uma placa de petri de alimentação. Um lado de cada caixa é feito de vidro para permitir observação.
- Faça a alimentação de pratos petri. Pegue a tampa de uma placa de Petri de 35 mm, coloque cera dentro da superfície e coloque a parte inferior da placa de Petri na cera. Segure a placa na caixa.
- Usando uma seringa de 5 mL, injete solução de sacarose de 1M entre a tampa e a parte inferior do prato. As abelhas podem rapidamente colocar suas proboscises entre as duas placas e permanecerão secas por um período de incubação de 48 h. Coloque um prato em cada caixa.
- Para preparar o sistema de microinjeção, encha os capilares com óleo mineral sem bolhas de ar. Coloque os capilares no suporte injetor do sistema de microinjeção. Para carregar o capilar com a solução de injeção desejada, coloque uma película hidrofóbica em uma superfície plana e pipete a solução sobre ele. Injete o óleo dos capilares e substitua a mistura pelo mix RNP correspondente.
- Usando o sistema de microinjeção, injete 345 nL de solução de mistura RNP diretamente no ocelli mediano de cada abelha.
- Para injeção RDL-CRISPR-Cas9, injete oito abelhas com o RNPRDLmix preparado conforme descrito na etapa 4.4.2. Alimente-os 1M sacarose após a injeção. Solte-as na caixa branca (experimental) com o pente pequeno e alimentando a placa de Petri por 48 h. Use outras oito abelhas como controles sem qualquer injeção, alimente-as e solte-as na caixa preta (controle).
- Para mGlutR1 CRISPR-Cas9, injete nove abelhas com o RNPmGlutR1mix preparado como descrito na etapa 4.4.3. Para o controle injetar oito abelhas com mistura de RNA sem guia (RNAmix_noguide) preparada como descrito na etapa 4.4.4. Alimente-os 1M sacarose após a injeção. Solte abelhas de seus suportes para a caixa branca (condição experimental) ou a caixa preta (controle) preparada conforme descrito na seção 5.1.
- Coloque as caixas em um recipiente de poliestireno com papel molhado dentro para umidade. Deixe as abelhas por 48 h e observe 2x por dia para garantir que elas tenham comida suficiente e boa umidade.
- Disseque o cérebro de cada abelha após 48 h (passo 3.1) e processo de imunocitoquímica conforme descrito na seção 3. Para imunostainamentos anti-mGlutR1 use a etapa 3.11 e para imunostainamentos anti-RDL use a etapa 3.12.
- Realizar imagens confocais para avaliar o nível de fluorescência nas seções cerebrais manchadas imunológicas.
- Para avaliar a redução da proteína em cérebros imunorotulados, use a coleta de imagens confocais no mesmo nível de ganho tanto para o controle quanto para cérebros injetados.
6. qPCR-based Drop-off Ensaio para avaliar o DNA RDL genômica modificado 48 H após injeção CRISPR Cas9 RNPRDLmix
- Projete os primers, a sonda de controle e a sonda de entrega para avaliar a quantidade de DNA modificada pela injeção CRISPR-Cas9. Cada primer é projetado para que ele flanqueie pelo menos um guia CRISPR-Cas 9 e o tamanho do amplicon é de 132 bp. As cartilhas e sondas utilizadas são dadas abaixo:
RDL1_For: CTCGGAGTGACCACCGT
RDL1_Rev: CAACGAGGCGAACACCAT
RDL1_control_probe: /5HEX/CGA+C+G+TTT+A+C+CT/3IABkFQ/
RDL1_Drop-off_probe: /56-FAM/CCTA+C+G+T+CA+A+GT/3IABkFQ/- Certifique-se de que os primers correspondam a sequências únicas que são específicas para a área. Certifique-se de que a sonda de entrega seja projetada para a área que se sobrepõe ao guia RDL-CRISPR-Cas9.
- Para testar se há uma modificação do gDNA na área de guia, injete 12 abelhas no ocelli com a mistura RNP_RDL descrita na etapa 5.3.1 e use oito abelhas não injetadas como controles.
- Disseque os cérebros das abelhas sem os lóbulos ópticos 48 h após as injeções e extraia o gDNA de cada cérebro injetado e controle de abelhas (sem lóbulos ópticos) usando o kit seguindo o protocolo do fabricante (ver Tabela de Materiais).
- Avalie a quantidade, qualidade e pureza do gDNA extraído usando um espectrofotômetro.
- Quantifique a expressão relativa de gDNA modificado da AmRDL usando o protocolo de entrega baseado em qPCR e o ciclor PCR em tempo real.
- Resuspender os oligos para 100 μM, diluir os primers a 10 μM, e as sondas para 5 μM.
- Configure a reação pcr na placa de 96 poços, como mostrado aqui para uma amostra de gDNA (3 repetições): 10 μL de mix mestre (ver Tabela de Materiais),1 μL de primer dianteiro, 1 μL de primer reverso, 1 μL de sonda de controle, 1 μL de sonda de entrega, 2 μL de gDNA (50 ng), 4 μL de água livre de nuclease para trazer a reação final a 20 μL.
NOTA: Os controles são a solução em vez das amostras e da água em vez das sondas. - Configure o programa de ciclismo da seguinte forma para uma motodor PCR em tempo real: 95 °C por 3 min seguido por 40 ciclos de 95 °C para 15 s, 60 °C para 1 min.
- Avalie a modificação genética relativa usando o método 2-ΔΔCt, onde
 E
E
7. Quantificação relativa do RNA RDL 48 H após injeção de RNPRDLmix
- Para testar se houver uma redução do RDL mRNA, injete 20 abelhas no ocelli com RNP_RDL mistura conforme descrito na etapa 5.3.1 e use 12 abelhas não injetadas como controles. Dissecem os cérebros das abelhas (sem lóbulos ópticos) 48 h após as injeções, extraem o mRNA total de cada abelha injetada e se separem usando o protocolo do fabricante (ver Tabela de Materiais).
- Remova qualquer resíduo de DNA restante na amostra usando um kit sem DNA (ver Tabela de Materiais).
- Avalie a qualidade e a pureza do RNA extraído usando um espectrômetro.
- Quantifique a expressão da AmRDL usando um kit RT-PCR verde fluorescente disponível comercialmenteemuma motocicleta PCR em tempo real usando o protocolo do fabricante para uma placa de poço 384.
NOTA: Neste experimento, foram utilizadas cartilha siciadas anteriormente. Para AmRDL (AmRDL_F GGTCGATGGGCTACTACCTG; AmRDL_R Primers TCGATCGACTTGACGTAGGA)22 e actin como um gene de referência [AmActin_F TGCCAACACTGTCCTTCTG; AmActin_R GAATTGACCCACCAATCCA]23. A expressão genética relativa foi calculada usando o método 2-ΔΔCt (passo 6.6).
8. qPCR Ensaio de entrega baseado para avaliar o DNA genômico modificado 48 H após injeção RNPmGlutR1mix
- Projete as cartilhas, controle e sondas de entrega para avaliar a quantidade de DNA modificada pela injeção CRISPR-Cas9. Projete os primers para que flanqueiem pelo menos um guia CRISPR-Cas 9 e o tamanho do amplicon seja de 96 bp.
mGlutR1_For: GGTGAAACGACGACGGa
AmGlurR1_Rev: GGAGAGAGGGAGCGAGAA
AmGlurR1_control_probe: /5HEX/CGAGG+G+AAA+CGA+GT/3IABkFQ/
AmGlurR1_Drop-off_probe: /56-FAM/CGA+C+A+C+CG+TC/3IABkFQ/
NOTA: Certifique-se de que os primers correspondam a sequências únicas que são específicas para a área que deveria ter sido modificada. A sonda de entrega foi projetada para a área que se sobrepôs ao guia CRISPR-Cas9. - Para testar se há uma modificação do gDNA na área de guia, injete 12 abelhas no ocelli com RNP_ mix GlutR1 conforme descrito na etapa 5.3.1 e use oito abelhas não injetadas como controles.
- Disseque os cérebros das abelhas (sem lóbulos ópticos) 48 h após as injeções e extraia o gDNA de cada cérebro de abelha injetado e controle (sem lóbulos ópticos) usando o protocolo do fabricante (ver Tabela de Materiais).
- Avalie a quantidade, qualidade e pureza do gDNA extraído usando um espectrofotômetro.
- Quantifique a modificação relativa do gDNA AmGlutR1 usando o protocolo de entrega qPCR e para o ciclor PCR em tempo real conforme descrito na etapa 6.5.
- Avalie a modificação genética relativa usando o método 2-ΔΔCt, onde
 E
E
9. Quantificação do RNA mGlutR1 48 h Após injeção RNPmGlutR1mix
- Para testar se há uma redução no mGlutR1 RNA mRNA, injete seis abelhas no ocelli com a mistura RNP_RDL conforme descrito na etapa 5.3.2 e use seis abelhas não injetadas como controles. Dissecem os cérebros das abelhas (sem lóbulos ópticos) 48 h após as injeções, extraem o mRNA total de cada abelha injetada e se separem usando o protocolo do fabricante para isolamento de RNA (ver Tabela de Materiais).
- Remova qualquer resíduo de DNA restante na amostra usando um kit sem DNA (ver Tabela de Materiais).
- Avalie a qualidade e a pureza do RNA extraído usando um espectrômetro.
- Quantifique a expressão do mGlutR1 usando um kit SYBR Green RT-PCR (ver Tabela de Materiais)em uma motocicleta PCR em tempo real com o protocolo fornecido para o kit de poço384. Use os seguintes primers para mGlutR1 (mGlut_F CCTCCTCAACGTCTCCTTCATA; mGlut_R TGCCGTTGTGTTCCGATTT) e primers actin como gene de referência [AmActin_F TGCCAACACTGTCCTTCTG; AmActin_R GAATTGACCCACCAATCCA]. Calcule a expressão genética relativa usando o método 2-ΔΔCt (passo 8.6).
Resultados
Testes de anticorpos anti-RDL
Os anticorpos foram produzidos contra os conjugados de peptídeos RDL, como mostrado na Figura 1A. O primeiro passo para caracterizar os anticorpos anti-RDL é verificar o homogeneizado da proteína extraída do cérebro da abelha usando uma mancha ocidental com anticorpos anti-RDL e um anticorpo secundário igg de cabra rotulado por HRP(Figura 1A,inserção). Ambos os anticorpos anti-RDL reconheceram a banda localizada em ~50-60 kD (seta), correspondendo ao peso estimado das proteínas isoformes de subunidade RDL. Para demonstrar que anticorpos anti-RDL reconheceram o peptídeo nas fatias cerebrais, usamos um controle de preaversão(Figura 1B,C). Quando os anticorpos foram preincubados com peptídeos conjugados, a coloração na seção estava ausente. Isso demonstrou que os anticorpos anti-RDL reconheceram o peptídeo conjugado contra o qual foram levantados. Para demonstrar que os anticorpos anti-RDL reconhecem a proteína no tecido cerebral fixo, usamos o CRISPR-Cas9 para derrubar o gene RDL que produz a proteína RDL nas células. Figura 1D1-3, mostra controle seções cerebrais de abelhas frontals que foram rotuladas com anticorpos anti-RDL. Esta abelha não foi injetada com RDL-CRISPR-Cas9 RNP. Na Figura 1D1,anti-RDL rotula neuropilpiles na seção frontal do cérebro das abelhas. A mesma seção frontal na Figura 1D2 mostra a ausência de fluorescência da ATTO550, pois o complexo RDL-CRISPR-Cas9 não foi injetado.
A Figura 1E1-E3 mostra uma seção cerebral de uma abelha injetada com RDL-CRISPR-Cas 9 e depois processada com a mesma quantidade de anticorpos que o cérebro de controle na Figura 1D1-D3. A coloração anti-RDL foi significativamente reduzida em todo o cérebro 48 h após a injeção, e a distribuição da coloração ATTO550 no cérebro (Figura 1E2) mostra o sucesso das injeções RDL-CRISPR-Cas 9 no ocelli mediano da abelha. As múltiplas células dispersas do cérebro exibem ATTO550. A injeção bem sucedida de RDL-CRISPR-Cas 9 reduziu a expressão proteica em comparação com o controle (Figura 1E1-3). De oito cérebros de abelhas imunosmanchados, apenas um cérebro tinha um alto nível de distribuição do RDL-CRISPR-Cas9 nas células do corpo de cogumelo, protocerebrum e lobo antenal, enquanto outros cérebros tinham coloração celular com ATTO550 no calyx corporal de cogumelo, complexo central, mas não lobe antenal. É importante notar que nestas abelhas, a redução da imunocoloração anti-RDL não foi tão dramática quanto no cérebro mostrado na Figura 1E1.
Em seguida, para estimar o nível do gDNA RDL modificado nas abelhas 48 h após a injeção RDL-CRISPR-Cas9, realizamos um teste de entrega qPCR, onde a sonda de entrega foi projetada para combinar com a área de um dos gRNAs RDL. Nesses experimentos, nos cérebros das abelhas injetados com RDL-CRISPR-Cas 9, a redução relativa da fluorescência correspondia ao número de gDNA modificado nas amostras. Em nossos testes, a área correspondente a este guia no gDNA em 12 abelhas injetadas com RDL-CRISPR-Cas9 foram de 64 % ± (média ± 30 %SD) em comparação com gDNA em abelhas não injetadas(Figura 3A).
Em seguida, para estimar o nível do RNA RDL nas abelhas 48 h após a injeção, realizamos qRT-PCR em um grupo separado de abelhas(Figura 3B). Comparamos o nível de RNA RDL de abelhas injetadas RDL-CRISPR-Cas 9 (n = 19) com o nível de RNA em abelhas que não foram injetadas (n = 12). Nesses experimentos, a redução relativa do RDL mRNA foi de 59% ±(média ± 15% SE) em comparação com o nível de RNA em abelhas não injetadas. Quando examinamos o nível de RNA RDL em cada abelha individualmente, apenas 13 abelhas em 19 abelhas apresentaram uma redução significativa do RNA. Esses dados indicam que a injeção de abelhas injetadas RDL-CRISPR-Cas9 através do ocelli pode nem sempre atingir um grande número de células cerebrais, o que confirma os dados com abelhas injetadas RDL-CRISPR-Cas9 manchadas de RDL-CRISPR-Cas9. Nestas preparações, apenas uma abelha de 8 tinha RDL CRISP-Cas9 em muitas células cerebrais (corpo de cogumelo, protocerebrum e lobo antenal) em comparação com outros cérebros de abelhas, onde a distribuição do RDL-CRISPR-Cas9 estava concentrada em células no protocerebrum (calyx corpo de cogumelo e complexo central), mas não o lóbulo antenal(Figura 4A-D).
Testes anticorpos anti-mglutr1
Usamos os anticorpos anti-mGlutR1 produzidos em coelho contra peptídeos conjugados específicos do melanogaster Drosophila (Figura 2A). A sequência deste peptídeo mostra uma identidade de 94% com o peptídeo de abelha (CLSDKTRFDYFARTVPPD) Figura 2A. Primeiro, verificamos os anticorpos contra a proteína cerebral das abelhas usando imunocoagulante. O cérebro das abelhas homogeneado foi separado por 10% SDS-PAGE e eletrophoreticamente transferido para uma membrana de nitrocelulose e manchado com anti-mGlutR1. A inserção na Figura 2A mostra duas bandas com pesos estimados (103 e 83 kD) correspondentes a duas isoformas. Quando testamos este anticorpo em cérebros de abelhas, descobrimos que eles rotulam perfis neuropilar e células nas seções cerebrais das abelhas como ilustrado na Figura 2B,D. Após a preacisão do anticorpo anti-mGlutR1 com peptídeo conjugado-mGlutR1, a coloração específica desapareceu na fatia cerebral das abelhas(Figura 2C). Isso confirma que anticorpos anti-mGlutR1 reconhecem o peptídeo (Figura 2C). Em seguida, injetamos uma mistura de mGlutR1-CRISPR-Cas9 no ocelli mediano e usamos o noguideRNA de controle. Nas abelhas de controle (n = 7), a fluorescência da ATTO550 não estava concentrada nas células. Alguns cérebros tinham fluorescência dispersa etiquetando ATTO550. Assim, a preparação de controle na Figura 2D1-3 mostra a coloração anti-mGlutR1 no cérebro, mas não a fluorescência ATTO550. Quando mGlutR1-CRISPR-Cas9 foi injetado no ocelli e tomado por muitas células, o nível de fluorescência dos anticorpos secundários foi significativamente reduzido na área que capta o mGlutR1RNP funcional (Figura 2E1-3). As abelhas foram monitoradas por 48 h, e uma abelha de cada condição experimental foi encontrada morta. Assim, neste experimento, verificamos sete abelhas de controle e oito abelhas CRISPR-Cas9. Todas as abelhas injetadas com CRISPR-Cas9 tinham células que atiravam mGlutR1-CRISPR-Cas9. A maioria dessas células estavam no calyx do corpo de cogumelo, complexo central e protocerebrum posterior. Apenas duas em cada sete apresentaram rotulagem ATTO550 em muitas células do corpo de cogumelo, complexo central e o lobo antenal. Um exemplo de uma dessas abelhas é mostrado na Figura 2E. A redução do nível de coloração mGlutR1 nestas preparações foi significativa. As outras cinco abelhas têm rotulagem ATTO550 correspondente à entrega bem sucedida do mGlutR1 CRISPR-Cas9 no corpo do cogumelo e protocerebrum posterior, mas não nos lóbulos antenais.
Em seguida, para estimar o nível do gDNA mGlutR1 modificado nas abelhas 48 h após a injeção, realizamos um teste de entrega baseado em qPCR, onde a sonda de entrega foi projetada para estar na área perto do guia mGlutR1. Nesses experimentos, nos cérebros das abelhas injetados com mGlutR1-CRISPR-Cas 9, a modificação relativa do gDNA em 12 abelhas foi de 59% ± (média ± 33 %SD) em comparação com gDNA em abelhas não injetadas(Figura 3A).
Esses resultados também foram confirmados por testes qRT-PCR em um grupo diferente de abelhas, onde estimamos os níveis de RNA mGlutR1 usando qRT-PCR nas abelhas 48 h após injeções com RNPmGlutR1mix(Figura 3B). Comparamos o nível de RNA mGlutR1 das abelhas injetadas mGlutR1-CRISPR-Cas9 (n = 6) com os níveis de RNA em abelhas que não foram injetadas (n = 6). Nesses experimentos, a redução relativa do mGlutR1 mGlutR1 em abelhas injetadas foi de 53% ±(média ± 18% SE) em comparação com abelhas não injetadas(Figura 3B).
A seção de quatro abelhas diferentes que expressavam o RNP RDL-CRISPR-Cas9 na célula kenyon de corpo de cogumelo é mostrada na Figura 4A-D. O exemplo de abelha com fluorescência ATTO50 no corpo do cogumelo e o lobo antenal é mostrado na Figura 4E,F.
| Guias | Seqüências | gRNA | Rnp |
| [guideRNA:tracrRNA] | [gRNA:Cas9 Nuclease] | ||
| RDL_Guide1 | ACCGTAACGCGCCGCGCT | GRDL1 | RRDL1 |
| RDL_Guide2 | AACGTCGATCGACTTGACGT | GRDL2 | RRDL2 |
| RDL_Guide3 | CCATGACGAAAACACGTGCCC | GRDL3 | RRDL3 |
| mGlu_Guide 1 | CGAAAGTTATCTGACGGTGTGTGT | GMGL1 | RMGL1 |
| mGlu_Guide 2 | TTCAACGAGAGCAAAGTTCAT | GMGL2 | RMGL2 |
| mGlu_Guide 3 | GCAAACGTCGGTAGGAGTGA | GMGL3 | RMGL3 |
Tabela 1: Sequências nucleotídeas de guias projetados para RDL e mGlutR1.
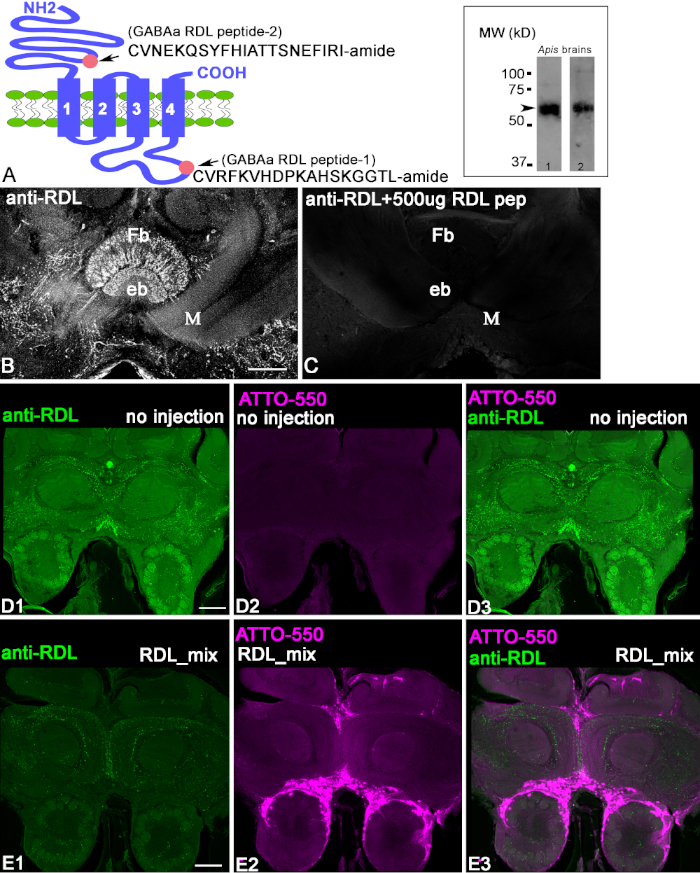
Figura 1: Caracterização de anticorpos anti-RDL. (A)Esquemática da subunidade RDL, onde os círculos rosas indicam a localização do peptídeo 2 (cvnekqsyfhiattsnefiri-amide) no N-terminus e peptídeo 1 (CVRFKVHDPKAHSKGGTL-amide intracelular) no Terminal C. A inserção em A mostra as bandas na mancha ocidental de extratos cerebrais de abelhas processados com anticorpos anti-RDL correspondentes (um com pep1 anti-RDL e pep2 anti-RDL). Cada imunoblot mostra o tamanho aparente da proteína ~50-60 kD, correspondendo aos pesos estimados das várias isoformes das subunidades RDL. (B,C) Preaferionação dos anticorpos anti-RDL com peptídeo conjugado 1. A imagem em C mostra uma redução na coloração na seção quando os anticorpos foram preincubados com peptídeo conjugado 1. O corpo em forma de ventilador (Fb) e o corpo elipsoide (eb) são estruturas complexas centrais no cérebro. M = lobo medial do corpo de cogumelo. (D1-3) A coloração anti-RDL de um controle, seção de cérebro de abelha não injetada após 48 h. Green indica o perfil positivo anti-RDL no cérebro. (D2) Esta abelha não foi injetada e não contém fluorescência ATTO550. (D3) Imagens mescladas de D1 e D3. (E1-3) A injeção de RDL-CRISPR-Cas9 reduziu a coloração anti-RDL após 48 h. (E2) fluorescência ATTO550 nos núcleos celulares indicou a entrega bem sucedida RDL-CRISPR-Cas9. (E3) A imagem mesclada de anti-RDL (verde) e ATTO550 (vermelho). Barra de escala = 100 μm (B-E). Clique aqui para ver uma versão maior deste valor.
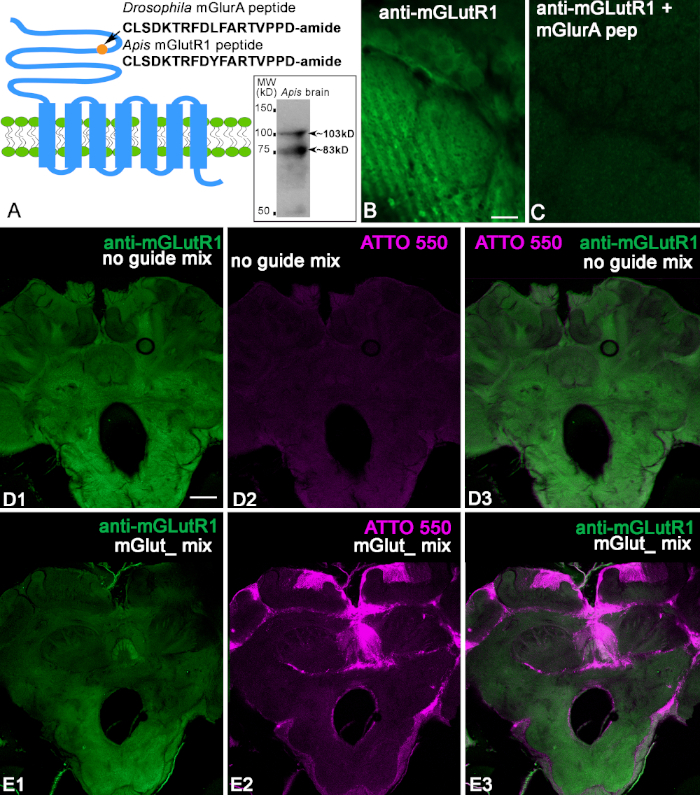
Figura 2: Caracterização de anticorpos anti-mGlutR1. (A)Esquema de GCPR mGluR que mostra o peptídeo de melanogaster Drosophila usado para imunização. Para comparação, o peptídeo Apis mellifera é mostrado abaixo. O círculo indica a localização do peptídeo no terminal N do domínio extracelular do receptor mGlutR1. A inserção em A mostra que os anticorpos anti-mGlutR1 reconheceram duas bandas na mancha ocidental de cérebros de abelhas ~103 kD e ~83 kD que correspondem aos pesos estimados de isoformas conhecidas. (B,C) Controle de preadição do anticorpo anti-mGlutR1 em duas seções consecutivas do lobo-antena glomeruli. A imagem do anti-mGlutR1 na seção glomeruli antenal em C mostra a redução da coloração como resultado da preincubação do peptídeo anti-mGlutR1 com o anticorpo anti-mGlutR1. Esse procedimento faz com que o anticorpo se precipitasse fora da solução, o que abolia a coloração em comparação com B (controle, ausência do peptídeo na preincubação). (D1) mostra a coloração de anti-mGlutR1 em uma fatia cerebral de abelha após uma injeção de controle no ocelus mediano. Esta injeção não tinha o gRNA mGlutR1 que permite a derrubada de receptores mGlutR1 pelo CRISPR-Cas9, e assim a coloração do anticorpo anti-mGlutR1 não foi reduzida (verde). (D2) A ausência de fluorescência ATTO550 indica a ausência de CRISPR-Cas9 funcional no cérebro. (D3) Imagens mescladas de anti-mGlutR1 e ATTO550. (E1-E3) mostra a coloração de anti-mGlutR1 em uma seção cerebral onde mGlutR1 foi permanentemente derrubado 48 h após injeção com mGlutR1-CRISPR-Cas 9 no ocelus mediano. Assim, a coloração neste cérebro é muito reduzida devido ao nocaute bem sucedido do mGlutR1 em muitas células. (E2) A coloração ATTO550 em muitos núcleos celulares nos cérebros das abelhas indica que a injeção de mGlut1-CRISPR-Cas 9 foi bem sucedida. (E3) Imagem mesclada de ATTO550 (vermelho) e anti-mGlutR1 (verde). Barra de escala = 10 μm (B,C); 100 μm (D,E). Clique aqui para ver uma versão maior deste valor.
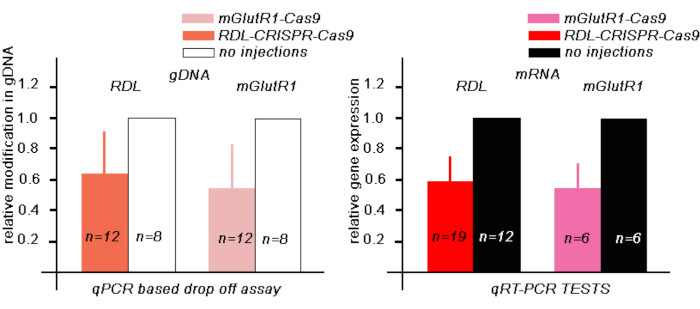
Figura 3: Avaliação de gDNA modificado e expressão de mRNA de RDL e mGlutR1 mRNA em cérebros de abelha48 h após injeção com 345 nL de RPN CRISPR-Cas9 correspondente. (A)O teste de ensaio baseado em qPCR para avaliar a quantidade de gDNA com uma área modificada foi calculado usando o método 2-ΔΔCt e normalizado contra o controle, cérebros não injetados. Os dados são expressos como média + SD . (B) O teste TheqRT-PCR foi usado para avaliar a quantidade de mRNA em abelhas injetadas e não injetadas crispr-cas9. Amactin foi usado como um gene de referência. A expressão genética relativa foi calculada usando 2 métodos-ΔΔCt e normalizada contra o controle, cérebros não injetados. Os dados são expressos como média + SE. Clique aqui para ver uma versão maior deste valor.
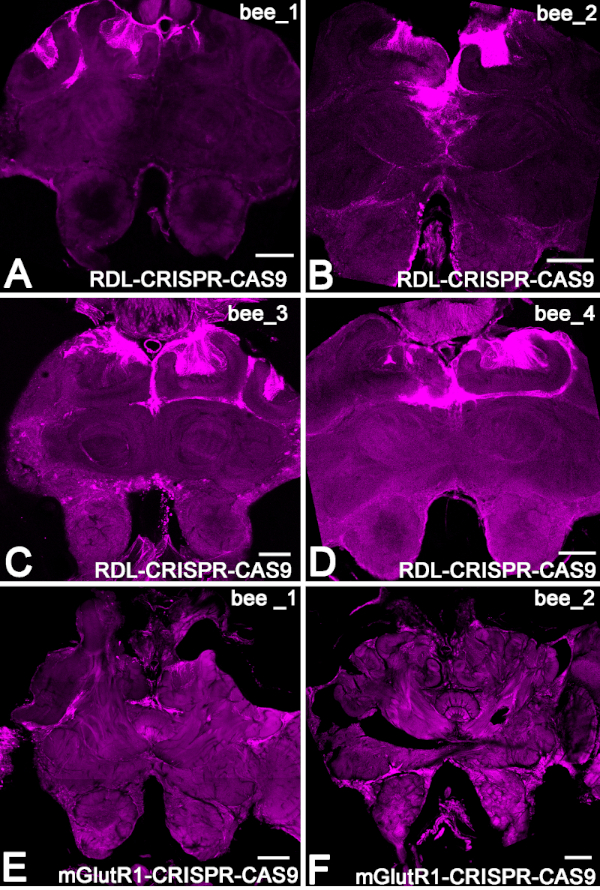
Figura 4: Exemplo da distribuição de RNP CRISPR-Cas9 em cérebros de abelhas via fluorescência ATTO550. (A-D) As seções cerebrais de quatro abelhas diferentes que expressaram o RNP RDL-CRISPR-Cas9 na célula Kenyon do corpo de cogumelo. (E,F) Exemplo de dois cérebros 48 h após injeção com RNPmGlutR1 CRISPR-Cas9. Barra de escala = 150 μm (A-F). Clique aqui para ver uma versão maior deste valor.
Discussão
Caracterização do anti-RDL e anti-mGlutR1
Primeiro, caracterizamos os anticorpos anti-RDL e anti-mGlutR1 por imunoblote e pré-adsorção nas fatias de cérebros fixos de abelhas. Cada anticorpo foi feito para reconhecer todas as suas isoformas conhecidas, e a análise ocidental mostra que eles reconhecem bandas que correspondem aos seus pesos moleculares previstos. Em seguida, ambos os anticorpos foram bloqueados pelo peptídeo conjugado contra o qual foram produzidos em seções cerebrais de abelhas.
Um dos primeiros objetivos em nosso estudo foi estabelecer que os anticorpos produzidos contra o peptídeo conjugado específico são específicos para sua proteína no tecido cerebral fixo. Para isso, aproveitamos o sistema CRISPR-Cas9. Projetamos guias específicos para rdl de abelhas e mGlutR1 e usamos cada um deles para fazer CRISPR-Cas9 rotulado com a sonda fluorescente ATTO550. Para cada receptor, injetamos uma mistura de três ribonucleoproteínas CRISPR-Cas9 diferentes no ocelli para reduzir a quantidade da proteína direcionada no cérebro de abelha adulta, eliminando o gene correspondente em células que tomaram nosso sistema Cas9 projetado. Em nosso estudo, realizamos esse passo.
Um dos primeiros passos cruciais para o sucesso desses experimentos é projetar o guia apropriado RNAs. Recomendamos projetar até cinco RNAs guias, localizados no início, meio e o fim da sequência genética. Em nosso trabalho preliminar, testamos em várias combinações em três a cinco abelhas. Também tentamos diferentes concentrações de injeções, bem como tempos após a injeção, e várias misturas de RNP nas injeções. Dissecamos cérebros e os processamos usando anticorpos anti-RDL e anti-mGlutR1. Nestes testes iniciais, estabelecemos a combinação apropriada, o tempo pós-injeção, bem como a concentração e quantidade de CRISPR-Cas9 para injeção. Estes testes iniciais foram a base para a criação dos experimentos que descrevemos em detalhes aqui.
O objetivo era duas vezes: 1) demonstrar em uma abelha que nossa coloração de anticorpos foi reduzida após o tratamento com CRISPR-Cas9 e 2) para trabalhar as melhores condições experimentais para estudos comportamentais. Assim, mostramos que se muitos núcleos celulares contiverem crispr-cas9 48 h após a injeção, a redução da coloração anti-RDL e anti-mGlutR1 é significativa. Além disso, isso demonstra que os anticorpos testados reconhecem especificamente a proteína mGlut1 e RDL na preparação cerebral da abelha e que podem ser usadas para estudos de localização no cérebro de abelhas.
Configuração experimental CRISPR-Cas9 para estudo comportamental
Em seguida, montamos os experimentos para que o CRISPR-Cas9 pudesse ser usado em estudos comportamentais. Oito ou nove abelhas foram coletadas para controle e tratamentos experimentais. Eles foram testados comportamentalmente antes e depois da injeção, e então seus cérebros foram processados para ATTO550 e/ou imunocitoquímica para determinar as regiões cerebrais que mostraram redução da proteína-alvo. Aqui é essencial notar que o número de abelhas levadas para um conjunto de experimentos foi limitado a não mais do que 8-9 abelhas para o controle e condições experimentais. Desta forma, ambas as condições podem ser testadas no mesmo dia. Além disso, uma vez que preparamos as misturas CRISPR-Cas9 para injeção, nunca as congelamos. A mistura CRISPR-Cas9 não mudou de potência quando usada 3 dias seguidos e mantida em 4-8 °C. No entanto, não o testamos depois de 3 dias.
Como descrevemos na seção resultados para ambos os conjuntos de injeções experimentais e para ambos os anticorpos, apenas três abelhas de 16 testadas mostraram uma grande distribuição ATTO550 no corpo de cogumelos, protocerebrum e lóbulos antenais. Em todas as outras abelhas, a distribuição do CRISPR-Cas9 foi limitada ao corpo de cogumelo, complexo central e/ou protocerebrum posterior. É essencial entender para qualquer estudo comportamental que usando este método de injeção a redução da proteína-alvo será restrita apenas ao corpo de cogumelo na maioria das abelhas. Não se estenderá ao lóbulo antena ou gânglio subesofágico. Assim, a técnica de injeção que usamos é adequada para estudar o efeito da redução de receptores no corpo de cogumelo e complexo central em experimentos comportamentais, enquanto um método diferente de introdução do CRISPR-Cas9 será mais apropriado para estudar outras regiões cerebrais.
Em conclusão, nosso estudo demonstrou a aplicação bem sucedida do CRISPR-Cas9 como um controle para a coloração de anticorpos no cérebro. Para ambos os anticorpos (anti-RDL e anti-mGlutR1), quando a captação de mGlutR1-CRISPR-Cas9 ou RDL-CRISPR-Cas9 foi bem sucedida, o nível de coloração de anticorpos correspondente também foi reduzido significativamente. Além disso, é essencial notar que a injeção no ocelli levou a uma distribuição de CRISPR-Cas9 no cérebro que não era homogênea. A distribuição variou de uma área mínima ao redor do corpo de ocelli e cogumelo até muitas células em todo o cérebro. A variabilidade da captação mGlutR1 ou RDL-CRISPR-Cas9 pelas células provavelmente deveu-se à variação das injeções. Nossos dados mostram que o sistema CRISPR-Cas9 funciona em abelhas, mas o método de injeção precisa ser melhorado para reduzir a variabilidade da captação CRISPR-Cas9 em cérebros individuais de abelhas. Dentro dessas restrições, agora é possível empregar essa técnica para manipular genes em abelhas adultas para experimentos comportamentais.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi apoiado pelos seguintes prêmios à BHS: Human Frontiers Science Program; NIH NIGMS (R01 GM113967); NsF Ideas Lab (1556337). O peptídeo e os anticorpos do DmGluRA foram projetados no laboratório Dr. Serge Birman (Marselha, Luminy, França) quando o EI foi apoiado pelo Programa d'Urgence FRM/Postdocs UFP20060306548 do Derramamento de Fondation la Recherche Medicale. Somos gratos por Daniela Junqueira Marosi e Alex Hanter, da Integrated DNA Technology (IDT), pela ajuda com o design de guias RDL e ensaios de entrega qPCR.
Materiais
| Name | Company | Catalog Number | Comments |
| Acetic Acid | Sigma-Aldrich | A6283_100 mL | western blotting |
| Acrylamide-bis Acrylamide | Bio-Rad | 500 mL 1610156 | western blotting |
| Agarose | Sigma-Aldrich | A0169-250g | used with distilled water to fix honey bee brain in blocks |
| Alexa Fluor 488 AffiniPure F(ab')2 Fragment Donkey Anti-Rabbit IgG (H+L) | Jackson Research Laboratories | 711-546-152 | secondary antibody to reveal the primary antibodies from rabbit |
| Alt-R CRISPR-cas9 tracrRNA-5'ATTO | IDT | 1075934 | with desinged guide RNA, it creates gRNA |
| Alt-R S.p. Cas9 nuclease V3 | IDT | 1081058 | enzyme used to make ribonucleoprotein for CRISPR system |
| Ammonium Persulfate | Bio-RAD | 10 g 1610700 | western blotting |
| Anti-mGlutR1 antibodies | Covalab (FRANCE)/21st Century Biochemical | characterized by authors in present paper | Used for primary incubation of mGlut 1 in honey bees |
| Anti-RDL antibodies | 21st Century Biochemical | characterized by authors in present paper | Used for primary incubation of RDL in honey bees |
| Aprotinin | Sigma-Aldrich | Y0001154 | protease inhibitor |
| Barraquer Iris Scissors 7mm Blade Sharp point | World Precision Instruments | 14128-G | used for dissection honey bee brain |
| Benzamidine | Sigma-Aldrich | 12072 | protease inhibitor |
| Blade (breakable) for blade holder | Fine Science Tool | 10050-00 | dissection for western blotting |
| Blade holder and breaker | Fine Science Tool | 15309 | dissection for western blotting |
| Borosilicate glass capillaries | World Precision Instruments | 1B100 F | for injection procedure |
| Chemiluminescent western blot detection substrate | Bio-Rad | 1705062 | western blotting |
| Chloroform | Sigma-Aldrich | 472476-50mL | RNA isolation |
| Dithiothreitol | Bio-Rad | 1610611 | western blotting |
| DNA easy kit | QIAGEN | 69504 | DNA extractionuj |
| DNA-free kit | INVITROGEN | AM1907 | Used to remove DNA |
| Eppendorf Research plus pipette, 3-pack | Sigma-Aldrich | Z683884 | |
| Falcon 24 Well Polystyrene Multiwell | Falcon | 351147 | Multiwell |
| Flat Bottom Embedding Capsules, Polyethylene | Electron Microscopy Science | 70021 | basket for brain sections |
| Forceps Dumont #5 (pair) | Fine Science Tool | 11254-20 | for dissection of honey bee brain from the head |
| Forceps Dumont #5S (pair) | Fine Science Tool | 11252-00 | to clean up the brain from trachea befor dissection |
| Gene Expression Master Mix | Integrated DNA Technologies | 1055770 | qPCR drop off assay |
| Glutaraldehyde | EMS | 16220 | used for preparation of peptide conjugates for control peabsorption |
| Glycerol | Sigma-Aldrich | G5516-500 mL | western blotting, embedding media |
| Glycine | Bio-Rad | 1610718 | western blotting |
| Hydrophobic filtered nylonmesh | Spectrum Labs | 145910 | for bottom of basket for brain sections |
| Isopropyl alcohol | Sigma-Aldrich | I9030-50mL | RNA isolation |
| Keyhole limpet hemocyanin | Sigma-Aldrich | H7017 | used for preparation of conjugates for control |
| LSM800 cofocal microscope | Zeiss | ||
| mGlu_Guide 1 | IDT | designed guide RNA for CRISPR-Cas 9. Target mGlutR1 genome sequences | |
| mGlu_Guide 2 | IDT | designed guide RNA for CRISPR-Cas 9. Target mGlutR1 genome sequences | |
| mGlu_Guide 3 | IDT | designed guide RNA for CRISPR. Target mGlutR1 genome sequences | |
| methanol | Sigma-Aldrich | 34860 | western blotting |
| 96-well PCR microplate | Applied biosystem | 4346907 | qPCR drop off assay and qRT-PCR |
| 384-well PCR microplate | Sigma-Aldrich | Z374911 | qRT-PCR |
| Mineral oil | Sigma-Aldrich | M5904_500mL | for injection procedure |
| Model p87 Flaming Brown Micropipette Puller | Sutter Instrument Co. | Capillary Preparation | |
| Mowiol 4-88 | Sigma-Aldrich | 81381 | for embedding solution |
| Nanoliter 2000 | World Precision Instruments Nanoliter | Injection Apparatus | |
| Normal Donkey Serum | Jackson Research Laboratories | AB_2337258 | blocking agent for immunocytochemistry |
| Non-immune Goat Serum | Invitrogen | 50-062Z 100 mL | blocking agent for immunoblotting |
| Nuclease-free buffer | IDT | 1072570 | Nuclease-free buffer that is used in the preparation of CRISPR-Cas9 injection |
| Nuclease-free water | IDT | AM9337 | nuclease -free water that is used in preparation of CRISPR-Cas 9 system |
| OrbitalShaker Mp4 | Genemate | ||
| Paraformaldehyde | Sigma-Aldrich | 158127-500 g | used with PBS to make fixative for bee brains |
| Phenylmethylsulphonyl fluoride (PMSF) | Sigma-Aldrich | P7626-250 mg | protease inhibitor |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417-100TAB | Used with Paraformaldehyde as fixative; buffer for antibodies |
Referências
- Harms, D. W., et al. Mouse Genome Editing Using the CRISPR/Cas System. Current Protocols in Human Genetics. 83 (15), 7-27 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Kouranova, E., et al. CRISPRs for Optimal Targeting: Delivery of CRISPR Components as DNA, RNA, and Protein into Cultured Cells and Single-Cell Embryos. Human Gene Therapy. 27 (6), 464-475 (2016).
- Schubert, M., et al. Fluorescently labeled tracrRNA provides efficient genome editing while allowing cellular microscopy and FACS analysis. Genome Editing. , 1-3 (2017).
- Kucharski, R., Mitri, C., Grau, Y., Maleszka, R. Characterization of a metabotropic glutamate receptor in the honeybee (Apis mellifera): implications for memory formation. Invertebrate Neuroscience. 7 (2), 99-108 (2007).
- Aronstein, K., Auld, V., Ffrench-Constant, R. Distribution of two GABA receptor-like subunits in the Drosophila CNS. Invertebrate Neuroscience. 2 (2), 115-120 (1996).
- Thompson, M., Steichen, J. C., ffrench-Constant, R. H. Conservation of cyclodiene insecticide resistance-associated mutations in insects. Insect Molecular Biology. 2 (3), 149-154 (1993).
- Chung, B. Y., Kilman, V. L., Keath, J. R., Pitman, J. L., Allada, R. The GABA(A) receptor RDL acts in peptidergic PDF neurons to promote sleep in Drosophila. Current Biology. 19 (5), 386-390 (2009).
- Taylor-Wells, J., Hawkins, J., Colombo, C., Bermudez, I., Jones, A. K. Cloning and functional expression of intracellular loop variants of the honeybee (Apis mellifera) RDL GABA receptor. Neurotoxicology. 60, 207-213 (2017).
- Jones, A. K., Sattelle, D. B. The cys-loop ligand-gated ion channel superfamily of the honeybee, Apis mellifera. Invertebrate Neuroscience. 6 (3), 123-132 (2006).
- Dupuis, J. P., et al. Homomeric RDL and heteromeric RDL/LCCH3 GABA receptors in the honeybee antennal lobes: two candidates for inhibitory transmission in olfactory processing. Journal of Neurophysiology. 103 (1), 458-468 (2010).
- Farooqui, T., Robinson, K., Vaessin, H., Smith, B. H. Modulation of early olfactory processing by an octopaminergic reinforcement pathway in the honeybee. Journal of Neuroscience. 23 (12), 5370-5380 (2003).
- Guo, X., Wang, Y., Sinakevitch, I., Lei, H., Smith, B. H. Comparison of RNAi knockdown effect of tyramine receptor 1 induced by dsRNA and siRNA in brains of the honeybee, Apis mellifera. Journal of Insect Physiology. 111, 47-52 (2018).
- Roth, A., et al. A genetic switch for worker nutrition-mediated traits in honeybees. PLOS Biology. 17 (3), e3000171 (2019).
- Kohno, H., Suenami, S., Takeuchi, H., Sasaki, T., Kubo, T. Production of Knockout Mutants by CRISPR/Cas9 in the European Honeybee, Apis mellifera L. Zoological Science (BIOONE). 33 (5), 505-512 (2016).
- Hu, X. F., Zhang, B., Liao, C. H., Zeng, Z. J. High-Efficiency CRISPR/Cas9-Mediated Gene Editing in Honeybee Apis mellifera Embryos. G3: Genes, Genomes, Genetics. 9 (5), 1759-1766 (2019).
- Smith, B. H., Burden, C. M. A Proboscis Extension Response Protocol for Investigating Behavioral Plasticity in Insects: Application to Basic, Biomedical, and Agricultural Research. Journal of Visualized Experiments. (91), e51057 (2014).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Rodriguez, J., Deinhardt, F. Preparation of a semipermanent mounting medium for fluorescent antibody studies. Virology. 12, 316-317 (1960).
- . Custom Alt-R® CRISPR-Cas9 guide RNA Available from: https://www.idtdna.com/site/order/designtool/index/CRISPR_CUSTOM (2019)
- Bonnafe, E., et al. Effect of a thymol application on olfactory memory and gene expression levels in the brain of the honeybee Apis mellifera. Environmental Science and Pollutant Research (International). 22 (11), 8022-8030 (2015).
- Wang, Y., et al. Regulation of behaviorally associated gene networks in worker honeybee ovaries. Journal of Experimental Biology. 215 (Pt 1), 124-134 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados