Method Article
Test anticorpale sui recettori anti-RDL e anti-mGlutR1 nelle sezioni del cervello di Honeybee utilizzando CRISPR-Cas9
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Presentato qui è un protocollo per utilizzare il sistema CRISPR-Cas9 per ridurre la produzione di una proteina nel cervello delle api adulte per testare la specificità degli anticorpi.
Abstract
Cluster Regolarmente Interspaced Short Palindromic Repeats (CRISPR)/CRISPR-associated protein 9 (Cas9) è una tecnica di editing genico ampiamente utilizzata negli studi sulla funzione genica. Usiamo questo metodo in questo studio per verificare la specificità degli anticorpi sviluppati contro l'insetto GABAA recettore Resistenza alla dieldrina (RDL) e un recettore del glutammato metabotropico mGlutR1 (mGluRA). Gli anticorpi sono stati generati nei conigli contro i peptidi coniugati specifici per i moscerini della frutta (Drosophila melanogaster) e alle api melappe (Apis mellifera). Abbiamo usato questi anticorpi nelle sezioni del cervello delle api per studiare la distribuzione dei recettori nel cervello delle api. Gli anticorpi sono stati purificati contro il peptide e testati con immunoblotting e il metodo classico di preadsorption con peptidi coniugato per dimostrare che gli anticorpi sono specifici per il corrispondente peptide coniugati contro cui sono stati sollevati. Qui abbiamo sviluppato la tecnica CRISPR-Cas9 per testare la riduzione dei bersagli proteici nel cervello 48 h dopo l'iniezione CRISPR-Cas9 con RNA guida progettati per il recettore corrispondente. Il metodo CRISPR-Cas9 può essere utilizzato anche nelle analisi comportamentali nelle api adulte quando uno o più geni devono essere modificati.
Introduzione
Il sistema CRISPR/Cas9 recentemente scoperto è un potente strumento che è stato utilizzato per modificare il DNA genomico in vari sistemi modello e organismi. Ha accelerato la ricerca biomedica e importanti scoperte tecnologiche rendendo la modificazione del genoma più efficiente e robusta rispetto ai metodi precedenti1. Originario dei batteri S. pyogenes, il sistema si basa su un endonuclease Cas9, la cui attività porta a rotture a doppio filamento (DSB) nel DNA e a un RNA guida (gRNA) che indirizza la proteina Cas9 verso una posizione specifica dipendente dalla sequenza2. Le rotture a doppio filamento generate da CRISPR/Cas9 possono essere riparate tramite l'unione finale non omologa (NHEJ), un processo soggetto a errori che può portare a frameshift, o riparazione diretta omemologia quando è presente un modello di donatore. Il gRNA stesso è costituito da un RNA CRISPR (crRNA) specifico del bersaglio e da un crRNA transattivauniversale universale (tracrRNA) che può essere sintetizzato chimicamente e consegnato con nuclease Cas9 purificata come complesso di ribonucleoproteina (RNP)2,3. L'etichettatura fluorescente del gRNA o della nuclea leva di Cas9 può consentire il rilevamento e la visualizzazione intracellulare di componenti molecolari tramite microscopia fluorescente4.
Nel nostro lavoro attuale, approfittiamo del sistema CRISPR-Cas9 per ridurre i livelli proteici nel cervello delle api adulte. Abbiamo studiato il recettore del glutammato metabotropico (mGluR) e gli anticorpi recettorianti anti-mGlutR1 e la sottounità del recettore GABAA RDL e anticorpi anti-RDL. Abbiamo sviluppato un metodo semplice per ridurre la quantità di proteine nel cervello dell'ape adulta e l'abbiamo usata per guidare ulteriori test degli anticorpi sviluppati contro le proteine corrispondenti. Il monitoraggio della fluorescenza di CRISPR-Cas9 ci ha permesso di stimare le aree e le cellule coinvolte nella riduzione della proteina.
Utilizzando questo metodo, abbiamo anche caratterizzato gli anticorpi anti-mGlutR1 che sono stati fatti nei conigli contro il peptide coniugato. Il genoma delle api codifica un AmGluRA altamente conservato (chiamato mGlutR1 secondo la nomenclatura NCBI) il recettore del glutammato metabotropico5. Il gene honeybee mGlutR1 ha quattro varianti di giunzione previste secondo il database NCBI. È stato riferito che è espresso nel sistema nervoso centrale (CNS) di entrambi gli stadi pupali e adulti delle api ed è coinvolto nella formazione della memoria a lungo termine5. Gli anticorpi sviluppati contro mGlutR1 possono essere uno strumento essenziale per studiare il sistema glutamategico nel processo di apprendimento e memoria nelle api.
Nei nostri studi, abbiamo anche caratterizzato anticorpi anti-RDL sviluppati in conigli immunizzati con peptidi coniugati dalla sottounità recettore Apis mellifera RDL. Il gene delle api melfale Rdl, AmRdl (XM_006565102.3, database NCBI), ha 14 varianti di giunzione previste. Un frammento parzialmente clonato è stato segnalato nel database NCBI AF094822.1. La funzione del recettore RDL e la sua fisiologia è ben studiata negli insetti6,7,8, comprese le api9,10,11. Anticorpi sviluppati contro anti-RDL può essere uno strumento essenziale per studiare il sistema GABAergic nel processo di apprendimento e memoria nelle api.
Uno studio precedente sul ruolo dei recettori della poltopamina e della tiramina ha usato RNAi iniettato nel cervello con un successivo test della quantità di proteine da macchia occidentale12,13. Tuttavia, l'RNAi presenta alcune limitazioni significative. C'è solo un breve lasso di tempo dopo l'iniezione di RNAi all'interno della quale si verifica una riduzione della proteina13. CRISPR-Cas9 è stato utilizzato molto recentemente negli embrioni di api per cancellare o modificare i geni in tutto l'animale14,15,16. Abbiamo segnalato l'uso di CRISPR-Cas9 per ridurre la quantità di proteine nell'ape adulta. Abbiamo sviluppato questo approccio per le api a causa della capacità di accoppiarlo a studi comportamentali di apprendimento e memoria in condizioni di laboratorio controllate17.
Nel presente lavoro, abbiamo sviluppato anticorpi contro due recettori e li abbiamo testati sulle sezioni adulte del cervello delle api dopo che la proteina è stata ridotta con l'iniezione CRISPR-Cas9. Allo stesso tempo, abbiamo stabilito un progetto sperimentale che permette l'uso del metodo per esperimenti comportamentali.
Protocollo
Il protocollo qui descritto segue le linee guida per la cura degli animali dell'Arizona State University.
1. Isolamento proteico totale dal cervello di Apis mellifera
NOTA: Utilizzare Apis mellifera Nuovo mondo Carniolan rinunciatori di età sconosciuta per questo esperimento.
- Posizionare uno schermo in rete in alluminio sopra l'ingresso dell'alveare per catturare le api forager17. Catturare ogni ape in una fiala con un piccolo foro in ogni tappo. Posizionare le fiale contenenti le api nel ghiaccio per abbassare la temperatura corporea e immobilizzarle. Lasciare le api nel ghiaccio per non più di 3 min.
- Fissare le api immobilizzate in supporti metallici precedentemente preparati. Assicurarsi che i supporti metallici siano costruiti in modo che l'ape possa essere fissata con piccoli pezzi di nastro adesivo, ma che abbiano ancora torace posteriore, ali e teste esposte.
AVVISO: Assicurarsi che le api siano completamente immobilizzate prima di tentare di posizionarle nei supporti. - Nutrire le api con una soluzione di saccarosio da 1 M utilizzando una siringa da 5 mL fino a quando non hanno più fame. Mettere le api fissate in una scatola con un tovagliolo di carta bagnato per garantire un ambiente umido.
- Dissezionare rapidamente il cervello dell'ape tagliando la testa con le forbici Barraquer Iris (vedi Tabella dei materiali)e utilizzare le forbici per aprire la testa dalla parte anteriore.
- Tagliare il cervello dalla capsula della testa, prendere il cervello con #5 forza, e metterlo in 100 . Il buffer di lisi è costituito da 120 mM Tris-HCl, 2% solfato di sodio dodecyl (SDS), 5% glicerolo, 0,2 mM dithiothreitol, 1% Triton X-100, e 1-5 g/mL degli inibitori della proteasi PMSF (phenylmethylsulphonylfluoride), aprotinin, benzamidine (pH 6,8) a 4 gradi centigradi. Omogeneizzare il cervello nella soluzione di lisi girando in un pestello per circa 2 min.
- Centrifugare il campione a 12.000 x g per 20 min. Aspirate 90 -L del supernatante e scartare il pellet.
- Prendere 1 lofile del supernatante per quantizzare la proteina totale utilizzando un fluorimetro. La concentrazione approssimativa della proteina totale era tra 2-3 mg/mL per ape. Prendere 10 l del supernatante e aggiungere 10 L del buffer di lisi e 10 l un buffer 6x Laemmli18. Girare brevemente e far bollire per 3 min, quindi raffreddare sul ghiaccio. Girare per 1 min a 10.000 x g per rimuovere tutti i detriti. Un decimo di un cervello d'api contiene circa 25 ng di proteine totali.
2. Ovest Blotting19
- Fare 30 mL di gel da corsa 10% contenente 12,15 mL di acqua distillata ultrapura, 7,5 mL di 1,5 M Tris-HCl (pH 8,8), 0,3 mL di 10% SDS, 10 mL di 30% soluzione acrilamide-bis acrilamide, 0,15 mL di 10% di ammonio persulfato (APS) e 20 l di tetrathylethylenedia (TEMED ).
- Lanciare il gel tra due lastre di vetro separate da distanziali.
- Crea un gel di impilamento da 20 mL contenente 12,1 mL di acqua distillata ultrapura, 5,0 mL di 0,5 M Tris-HCl (pH 6,8), 0,2 mL di 10% SDS, 2,6 mL di acrilammide acrilico, 0,1 mL del 10% APS e 20 gradi di TEMED.
- Quando il gel in esecuzione solidifica versare un gel di impilamento. Aggiungere con attenzione il separatore di plastica per lanciare la corsia di carico, evitando bolle. Attendere 15-30 min, fino a quando il gel è solidificato.
- Iniziare a caricare il gel utilizzando 5 - L di standard proteici. Caricare 20 l untempo della miscela di lisa da passo 1,8 per corsia, corrispondente a 1/15 dollari di un cervello d'api o 16 ng di proteine totali per corsia. Eseguire i campioni per 3,5-4 h totale a 16 mA nel gel impilamento e 32 mA nel gel in esecuzione. Fermati quando il colorante lascia il gel.
- Trasferire le proteine su membrane di nitrocellulosa nel buffer di trasferimento (25 mM MM Tris-HCl, 192 mM glicina, 15% di metanolo) a 0,45 mA per 1 h 30 min a 4 gradi centigradi.
- Per valutare l'efficienza del trasferimento di proteine dopo SDS-PAGE prima dell'immunoblotting, aggiungere la soluzione di colorazione Ponceau S (aggiungere 0,1 g di Ponceau S e 5 mL di acido acetico all'acqua ad un volume finale di 100 mL). Conservare a 4 gradi centigradi per 1 min e risciacquare rapidamente con acqua distillata.
- Etichettare ogni corsia con una penna a sfera, tagliare la membrana contenente due corsie con omogeneo cerebrale e una corsia con marcatore proteico e posizionare ciascuno in una scatola di incubazione macchia occidentale. Lavare 3x per 5 min ciascuno in salina tampone di fosfato (PBS) contenente 0,1% Tween 20 (PBS-Tw).
- Bloccare la membrana con 10% NGS (1 mL di normale siero di capra a 10 mL di PBS-Tw) in una scatola di incubazione macchia occidentale per 1 h. Fare una diluizione anti-mGlutR1 (5 lofanti in 10 mL di 10% NGS). Effettuare diluizioni anti-RDL1 e anti-RDL2 (5 - L di anticorpi in 10 mL di 10% NGS ciascuno). Sostituire la soluzione di blocco in ogni scatola con gli anticorpi diluiti e lasciare per una notte (16-24 h) a 4 gradi centigradi.
- Lavare la membrana 3x per 5 min ciascuno in PBS-Tw. Incubare la membrana con anticorpi secondari coniugati IgG HRP anti-coniglio a 1:10,000 nel 10% NGS PBS-Tw per 2 h a temperatura ambiente (RT). Lavare le membrane 3x in PBS-Tw, quindi 1x in PBS.
- Rilevare le bande utilizzando il substrato HRP chemiluminescente occidentale. Mescolare due substrati 1:1 (v/v) a RT, mettere tutte le membrane nella stessa scatola e coprirle con il mix di substrati per 2 min (in una stanza buia con una luce rossa) a RT. Procedere al rilevamento delle proteine utilizzando una pellicola di autoradiografia con più volte di esposizione. Di solito un anticorpo viene testato su una membrana contenente la stessa diluizione dell'omogeneo cerebrale su due o tre corsie e una corsia con il marcatore di peso.
3. Procedure immunocitochimiche
- Per sezionare i cervelli delle api per l'immunocitochimica, immobilizza le api melpote nel ghiaccio per 30 s. Dopo che le api sono immobilizzate, decapitare l'ape con le forbici e mettere la testa in una soluzione del 4% di paraformaldeide in PBS. Lavorare sotto il cofano dei fumi.
AVVISO: Il corpo deve essere smaltito con cura perché l'addome può ancora pungiglione dopo la decapitazione. - Rimuovere con attenzione ma rapidamente le antenne, gli occhi composti e tagliare tutto intorno all'esoscheletro superiore con le forbici Barraquer Iris. Lasciare riposare le teste nella soluzione fissativa per 10 min. Rimuovere il resto dell'esoscheletro della testa e tagliare tutte le trachea rimanenti.
- Mettere ogni cervello in un tubo di microcentrifuga da 1,5 mL contenente almeno 1 mL di soluzione di paraformaldeide del 4% e lasciare la notte durante la notte a 4-8 gradi centigradi.
- Fare una soluzione agarose 7.6% mescolando 3.8 g di agarose e 50 mL di acqua distillata in una fiaschetta Erlenmeyer. Microonde la soluzione fino a quando l'agarose si liquifa.
NOTA: Un piccolo pezzo di carta velina può essere posizionato nell'apertura del pallone per evitare che la soluzione agarose si sovraccarici durante il riscaldamento. - Posizionare i cervelli fissi delle api (3-4 cervelli) in una parabola Petri da 35 mm e rimuovere l'eccesso fissativo con carta velina. Versare la soluzione agarose liquido sul cervello. Orientare i cervelli nell'agarose in modo che i lobi dell'antenna siano rivolti verso l'alto. Lasciare raffreddare e solidificare l'agarose.
- Dopo che l'agarose si è solidificato, tagliare i blocchi di agarose ciascuno contenente un cervello.
- Per la sezionamento vibratoma, preparare una piastra 24 pozzo con ogni pozzo contenente un cesto con una rete idrofobica sul fondo. Riempire ogni pozzo con 600 gradi l di PBS.
- Tagliare ogni blocco in sezioni trasversali da 70 m utilizzando la macchina vibratoma e posizionare le sezioni nel cestello contenente PBS.
NOTA: Assicurarsi che le sezioni dello stesso cervello siano collocate nello stesso cesto. - Lavare le sezioni cerebrali 6x per 10 min ciascuna con PBS-TX per garantire che nessun fissatore rimanga nelle sezioni. Posizionare la piastra multiwell su uno shaker orbitale e lavare il cervello a 210 rpm. Prima di ogni lavaggio, assicurarsi di sostituire la soluzione PBS-TX con una soluzione PBS-TX nuova. Blocco con siero d'asino normale dell'1% durante l'ultimo lavaggio.
- Per testare l'anticorpo primario anti-mGlutR1, preparare una diluizione 1:112 degli anticorpi anti-mGlutR1 aggiungendo 9 mL di PBS-TX a 80 anticorpi anti-mGlutR1 in un tubo di centrifuga di 15 mL. Vorticare il tubo brevemente per mescolare accuratamente. La diluizione di lavoro degli anticorpi è stata determinata negli esperimenti preliminari.
- Per testare l'anticorpo primario anti-RDL, preparare una diluizione 1:100 di anticorpi anti-RDL aggiungendo 30 -L di peptide anti-RDL 1, 30 L di peptide anti-RDL 2 e 6 mL di centri-TX in un tubo da 15 mLfuge. Vorticare il tubo brevemente per mescolare accuratamente. La diluizione di lavoro degli anticorpi è stata determinata negli esperimenti preliminari.
- Aggiungere 800 gradi di soluzione anticorpale ad ogni pozzo nella piastra. Coprire la piastra multiwell e avvolgerla in un foglio di alluminio per evitare la degradazione dall'esposizione alla luce. Posizionare la piastra avvolta in un foglio di alluminio su uno shaker orbitale e agitare a 210 rpm per 2 h. Quindi lasciare per una notte a RT senza agitare.
- Dopo che le sezioni cerebrali sono state lasciate durante la notte, lavare con PBS-TX per 10 min. Ripetere la fase di lavaggio 6x.
- Preparare gli anticorpi secondari (anti-coniglio dall'asino) facendo una diluizione 1:225 di anticorpi secondari aggiungendo 40 l di anticorpi secondari a 9 mL di PBS-TX.
- Aggiungere 800 l della diluizione dell'anticorpo secondario ad ogni pozzo. Coprire la piastra e avvolgerla in un foglio di alluminio. Posizionare la piastra avvolta in un foglio di alluminio sullo shaker orbitale e agitare a 210 rpm per 2 h. Poi lasciarlo per una notte a RT.
- Lavare le sezioni cerebrali 3x per 10 min ciascuna con PBS-TX e 3x con una normale soluzione PBS.
- Per incorporare le sezioni nelle diapositive, preparare la soluzione di incorporamento supporti/glicerol di montaggio modificata da Rodriguez et al.20. Aggiungere 5 g del supporto di montaggio in PBS da 20 mL e mescolare per 16 h con un agitatore magnetico. Aggiungere 10 ml di glicerolo e mescolare per altri 16 h con un agitatore magnetico. Centrifuga per 15 min a 4.000 x g, prendere il supernatante omogeneo liquido e l'aliquota in tubi da 1 mL. Mantenere a -20 gradi centigradi
- Incorporare le sezioni sulle diapositive con una goccia del supporto di montaggio preparato nel passaggio 3.17, assicurandosi che ogni diapositiva contenga sezioni di un cervello.
- Controllo preadsorption of immunostaining con peptidi coniugati
- Per i peptidi anti-RDL e coniugati, incubare la diluizione di lavoro degli anticorpi anti-RDL con il corrispondente coniugamento peptide durante la notte alla RT sullo shaker: condizione 1) 500 g di peptide coniugato con il buco della serratura Limpet Hemocyanin (KLH) via glutaralhydede; condizione 2) senza coniugare.
- Centrifugare ogni miscela per 10 min a 10.000 x g e raccogliere il supernatante da entrambe le condizioni (passaggio 3.19.1).
- Incubare le sezioni seriali del cervello delle api melpote con ogni supernatante e processo con gli anticorpi secondari descritti sopra. Le sezioni seriali sono sezioni che si susseguono durante le procedure di sezionamento vibratome, quindi la stessa parte del cervello sarà esposta a controlli positivi e negativi.
- Per l'anti-mGlutR1 e il peptide coniugato, incubare la diluizione di lavoro degli anticorpi anti-mGlutR1 a RT con il peptide coniugato KLH contenente il peptide 10-4 M (condizione 1) e senza coniugato (condizione 2).
- Centrifuga ogni miscela per 10 min a 10.000 x g e raccogliere il supernatante da entrambi i controlli.
- Incubare le sezioni seriali del cervello delle api melpote con supernatali da entrambe le condizioni e il processo con gli anticorpi secondari (passaggi 3.14-3.15).
- Incorporare sezioni da entrambe le condizioni nella diapositiva utilizzando il supporto di incorporamento (passaggio 3.17).
4. Test di RDL e mGlutR1 Espressione proteica di immunocitochimica (sezione 3) in Honeybee Brain After Injection del corrispondente sistema CRISPR-Cas9
- Progettare le guide utilizzando uno strumento di progettazione CRISPR-Cas9 online21 utilizzando le sequenze genomiche di DNA di AmRdl (XM_006565102.3) e le sequenze di mGlutR1 (XM_006566244.3) (Tabella 1). Ordina come crRNA Cas9 e Cas-9 tracrRNA con il coloranti fluorescente ATTO550 alla fine 5'. Ordinare Cas9 Nuclease V3 (vedere Tabella dei materiali).
- Preparare i componenti per il sistema CRISPR-Cas 9 effettuando uno stock di 100 m di ogni crRNA guida e tracrRNA utilizzando acqua priva di nuclease. Aliquota e conservare a -20 gradi centigradi. Preparare la concentrazione di lavoro della soluzione Cas-9 aggiungendo 2,5 l di Cas 9 nuclease V3 (10 mg/mL) a 47,5 l di buffer libero nucleotide per ottenere una concentrazione finale di 0,5 g/l. Fare gRNA e RNP per l'iniezione.
- Preparare separatamente la formazione complessa di gRNA per ogni guida RNA (guideRNA:tracrRNAAT550)
- Etichettare una provetta con il nome del gRNA, aggiungere 92 L di nucleotide free buffer, 4 L di 100M CRISPR-Cas9 tracrRNA- 5'ATTO550 e 4 L della soluzione di crRNA guida. Mescolare delicatamente e girare.
NOTA: Esempio di etichettatura dei tubi gRNA per RDL: GRDL1, GRDL2, GRDL3 (corrispondente alle guide RDL RNA nella tabella 1). Esempio di etichettatura dei tubi gRNA per mGlutR1, GMGL2, GMGL3 (Tabella 1). - Riscaldare la soluzione a 95 gradi centigradi per 5 minuti per creare gRNA. Raffreddare il composto a RT per 10 min.
- Etichettare una provetta con il nome del gRNA, aggiungere 92 L di nucleotide free buffer, 4 L di 100M CRISPR-Cas9 tracrRNA- 5'ATTO550 e 4 L della soluzione di crRNA guida. Mescolare delicatamente e girare.
- Preparare la formazione complessa di RNP (gRNA: S.p Cas9Nuclease), miscele di consegna e controllo.
- Etichettare una provetta con il nome del PNR, aggiungere 6 L di soluzione gRNA e 6 .l di 0,5 g/L S.p Cas9 Nuclease V3. Mescolare delicatamente e incubare le soluzioni a 37 gradi centigradi per 10 min prima dell'iniezione.
NOTA: Esempio di etichette di tubi RNP: RRDL1, RRDL2, RRDL3. - Fare un RNPRDLmix. Per preparare la miscela finale utilizzata per le iniezioni, aggiungere 4 l' l di ogni RNP da RDL insieme. Mix RNP/RDL : 4 RRDL1 luna 1 x 4 RRDL lunatiera 4 RRDL.
- Fare un RNPmGlutR1mix. Mescolare 4 - L di ogni RNP da mGlutR1 insieme per preparare la miscela finale utilizzata per le iniezioni (RNPmGlutR1mix) : 4 RMG1 , 4 RMG2 , 4 RMG3 .
NOTA: Esempio di etichette di tubi RNP: RMG1, RMG2, RMG3. - Fare una soluzione di controllo noguideRNA mescolando 4 luna di tracrRNA, 92 l di buffer e 4 l of water invece di guideRNA (vedere la sezione 4.3). Mescolare 6 l di questa soluzione noguideRNA e 6 ll di 0,5 g/L Cas9 Nuclease V3 (passaggio 4.4.1) per produrre la soluzione di iniezione di controllo.
- Etichettare una provetta con il nome del PNR, aggiungere 6 L di soluzione gRNA e 6 .l di 0,5 g/L S.p Cas9 Nuclease V3. Mescolare delicatamente e incubare le soluzioni a 37 gradi centigradi per 10 min prima dell'iniezione.
5. Procedura di iniezione
- Immobilizza ogni ape come descritto nel passaggio 1.3 e alimentale come nel passaggio 1.4. Successivamente, preparare due serie di due scatole di legno per rilasciare le api in dopo l'iniezione: una scatola bianca (condizione sperimentale) e una scatola nera (controllo). Ogni scatola deve contenere un piccolo pettine e un piatto Petri che si nutre. Un lato di ogni scatola è fatto di vetro per consentire l'osservazione.
- Fare alimentazione piatti Petri. Prendere il coperchio di un piatto Petri 35 mm, posizionare la cera all'interno della superficie e posizionare la parte inferiore del piatto Petri sulla cera. Fissare la piastra nella scatola.
- Utilizzando una siringa da 5 mL, iniettare una soluzione di saccarosio da 1 M tra il coperchio e la parte inferiore del piatto. Le api possono mettere rapidamente le loro proboscide tra le due piastre e rimarranno asciutte per un periodo di incubazione di 48 h. Mettere un piatto in ogni scatola.
- Per preparare il sistema di microiniezione, riempire i capillari con olio minerale senza bolle d'aria. Posizionare i capillari nel titolare dell'iniettore del sistema di microiniezione. Per caricare il capillare con la soluzione di iniezione desiderata, posizionare una pellicola idrofobica su una superficie piana e pipettare la soluzione su di esso. Iniettare l'olio dei capillari e sostituire la miscela con la corrispondente miscela RNP.
- Utilizzando il sistema di microiniezione, iniettare 345 nL della soluzione della miscela RNP direttamente nell'ocelli mediano di ogni ape.
- Per l'iniezione RDL-CRISPR-Cas9, iniettare otto api con il RNPRDLmix preparato come descritto al passaggio 4.4.2. Dar loro da mangiare 1M di saccarosio dopo l'iniezione. Rilasciali nella scatola bianca (sperimentale) con il piccolo pettine e alimentando la parabola Petri per 48 h. Usa altre otto api come controlli senza iniezioni, nutrile e rilasciale nella scatola nera (controllo).
- Per mGlutR1 CRISPR-Cas9, iniettare nove api con il RNPmGlutR1mix preparato come descritto nel passaggio 4.4.3. Per il controllo iniettare otto api con miscela di RNA senza guida (RNAmix_noguide) preparati come descritto nel passaggio 4.4.4. Dar loro da mangiare 1M di saccarosio dopo l'iniezione. Rilasciare le api dai loro supporti nella scatola bianca (condizione sperimentale) o nella scatola nera (controllo) preparata come descritto nella sezione 5.1.
- Mettere le scatole in un contenitore di polistirolo con carta bagnata all'interno per l'umidità. Lasciare le api per 48 h e osservare 2volte al giorno per assicurarsi che abbiano abbastanza cibo e buona umidità.
- Dissezionare il cervello di ogni ape dopo 48 h (passaggio 3.1) e il processo per l'immunocitochimica come descritto nella sezione 3. Per gli immunostaininganti anti-mGlutR1 utilizzare il passaggio 3.11 e per gli immunostaining anti-RDL utilizzare il passaggio 3.12.
- Eseguire l'imaging confocale per valutare il livello di fluorescenza nelle sezioni del cervello macchiate di immuno.
- Per valutare la riduzione delle proteine nei cervelli immunoetichettati, utilizzare la raccolta di immagini confocali allo stesso livello di guadagno sia per il controllo che per il cervello iniettato.
6. Analisi di drop-off basata su qPCR per valutare il GENOMICo modificato RDL DNA 48 H dopo l'iniezione CRISPR Cas9 RNPRDLmix
- Progettare i primer, la sonda di controllo e la sonda drop-off per valutare la quantità di DNA modificato dall'iniezione CRISPR-Cas9. Ogni primer è progettato in modo che fiancheggi almeno una guida CRISPR-Cas 9 e la dimensione dell'amplificatore è di 132 bp. I primer e le sonde utilizzate sono indicati di seguito:
RDL1_For: CTCGGAGTGACCACCGT
RDL1_Rev: CAACGAGGCGAACACCAT
RDL1_control_probe: /5HEX/CGA
RDL1_Drop off_probe: /56-FAM/CCTA- Assicurarsi che i primer corrispondano a sequenze univoche specifiche dell'area. Assicurarsi che la sonda drop-off sia progettata per l'area che si sovrappone alla guida RDL-CRISPR-Cas9.
- Per verificare se c'è una modifica del gDNA nell'area guida, iniettare 12 api negli ocelli con il mix RNP_RDL descritto nel passaggio 5.3.1 e utilizzare otto api non iniettate come controlli.
- Dissezionare i cervelli delle api senza i lobi ottici 48 h dopo le iniezioni ed estrarre il gDNA di ogni cervello iniettato e controllare le api (senza lobi ottici) utilizzando il kit seguendo il protocollo del produttore (vedi Tabella dei materiali).
- Valutare la quantità, la qualità e la purezza del gDNA estratto utilizzando uno spettrofotometro.
- Quantificare l'espressione relativa del gDNA modificato di AmRDL utilizzando il protocollo drop-off basato su qPCR e il ciclore PCR in tempo reale.
- Risospendere gli oligos a 100 m, diluire i primer a 10 M, e le sonde a 5 M.
- Impostare la reazione PCR nella piastra 96, come mostrato qui per un campione di gDNA (3 ripetizioni): 10 l di master mix (vedi Tabella dei materiali),1 L di primer in avanti, 1 L di primer inverso, 1 L di sonda di controllo, 1 l una di sonda di drop-off, 2 l di gDNA (50 ng), 4 lof acque senza nuclease per portare il volume di reazione finale a 20 l.
NOTA: i controlli sono la soluzione al posto dei campioni e dell'acqua al posto delle sonde. - Impostare il programma di ciclismo come segue per un ciclore PCR in tempo reale: 95 s per 3 min seguito da 40 cicli di 95 s per 15 s, 60 gradi centigradi per 1 min.
- Valutare la modifica del gene relativo utilizzando il metodo 2--zCt, dove
 E
E
7. Quantificazione relativa di RDL RNA 48 H dopo l'iniezione di RNPRDLmix
- Per verificare se c'è una riduzione dell'mRNA RDL, iniettare 20 api negli ocelli con RNP_RDL mix come descritto nel passaggio 5.3.1 e utilizzare 12 api non iniettate come controlli. Estrarre i cervelli delle api (senza lobi ottici) 48 h dopo le iniezioni, estrarre l'mRNA totale da ogni ape iniettata e separarli utilizzando il protocollo del produttore (vedere Tabella dei materiali).
- Rimuovere i residui di DNA rimanenti nel campione utilizzando un kit privo di DNA (vedere Tabella dei materiali).
- Valutare la qualità e la purezza dell'RNA estratto utilizzando uno spettrofotometro.
- Quantificare l'espressione di AmRDL utilizzando un kit RT-PCR verde fluorescente disponibile in commercio (Tabella dei materiali) su un cycler PCR in tempo reale utilizzando il protocollo del produttore per una piastra ben 384.
NOTA: In questo esperimento sono stati utilizzati i primer pubblicati in precedenza. Per AmRDL (AmRDL_F GGTCGATGGTACTACCTG; AmRDL_R TCGATCGACTTGACGTAGGA)22 e primer actin come gene di riferimento [AmActin_F TGCCAACACTGTCCTTTCTG; AmActin_R GAATTGACCCACCAATCCA]23. L'espressione genica relativa è stata calcolata utilizzando il metodo 2--zCt (passaggio 6.6).
8. QPCR Based drop-off Analisi per valutare il DNA genomico modificato 48 H dopo l'iniezione di RNPmGlutR1mix
- Progettare i primer, il controllo e le sonde drop-off per valutare la quantità di DNA modificato dall'iniezione CRISPR-Cas9. Progettare i primer in modo che fiancheggiano almeno una guida CRISPR-Cas 9 e la dimensione dell'amplificatore è 96 bp.
mGlutR1_For: GGTGAAACGAACGACGGA
AmGlurR1_Rev: GGAGAGAGGGAGCGAGA
AmGlurR1_control_probe: /5HEX/CGAGG
AmGlurR1_Drop off_probe: /56-FAM/CGA
NOTA: Assicurarsi che i primer corrispondano a sequenze univoche specifiche dell'area che avrebbe dovuto essere modificata. La sonda drop-off è progettata per l'area che si sovrapponeva alla guida CRISPR-Cas9. - Per verificare se c'è una modifica del gDNA nell'area guida, iniettare 12 api negli ocelli con RNP_ mix GlutR1 come descritto nel passaggio 5.3.1 e utilizzare otto api non iniettate come controlli.
- Dissezionare i cervelli delle api (senza lobi ottici) 48 h dopo le iniezioni ed estrarre il gDNA da ogni cervello iniettato e controllare le api (senza lobi ottici) utilizzando il protocollo del produttore (vedi Tabella dei materiali).
- Valutare la quantità, la qualità e la purezza del gDNA estratto utilizzando uno spettrofotometro.
- Quantificare la modifica relativa di gDNA AmGlutR1 utilizzando il protocollo drop-off qPCR e per il cycler PCR in tempo reale come descritto nel passaggio 6.5.
- Valutare la modifica del gene relativo utilizzando il metodo 2--zCt, dove
 E
E
9. Quantificazione di mGlutR1 RNA 48 h Dopo l'iniezione di RNPmGlutR1mix
- Per verificare se c'è una riduzione dell'mRNA mGlutR1, iniettare sei api negli ocelli con il mix di RNP_RDL come descritto nel passaggio 5.3.2 e utilizzare sei api non iniettate come controlli. Estrarre i cervelli delle api (senza lobi ottici) 48 h dopo le iniezioni, estrarre l'mRNA totale da ogni ape iniettata e separarli utilizzando il protocollo del produttore per l'isolamento dell'RNA (vedere Tabella dei materiali).
- Rimuovere i residui di DNA rimanenti nel campione utilizzando un kit privo di DNA (vedere Tabella dei materiali).
- Valutare la qualità e la purezza dell'RNA estratto utilizzando uno spettrofotometro.
- Quantificare l'espressione di mGlutR1 utilizzando un kit SYBR Green RT-PCR (vedere Tabella dei materiali) su un cycler PCR in tempo reale con il protocollo fornito per il kit 384 well. Utilizzare i seguenti primer per mGlutR1 (mGlut_F CCTCCTCAACGTCTCTCTCTTCATA; mGlut_R TGCCGTTGTTGTTGTTGTTCCGATTT) e i primer actin [AmActin_F TGCCAACACTGTCCTTCTG; AmActin_R GAATTGACCCACCAATCCA]. Calcolare l'espressione genica relativa utilizzando il metodo 2- - Ct (passaggio 8.6).
Risultati
Test anticorpali anti-RDL
Gli anticorpi sono stati prodotti contro il coniugato peptide RDL come mostrato nella Figura 1A. Il primo passo nella caratterizzazione degli anticorpi anti-RDL consiste nel controllare l'omogeneità della proteina estratta dal cervello delle api utilizzando una macchia occidentale con anticorpi anti-RDL e un anticorpo secondario IgG con etichetta su HRP(Figura 1A), inserto). Entrambi gli anticorpi anti-RDL riconobbero la banda situata a 50-60 kD (freccia), corrispondente al peso stimato delle proteine isoformi della sottounità RDL. Per dimostrare che gli anticorpi anti-RDL riconoscevano il peptide nelle fette cerebrali, abbiamo usato un controllo di preadsorption (Figura 1B,C). Quando gli anticorpi sono stati preincubati con peptidi coniugati, la colorazione sulla sezione era assente. Ciò ha dimostrato che gli anticorpi anti-RDL riconoscevano il peptide coniugato contro il quale venivano sollevati. Per dimostrare che gli anticorpi anti-RDL riconoscono la proteina nel tessuto cerebrale fisso, abbiamo usato CRISPR-Cas9 per eliminare il gene RDL che produce la proteina RDL nelle cellule. Figura 1D1-3, mostra le sezioni del cervello frontale di controllo che sono stati etichettati con anticorpi anti-RDL. Questa ape non è stata iniettata con RDL-CRISPR-Cas9 RNP. Nella figura 1D1, anti-RDL etichetta neuropils nella sezione frontale del cervello delle api. La stessa sezione frontale nella figura 1D2 mostra l'assenza di fluorescenza da ATTO550, perché il complesso RDL-CRISPR-Cas9 non è stato iniettato.
Figura 1E1-E3 Mostra una sezione del cervello da un'ape iniettata con RDL-CRISPR-Cas 9 e quindi elaborata con la stessa quantità di anticorpi del cervello di controllo nella figura 1D1-D3. La colorazione anti-RDL è stata significativamente ridotta in tutto il cervello 48 h dopo l'iniezione, e la distribuzione della colorazione ATTO550 nel cervello (Figura 1E2) mostra il successo delle iniezioni RDL-CRISPR-Cas 9 nell'ocelli mediano delle api. Le molteplici cellule sparse del cervello presentano ATTO550. La riuscita iniezione di RDL-CRISPR-Cas 9 ha ridotto l'espressione proteica rispetto al controllo (Figura 1E1-3). Da otto cervelli di api immunostained, solo un cervello aveva un alto livello di distribuzione del RDL-CRISPR-Cas9 nelle cellule del corpo dei funghi, del protocerebrum e del lobo antenne, mentre altri cervelli avevano una colorazione cellulare con ATTO550 nel calice del corpo dei funghi, complesso centrale, ma non del lobo delle antenne. È importante notare che in queste api, la riduzione anti-RDL immunostaining non era così drammatica come nel cervello mostrato Figura 1E1.
Successivamente, per stimare il livello del gDNA RDL modificato nelle api 48 h dopo l'iniezione RDL-CRISPR-Cas9, abbiamo eseguito un test di drop-off qPCR, in cui la sonda di drop-off è stata progettata per corrispondere all'area di uno dei gRNA RDL. In questi esperimenti, nel cervello delle api iniettato con RDL-CRISPR-Cas 9, la riduzione relativa della fluorescenza corrispondeva al numero del gDNA modificato nei campioni. Nei nostri test l'area corrispondente a questa guida in gDNA in 12 api iniettate con RDL-CRISPR-Cas9 erano 64 % (media 30 % SD) rispetto a gDNA in api non inonettate (Figura 3A).
Successivamente, per stimare il livello dell'RNA RDL nelle api 48 h dopo l'iniezione, abbiamo eseguito qRT-PCR in un gruppo separato di api (Figura 3B). Abbiamo confrontato il livello di RNA RDL di PSL-CRISPR-Cas 9 api iniettate (n - 19) con il livello di RNA nelle api che non sono state iniettate (n n : 12). In questi esperimenti, la relativa riduzione dell'mRNA RDL è stata del 59% (media del 15% SE) rispetto al livello di RNA nelle api non iniettate. Quando abbiamo esaminato il livello di RNA RDL in ogni ape individualmente, solo 13 api su 19 api hanno mostrato una significativa riduzione dell'RNA. Questi dati indicano che l'iniezione di RDL-CRISPR-Cas9 attraverso gli ocelli potrebbe non sempre raggiungere un gran numero di cellule cerebrali, che conferma i dati con RDL immuno-colorato RDL-CRISPR-Cas9 iniettato api. In questi preparati, solo un'ape su 8 aveva RDL CRISP-Cas9 in molte cellule cerebrali (corpo del fungo, protocerebrum e lobo antenne) rispetto ad altri cervelli delle api, dove la distribuzione del RDL-CRISPR-Cas9 era concentrata nelle cellule nel protocerebrum (calice del corpo del fungo e complesso centrale) ma non il lobo delle antenne (Figura 4A-D).
Test anticorpi anti-mGlutR1
Abbiamo usato gli anticorpi anti-mGlutR1 prodotti nel coniglio contro peptidi coniugati specifici della Drosophila melanogaster (Figura 2A). La sequenza di questo peptide mostra un'identità del 94% con il peptide apile (CLSDKTRFDYFARTVPPD) Figura 2A. In primo luogo, abbiamo controllato gli anticorpi contro la proteina cerebrale delle api usando l'immunoblotting. L'omogenea cerebrale delle api è stata separata dal 10% di SDS-PAGE e trasferita elettrofoeticamente da una membrana di nitrocellulosa e macchiata con anti-mGlutR1. L'inserto nella Figura 2A mostra due bande con pesi stimati (103 e 83 kD) corrispondenti a due isoformi. Quando abbiamo testato questo anticorpo sul cervello delle api, abbiamo scoperto che etichettano i profili e le cellule del neuropilar nelle sezioni del cervello delle api, come illustrato nella Figura 2B,D. Dopo la preadsorption dell'anticorpo anti-mGlutR1 con peptide coniugato-mGlutR1 , la colorazione specifica scomparve nella fetta del cervello delle api (Figura 2C). Ciò conferma che gli anticorpi anti-mGlutR1 riconoscono il peptide (Figura 2C). Successivamente, abbiamo iniettato un mix di mGlutR1-CRISPR-Cas9 nell'ocelli mediano e abbiamo usato il noguideRNA di controllo. Nelle api di controllo (n - 7), la fluorescenza da ATTO550 non era concentrata nelle cellule. Alcuni cervelli avevano manifestazione difluorescenza diffusa ATTO550 etichettatura. Così, la preparazione del controllo in Figura 2D1-3 Mostra colorazione anti-mGlutR1 nel cervello, ma non la fluorescenza ATTO550. Quando mGlutR1-CRISPR-Cas9 è stato iniettato negli ocelli e assorbito da molte cellule, il livello di fluorescenza degli anticorpi secondari è stato significativamente ridotto nell'area che riprende il mGlutR1RNP funzionale (Figura 2E1-3). Le api sono state monitorate per 48 h, e un'ape per ogni condizione sperimentale è stata trovata morta. Così, in questo esperimento, abbiamo controllato sette api di controllo e otto api CRISPR-Cas9. Tutte le api iniettate con CRISPR-Cas9 avevano cellule che prendevano mGlutR1-CRISPR-Cas9. La maggior parte di queste cellule erano nel calice del corpo del fungo, complesso centrale, e protocererem posteriore. Solo due api su sette hanno mostrato l'etichettatura ATTO550 in molte cellule nel corpo del fungo, complesso centrale, e il lobo delle antenne. Un esempio di una di queste api è illustrato nella Figura 2E. La riduzione del livello di colorazione mGlutR1 in questi preparati è stata significativa. Le altre cinque api hanno l'etichettatura ATTO550 corrispondente alla consegna di successo del mGlutR1 CRISPR-Cas9 nel corpo del fungo e nel protocerebrum posteriore, ma non nei lobi delle antenne.
Successivamente, per stimare il livello del mGlutR1 gDNA modificato nelle api 48 h dopo l'iniezione, abbiamo eseguito un test di drop-off basato su qPCR, in cui la sonda drop-off è stata progettata per trovarsi nell'area vicino alla guida mGlutR1. In questi esperimenti, nel cervello delle api iniettato con mGlutR1-CRISPR-Cas 9, la relativa modifica del gDNA in 12 api era del 59% (media 33 % SD) rispetto al gDNA nelle api non iniettate (Figura 3A).
Questi risultati sono stati confermati anche dai test qRT-PCR in un diverso gruppo di api, dove abbiamo stimato i livelli di RNA mGlutR1 utilizzando qRT-PCR nelle api 48 h dopo le iniezioni con RNPmGlutR1mix (Figura 3B). Abbiamo confrontato il livello di mGlutR1 RNA delle api iniettate mGlutR1-CRISPR-Cas9 (n - 6) con i livelli di RNA nelle api che non sono state iniettate (n . 6). In questi esperimenti, la riduzione relativa dell'mRNA mGlutR1 nelle api iniettate è stata del 53% (media 18% SE) rispetto alle api non iniettate (Figura 3B).
La sezione di quattro api diverse che ha espresso il RNP RDL-CRISPR-Cas9 nella cella Kenyon del corpo fungo è mostrato figura 4A-D. L'ape di esempio con fluorescenza ATTO550 nel corpo del fungo e il lobo dell'antenna è illustrato Nella Figura 4E, F.
| Guide | Sequenze | gRNA | Rnp |
| [guidaRNA:tracrRNA] | [gRNA:Cas9 Nuclease] | ||
| RDL_Guide1 | ACCGTAACGCGCCCCCGCT | GRDL1 | RRDL1 |
| RDL_Guide2 | AACGTCGATCGACTTGACGT | GRDL2 | RRDL2 |
| RDL_Guide3 | CCATGAGAAACACGTGCCCCCCCCC | GRDL3 | RRDL3 |
| mGlu_Guide 1 | CGAAAGTTATCTCTGAGGTGT | GMGL1 | RMGL1 |
| MGlu_Guide 2 | TTCAACGAGACAAGTTCAT | GMGL2 | RMGL2 |
| mGlu_Guide 3 | GCAAACGTCGGTAGGAGTGA | GMGL3 | RMGL3 |
Tabella 1: Sequenze nucleotide di guide progettate per RDL e mGlutR1.
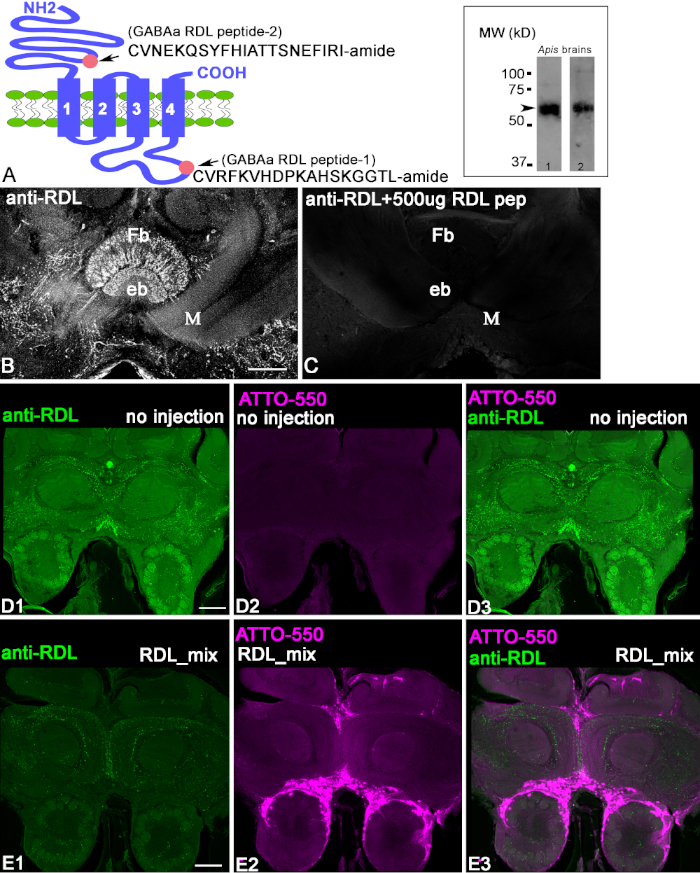
Figura 1: Caratterizzazione degli anticorpi anti-RDL. (A) Schematico della sottounità RDL, dove i cerchi rosa indicano la localizzazione del peptide 2 (extracellulare CVNEKQSYFHIATTSNEFIRI-amide) nel N-terminus and peptide 1 (intracellulare CVRFKVHDPKAHSKGS-amide) nel termine C. L'inserto in A mostra le bande nella macchia occidentale di estratti cerebrali di api craniche elaborate con anticorpi anti-RDL corrispondenti (uno con pep1 anti-RDL e pep2 anti-RDL). Ogni immunoblot mostra la dimensione apparente della proteina 50-60 kD, corrispondente ai pesi stimati delle varie isoforme delle sottounità RDL. (B, C) Preadsorption degli anticorpi anti-RDL con peptide coniugato 1. L'immagine in C mostra una riduzione delle macchie nella sezione quando gli anticorpi sono stati preincubati con peptide coniugato 1. Il corpo a forma di ventaglio (Fb) e il corpo ellissoide (eb) sono strutture complesse centrali nel cervello. M - lobo mediale del corpo del fungo. (D1-3) Colorazione anti-RDL di un controllo, sezione del cervello di api non iniettato dopo 48 h. Verde indica il profilo positivo anti-RDL nel cervello. (D2) Questa ape non è stata iniettata e non contiene fluorescenza ATTO550. (D3) Immagini unite da D1 e D3. (E1-3) L'iniezione di RDL-CRISPR-Cas9 ha ridotto la colorazione anti-RDL dopo la fluorescenza di 48 h. (E2) nei nuclei cellulari ha indicato il successo della consegna RDL-CRISPR-Cas9. (E3) Immagine unita di anti-RDL (verde) e ATTO550 (rosso). Barra della scala: 100 m (B-E). Fare clic qui per visualizzare una versione più grande di questa figura.
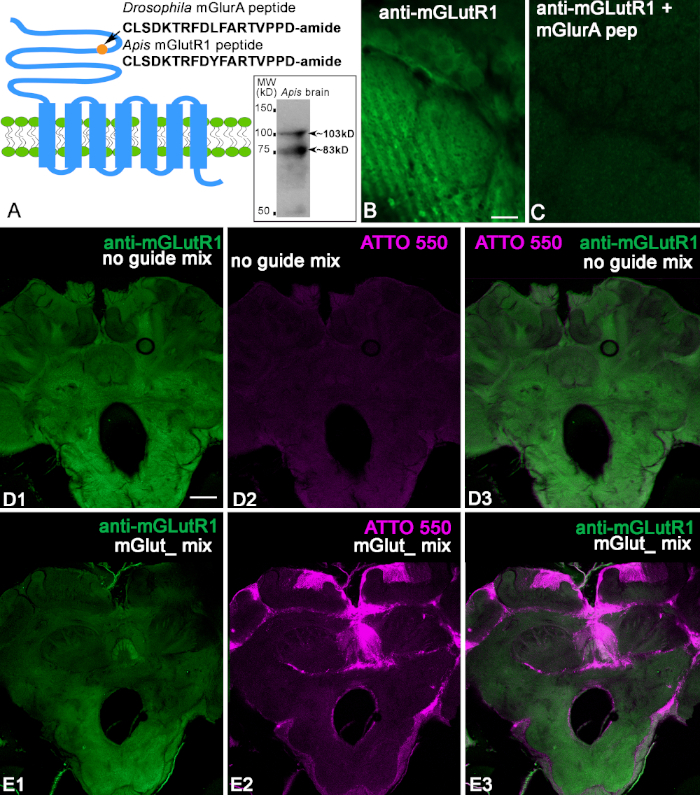
Figura 2: Caratterizzazione degli anticorpi anti-mGlutR1. (A) Schematica di GCPR mGluR che mostra il peptide Drosophila melanogaster utilizzato per l'immunizzazione. Per confronto, il peptide Apis mellifera è mostrato di seguito. Il cerchio indica la localizzazione del peptide nel capoN del dominio extracellulare del recettore mGlutR1. L'inserto in A mostra che gli anticorpi anti-mGlutR1 riconoscevano due bande nella macchia occidentale dei cervelli delle api, 103 kD e 83 kD che corrispondono ai pesi stimati delle isoforme conosciute. (B, C) Controllo della preadsorbioni dell'anticorpo anti-mGlutR1 in due sezioni consecutive del lobo dell'antenna glomeruli. L'immagine dell'anti-mGlutR1 nella sezione glomeruli antenna in C mostra la riduzione della colorazione come risultato della preincubazione del peptide anti-mGlutR1 con l'anticorpo anti-mGlutR1. Questa procedura fa precipitare l'anticorpo fuori soluzione, che abolisce la colorazione rispetto a B (controllo, assenza del peptide nella preincubazione). (D1) mostra la colorazione dell'anti-mGlutR1 in una fetta di cervello delle api dopo un'iniezione di controllo nell'ocellus mediano. Questa iniezione mancava del gRNA mGlutR1 che consente il knock down dei recettori mGlutR1 da CRISPR-Cas9, e quindi la colorazione dell'anticorpo anti-mGlutR1 non è stata ridotta (verde). (D2) L'assenza di fluorescenza ATTO550 indica l'assenza di CRISPR-Cas9 funzionale nel cervello. (D3) Immagini unite di anti-mGlutR1 e ATTO550. (E1-E3) mostrano la colorazione di anti-mGlutR1 in una sezione cerebrale dove mGlutR1 è stato definitivamente abbattuto 48 h dopo l'iniezione con mGlutR1-CRISPR-Cas 9 nell'ocellus mediano. Così, la colorazione in questo cervello è notevolmente ridotta a causa del successo knockout del mGlutR1 in molte cellule. (E2) La colorazione ATTO550 in molti nuclei cellulari nel cervello delle api indica che l'iniezione di mGlut1-CRISPR-Cas 9 ha avuto successo. (E3) Immagine unita di ATTO550 (rosso) e anti-mGlutR1 (verde). Barra della scala: 10 m (B, C); 100 m (D,E). Fare clic qui per visualizzare una versione più grande di questa figura.
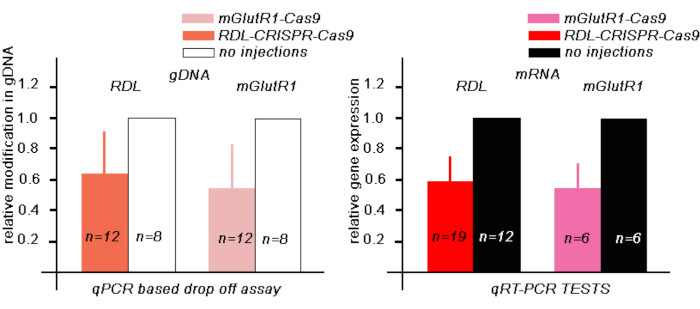
Figura 3: Valutazione del gDNA modificato e espressione di mRNA di RDL e mGlutR1 mRNA nei cervelli delle api 48 h dopo l'iniezione con 345 nL del corrispondente RPN CRISPR-Cas9. (A) Il test di analisi di drop-off basato su qPCR per valutare la quantità di gDNA con un'area di modifica è stato calcolato utilizzando il metodo 2-ZCt e normalizzato contro il controllo, cervelli non iniettati. I dati sono espressi come media(B ) Il test TheqRT-PCR è stato utilizzato per valutare la quantità di mRNA nelle api iniettate e non iniettate CRISPR-Cas9. AmActin è stato usato come gene di riferimento. L'espressione genica relativa è stata calcolata utilizzando 2 metodidi z-zct e normalizzata contro il controllo, cervelli non iniettati. I dati sono espressi come media : SE. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
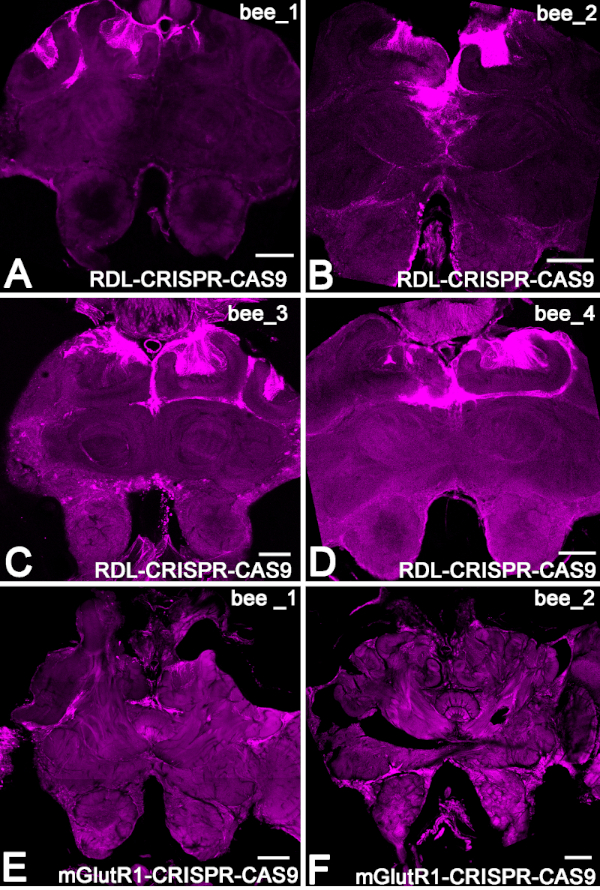
Figura 4: Esempio della distribuzione di RNP CRISPR-Cas9 nel cervello delle api tramite la fluorescenza ATTO550. (A-D) Le sezioni cerebrali di quattro api diverse che hanno espresso il RNP RDL-CRISPR-Cas9 nella cellula Kenyon del corpo del fungo. (E, F) Esempio di due cervelli 48 h dopo l'iniezione con RNPmGlutR1 CRISPR-Cas9. Barra della scala: 150 m (A-F). Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Caratterizzazione dell'anti-RDL e dell'anti-mGlutR1
In primo luogo, abbiamo caratterizzato gli anticorpi anti-RDL e anti-mGlutR1 da immunoblot e pre-adsorbiente sulle fette di cervelli fissi delle api. Ogni anticorpo è stato fatto per riconoscere tutte le sue isoformi conosciute, e l'analisi occidentale mostra che riconoscono le bande che corrispondono ai loro pesi molecolari previsti. Successivamente, entrambi gli anticorpi sono stati bloccati dal peptide coniugato contro il quale sono stati prodotti su sezioni cerebrali di api melpote.
Uno dei primi obiettivi del nostro studio è stato quello di stabilire che gli anticorpi prodotti contro il peptide coniugato specifico sono specifici per la sua proteina nel tessuto cerebrale fisso. A tale scopo, abbiamo approfittato del sistema CRISPR-Cas9. Abbiamo progettato guide specifiche per honeybee RDL e mGlutR1 e le abbiamo utilizzate per realizzare CRISPR-Cas9 etichettato con la sonda fluorescente ATTO550. Per ogni recettore, abbiamo iniettato una miscela di tre diverse ribonucleoproteine CRISPR-Cas9 negli ocelli per ridurre la quantità di proteine mirate nel cervello delle api adulte eliminando il gene corrispondente nelle cellule che hanno assunto il nostro sistema Cas9 progettato. Nel nostro studio, abbiamo compiuto questo passaggio.
Uno dei primi passi cruciali per il successo di questi esperimenti è la progettazione degli RNA guida appropriati. Si consiglia di progettare fino a cinque RNA guida, situati all'inizio, al centro e alla fine della sequenza genica. Nel nostro lavoro preliminare, li abbiamo testati in varie combinazioni su tre o cinque api. Abbiamo anche provato diverse concentrazioni di iniezioni, così come i tempi dopo l'iniezione, e varie miscele di RNP nelle iniezioni. Abbiamo sezionato i cervelli e li abbiamo elaborati usando anticorpi anti-RDL e anti-mGlutR1. In questi test iniziali, abbiamo stabilito la combinazione appropriata, il tempo di post-iniezione, così come la concentrazione e la quantità di CRISPR-Cas9 per l'iniezione. Questi test iniziali sono stati la base per la creazione degli esperimenti che abbiamo descritto in dettaglio qui.
L'obiettivo era duplice: 1) dimostrare in un'ape che la nostra colorazione anticorpale è stata ridotta dopo il trattamento con CRISPR-Cas9 e 2) per lavorare attraverso le migliori condizioni sperimentali per gli studi comportamentali. Così, mostriamo che se molti nuclei cellulari contengono CRISPR-Cas9 48 h dopo l'iniezione, la riduzione della colorazione anti-RDL e anti-mGlutR1 è significativa. Inoltre, questo dimostra che gli anticorpi testati riconoscono specificamente la proteina mGlut1 e RDL nella preparazione del cervello delle api melfale e che possono essere utilizzati per studi di localizzazione nel cervello delle api.
Impostazione sperimentale CRISPR-Cas9 per lo studio comportamentale
Successivamente, abbiamo impostato gli esperimenti in modo che CRISPR-Cas9 potrebbe essere utilizzato in studi comportamentali. Sono state raccolte otto o nove api melpote per il controllo e i trattamenti sperimentali. Sono stati testati comportamentalmente prima e dopo l'iniezione, e poi i loro cervelli sono stati elaborati per ATTO550 e/o immunocitochimica per determinare le regioni cerebrali che hanno mostrato la riduzione della proteina bersaglio. Qui è essenziale notare che il numero di api prese per una serie di esperimenti è stato limitato a non più di 8-9 api per le condizioni di controllo e sperimentali. In questo modo entrambe le condizioni potrebbero essere testate lo stesso giorno. Inoltre, una volta preparate le miscele CRISPR-Cas9 per l'iniezione, non le abbiamo mai congelate. La miscela CRISPR-Cas9 non ha cambiato la potenza quando viene utilizzata 3 giorni di fila e mantenuta a 4-8 gradi centigradi. Tuttavia, non abbiamo provato dopo 3 giorni.
Come abbiamo descritto nella sezione Risultati per entrambi i set di iniezioni sperimentali e per entrambi gli anticorpi, solo tre api da 16 testate hanno mostrato una grande distribuzione ATTO550 nel corpo del fungo, nel protocerebrum e nei lobi delle antenne. In tutte le altre api, la distribuzione di CRISPR-Cas9 era limitata al corpo del fungo, al complesso centrale e/o al protocerere posteriore. È essenziale capire per qualsiasi studio comportamentale che utilizzando questo metodo di iniezione la riduzione della proteina bersaglio sarà limitata solo al corpo del fungo nella maggior parte delle api. Non si estenderà al lobo dell'antenna o al ganglio subesofageo. Così, la tecnica di iniezione che usiamo è adatta per studiare l'effetto della riduzione dei recettori nel corpo del fungo e complesso centrale in esperimenti comportamentali, mentre un metodo diverso di introduzione CRISPR-Cas9 sarà più appropriato per studiare altre regioni del cervello.
In conclusione, il nostro studio ha dimostrato il successo dell'applicazione di CRISPR-Cas9 come controllo per la colorazione degli anticorpi nel cervello. Per entrambi gli anticorpi (anti-RDL e anti-mGlutR1), quando l'assorbimento di mGlutR1-CRISPR-Cas9 o RDL-CRISPR-Cas9 ha avuto successo, anche il livello di colorazione degli anticorpi corrispondenti è stato ridotto in modo significativo. Inoltre, è essenziale notare che l'iniezione negli ocelli ha portato ad una distribuzione di CRISPR-Cas9 nel cervello che non era omogeneo. La distribuzione variava da un'area minima che circonda il corpo di ocelli e funghi a molte cellule in tutto il cervello. La variabilità dell'assorbimento mGlutR1-o RDL-CRISPR-Cas9 da parte delle cellule era probabilmente dovuta a variazioni nelle iniezioni. I nostri dati mostrano che il sistema CRISPR-Cas9 funziona nelle api, ma il metodo di iniezione deve essere migliorato per ridurre la variabilità dell'assorbimento di CRISPR-Cas9 nei singoli cervelli delle api. All'interno di queste restrizioni, è ora possibile impiegare questa tecnica per manipolare i geni nelle api adulte per esperimenti comportamentali.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dai seguenti premi a BHS: Human Frontiers Science Program; NIH NIGMS (R01 GM113967); Laboratorio di idee NSF (1556337). Il peptide e anticorpi per DmGluRA sono stati progettati nel laboratorio Dr. Serge Birman (Marsiglia, Luminy, Francia) quando IS è stato supportato dal Programma d'Urgence FRM/Postdocs UFP20060306548 dalla Fondation pour la Recherche Medicale. Siamo grati per Daniela Junqueira Marosi e Alex Hanter di Integrated DNA Technology (IDT) per la progettazione di guide RDL e analisi di drop-off qPCR.
Materiali
| Name | Company | Catalog Number | Comments |
| Acetic Acid | Sigma-Aldrich | A6283_100 mL | western blotting |
| Acrylamide-bis Acrylamide | Bio-Rad | 500 mL 1610156 | western blotting |
| Agarose | Sigma-Aldrich | A0169-250g | used with distilled water to fix honey bee brain in blocks |
| Alexa Fluor 488 AffiniPure F(ab')2 Fragment Donkey Anti-Rabbit IgG (H+L) | Jackson Research Laboratories | 711-546-152 | secondary antibody to reveal the primary antibodies from rabbit |
| Alt-R CRISPR-cas9 tracrRNA-5'ATTO | IDT | 1075934 | with desinged guide RNA, it creates gRNA |
| Alt-R S.p. Cas9 nuclease V3 | IDT | 1081058 | enzyme used to make ribonucleoprotein for CRISPR system |
| Ammonium Persulfate | Bio-RAD | 10 g 1610700 | western blotting |
| Anti-mGlutR1 antibodies | Covalab (FRANCE)/21st Century Biochemical | characterized by authors in present paper | Used for primary incubation of mGlut 1 in honey bees |
| Anti-RDL antibodies | 21st Century Biochemical | characterized by authors in present paper | Used for primary incubation of RDL in honey bees |
| Aprotinin | Sigma-Aldrich | Y0001154 | protease inhibitor |
| Barraquer Iris Scissors 7mm Blade Sharp point | World Precision Instruments | 14128-G | used for dissection honey bee brain |
| Benzamidine | Sigma-Aldrich | 12072 | protease inhibitor |
| Blade (breakable) for blade holder | Fine Science Tool | 10050-00 | dissection for western blotting |
| Blade holder and breaker | Fine Science Tool | 15309 | dissection for western blotting |
| Borosilicate glass capillaries | World Precision Instruments | 1B100 F | for injection procedure |
| Chemiluminescent western blot detection substrate | Bio-Rad | 1705062 | western blotting |
| Chloroform | Sigma-Aldrich | 472476-50mL | RNA isolation |
| Dithiothreitol | Bio-Rad | 1610611 | western blotting |
| DNA easy kit | QIAGEN | 69504 | DNA extractionuj |
| DNA-free kit | INVITROGEN | AM1907 | Used to remove DNA |
| Eppendorf Research plus pipette, 3-pack | Sigma-Aldrich | Z683884 | |
| Falcon 24 Well Polystyrene Multiwell | Falcon | 351147 | Multiwell |
| Flat Bottom Embedding Capsules, Polyethylene | Electron Microscopy Science | 70021 | basket for brain sections |
| Forceps Dumont #5 (pair) | Fine Science Tool | 11254-20 | for dissection of honey bee brain from the head |
| Forceps Dumont #5S (pair) | Fine Science Tool | 11252-00 | to clean up the brain from trachea befor dissection |
| Gene Expression Master Mix | Integrated DNA Technologies | 1055770 | qPCR drop off assay |
| Glutaraldehyde | EMS | 16220 | used for preparation of peptide conjugates for control peabsorption |
| Glycerol | Sigma-Aldrich | G5516-500 mL | western blotting, embedding media |
| Glycine | Bio-Rad | 1610718 | western blotting |
| Hydrophobic filtered nylonmesh | Spectrum Labs | 145910 | for bottom of basket for brain sections |
| Isopropyl alcohol | Sigma-Aldrich | I9030-50mL | RNA isolation |
| Keyhole limpet hemocyanin | Sigma-Aldrich | H7017 | used for preparation of conjugates for control |
| LSM800 cofocal microscope | Zeiss | ||
| mGlu_Guide 1 | IDT | designed guide RNA for CRISPR-Cas 9. Target mGlutR1 genome sequences | |
| mGlu_Guide 2 | IDT | designed guide RNA for CRISPR-Cas 9. Target mGlutR1 genome sequences | |
| mGlu_Guide 3 | IDT | designed guide RNA for CRISPR. Target mGlutR1 genome sequences | |
| methanol | Sigma-Aldrich | 34860 | western blotting |
| 96-well PCR microplate | Applied biosystem | 4346907 | qPCR drop off assay and qRT-PCR |
| 384-well PCR microplate | Sigma-Aldrich | Z374911 | qRT-PCR |
| Mineral oil | Sigma-Aldrich | M5904_500mL | for injection procedure |
| Model p87 Flaming Brown Micropipette Puller | Sutter Instrument Co. | Capillary Preparation | |
| Mowiol 4-88 | Sigma-Aldrich | 81381 | for embedding solution |
| Nanoliter 2000 | World Precision Instruments Nanoliter | Injection Apparatus | |
| Normal Donkey Serum | Jackson Research Laboratories | AB_2337258 | blocking agent for immunocytochemistry |
| Non-immune Goat Serum | Invitrogen | 50-062Z 100 mL | blocking agent for immunoblotting |
| Nuclease-free buffer | IDT | 1072570 | Nuclease-free buffer that is used in the preparation of CRISPR-Cas9 injection |
| Nuclease-free water | IDT | AM9337 | nuclease -free water that is used in preparation of CRISPR-Cas 9 system |
| OrbitalShaker Mp4 | Genemate | ||
| Paraformaldehyde | Sigma-Aldrich | 158127-500 g | used with PBS to make fixative for bee brains |
| Phenylmethylsulphonyl fluoride (PMSF) | Sigma-Aldrich | P7626-250 mg | protease inhibitor |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417-100TAB | Used with Paraformaldehyde as fixative; buffer for antibodies |
Riferimenti
- Harms, D. W., et al. Mouse Genome Editing Using the CRISPR/Cas System. Current Protocols in Human Genetics. 83 (15), 7-27 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Kouranova, E., et al. CRISPRs for Optimal Targeting: Delivery of CRISPR Components as DNA, RNA, and Protein into Cultured Cells and Single-Cell Embryos. Human Gene Therapy. 27 (6), 464-475 (2016).
- Schubert, M., et al. Fluorescently labeled tracrRNA provides efficient genome editing while allowing cellular microscopy and FACS analysis. Genome Editing. , 1-3 (2017).
- Kucharski, R., Mitri, C., Grau, Y., Maleszka, R. Characterization of a metabotropic glutamate receptor in the honeybee (Apis mellifera): implications for memory formation. Invertebrate Neuroscience. 7 (2), 99-108 (2007).
- Aronstein, K., Auld, V., Ffrench-Constant, R. Distribution of two GABA receptor-like subunits in the Drosophila CNS. Invertebrate Neuroscience. 2 (2), 115-120 (1996).
- Thompson, M., Steichen, J. C., ffrench-Constant, R. H. Conservation of cyclodiene insecticide resistance-associated mutations in insects. Insect Molecular Biology. 2 (3), 149-154 (1993).
- Chung, B. Y., Kilman, V. L., Keath, J. R., Pitman, J. L., Allada, R. The GABA(A) receptor RDL acts in peptidergic PDF neurons to promote sleep in Drosophila. Current Biology. 19 (5), 386-390 (2009).
- Taylor-Wells, J., Hawkins, J., Colombo, C., Bermudez, I., Jones, A. K. Cloning and functional expression of intracellular loop variants of the honeybee (Apis mellifera) RDL GABA receptor. Neurotoxicology. 60, 207-213 (2017).
- Jones, A. K., Sattelle, D. B. The cys-loop ligand-gated ion channel superfamily of the honeybee, Apis mellifera. Invertebrate Neuroscience. 6 (3), 123-132 (2006).
- Dupuis, J. P., et al. Homomeric RDL and heteromeric RDL/LCCH3 GABA receptors in the honeybee antennal lobes: two candidates for inhibitory transmission in olfactory processing. Journal of Neurophysiology. 103 (1), 458-468 (2010).
- Farooqui, T., Robinson, K., Vaessin, H., Smith, B. H. Modulation of early olfactory processing by an octopaminergic reinforcement pathway in the honeybee. Journal of Neuroscience. 23 (12), 5370-5380 (2003).
- Guo, X., Wang, Y., Sinakevitch, I., Lei, H., Smith, B. H. Comparison of RNAi knockdown effect of tyramine receptor 1 induced by dsRNA and siRNA in brains of the honeybee, Apis mellifera. Journal of Insect Physiology. 111, 47-52 (2018).
- Roth, A., et al. A genetic switch for worker nutrition-mediated traits in honeybees. PLOS Biology. 17 (3), e3000171 (2019).
- Kohno, H., Suenami, S., Takeuchi, H., Sasaki, T., Kubo, T. Production of Knockout Mutants by CRISPR/Cas9 in the European Honeybee, Apis mellifera L. Zoological Science (BIOONE). 33 (5), 505-512 (2016).
- Hu, X. F., Zhang, B., Liao, C. H., Zeng, Z. J. High-Efficiency CRISPR/Cas9-Mediated Gene Editing in Honeybee Apis mellifera Embryos. G3: Genes, Genomes, Genetics. 9 (5), 1759-1766 (2019).
- Smith, B. H., Burden, C. M. A Proboscis Extension Response Protocol for Investigating Behavioral Plasticity in Insects: Application to Basic, Biomedical, and Agricultural Research. Journal of Visualized Experiments. (91), e51057 (2014).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Rodriguez, J., Deinhardt, F. Preparation of a semipermanent mounting medium for fluorescent antibody studies. Virology. 12, 316-317 (1960).
- . Custom Alt-R® CRISPR-Cas9 guide RNA Available from: https://www.idtdna.com/site/order/designtool/index/CRISPR_CUSTOM (2019)
- Bonnafe, E., et al. Effect of a thymol application on olfactory memory and gene expression levels in the brain of the honeybee Apis mellifera. Environmental Science and Pollutant Research (International). 22 (11), 8022-8030 (2015).
- Wang, Y., et al. Regulation of behaviorally associated gene networks in worker honeybee ovaries. Journal of Experimental Biology. 215 (Pt 1), 124-134 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon