É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Interrogatório paralelo de β-Arrestin2 Recrutamento para Triagem de Ligand em escala GPCR usando ensaio PRESTO-Tango
Neste Artigo
Resumo
Dado que os GPCRs são alvos atraentes para drogados, a triagem de ligantes GPCR é, portanto, indispensável para a identificação de compostos de chumbo e para estudos de desordeiro. Para esses esforços, descrevemos o PRESTO-Tango, uma plataforma de recursos de código aberto usada para o perfil simultâneo de recrutamento transitório β-arrestin2 em aproximadamente 300 GPCRs usando um ensaio de repórter baseado em TEV.
Resumo
Como a maior e mais versátil superfamília genética e mediadora de uma gama de vias de sinalização celular, os receptores acoplados à proteína G (GPCRs) representam um dos alvos mais promissores para a indústria farmacêutica. Em medida, o projeto, a implementação e a otimização dos ensaios de triagem de ligadores GPCR são cruciais, pois representam ferramentas de controle remoto para a descoberta de medicamentos e para manipular a farmacologia e os resultados do GPCR. No passado, ensaios dependentes de proteína G tipificavam essa área de pesquisa, detectando eventos induzidos por ligantes e quantificando a geração de mensageiros secundários. No entanto, desde o advento da seletividade funcional, bem como uma maior consciência de várias outras vias independentes da proteína G e as limitações associadas aos ensaios dependentes da proteína G, há um maior impulso para a criação de alternativas Ensaios de triagem de ligais GPCR. Para este esforço, descrevemos a aplicação de um desses recursos, a plataforma PRESTO-Tango, um sistema baseado em repórteres de luciferase que permite o interrogatório paralelo e simultâneo do GPCR-ome humano, um feito que foi previamente considerado tecnicamente e economicamente inviáveis. Com base em um ensaio de recrutamento β-arrestin2 independente de proteína G, a universalidade do tráfico e sinalização mediados por β-arrestin2 em GPCRs faz do PRESTO-TANGO uma ferramenta adequada para estudar aproximadamente 300 GPCRs humanos não olfativos, incluindo aproximadamente 100 receptores órfãos. A sensibilidade e a robustez do PRESTO-Tango tornam-no adequado para telas primárias de alto desempenho usando bibliotecas compostas, empregadas para descobrir novos alvos de GPCR para drogas conhecidas ou para descobrir novos ligantes para receptores órfãos.
Introdução
Os receptores acoplados à proteína G (GPCRs) constituem a maior e mais diversificada família de proteínas transmembranas, operando como interfaces de comunicação entre uma célula e seu ambiente1. A versatilidade dos GPCRs é destacada por sua capacidade de detectar uma variedade diversificada de ligantes – de neurotransmissores a nucleotídeos, peptídeos a fótons, e muito mais – bem como sua capacidade de regular numerosas cascatas de sinalização a jusante envolvidas no crescimento celular, migração, diferenciação, apoptose, disparo celular, etc.2,3. Considerando sua ubiquidade e envolvimento em uma infinidade de processos fisiológicos, essa família receptora é de extrema importância terapêutica, demonstrada pelo fato de que mais de um terço dos medicamentos prescritos atualmente disponíveis têm como alvo as RPCRs4. No entanto, essas terapêuticas existentes visam apenas um pequeno subconjunto da superfamília (estimado em 10%), e a farmacologia de muitos GPCRs permanece inelucidada. Além disso, existem mais de 100 GPCRs como receptores órfãos, pois não foram combinados com um ligante endógeno5. Assim, a triagem de ligantes gpcr é fundamental na desordeirização e desenvolvimento de medicamentos, pois pavimenta o caminho para a descoberta e otimização de chumbo, e possivelmente para a fase de ensaio clínico.
Os métodos de triagem de ligantes GPCR tradicionalmente caíram em uma das duas categorias, ensaios funcionais independentes de proteína G ou g-proteína6. A sinalização GPCR é regulada por proteínas G heterotriméricas (Gαβγ), que são ativadas pela troca de GTP por PIB vinculado à subunidade Gα7. Os sinais do receptor ativado são transprovocados por proteínas G através de mensageiros secundários, como cAMP, Cálcio, DAG e IP3, para mediar a sinalização a jusante em efeitos a jusante8. A natureza das consequências funcionais da sinalização da proteína G tem sido explorada para criar ensaios baseados em células que refletem a ativação do receptor. Esses métodos, que medem eventos proximais (diretos) ou distais (indiretos) na sinalização de proteína G, são mais utilizados para a triagem de ligantes GPCR e têm sido empregados principalmente em estudos de desordeiro6. Exemplos de ensaios que medem diretamente a ativação de proteína G mediada pelo GPCR incluem o ensaio de ligação [35S]GTPγS, que mede a vinculação de um GTP radiorotulado e não hidrolisavel análogo à subunidade Gα, e sondas de transferência de energia de ressonância förster/bioluminescência (FRET/BRET, respectivamente) para monitorar as interações GPCR-Gα e Gα/Gγ, que vêm ganhando mais tração ao longo dos anos9,10. Ensaios que monitoram eventos distais são as ferramentas mais utilizadas para a criação de perfis de GPCR; por exemplo, os ensaios cAMP e IP1/3 medem o acúmulo intracelular de mensageiros secundários dependentes da proteína G, enquanto os ensaios de fluxo e repórter [Ca2+]envolvendo elementos de resposta específicos implicados na ativação da proteína G (CRE, NFAT-RE, SRE, SRF-RE) examinam eventos mais a jusante da cascata de sinalização11. Embora a maioria dos ensaios acima mencionados possa ser realizada em um nível de alta duração, são bastante sensíveis, e possuem certas vantagens específicas do ensaio (por exemplo, discriminação entre agonistas completos/parciais, antagonistas neutros e agonistas inversos no caso da ligação GTPγS, ou funcionalidade de ensaio em células vivas como [Ca2+] e IP1/3)6, infelizmente não existem métodos dependentes de proteína G condizentes com o interrogatório de todo o GPCR-ome drogado. Isso se deve em grande parte ao acoplamento nativo de múltiplas subfamílias de proteína G aos GPCRs, resultando em sinalização em várias cascatas e o acoplamento desconhecido de proteína G em GPCRs órfãos. Para mitigar esse problema, foram desenvolvidos ensaios para forçar o acoplamento promíscuo de proteína G através de uma única leitura de sinalização comum, como cAMP, e Ca2+,embora a maioria deles seja de baixa produção12.
Um aspecto importante do ciclo de vida do GPCR é o término da sinalização dependente da proteína G, que ocorre em grande parte através do recrutamento de β-arrestins que induz a dissociação da proteína G, e, em última análise, dessensibilização do receptor, que é direcionado para a internalização revestida de clatina13. As isoformas mais expressas de β-arrestin são as β-arrestin1 e β-arrestin2 não-visuais, também denotadas como prisões em-2 e prisões em-3, respectivamente14. Insira ensaios baseados em células independentes de proteína G, que adicionam uma nova dimensão à triagem de ligantes GPCR; tráfico de receptores, célula inteira sem rótulos e β-arrestem em ensaios de recrutamento são todos exemplos notáveis. Os ensaios de tráfico do GPCR empregam ligantes rotulados com fluoroforo ou anticorpos co-internalizados direcionados ao receptor15, enquanto os ensaios celulares inteiros sem rótulos usam biosensores que traduzem alterações celulares induzidas por ligantes em saídas quantificáveis, como sinais elétricos ou ópticos16. Notavelmente, o quintessencial GPCR-β-arrestin interações forma o β-arrestin recrutamento como uma ferramenta atraente no repertório de ensaios funcionais17. O sistema Tango, desenvolvido pela primeira vez por Barnea et al. apenas uma década atrás, envolve a introdução de três elementos genéticos exógenos: uma fusão de proteínas constituída por β-arrestin2 com um vírus etch protease (TEVp), um transativador tetraciclina (TTA) que é amarrado a um GPCR através de um tabaco e tch vírus protease site (TEVcs) e é precedido por uma seqüência do C-terminus do receptor de vasopressina V2 (cauda V2) para promover a prisão no recrutamento, e um gene de luciferase repórter cuja transcrição é desencadeada pelo translocação do fator de transcrição tTA para o núcleo, que é libertado da ancoragem da membrana após β-arrestin2 recrutamento (Figura 1)18. Leituras quantitativas de ativação do GPCR e recrutamento β-arrestin2 podem ser posteriormente determinadas pela leitura para luminescência. Uma distinção notável é que, embora o tráfico de receptores e os métodos celulares inteiros sem rótulos sejam relativamente baixos, o Tango tem várias vantagens, incluindo leitura seletiva específica do receptor alvo e sensibilidade devido à integração de sinais, o que o torna um candidato adequado para a triagem de ligantes em uma escala maior18.
Em vista dessas características estratégicas, Kroeze et al. desenvolveram presto-tango (Parallel Receptor-ome Expression and Screening via Transcriptional Output-Tango), uma plataforma de código aberto de alto throughput que usa a abordagem Tango para traçar o perfil do GPCR-ome drogado de forma paralela e simultânea19. Explorando o recrutamento "promíscuo" de β-arrestin2 para quase todos os GPCRs, presto-tango é o primeiro de seu tipo em termos de ensaios funcionais baseados em células, permitindo uma rápida triagem "primeira rodada" de pequenos compostos de moléculas em quase todos os GPCRs não olfativos, incluindo órfãos, independente do acoplamento subfamiliar de proteína G.
Protocolo
1. Triagem primária: cultura celular e semeada de placas
- Para preparar placas revestidas de poli-L-lysina (PLL), dispense 20 μL/poço de uma solução de 25 μg/mL de pll em placas inferiores ópticas brancas ou pretas de 384 poços usando uma pipeta multicanal eletrônica ou um dispensador de reagente. Incubar as placas à temperatura ambiente por 0,5-2 h.
NOTA: Se usar as placas pretas de 384 poços, espere que o sinal de fundo seja menor em comparação com as placas brancas. Placas pretas são recomendadas para reduzir o sangramento da luminescência entre poços adjacentes. - Para preservar as placas revestidas e lavar o excesso de PLL, remova o PLL, apertando-o sobre a pia, toque seco sobre uma toalha de papel e adicione 40 μL/poço de solução diluída de 1x de antibiótico-antimicocético usando uma pipeta multicanal eletrônica ou um dispensador de reagente. Armazene as placas revestidas de PLL a 4 °C até que esteja pronta para a semeadura da placa.
- Manter células HTLA (gentilmente fornecidas pelo Dr. Richard Axel) – uma linha de células renais embrionárias humanas (HEK293T) expressando silenciosamente β-arrestin2-TEV e luciferase impulsionada por tTA – em completo o meio modificado de Dulbecco (DMEM) complementado com 5% de Soro Bovino Fetal, 5% de Soro de Bezerro Bovino, 2,5 μg/mL de puromicina, 50 μg/mL de higoricina, 100 u/mL penicilina e 100 μg/mL de estreptomicina a 37 °C em uma incubadora umidificada contendo 5% de CO2.
- Cultura Células HTLA em pratos de 150 mm e passam células duas vezes por semana em um fator de diluição de 1:10, com número ideal de passagem celular de 5-25. Certifique-se de que um número suficiente de pratos de 150 mm são confluentes no dia da semeada de placas de 384 poços, dependendo da escala da tela primária.
NOTA: O uso de células HTLA maiores que a passagem 25 pode resultar em viabilidade reduzida, produzindo resultados subótimos. - Para semear células HTLA para a tela primária, enxágue suavemente o prato confluente de 150 mm com 1x de solução salina tamponada por fosfato (PBS), pH 7.4. Desconecte as células com aproximadamente 6 mL de 0,05% de Trypsin/0,53 mM EDTA, e transfira para um tubo de centrífuga contendo pelo menos a mesma quantidade do meio águia modificado (DMEM) de Dulbecco completo para neutralizar a tripsina.
- Gire as células HTLA a 500 x g por 3 min e resuspenda a pelota celular a uma densidade de 0,22 x 106 células/mL em DMEM completo, omitindo a adição de 2,5 μg/mL de puromicina e 50 μg/mL de higrifina, pois podem diminuir a eficácia da transfecção.
- Incubar as placas revestidas de PLL de 384 poços a 37 °C para aquecê-las antes de semea-las. Remova a solução de armazenamento de 1x antimicótico antibiótico da placa revestida de PLL de 384 poços, apertando a placa sobre a pia e batendo-a sobre uma toalha de papel para secar.
- Células de sementes nas placas revestidas de PLL de 384 poços a uma densidade final de 10.000 células/poço, dispensando 45 μL da suspensão 0,22 x 106 células/mL HTLA usando um tubo multicanal eletrônico. Incubar placas a 37 °C durante a noite. Se uma transfecção no mesmo dia for preferível, as células de sementes com uma densidade de 16.000 células/bem e realizam a transfecção 4h depois.
NOTA: Para alta eficiência de transfecção, a confluência celular de 50 a 70% é ótima.
2. Triagem primária: preparação e transfecções de placas de DNA
- Para preparar a placa de origem de DNA de 384 poços para transfecção, conforme mostrado na Figura 2,distribua os cDNAs plasmáticos que codificam as construções GPCR-Tango de interesse em uma placa de 96 poços, com um GPCR/poço diferente. O DNA plasmático deve ser suspenso em tampão tris-EDTa (TE) de 0,1x a uma concentração de 50 ng/μL.
NOTA: As placas de DNA de 96 poços podem ser seladas e armazenadas a -20 °C, e reutilizadas para múltiplos experimentos de triagem. Todas as construções GPCR-Tango de codificação de cDNA estão disponíveis comercialmente (veja a Tabela de Materiais) e são clonadas no plasmídeo de neomicina pcDNA3.1. O Kit GPCR PRESTO-Tango consiste em quatro placas de 96 poços, que incluem 80 GPCRs cada, um par de poços com um vetor vazio como controles negativos, e poços de controle positivos que seguram o receptor de dopamina D2 (DRD2), e poços que carregam um plasmídeo codificando uma proteína fluorescente (YFP) para rastrear a eficiência da transfecção. - Usando uma pipeta multicanal, transfira manualmente a solução de DNA do poço de 96 para uma placa de fonte de DNA de 384 poços, adicionando 10 μL por 384-well. Para garantir que cada condição do experimento seja ensaio em quadruplicado, metade da placa de DNA de 96 poços (linhas A-D ou E-H) cobrirá uma placa completa de 384 poços distribuindo cada GPCR em dois quadrantes (primeiro quadrante = - composto, segundo quadrante = + composto), de tal forma que o mesmo GPCR será transfeccionado em 8 poços da placa de 384 poços (ver Figura 2 como guia).
- Montar os seguintes reagentes de transfecção necessários para o método de precipitação do fosfato de cálcio, conforme descrito por Jordan et al.20: 0,1x tampão TE (1 mM Tris-HCl e 0,1 mM EDTA); Solução CaCl2 de 2,5 M; 2x tampão hepes, pH 7.05 (50 mM HEPES, 280 mM NaCl, 1,5 mM Na2HPO4). Esterilize todas as soluções por filtragem e armazene a 4 °C. No dia da transfecção, permita que os reagentes atinjam a temperatura ambiente antes do uso.
- Diluir a solução de estoque cacl2 de 2,5 M em 0,1x TE (diluição 1:8) para uma concentração final de 0,313 M CaCl2 e vórtice. Transfira 40 μL de 0,313 M CaCl2 para a placa de fonte de DNA de 384 poços e misture-se encantando para cima e para baixo com uma pipeta multicanal portátil ou um pipettor de bancada automatizado de 384 canais.
- Adicione 50 μL de tampão de hepes 2x à placa de origem de DNA de 384 poços, misture novamente encantando para cima e para baixo e deixe ficar por 1 min; cada 384 poços terá uma quantidade adequada de mistura de DNA/transfecção para a transfecção de nove placas de 384 poços, dependendo do número de compostos que precisam ser testados. Transfira 10 μL da mistura de DNA/transfecção da placa de origem do DNA de 384 poços para as células HTLA semeadas e incubar as placas durante a noite a 37 °C.
3. Triagem primária: Estimulação celular
- Vinte e quatro horas depois, decante a mídia celular transfeccionada, apertando suavemente a placa de 384 poços sobre a pia e gravando-a sobre uma toalha de papel, ou com uma cabeça de aspirador. Adicione lentamente 40 μL de mídia faminta (DMEM suplementado com 1% de soro bovino fetal dialisado (dFBS) e 1x antibiótico/antimicótico), tomando cuidado para evitar tocar diretamente nas células.
- Pipet 20 μL do composto de juros a uma concentração de 3x (a concentração final da droga na placa celular será de 1x) nas linhas alternadas com (+) estimulação, e 20 μL de tampão do veículo para as linhas alternadas sem (-) composto. Volte a placa celular a 37 °C em 5% CO2 e incubar por pelo menos 16 horas.
4. Triagem primária: Leitura de luminescência
- Prepare o reagente Glo, modificado de Baker e Boyce21: 108 mM Tris-HCl; 42 mM Tris-Base, 75 mM NaCl, 3 mM MgCl2, 5 mM Dithiothrei-tol (DTT), 0,2 mM Coenzyme A, 0,14 mg/ml D-Luciferin, 1,1 mM ATP, 0,25% v/v Triton X-100, 2 mM Hidrosulfite de sódio.
NOTA: As soluções de estoque dos reagentes podem ser feitas com antecedência, exceto a D-Luciferin, que é sempre recém-adicionada ao reagente GLO em sua forma em pó. Se foram utilizadas placas pretas, a quantidade de D-Luciferin pode ser aumentada até 0,25 mg/mL. - Às 16-24 horas após a estimulação, decante a mídia celular transinfectada, apertando suavemente a placa de 384 poços sobre a pia e gravando-a sobre uma toalha de papel. Adicione 20 μL/poço de reagente Glo e incubar a placa em temperatura ambiente por 5-20 min. Leia as placas usando um contador de luminescência de microplacas, com um tempo de integração de 1 s/well.
5. Triagem primária: Análise de dados
- Exportar os arquivos salvos do contador de luminescência como uma planilha; os resultados serão registrados em unidades de luminescência relativa (RLU). Com base no layout da placa de 384 poços, calcule a ativação (mudança de dobra) de cada receptor usando a seguinte fórmula:
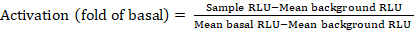
NOTA: Aqui a Amostra RLU refere-se ao valor de cada um dos quatro poços replicados do quadrante estimulado (+ composto), RLU médio de fundo é a média dos controles negativos na placa, e RLU basal médio refere-se à média do quadrante não tratado desse mesmo receptor (- composto). Além disso, calcule o desvio padrão dos 4 pontos de dados para verificar a qualidade dos resultados. Recomenda-se realizar uma transformação log2 na média das alterações da dobra para corrigir qualquer heteroskedasticidade; a base log2 é uma escolha prática para ajudar a identificar hits positivos. Empiricamente definir os limiares positivos de acerto; deve-se notar que alguns receptores podem ter um aumento de até 2 vezes e até 40 vezes para outros com agonista completo. - Com base nos resultados, selecione os GPCRs que são potenciais acertos positivos para a triagem secundária.
6. Triagem secundária: Semeamento celular e transfecções
- Subcultura Células HTLA em pratos de 100 mm a uma densidade celular total de 5 x 106 células em 11 mL de mídia completa (4,55 x 105/mL) e incubam a 37 °C por 24 horas. Se uma transfecção no mesmo dia for preferível, as células de sementes com uma densidade de 7,5 x 106 células e realizam a transfecção 4h depois.
- Pré-aquecimento dos reagentes necessários para precipitação de fosfato de cálcio à temperatura ambiente. Combine 450 μL de tampão TE 0,1x com 50 μL de 2,5 M CaCl2 e vórtice rápido; essas quantidades são específicas para um prato de 100 mm, com base no volume de meio de crescimento que ele mantém.
- Em um tubo, adicione 500 μL da solução TE/CaCl2 a 10 μg de cDNA GPCR e vórtice. Adicione 500 μL de solução tampão de hepes 2x no tubo, agite vigorosamente (não vórtice) e incuba r$ 1 min.
NOTA: 1 μg de qualquer codificação plasmídea de uma proteína fluorescente (por exemplo, YFP, mCherry, etc.) pode ser co-transfect com 9 μg de cDNA GPCR para um total de 10 μg. A proteína fluorescente é usada para rastrear a eficiência da transfecção, e essa quantidade mínima não interferirá com o ensaio. - Imediatamente após a curta incubação, dispense a solução de 1 mL dropwise sobre as células. Balance suavemente a placa para frente e para trás para distribuir uniformemente o precipitado, tomando cuidado para não girar a placa, e incubar a 37 °C por 24 horas.
- No dia seguinte, observe a eficiência da transfecção olhando para a expressão da proteína fluorescente um imager de células fluorescentes; transfecções superiores a 50% de cobertura são ideais.
- Incubar as placas revestidas de PLL de 384 poços na incubadora a 37 °C para aquecê-la antes de semea-la. Remova a solução de armazenamento de 1x antimicótico antibiótico da placa revestida de PLL de 384 poços, apertando a placa sobre a pia e batendo-a sobre uma toalha de papel para secar.
- Enxágüe suavemente as células transfeccionadas com solução verseno (1X PBS, pH 7.4; 0.53 mM EDTA) e desconecte adicionando 3 mL de 0,05% de tripsina/0,53 mM EDTA ao prato. Transfira o conteúdo para um tubo de centrífuga contendo pelo menos uma quantidade igual de DMEM completo para neutralizar a tripsina.
- Gire as células a 500 x g por 3 min e resuspenda as células a uma densidade de 0,4 x 106 células/mL em mídia faminta. Células de sementes nas placas revestidas de PLL de 384 poços a uma densidade final de 25.000 células/poço, dispensando 45 μL da suspensão celular usando um tubo eletrônico multicanal. Retorne as placas aos 37 °C por um mínimo de 4h, permitindo que as células se fixem corretamente aos poços antes de prosseguirem para a estimulação.
7. Triagem secundária: Preparação da placa de drogas para curva de dose de 16 pontos (meio log)
- Em uma placa de 96 poços, adicione 270 μL de tampão de droga 1X HBSS (1x Hank's Balanced Salt Solution [HBSS], 20 mM HEPES pH 7.4, 1x antibiótico-antimicotic), excluindo a última linha (linha H) da placa, conforme mostrado na Figura 4.
NOTA: Para peptídeos, moléculas coloidais e compostos solúveis em água, sugere-se a adição de 0,1-1% de BSA. Para prevenir a oxidação de drogas, até 0,01% de ácido ascórbico também pode ser adicionado. - A partir do estoque de drogas, prepare uma solução de droga (chamada de concentração "Alta") calculando uma concentração final de 3x (a concentração final da droga na placa celular será de 1x). Como exemplo, para uma curva de dose-resposta com 10 μM como sua maior concentração, prepare a concentração "Alta" a 30 μM. Pipet 300 μL de concentração "Alta" em poços na linha H.
- Em outro tubo, prepare a concentração "Baixa", que representa a concentração "Alta" dividida por 3,16 (meio-tronco). Com base no exemplo anterior, a concentração "Baixa" seria de 9,49 μM. Pipet 300 μL de concentração "Baixa" em poços na linha H, adjacentes aos poços "High".
NOTA: O número total de 96 poços necessários na linha H dependerá do número de células e condições de estimulação. Quatro poços (dois "Alto" e dois "Baixo") terão ampla solução de drogas para estimular uma placa de poço 384 inteira. - Realize uma diluição serial pipetando 30 μL de solução de droga dos poços "Alto" e "Baixo" da linha H até a linha anterior (linha G) e misture manualmente pipetando para cima e para baixo, ou como recomendado, usando uma pipeta multicanal eletrônica com a função "Pipette e Mix". Repita este passo até a primeira e mais diluída linha (linha A), enquanto descarta as pontas entre as diluições.
NOTA: Se desejar, as diluições seriais podem ser interrompidas antes da linha A, representando um controle interno sem droga, ou seja, um "verdadeiro zero". - Utilizando a Figura 4 como referência, estimule as células transfeccionadas pipetando 20 μL das diluições da coluna "Baixa" da placa de 96 poços para as linhas A-O da placa de 384 poços previamente semeada, bem como 20 μL das diluições da coluna "Alto" para poços B-P. Incubar a placa a 37 °C para um mínimo de 16 h.
8. Triagem secundária: Leitura de luminescência e análise de dados
- Às 16-24 horas após a estimulação, decante a mídia celular transinfectada, apertando suavemente a placa de 384 poços sobre a pia e gravando-a sobre uma toalha de papel. Adicione 20 μL/poço de reagente Glo e incubar a placa em temperatura ambiente por 5-20 min. Leia as placas usando um contador de luminescência de microplacas, com um tempo de integração de 1 s/well.
- Exportar os arquivos salvos do contador de luminescência como uma planilha; os resultados serão registrados em unidades de luminescência relativa (RLU). Transfira os dados da placa de 384 poços para um software estatístico para analisar os resultados usando sua análise XY incorporada para ajuste de curva de regressão não linear. Selecione a função de estimulação de dose-resposta incorporada de 3 parâmetros "Log(agonist) vs. response (três parâmetros)",

NOTA: Aqui Topo e Baixo são platôs nas unidades do eixo Y, respectivamente a resposta máxima e o nível basal, EC50 é a concentração do agonista que gera 50% de resposta entre superior e inferior, e X refere-se à concentração de tronco do agonista. Este modelo assume que a curva dose-resposta tem uma inclinação padrão de Colina de 1.
Resultados
Utilizando-se o protocolo PRESTO-Tango aqui apresentado, um extrato de granulo de cromafina (CG) foi examinado contra 168 alvos não olfativos do GPCR, sendo a maioria receptores órfãos. O perfil do referido extrato foi realizado examinando a mobilização β-arrestin2 nos receptores escolhidos, com base no princípio desenhado por Barnea et al.18 (Figura 1). O cDNA plasmático dos GPCRs de interesse foi retirado do Kit GPCR PRESTO-Tango e montado em duas 96 placas-...
Discussão
Os GPCRs conformacionalmente dinâmicos são potências de transdução de sinal. As propriedades fisioquímicas dos bolsos de ligação desses receptores heptaéricos, bem como sua relevância fisiológica ressaltam a necessidade de ferramentas de triagem de ligantes GPCR. Como apresentado acima, o ensaio PRESTO-Tango é rápido, sensível e fácil de usar, emprestando-se ao desenvolvimento de medicamentos. Este ensaio não só mede a ativação induzida por agonistas, mas também pode ser usado para quantificar a ativi...
Divulgações
As authoras não declaram interesses concorrentes.
Agradecimentos
Este trabalho foi apoiado pelos Institutos Canadenses de Pesquisa em Saúde (CIHR grant #MOP142219).
Materiais
| Name | Company | Catalog Number | Comments |
| 384 Well Optical Bottom Plates, Polystyrene Polymer Base, Cell Culture Treated, BLACK, with lid, Sterile | NUNC | 12-566 | |
| 384 Well Optical Bottom Plates, Polystyrene Polymer Base, Cell Culture Treated, White, with lid, Sterile | NUNC | 12-566-1 | |
| 384 Well Round Bottom, Polypropylene, Non-Treated, Blue, non-sterile, without lid | ThermoFisher | 12-565-390 | |
| Antibiotic-Antimycotic | Wisent | 450-115-EL | |
| D-Luciferin, sodium salt | GoldBio | LUCNA | |
| DMEM with L-Glutamine, 4.5g/L Glucose and Sodium Pyruvate | Corning | 10-013-CV | |
| Eppendorf Xplorer, 12-channel, variable, 15–300 µL | Eppendorf | 4861000155 | |
| Eppendorf Xplorer, 12-channel, variable, 5–100 µL | Eppendorf | 4861000139 | |
| Matrix Platemate 2x3 | ThermoFisher | 801-10001 | |
| MicroBeta 1450 Wallac | Perkin Elmer | ||
| Penicilin-Streptomycin | Wisent | 450-201-EL | |
| Poly-L-Lysine hydrobromide | Millipore-Sigma | P2636-500MG | |
| Roth Lab PRESTO-Tango GPCR Kit | Addgene | Kit #1000000068 |
Referências
- Liapakis, G., et al. The G-protein coupled receptor family: actors with many faces. Current pharmaceutical design. 18 (2), 175-185 (2012).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nature Reviews Molecular Cell Biology. 3 (9), 639-650 (2002).
- Kroeze, W. K., Sheffler, D. J., Roth, B. L. G-protein-coupled receptors at a glance. Journal of cell science. 116, 4867-4869 (2003).
- Rask-Andersen, M., Almén, M. S., Schiöth, H. B. Trends in the exploitation of novel drug targets. Nature Reviews Drug Discovery. 10 (8), 579-590 (2011).
- Ngo, T., et al. Identifying ligands at orphan GPCRs: current status using structure-based approaches. British journal of pharmacology. 173 (20), 2934-2951 (2016).
- Zhang, R., Xie, X. Tools for GPCR drug discovery. Acta pharmacologica Sinica. 33 (3), 372-384 (2012).
- Kimple, A. J., Bosch, D. E., Giguere, P. M., Siderovski, D. P. Regulators of G-Protein Signaling and Their G Substrates: Promises and Challenges in Their Use as Drug Discovery Targets. Pharmacological Reviews. 63 (3), 728-749 (2011).
- Wettschureck, N., Offermanns, S. Mammalian G Proteins and Their Cell Type Specific Functions. Physiological Reviews. 85 (4), 1159-1204 (2005).
- Denis, C., Saulière, A., Galandrin, S., Sénard, J. -. M., Galés, C. Probing heterotrimeric G protein activation: applications to biased ligands. Current Pharmaceutical Design. 18 (2), 128-144 (2012).
- Yin, H., et al. Lipid G protein-coupled receptor ligand identification using beta-arrestin PathHunter assay. The Journal of Biological Chemistry. 284 (18), 12328-12338 (2009).
- Cheng, Z., et al. Luciferase Reporter Assay System for Deciphering GPCR Pathways. Current Chemical Genomics. 4, 84-91 (2010).
- Roth, B. L., Kroeze, W. K. Integrated Approaches for Genome-wide Interrogation of the Druggable Non-olfactory G Protein-coupled Receptor Superfamily. The Journal of Biological Chemistry. 290 (32), 19471-19477 (2015).
- Jean-Charles, P. -. Y., Kaur, S., Shenoy, S. K. G Protein-Coupled Receptor Signaling Through β-Arrestin-Dependent Mechanisms. Journal of Cardiovascular Pharmacology. 70 (3), 142-158 (2017).
- Smith, J. S., Rajagopal, S. The β-Arrestins: Multifunctional Regulators of G Protein-coupled Receptors. The Journal of Biological Chemistry. 291 (17), 8969-8977 (2016).
- Böhme, I., Beck-Sickinger, A. G. Illuminating the life of GPCRs. Cell Communication and Signaling. 7 (1), 16 (2009).
- Fang, Y. Label-Free Receptor Assays. Drug discovery today. Technologies. 7 (1), 5-11 (2011).
- Wang, T., et al. Measurement of β-Arrestin Recruitment for GPCR Targets. Assay Guidance Manual. Eli Lilly & Company and the National Center for Advancing Translational Sciences. , (2004).
- Barnea, G., et al. The genetic design of signaling cascades to record receptor activation. Proceedings of the National Academy of Sciences of the United States of America. 105 (1), 64-69 (2008).
- Kroeze, W. K., et al. PRESTO-Tango as an open-source resource for interrogation of the druggable human GPCRome. Nature Structural & Molecular Biology. 22 (5), 362-369 (2015).
- Jordan, M., Schallhorn, A., Wurm, F. M. Transfecting Mammalian Cells: Optimization of Critical Parameters Affecting Calcium-Phosphate Precipitate Formation. Nucleic Acids Research. 24 (4), 596-601 (1996).
- Baker, J. M., Boyce, F. M. High-throughput functional screening using a homemade dual-glow luciferase assay. Journal of Visualized Experiments. (88), 50282 (2014).
- Li, H., Eishingdrelo, A., Kongsamut, S., Eishingdrelo, H. G-protein-coupled receptors mediate 14-3-3 signal transduction. Signal Transduction and Targeted Therapy. 1 (1), 16018 (2016).
- Walther, C., Ferguson, S. S. G. Minireview: Role of intracellular scaffolding proteins in the regulation of endocrine G protein-coupled receptor signaling. Molecular Endocrinology. 29 (6), 814-830 (2015).
- Gurevich, E. V., Benovic, J. L., Gurevich, V. V. Arrestin2 expression selectively increases during neural differentiation. Journal of Neurochemistry. 91 (6), 1404-1416 (2004).
- Shaikh, S., Nicholson, L. F. B. Optimization of the Tet-On system for inducible expression of RAGE. Journal of Biomolecular Techniques JBT. 17 (4), 283-292 (2006).
- Blau, H. M., Rossi, F. M. Tet B or not tet B: advances in tetracycline-inducible gene expression. Proceedings of the National Academy of Sciences of the United States of America. 96 (3), 797-799 (1999).
- Roche, O., et al. Development of a Virtual Screening Method for Identification of "Frequent Hitters" in Compound Libraries. Journal of Medicinal Chemistry. 45 (1), 137-142 (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados