É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Medindo complexos de transferência de elétrons mitocondrial em tecido cardíaco previamente congelado da prole da porca: um modelo para avaliar alterações bioenergetics mitocondrial induzidas pelo exercício
Neste Artigo
Resumo
A preparação de amostras enriquecidas com mitocôndrias de tecidos sólidos arquivados previamente congelados permitiu que os pesquisadores realizassem avaliações funcionais e analíticas de mitocôndrias em várias modalidades experimentais. Este estudo demonstra como preparar preparações enriquecidas com mitocôndrias a partir de tecido cardíaco congelado e realizar avaliações analíticas de mitocôndrias.
Resumo
O perfil do complexo de transferência de elétrons mitocondrial (ETC) é modificado no tecido cardíaco da prole nascido em uma porca exercida. A hipótese proposta e testada foi que um exercício materno regular de uma porca durante a gravidez aumentaria a eficiência mitocondrial da bioenergetic do coração da prole. Essa hipótese foi testada por mitocôndrias isolando utilizando um procedimento de isolamento leve para avaliar perfis mitocondriais etc e supercomplexo. O procedimento aqui descrito permitiu o processamento de tecidos cardíacos arquivados previamente congelados e eliminou a necessidade de preparação de mitocôndrias frescas para a avaliação de complexos etc mitocondrial, supercomplexos e perfis de atividades complexas etc. Este protocolo descreve a medição ideal do complexo proteico ETC em imunoblotting baseado em anticorpos multiplexados e avaliação super complexa usando eletroforese de gel azul-nativa.
Introdução
O objetivo deste protocolo foi fornecer passos detalhados para obter a preparação enriquecida com mitocôndrias a partir de tecido cardíaco previamente congelado com uma nova tecnologia de interrupção mecânica de baixa energia do tecido que melhora a lise tecidual e a extração de mitocôndrias. Com este método, a melhoria da eficiência extração sem gerar alto estresse de tesoura ou alta temperatura e curto tempo de homogeneização (10-12 s) tornam-se alcançáveis1.
Obter mitocôndrias a partir de tecido congelado arquivado é um ativo valioso para realizar estudos funcionais2 e bioquímicos3, caso contrário, não facilmente repetível sob as condições experimentais exatas. Um clássico homogeneizador de vidro de pilão Potter-Elvehjem Teflon ou homogeneizador de dounce tem sido usado e ainda está sendo usado em laboratórios de pesquisa para homogeneizar tecidos moles como fígado, rim e cérebro. No entanto, homogeneizar tecidos duros como músculo e coração requer mais tempo de homogeneização, tratamento enzimático, homogeneização de alta velocidade e ampla experiência do usuário. Isso é desvantajoso para extrair organelas intactas, como mitocôndrias de tecido duro, como músculo e coração. O método descrito neste protocolo é usado para obter uma preparação enriquecida com mitocôndrias de alto rendimento para analisar complexos proteicos da cadeia de transporte de elétrons mitocondrial (ETC) e sua formação supercomplexa em tecidos cardíacos colhidos de descendentes nascidos a porcas exercitadas e sedentárias, congelados em nitrogênio líquido e armazenados a -80 °C para uso futuro. Este método permite que o usuário isole a preparação enriquecida de mitocôndrias de tecidos arquivados previamente congelados.
A exposição nanomaterial externa a roedores gestantes pode afetar negativamente a função cardíaca e a respiração mitocondrial e bioenergésicos na prole durante a gestação4. No entanto, as alterações positivas induzidas pelo exercício aeróbico nos bioenergésicos do miócito fetal durante a gravidez ainda não foram documentadas. No entanto, estudos emergentes fornecem evidências de que o exercício aeróbico materno durante a gravidez tem uma influência positiva na função cardíaca fetal5. Para fornecer mais evidências, foi realizada uma análise dos efeitos longitudinais do exercício materno nos complexos da cadeia respiratória mitocondrial cardíaca (ou seja, Complexo I através do Complexo V) durante a gravidez.
Este estudo tem relevância significativa para a saúde, uma vez que os resultados podem sugerir que o exercício materno melhora a eficiência da produção de energia nas mitocôndrias cardíacas da prole. Neste estudo, as porcas (porcas fêmeas) foram utilizadas como modelo animal por duas razões: (i) a fisiologia cardíaca é semelhante à humana6, e (ii) a colheita de tecido cardíaco de descendentes de diferentes pontos de tempo é viável sob aprovação institucional da IACUC. O estudo proposto tem como objetivo responder a muitas das questões fundamentais que ligam o exercício materno e seus potenciais efeitos positivos na composição celular e bioquímica do tecido cardíaco da prole. Esta abordagem requer técnicas suaves, mas eficazes de isolamento de mitocôndrias de tecido cardíaco previamente congelado obtido a partir dos longos e caros estudos longitudinais que abordaram as questões das alterações bioenergésticas dentro dos miócitos cardíacos fetais durante a gravidez. O método descrito neste estudo permite utilizar grandes somas de tecido arquivado previamente congelado para preparação enriquecida com mitocôndrias para estudos analíticos e funcionais. O estudo também ajudará a preencher a lacuna de conhecimento nesse campo, fornecendo dados preliminares, o que poderia levar a estudos futuros determinando os efeitos do exercício materno sobre a saúde do coração no útero e além.
Protocolo
Os tecidos cardíacos da prole congelada foram recebidos do Dr. Sean Newcomer juntamente com a carta de aprovação institucional da IACUC. Os tecidos cardíacos foram obtidos a partir de um estudo longitudinal de longo prazo, congelados em nitrogênio líquido, e armazenados a -80 °C para uso futuro. Todos os protocolos relativos ao processamento de tecido cardíaco da prole seguiram as diretrizes dos comitês da Universidade de Kansas City IBC e da IACUC.
1. Preparação de tampões e reagentes
NOTA: Prepare todas as amostras de acordo com as diretrizes do fabricante. Use água ultra-purificada ou equivalente em todas as receitas e etapas de protocolo. Use equipamentos de proteção pessoal (gloats de laboratório, máscara facial, luvas e óculos/escudo facial) durante este procedimento. Os volumes de buffer são adequados para o processamento de seis amostras de tecido.
- Prepare 1,5 L tampão de lavagem de tecido combinando 154,035 g de sacarose (final de 0,3 M), 3,904 g de HEPES (final de 10 mM), 0,111 g de EDTA (final de 0,2 mM). Adicione os ingredientes a 1,0 L de água enquanto estiver na placa de mexida, ajuste o pH para 7,2 com HCl diluído e maquiagem para o volume final a 1,5 L.
NOTA: São necessários aproximadamente 200 mL de tampão de lavagem por amostra (50-300 mgs). Seis amostras de tecido podem ser processadas em um dia. Use o tampão de lavagem restante para fazer um tampão de isolamento. - Prepare o tampão de isolamento do tampão de lavagem descrito anteriormente na etapa 1.1 adicionando 210 mg de BSA em 210 mL de tampão de lavagem. Ajuste o pH para 7.4 com solução NaOH diluída.
NOTA: Aproximadamente 35 mL de tampão de isolamento são necessários por amostra de tecido; portanto, 210 mL de tampão de isolamento são necessários para seis amostras. Prepare este buffer adicionando nova solução BSA (final de 1mg/mL) no dia da preparação. A BSA é usada para isolar mitocôndrias à medida que melhora as propriedades mitocondriais removendo desacopladores naturais, como ácidos graxos livres. Verificou-se que a BSA tem atividade antioxidante aumentando a taxa de oxidação de substratos, especialmente o succinato, nas mitocôndrias cerebrais7. Alternativamente, a albumina de soro bovino pode ser substituída por análogos sem ácido mais baratos, como ovalbumin. - Prepare 150 mL de tampão de resuspensão combinando 0,011 g de EDTA (concentração final de 0,2 mM EDTA ou use 150 μL de 200 mM de estoque EDTA solução para 150 mL de tampão de resuspensão), 12,836 g de sacarose (concentração final de sacarose de 0,25 M) e 0,236 g de Tris (final tris-HCl de 10 mM). Ajuste o pH para 7,8 com NaOH de 0,1 N. Adicione 60 μL do coquetel inibidor de protease pouco antes de usar.
- Prepare a solução de trypsin dissolvendo 7,5 mg de trippsina em 3,0 mL de 1 mM HCl (2,5 mg de trippsin/mL final). Esta quantidade é suficiente para uma amostra.
NOTA: Prepare a solução de trippsina com base no número de amostras de tecido previstas para serem processadas. - Prepare a solução inibidora de trippsina da soja combinando 13,0 mg de inibidor de trippsina em 20 mL de tampão de isolamento contendo BSA (0,65 mg/mL de inibidor de trippsina final).
NOTA: Prepare uma nova solução inibidora de trippsina. - Prepare o reagente persulfeto de amônio adicionando 0,1 g de persulfito de amônio em 1 mL de água (final de persulfeto de amônio de 10%).
NOTA: O reagente persulfeto de amônio deve ser recém-preparado ou um grande volume pode ser aliquotado e mantido em um congelador de -20 °C. - Prepare a solução de ácido aminocagóico (ACA) combinando 0,131 g de ácido capróico 6-amino em 1,0 mL de água ultra-purificada e armazene-a em 4 °C (1 M 6-aminocaproic acid final).
- Prepare a solução de corante azul brilhante Coomassie dissolvendo 1 g de Coomassie G-250 em 0,5 mL de água, e 0,5 mL de ácido aminocaproico de 1 M (concentração final de solução de corante de 10%).
- Prepare 5% Digitonin para Blue native-PAGE (BN-PAGE). Calcule aproximadamente quanto digitonin é necessário na preparação da amostra mais algum volume extra para compensar os erros de pipetação (superestimar em 10 mgs). Para 15 mgs: Primeiro, diluir 15 mg de digitonina em 30 μL de 100% DMSO. Vórtice para ajudar o digitonin a entrar na solução. Em seguida, adicione os 90% restantes do ddH2O (270 μL). Aqueça suavemente para misturar a solução (vórtice) e depois esfrie-a no gelo.
- Prepare o buffer de ânodo page nativo. Adicione 50 mL de buffer de execução de 20x nativo PAGE a 950 mL de água para fazer um volume final de 1 L. Prepare o tampão de ânodo fresco para uso imediato. Use isso como um buffer de corrida de 1x.
- Prepare um tampão de cátodo azul escuro. Dissolver 0,0160 g de Coomassie brilhante azul G-250 em 80 mL de tampão de ânodo page nativo; misturar bem.
NOTA: Prepare um novo tampão de cátodo para uso imediato. Apenas 60 mL podem ser necessários, mas fazer um extra de 20 mL no caso de qualquer vazamento. - Prepare o tampão de cátodo azul claro. Adicione 20 mL de tampão de cátodo azul escuro em 180 mL de tampão de ânodo page nativo e misture bem.
NOTA: Este buffer só pode ser usado uma vez. - Prepare 5% Coomassie G-250 como aditivo amostral. Diluir 200 μL de 10% de estoque coomassie já feito (passo 1.8) com 200 μL de 1 M ACA e armazenar a 4 °C.
- Prepare o tampão de amostra de 4x nativo PAGE combinando 40% glicerol, Ponceau S 0,04%, 25 mM Tris-HCl (pH 6.8), 192 mM de glicecina em 10 mL de volume final. Aliquot-los e armazenar a -20 °C no congelador.
2. Isolamento mitocôndria do tecido cardíaco congelado
NOTA: Realize o procedimento de isolamento das mitocôndrias em uma sala fria de 4 °C. No entanto, em caso de indisponibilidade da sala fria, use um banho de gelo de grande porte para realizar o procedimento.
- Retire amostras previamente congeladas de tecido cardíaco do congelador de -80 °C.
NOTA: Um máximo de quatro amostras de tecido pode ser confortavelmente processado para isolamento de mitocôndrias em um determinado dia. - Pese o tubo contendo a amostra do tecido cardíaco e regissue o peso. Prepare três béquers (cada 50 mL) contendo 40 mL de tampão de lavagem. Esfrie cada béquer no banho de sal gelado com quantidades iguais de gelo e sal misturados por 3-5 minutos antes de prosseguir para o próximo passo. Coloque as barras de agitação magnética em cada béquer e coloque o agitador em velocidade média.
NOTA: Não congele demais os béquers e use-os imediatamente logo após nuvens de cristais de gelo começarem a aparecer. - Coloque o tecido cardíaco congelado diretamente na solução gelada no primeiro béquer. Aguarde aproximadamente 5 minutos enquanto mexe e aplique uma leve pressão no tecido usando pinças para sair o máximo de sangue possível.
- Pesar o tubo vazio e subtrair do peso original (passo 2.2) para obter um peso líquido do tecido cardíaco.
- Retire o tecido com fórceps e esprema o coração com uma toalha de papel limpa de papel filtro para remover o sangue. Em seguida, coloque o tecido no segundo béquer. Aguarde aproximadamente 5 minutos enquanto mexe para remover o excesso de sangue.
- Seque suavemente o tecido em uma toalha de papel. Remova toda a gordura, sangue coagulado, aurículos e fáscia (tecido branco). Em seguida, laça o tecido ventricular.
- Desfie as amostras de tecido usando o triturador seguindo as etapas 2.7.1-2.7.9.
- Coloque o tecido cardíaco (~50-300 mgs) na câmara de triturador de tecido do lado do carneiro.
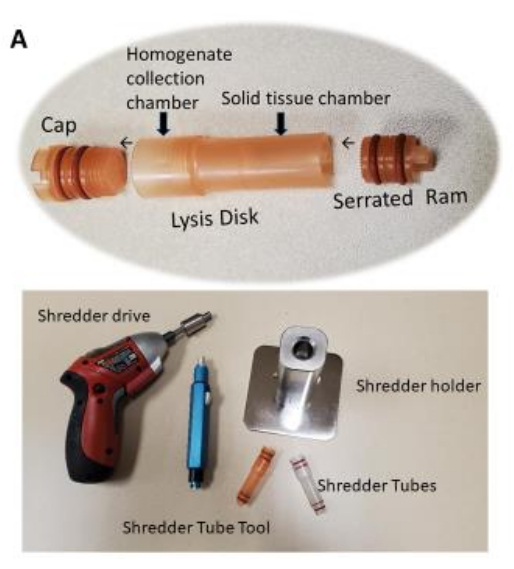
Figura 1: Câmara de triturador de tecido (tubo) para uso com o triturador. A homogeneização tecidual no tubo de triturador requer cerca de 10-12 s por amostra de tecido. A homogeneização do tecido é concluída uma vez que o tecido homogeneizado passa pelo disco perfurado para a câmara superior. Clique aqui para ver uma versão maior desta figura.
- Adicione ~0,2-0,8 mL de tampão de lavagem ao lado da tampa do tubo.
NOTA: Certifique-se de que o volume total da amostra e do tampão de lavagem combinados durante a trituração não exceda 0,7-0,8 mL. - Use a ferramenta do tubo para tampar a câmara de triturador com a tampa do triturador.
NOTA: Não aperte demais a tampa do triturador. - Coloque a câmara de triturador na unidade do suporte do triturador com o lado do carneiro para baixo. O carneiro vai engatar um encaixe no suporte do triturador.
- Inserir o pedaço do condutor do triturador; deixe a broca se envolver com a tampa do triturador. Certifique-se de que a broca está presa no lugar.
NOTA: Use sempre o condutor do triturador totalmente carregado. - Aplique manualmente pressão para baixo no condutor do triturador e na câmara de trituração até que o indicador de pressão no suporte do triturador atinja uma configuração de aproximadamente 2 no suporte do triturador.
NOTA: A configuração pode ser em um nível mais alto se o tecido desfiante for mais fibroso. - Execute o condutor do triturador por aproximadamente 10-12 s pressionando o gatilho do condutor do triturador, mantendo a configuração em 2.
- Olhe para o tubo para verificar se toda ou a maior parte da massa sólida do tecido é triturada através do disco de lise e passada para a câmara superior da câmara de triturador olhando para o tubo.
NOTA: Pode ser necessário triturar a amostra de tecido por um período mais longo. Basta devolver o tubo ao suporte do triturador e continuar o processo. A maioria dos tipos de amostras de tecido requerem apenas 10-12 s de trituração. Embora toda a trituração produza algum calor devido ao atrito, este curto tempo de processamento não aquecerá significativamente a amostra. - Depois de triturar a amostra, retire a câmara de triturador da unidade de suporte. Use a ferramenta do tubo para remover a tampa do triturador e coloque-a em uma superfície limpa para uso posterior.
NOTA: A tampa do triturador deve ser considerada contaminada com extrato amostral, e dependendo do tipo de amostra, pode estar contaminada com materiais infecciosos. Não use a mesma tampa de triturador para quaisquer outras amostras de tecido para evitar contaminação cruzada.
- Transfira o tecido cardíaco desfiado para o terceiro béquer com 40 mL de tampão de lavagem e mexa a barra e mexa o tecido por 5 minutos a uma velocidade média enquanto estiver na placa de mexida.
- Filtre a suspensão através de um monofilamento de malha de nylon (tamanho de poros de 74 μm) e descarte o filtrado. Lave o tecido desfiado no filtro 3x com 10 mL de tampão de lavagem.
NOTA: Certifique-se de que o monofilamento da malha de nylon está pré-molhado com água. - Transfira o tecido lavado e desfiado em um béquer de 50 mL com 20 mL de tampão de lavagem e coloque o béquer no banho de gelo no agitador magnético. Adicione 0,5 mL de solução de trippsina enquanto mexe constantemente por 15 min. Aproximadamente na metade da incubação (cerca de 7,5 min), transfira a suspensão do tecido para um homogeneizador portátil de vidro em vidro que conterá de 0 a 15 mL de volume.
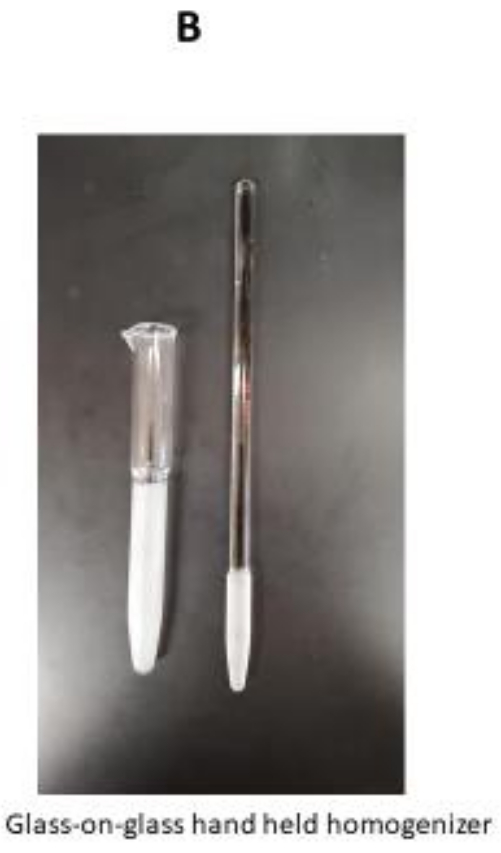
Figura 1: Câmara de triturador de tecido (tubo) para uso com o triturador. A homogeneização tecidual no tubo de triturador requer cerca de 10-12 s por amostra de tecido. A homogeneização do tecido é concluída uma vez que o tecido homogeneizado passa pelo disco perfurado para a câmara superior. Clique aqui para ver uma versão maior desta figura.
- Homogeneize suavemente com 4-5 tacadas para dispersar a suspensão sem produzir espuma. Coloque o homogeneizar em um béquer de 50 mL e misture delicadamente em uma placa de agitação por 15 min a 4 °C.
- Após 15 minutos de incubação, adicione 10 mL do tampão de isolamento que contém o inibidor de trippsina no béquer contendo o homogeneizar e incubar por 1 min a 4 °C enquanto mexe suavemente.
- Filtre a suspensão novamente através de um filtro de nylon e salve o filtrado em um tubo cônico de 50 mL.
- Colete a fração de partículas deixada no filtro de nylon e coloque-a no homogeneizador de vidro em vidro. Adicione 15-20 mL do tampão de lavagem e homogeneize suavemente manualmente por 25-30 s.
NOTA: O filtrado flui muito lentamente nesta etapa. Para aumentar o fluxo de filtragem, coloque a ponta do funil tocando o interior do tubo cônico. - Misture o homogeneizar com o filtrado, equilibre os tubos e centrífuga a 600 x g por 15 min a 4 °C.
- Filtre o supernatante resultante em um novo tubo cônico usando um filtro de nylon para evitar contaminação pela pelota. Execute a centrífuga novamente a 8.500 x g por 15 min a 4 °C.
NOTA: Use uma pipeta de transferência de plástico para remover o supernante e sacrifique 0,2 mL de supernadante do topo da pelota macia. - Descarte o supernatante e enxágue a pelota 2-3 vezes com 1 mL do tampão de isolamento. Certifique-se de descartar a camada de aro exterior branca fofa cada vez.
NOTA: Lave a pelota adicionando o tampão de isolamento usando uma pipeta com ponta de 1 mL ou uma nova pipeta de transferência. Adicione o tampão ao lado do tubo, não diretamente sobre a pelota, para evitar quebrar a pelota e evitar a contaminação do supernasciente. Em seguida, aspire o buffer com uma pipeta de transferência e repita o processo mais duas vezes. - Adicione 5 mL do tampão de isolamento a cada tubo e quebre a pelota usando uma pipeta com uma ponta de 1 mL ou uma nova pipeta de transferência.
- Diluir a suspensão com 20 mL de tampão de isolamento adicional e executar a centrífuga novamente a 8.500 x g por 15 min a 4 °C.
- Descarte o supernatante e resuspenque a pelota em 5 mL do buffer de resusus pendente. Em seguida, adicione 15 mL a mais do buffer para um total de 20 mL, e centrífuga a 8.500 x g por 15 min. a 4 °C. Descarte o supernatante. A pelota marrom resultante contém mitocôndrias intactas lavadas.
- Resuspend a pelota enriquecida com mitocôndrias em 250 μL do buffer de resususpending, incluindo inibidores de protease. Alíquota em 25 μL, congelamento rápido em nitrogênio líquido e armazenamento em um congelador profundo de -80 °C por vários anos.
NOTA: Poupe 5 μL de preparação enriquecida com mitocôndrias para o ensaio de análise de proteínas. Para um estudo BN-PAGE (Blue native-PAGE), aliquot a pelota resuspended em 50 μg de proteína mitocondrial por tubo. Centrifugar a 8.500 x g por 15 min a 4 °C. Aspire cuidadosamente o supernasce e armazene a pelota mitocondrial (50 μg de proteína/aliquot) em um congelador profundo de -80 °C por vários anos.
3. Avaliação do complexo de transferência de elétrons mitocondrial (ETC)
NOTA: As proteínas etc mitocondrial individuais (ou seja, Complex-I, II, III, IV, V) podem ser avaliadas pelo ensaio padrão de imunoblotação. Devido à variedade de amostras (quatro pontos de tempo: 48h, 3 meses,6 meses, 9 meses), duas condições experimentais (exercidas e sedentárias) e uma série de anticorpos imunoprobadores (cinco anticorpos), recomenda-se uma imunoblotting multiplexada. Realize imunoblotting multiplexed da seguinte forma.
- Resolva 50 μg de mitocôndria enriquecida amostra de proteína/bem em eletroforese de gel de poliacrilamida de 4%-20% gradiente (100 V, 90 min).
- Transfira as proteínas para uma membrana PVDF (75 V; 60 min)
- Bloqueie a membrana em uma solução tampão de bloqueio por um mínimo de 1h a 4 °C.
NOTA: O tempo de bloqueio pode ser estendido por até durante a noite a 4 °C. O tampão de bloqueio (Tabela de Materiais) contém ingredientes não-mamíferos (ou seja, proteínas de peixe) que são menos propensos a ter interações específicas de ligação com anticorpos, bem como outras proteínas mamíferas presentes em métodos típicos. - Teste a membrana com o coquetel de anticorpos incluindo anticorpos complexos I, II, III, IV, V anticorpos e incubar durante a noite em um agitador orbital a 4 °C.
- No dia seguinte, lave a membrana e a sonda com anticorpo secundário com marca IR por 2 h em um agitador orbital em RT (temperatura ambiente).
- Lave cinco vezes com 1x TBS-T (TBS contendo Interpol 20) e uma vez com 1x TBS (soro fisiológico tamponado tris).
NOTA: Para a última lavagem, não inclua detergente no buffer TBS. - Imagem da mancha em um analisador de imagem.
4. A avaliação do supercomplex mitocôndria por Blue Native Poly Acrylamide Gel Electrophoresis (BN-PAGE)
NOTA: Um perfil supercomplexo de ETC também pode ser avaliado utilizando a técnica BN-PAGE.
- Prepare o coquetel tampão de amostra para 20 μL por 50 μg de proteína de pelota enriquecida com mitocôndrias (7 μL de água + 5 μL de 4x nativa poliacrilamida gel eletroforese tampão de carga da amostra + 8 μL de 5% digitonina + 2 μL de Coomassie G-250).
NOTA: Faça um coquetel tampão de amostra de estoque dependendo do número de amostras disponíveis (50 μg de proteína/amostra). Escolha quantidades de digitonin apropriadas com base na concentração proteica da pelota enriquecida com mitocôndrias (Tabela 1)8. - Aqueça o estoque de digitonin a 95 °C por 3 min, misture e esfrie no gelo antes de fazer a mistura mestre.
- Adicione o aditivo da amostra Coomassie G-250 pouco antes de carregar as amostras no gel.
- Adicione 20 μL do coquetel tampão de amostra em um tubo que contém 50 μg de pellet de proteína mitocondrial. Solubilize/resuspenque a pelota misturando suavemente a pelota com uma pipeta sem gerar espuma.
- Incubar a mistura no gelo por 20 minutos para alcançar a solubilização máxima da pelota enriquecida com mitocôndrias. De tempos em tempos, aperte suavemente os tubos para melhorar a solubilização.
- Enquanto aguarda a incubação, prepare o buffer de ânodo page nativo/1x e o tampão de cátodo azul escuro.
- Centrifugar a pelota solubilizada enriquecida com mitocôndrias a 20.000 x g por 10 min a 4 °C.
- Após a centrifugação, colete 15 μL do supernatante em novos tubos colocados no gelo.
- Adicione a quantidade apropriada de 5% reagente Coomassie G-250 ao supernante obtido na etapa 4.8 (consulte a Tabela 1).
NOTA: O volume coomassie G-250 de 5% deve ser de tal forma que a concentração final do corante seja 1/4 da concentração de detergente. Realize esta etapa pouco antes de carregar as amostras no gel.
| Proteína | Digitonin/proteína | Tampão de amostra 4x | 5% Digitonin | Água | Volume final | 5% Coomassie G-250 |
| razão (g/g) | aditivo de amostra | |||||
| 50 μg | 8 | 5 | 8 | 7 | 20 | 2 |
| 50 μg | 4 | 5 | 4 | 11 | 20 | 1 |
Tabela 1: Preparação do coquetel tampão de amostra com duas diferentes relações detergente/proteína. Para alcançar a solubilização máxima das proteínas de membrana a partir da suspensão mitocondrial, a concentração de proteína de suspensão digitonin/mitocondrial deve ser ajustada entre 4-8 g de digitonina/g de proteína. No tecido cardíaco, 400 μg de digitonina para 50 μg de proteínas de suspensão mitocondrial (ou seja, 8 g de digitonin/g de proteína mitocôndria) foram usados para maximizar a solubilidade dos supercomplexos da suspensão das mitocôndrias.
- Despeje um gel de 3%-8%, remova o pente e lave os poços do gel com 1x tampão de corrida três vezes.
NOTA: O gel melhor polimeriza se derramado um dia antes. - Configure o sistema de eletroforese e coloque o gel no aparelho.
- Encha os poços de amostra com o tampão de cátodo azul escuro.
- Carregue 10 μL do padrão de proteína não manchada como marcador de peso molecular nativo no primeiro e último poço do gel.
- Carregue toda a quantidade do aditivo amostrante + Coomassie preparado na etapa 4.9 acima nos poços usando uma ponta de carregamento de gel de cabeça plana.
- Encha cuidadosamente a barragem de tampão de mini células com aproximadamente 60 mL de tampão de cátodo azul escuro sem perturbar as amostras nos poços e, em seguida, encha o tanque tampão com o buffer nativo PAGE 1x running buffer.
- Ligue a fonte de alimentação e eletroforese as amostras a 150 V por aproximadamente 30 minutos.
- Remova o tampão de cátodo de cor azul escuro da barragem tampão (câmara interna) com uma pipeta de transferência ou aspire os poços de amostra. Encha a câmara interna com 150-200 mL de tampão de cátodo azul claro e execute a eletroforese a 250 V por 60 min.
NOTA: O tempo de eletroforese pode ser determinado verificando quando a frente de corante migra apenas 0,5 cm acima da parte inferior do de gel. - Retire o gel do e coloque-o em um recipiente plástico limpo. Lave o gel com água destilada de boa qualidade por 15 minutos em um agitador/roqueiro orbital.
NOTA: Para remover o gel gradiente do, manuseie o de baixo, que é mais forte, para minimizar as chances de quebra. - Despeje a água, mergulhe o gel em uma quantidade suficiente de reagente de manchas Coomassie GelCode Blue e incubar por 1 h em um orbital/roqueiro à temperatura ambiente.
NOTA: Misture a solução de reagente de manchas azul (Tabela de Materiais) antes de ser usada girando suavemente a garrafa várias vezes. - Decante a solução de corante, adicione água destilada, enxágue várias vezes com água e, em seguida, coloque o orbital/roqueiro para desinining.
NOTA: Insira um pedaço de esponja (~3 cm x 0,5 cm) no recipiente de desinining de gel e deixe o gel para a desestabilização durante a noite. A esponja adsorbe as partículas de corante sem exigir múltiplas mudanças de água. - Analise o gel em um analisador de imagens.
5. Avaliação do supercomplex mitocôndria por análise de manchas de imunoblot
- Execute um novo conjunto de BN-PAGE sem colorir o gel no final da eletroforese.
- Siga o mesmo procedimento da seção 3 (avaliação do complexo de transferência de elétrons mitocondrial (ETC) para completar a imunoblotação.
Resultados
Seguindo o protocolo, foi preparado um bom rendimento da mistura de proteína enriquecida com mitocôndrias a partir do tecido cardíaco. Aproximadamente 15 mg/mL de mistura de proteína enriquecida com mitocôndrias foram obtidos a partir de uma média de 1,2 g de tecido cardíaco congelado colhido da prole da porca. Observações indicaram que menos de 0,5 g de tecido cardíaco congelado não produziu uma quantidade suficiente de mistura de proteína enriquecida mitocondrial para realizar um ensaio BN-PAGE. A quantidad...
Discussão
Os passos críticos para este protocolo estão indicados aqui. Em primeiro lugar, a homogeneização do tecido deve ser cuidadosamente tratada para que efeitos excessivos não sejam aplicados durante o processo de homogeneização do tecido. Deve-se utilizar um triturador de tecidos, que faz parte da tecnologia de ciclismo de pressão (PCT) para homogeneização inicial do tecido9. Esta etapa reduzirá o ciclo excessivo de avc do homogeneizador de vidro em vidro (Figura 1B
Divulgações
Os autores não declaram conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado financeiramente pela bolsa intramuros da Universidade de Kansas City para Abdulbaki Agbas e Summer Research Fellowship para Daniel Barrera. Os autores agradecem pelo trabalho editorial do Dr. Jan Talley.
Materiais
| Name | Company | Catalog Number | Comments |
| Amino caproic acid | Sigma/Aldrich | A2504-100G | |
| Anti-Hu Total OxPhos complex kit | Invitrogen | 458199 | |
| anti-VDAC antibody | abcam | ab15895 | use 1 µg/mL |
| Coomassie G-250 | ThermoSientific | 20279 | |
| Coomassie GelCode Blue | ThermoScientific | 24592 | |
| Digitonin | Cabiochem | 300410 | |
| Glass-Glass pestle homogenizer | VWR | KT885451-0020 | |
| Image Studio | LICOR | ||
| IR-Dye conjugated anti-Rabbit Ab | LICOR | LC0725 | |
| Multiwell plate reader | BioTek | Synergy HT | |
| Native molecular weight marker | ThermoFisher | BN2001 | |
| Nylon mesh monofilament | Small Part Inc | CMN-74 | |
| Orbital shaker | ThermoScientfic | ||
| PCT Shredder | Pressure Bioscience Inc | ||
| SEA BLOCK Blocking buffer | ThermoScienctific | 37527 | |
| Shredder PULSE Tube | Pressure Bioscience Inc | FT500-PS | |
| Table top centrifuge | Eppendorf | 5418 | |
| Trypsin | Amresco | M150-1G | |
| Trypsin inhibitor | Amresco | M191-1G | Requires fresh preparation |
Referências
- Gross, V. S., et al. Isolation of functional mitochondria from rat kidney and skeletal muscle without manual homogenization. Analytical Biochemistry. 418 (2), 213-223 (2011).
- Osto, C., et al. Measuring mitochondrial respiration in previously frozen biological samples. Current Protocols in Cell Biology. 89 (1), 116 (2020).
- Agbas, A., et al. Mitochondrial electron transfer cascade enzyme activity assessment in cultured neurons and select brain regions. Current Protocols in Toxicology. 80, 73 (2019).
- Hathaway, Q. A., et al. Maternal-engineered nanomaterial exposure disrupts progeny cardiac function and bioenergetics. American Journal of Physiology-Heart and Circulatory Physiology. 312 (3), 446-458 (2017).
- May, L. E., et al. Influence of maternal aerobic exercise during pregnancy on fetal cardiac function and outflow. American Journal of Obstetrics & Gynecology MFM. 2 (2), 100095 (2020).
- Ehler, W. J., et al. Avoidance of malignant hyperthermia in a porcine model for experimental open heart surgery. Laboratory Animal Science. 35 (2), 172-175 (1985).
- Panov, A. V., et al. Effect of bovine serum albumin on mitochondrial respiration in the brain and liver of mice and rats. Bulletin of Experimental Biology and Medicine. 149 (2), 187-190 (2010).
- Jha, P., Wang, X., Auwerx, J. Analysis of mitochondrial respiratory chain supercomplexes using blue native polyacrylamide gel electrophoresis (BN-PAGE). Current Protocols in Mouse Biology. 6 (1), 1-14 (2016).
- Pressure Biosciences Inc. . Isolation of Functional Mitochondria from Whole Rat Heart Using a PBI Shredder and Pressure Cycling Technology (PCT). , (2010).
- McLaughlin, K. L., et al. Novel approach to quantify mitochondrial content and intrinsic bioenergetic efficiency across organs. Scientific Reports. 10 (1), 17599 (2020).
- Hom, J., Sheu, S. S. Morphological dynamics of mitochondria--a special emphasis on cardiac muscle cells. Journal of Molecular and Cellular Cardiology. 46 (6), 811-820 (2009).
- Greggio, C., et al. Enhanced respiratory chain supercomplex formation in response to exercise in human skeletal muscle. Cell Metabolism. 25 (2), 301-311 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados