Method Article
Co-cultura e transdução de timócitos murinos em células estromais delta-like 4 expressando para estudo de oncogenes em leucemia de células T
Neste Artigo
Resumo
Este protocolo descreve o isolamento de timócitos duplo-negativos do timo de camundongo seguido de transdução retroviral e co-cultura no sistema de co-cultura de linhagem celular estromal de medula óssea delta-like 4 (OP9-DL4) para análise funcional adicional.
Resumo
Timócitos imaturos transduzidos de camundongos podem ser diferenciados em células T in vitro usando o sistema de co-cultura de linhagem celular estromal de medula óssea delta-like 4 (OP9-DL4). Como a transdução retroviral requer células em divisão para integração transgênica, OP9-DL4 fornece um ambiente in vitro adequado para o cultivo de células progenitoras hematopoéticas. Isso é particularmente vantajoso ao estudar os efeitos da expressão de um gene específico durante o desenvolvimento normal de células T e leucemogênese, pois permite que os pesquisadores contornem o demorado processo de geração de camundongos transgênicos. Para alcançar resultados bem-sucedidos, uma série de etapas coordenadas envolvendo a manipulação simultânea de diferentes tipos de células deve ser cuidadosamente realizada. Embora esses sejam procedimentos muito bem estabelecidos, a falta de uma fonte comum na literatura muitas vezes faz com que uma série de otimizações sejam necessárias, o que pode ser demorado. Este protocolo tem se mostrado eficiente na transdução de timócitos primários seguida de diferenciação em células OP9-DL4. Detalhado aqui está um protocolo que pode servir como um guia rápido e otimizado para o co-cultivo de timócitos transduzidos retroviricamente em células estromais OP9-DL4.
Introdução
A linhagem celular estromais de medula óssea OP9 fornece um sistema in vitro útil para a indução de linfopoiese a partir de várias fontes de progenitores1. Os primeiros estudos que utilizaram células OP9 demonstraram que a falta de expressão do fator estimulador de colônias de macrófagos (MCSF) contribuiu para a capacidade da linhagem celular OP9 de suportar a diferenciação de hematopoiese e células B a partir de células-tronco hematopoéticas derivadas da medula óssea (CTHs), como posteriormente também foi demonstrado para células-tronco embrionárias (CTEs)2,3,4,5 . Em estudos anteriores, a geração de células OP9 que expressam delta-like 1/4 (OP9-DL1/OP9-DL4) possibilitou a indução do comprometimento da linhagem de células T6 e demonstrou a capacidade de recapitular com sucesso a maturação tímica 7,8. Resumidamente, o desenvolvimento de células T tem sido descrito pela expressão sequencial de moléculas CD4 e CD8. Os timócitos imaturos são "duplo-negativos" (DN, CD4− CD8−) e podem ser subdivididos de acordo com a expressão superficial de CD44 e CD25. Os timócitos da ND diferenciam-se através do estágio imaturo single-positivo (ISP), caracterizado pela expressão de CD8 em camundongos e CD4 em humanos, seguido pelo estágio duplo-positivo (DP), caracterizado pela co-expressão de CD4 e CD8, e, finalmente, pelo estágio maduro single-positivo, caracterizado pela expressão de CD4 ou CD89. As HSCs expressam o receptor Notch1, que normalmente interage com o delta-like 4 (DL4) expresso nas células epiteliais tímicas para induzir a diferenciação da linhagem T10. Assim, o interesse no modelo OP9-DL1/4 tem aumentado progressivamente, levando ao uso extensivo dessa abordagem em uma ampla variedade de aplicações nas últimas duas décadas8,11,12,13. Embora DL1 e DL4 sejam capazes de suportar a diferenciação de células T in vitro, elas apresentam requisitos diferenciais in vivo, e alguns estudos sugerem que OP9_DL4 é mais eficiente do que OP9_DL1 em recapitular o ambiente tímico decamundongos7,14.
Dentre as potenciais aplicações do sistema OP9-DL1/4, há particular interesse na combinação deste sistema com a transdução de células DN ou CTHs com vetores retrovirais. Essa combinação é uma maneira eficaz de manipular a expressão gênica durante o desenvolvimento normal de células T e leucemogênese e tem se mostrado um método eficiente para induzir ou inibir a função de um gene de interesse15,16,17. Este modelo tem sido particularmente utilizado com sucesso para estudar a colaboração entre os oncogenes que conduzem a leucemia15 por ser flexível e permitir o exame dos efeitos de múltiplas combinações gênicas em um tempo razoável, em contraste com a geração de camundongos transgênicos. Além disso, modelos semelhantes foram usados anteriormente para avaliar os efeitos da introdução de oncogenes em células normais15,16,17. Além disso, a transdução retroviral requer células em divisão para integração transgênica18, e enquanto a transdução lentiviral superaria essa limitação eliminando a necessidade de células em divisão para integração transgênica, não conseguimos alcançar a transdução bem-sucedida de timócitos de DN usando vetores lentivirais. Assim, OP9-DL1/DL4 é uma ferramenta adequada para o crescimento de células progenitoras hematopoéticas.
O protocolo padrão para linfopoiese de timócitos transduzidos em OP9-DL4 envolve uma série de etapas coordenadas que devem ser cuidadosamente realizadas para alcançar um desfecho bem-sucedido. Embora sejam técnicas que vêm servindo bem a comunidade há muitos anos, muitas vezes os protocolos disponíveis na literatura são fragmentados. Como resultado, cada laboratório é forçado a adaptar e otimizar diferentes etapas do procedimento, o que pode ser demorado. Este protocolo descreve o isolamento de timócitos de DN do timo de camundongo, seguido de transdução retroviral e co-cultura em células estromais OP9-DL4 para posterior análise funcional. Esse protocolo estabelecido tem se mostrado eficiente e reprodutível na transdução de timócitos primários, seguida da diferenciação em células OP9-DL4 ou da indução de leucemia linfoblástica aguda de célulasT15.
Protocolo
Todos os experimentos com animais descritos foram aprovados pelo Comitê Institucional de Biossegurança (IBC) do NIH e pelo Comitê de Cuidados e Uso de Animais (ACUC). Consulte a Tabela de Materiais para obter detalhes relacionados a todos os reagentes e materiais utilizados neste protocolo. Consulte as diretrizes publicadas19 para obter mais detalhes sobre a cultura de células produtoras de retrovírus e os procedimentos de manutenção. Consulte a Figura 1 para obter uma visão geral do protocolo.
1. Início da cultura de células OP9-DL4 (Dia 1)
- Descongele rapidamente as células OP9-DL4 segurando o frasco para injetáveis e agitando suavemente em banho-maria a 37 °C. Transferir imediatamente as células para um tubo de centrífuga contendo 5 mL do meio OP9 (meio MEM-alfa, FBS a 10%, penicilina/estreptomicina 50 U/mL, 2-mercaptoetanol 55 μM, L-glutamina 2 mM) para remover os agentes crioprotetores.
- Centrifugar a 300 × g durante 5 min à temperatura ambiente. Descarte o sobrenadante e ressuspenda as células em 1 mL de meio OP9.
- Colocar 5 ml de meio OP9 num balão T25 e adicionar as células ressuspensas a este balão. Cultura a 37 °C e 5% CO2.
- Após 2 a 3 dias, subculte as células usando uma divisão 1:3 (passe as células todas as segundas, quartas e sextas-feiras).
Observação : dividir as células OP9-DL4 antes que elas atinjam a confluência. O frasco OP9-DL4 deve ser 80%–90% confluente nos dias 6–7 e nos dias 8–9+ para co-cultura com timócitos. Assim, é importante planejar quantas células OP9-DL4 serão necessárias nesses dias enquanto divide as células OP9-DL4.- Eliminar o meio do balão e lavar a monocamada com 1x PBS. Eliminar o PBS, adicionar 1 ml de tripsina a 0,25% e incubar durante 1–5 min a 37 °C ou até que as células se tenham deslocado do frasco. Bata o frasco para desalojar as células.
NOTA: (Opcional) O progresso da dissociação celular pode ser verificado por microscopia. - Adicionar 5 mL de meio OP9 para inativar a tripsina e ressuspender as células enxaguando a superfície coberta de células do frasco durante a ressuspensão.
- Centrifugar a suspensão celular a 300 × g durante 5 min à temperatura ambiente e ressuspender em 3 ml de meio OP9 (para uma divisão 1:3).
- Colocar 1 ml da suspensão celular num balão T25 que já contenha 5 ml de meio OP9 fresco. Congelar/descartar as células restantes.
- Para congelar as células OP9-DL4, ressuspendê-las em FBS 90%/DMSO 10% na proporção de 1 mL de meio de congelação para um frasco de células.
- Congelar em 1 ml de criópoles a -80 °C ou em azoto líquido, dependendo das necessidades futuras. Armazenar em nitrogênio líquido para armazenamento prolongado.
- Eliminar o meio do balão e lavar a monocamada com 1x PBS. Eliminar o PBS, adicionar 1 ml de tripsina a 0,25% e incubar durante 1–5 min a 37 °C ou até que as células se tenham deslocado do frasco. Bata o frasco para desalojar as células.
2. Início da cultura da linhagem celular produtora de retrovírus (Dia 1)
- Descongele rapidamente a linhagem celular produtora de retrovírus segurando o frasco para injetáveis e agitando suavemente em banho-maria a 37 °C. Transferir imediatamente as células para um tubo de centrífuga contendo 5 mL de meio celular produtor de retrovírus (RPC: DMEM, 10% FBS) para remover o agente crioprotetor.
- Centrifugar a 300 × g durante 5 min à temperatura ambiente. Descarte o sobrenadante e ressuspenda as células em 1 mL de meio RPC.
- Colocar 5 ml de meio RPC num balão T25 e adicionar as células ressuspensas a este balão. Cultura a 37 °C e 5% CO2.
- Passe as células a cada 2–3 dias em uma divisão de 1:5 a 1:8.
- Bata o frasco para desalojar as células e ressuspenda as células pipetando agressivamente.
NOTA: Estas células podem ser removidas da superfície do frasco por pipetagem agressiva sem tripsina. Alternativamente, tripsinize (0,05% tripsina/0,53 mM EDTA) até que as células se desprenda facilmente e possam ser facilmente pipetadas em uma suspensão unicelular.
- Bata o frasco para desalojar as células e ressuspenda as células pipetando agressivamente.
- Centrifugar a suspensão celular a 300 × g por 5 min e ressuspender em 5 mL de meio RPC (para uma divisão de 1:5).
- Colocar 1 ml da suspensão celular num balão T25 que já contenha 5 ml de meio RPC fresco. Congelar/descartar as células restantes. Congelar as células produtoras de retrovírus da mesma forma que descrito para as células OP9-DL4 (passos 1.4.4.1–1.4.4.2).
3. Início da cultura das células produtoras de retrovírus em placas de 6 poços (Dias 4–5)
- Semeando as células produtoras de retrovírus 18–24 h antes da transfecção na confluência de 70%–90% em cada poço de uma placa de cultura de tecido de 6 poços em 2 mL de meio RPC e incubar a 37 °C e 5% CO2.
NOTA: Transfecte dois poços das células produtoras de retrovírus para cada 2–5 × 105 a 1 × 106 timócitos que serão transduzidos.
4. Transfecção das células produtoras de retrovírus para gerar o retrovírus contendo os genes de interesse (Dias 5–6)
- Substitua o meio por meio RPC fresco 1 h antes da transfecção.
- Preparar misturas de lipofecção (para cada poço de uma placa de 6 poços – aumentar conforme necessário): Diluir 4 μg de DNA (2 μg de plasmídeo auxiliar (pCL-Eco) e 2 μg de plasmídeo de transferência (pMIG) em 250 μL de meio de soro reduzido. Misture delicadamente.
- Misturar 10 μL do reagente de transfecção com 250 uL do meio de soro reduzido do passo 4.2. Incubar durante 5 minutos à temperatura ambiente.
- Após 5 min de incubação, combinar o DNA diluído com o reagente de transfecção diluído (volume total = 500 μL). Misture delicadamente e incube por 20 a 25 minutos à temperatura ambiente.
- Adicione suavemente os 500 μL de misturas de reagentes de DNA/transfecção ao poço que contém as células produtoras de retrovírus, caindo sobre as células com um movimento circular. Misture suavemente balançando a placa para frente e para trás e incube a placa em uma incubadora de 37 °C por 16 a 24 h.
NOTA: As células subconfluentes (80%–90%) são mais adequadas para transfecção e potencialmente geram o título viral mais alto. Como as células produtoras de retrovírus se deslocam muito facilmente do frasco, evite movimentos bruscos ao manusear essas células.
5. Mudança do meio celular produtor de retrovírus (Dias 6–7)
- Substitua o meio velho aproximadamente 16 h após a transfecção por 2 mL de meio RPC fresco. Continuar a incubar as células a 37 °C durante 20–24 horas.
- Avaliar a eficiência da transfecção em microscópio de fluorescência (opcional).
NOTA: Neste protocolo, as células GFP-positivas são as células de interesse (ver Figura 2). Essa etapa depende do vetor retroviral selecionado para uso neste protocolo (se um gene repórter está presente na espinha dorsal).
6. Preparo de timócitos e depleção de células CD4+ e CD8+ (Dias 6–7)
- Colha o timo de um rato C57BL/6 com 4 a 6 semanas de idade. Para mais detalhes sobre a colheita do timo, consulte Xing e Hogquist20.
OBS: Os camundongos foram eutanasiados por inalação de CO2 seguida de deslocamento cervical. - Preparar uma suspensão unicelular de timócito colocando o timo em 5 mL de PBS em uma placa de Petri. Usando lâminas de vidro estéreis, coloque o timo entre as superfícies foscas das lâminas e esfregue suavemente as lâminas juntas, rolando o timo entre as duas lâminas. Enxágue as lâminas de vidro para coletar as células e descarte o tecido estromal tímico remanescente.
NOTA: O rendimento estimado de um timo é de 90 × 10 6–100 × 106 células, e aproximadamente 1% das células permanecerão após a depleção de CD4 e CD8. Use tecido do timo de camundongos mais jovens para um melhor rendimento de células, uma vez que a celularidade do timo de camundongo nas primeiras semanas após o nascimento aumenta rapidamente, atinge um platô em 4-6 semanas de idade e involui progressivamente21. Conte as células usando um contador automático de células sanguíneas ou qualquer método alternativo. - Filtrar a suspensão tímica através de um filtro de 30 μm ou 40 μm pipetando 5 ml da suspensão tímica através de um filtro celular. Centrifugar as células a 300 × g durante 10 min.
- Remova o sobrenadante e lise as hemácias com tampão de lise ACK adicionando (dependendo do tamanho da amostra) 1 mL de tampão por tubo por 1 min. Adicionar 5 mL de tampão de depleção celular (500 mL de PBS [pH 7,2], BSA 0,5%, EDTA 0,5M 2 mM, pH 8,0) para desativar o tampão de lise ACK, centrifugar a 300 × g por 10 min e ressuspender em 1–5 mL de tampão de depleção celular para contagem.
OBS: Separar no gelo antes da depleção (pré-depleção): 70 μL de suspensão celular para contagem (variando de acordo com o método de contagem celular escolhido) e 200 μL de suspensão celular para determinar a eficiência da depleção pela coloração para CD4 e CD8 e análise por citometria de fluxo22,23. - Conte as células e centrifugue a 300 × g durante 10 min. Ressuspender as células a 1 × 107/80 μL de tampão de depleção celular. Adicionar 10 μL de microesferas CD4 e CD8 por 1 × 107 células. Misture bem e incube por 15 min no escuro na geladeira (2–8 °C).
- Prepare uma coluna de depleção enxaguando-a com 2 mL de tampão de depleção e descartando o fluxo.
- Lavar as células do passo 6.6 adicionando 1–2 ml de tampão de depleção por 1 × 107 células e centrifugar a 300 × g durante 10 minutos. Retire e descarte o sobrenadante.
- Ressuspender até 1,25 × 108 células em 500 μL de tampão de depleção. Aplique a suspensão da célula à coluna e colete o fluxo (células não rotuladas). Lavar a coluna 2x com 1 ml de tampão e recolher o fluxo.
Observação : só adicionar novo buffer quando o reservatório de coluna está vazio. Separar o seguinte no gelo após a depleção (controle pós-depleção): 70 μL de suspensão celular para contagem (variando isso dependendo do método de contagem de células escolhido) e 1.000 μL de suspensão celular para determinar a eficiência da depleção por coloração para CD4 e CD8 e análise por citometria de fluxo22,23. - Controlar a eficiência da depleção dividindo os 200 μL de células coletadas antes da depleção em quatro tubos FACS: não corado, CD4 de coloração simples, CD8 de coloração simples e CD4/CD8 de dupla coloração (use as amostras não coradas e de coloração única para configurar os parâmetros de citometria de fluxo). Use os 1.000 μL de células coletadas após a depleção para corar para CD4 e CD8 e compare com a amostra de dupla coloração coletada antes da depleção (ver resultados típicos de depleção na Figura 3).
NOTA: Ajustar os volumes para coloração por citometria de fluxo de acordo com a recomendação do fabricante do anticorpo (por exemplo, 1 μL de anti-CD4-FITC + 0,5 μL de anti-CD8-PE por 1 x 106 células em 50 μL de tampão).
7. Cultura de timócitos em células OP9-DL4 (Dias 6–7)
- Colocar 2–5 × 105 a 1 × 10 6 timócitos pós-depleção em um frasco T25 de células OP9-DL4 confluentes 80%–90% em meio OP9 contendo citocinas (10 ng/mL de IL-7 recombinante e hFLT-3 recombinante). Cultura a 37 °C e 5% CO2. Crescer por aproximadamente 24 h em células OP9-DL4.
NOTA: Essa duração é necessária para tornar as células T transdutíveis (consulte um resultado típico na Figura 4). Reserve um frasco T25 da co-cultura de timócitos e células OP9-DL4 para ser usado como controle (não transduzido). Os timócitos não transduzidos serão corados juntamente com os timócitos transduzidos (passo 9.1) como controle negativo para avaliar a eficiência da transdução. Timócitos não transduzidos também podem ser usados para medir o efeito da expressão do transgene na diferenciação celular. Descarte o meio contendo citocinas após 1 mês.
8. Colher o retrovírus do sobrenadante e usá-lo para transduzir os timócitos (Dias 8–9)
- Coletar o sobrenadante contendo os retrovírus das células transfectadas inclinando a placa de 6 poços e posicionando uma seringa de 3 a 5 mL no fundo da placa enquanto puxa o êmbolo para aspirar o sobrenadante. Substitua o meio por 2 mL de meio RPC fresco. Continuar a incubar as células a 37 °C durante 20–24 h para a segunda transdução do passo 8.9.
- Filtrar o sobrenadante do retrovírus através de um filtro de seringa de 0,45 μm e recolher o filtrado num tubo de 50 ml.
NOTA: Não congele o sobrenadante retroviral. Use uma preparação de vírus recém-feita para transdução. - Coletar timócitos da cultura OP9-DL4 por pipetagem agressiva para remover os timócitos e as células OP9-DL4 da superfície do frasco. Filtrar a suspensão celular através de um filtro celular de 40 μm para remover a maioria das células OP9-DL4 e coletar o filtrado em um tubo de 50 mL.
NOTA: As células OP9 são muito aderentes. Embora a pipetagem agressiva possa remover algumas das células OP9-DL4 do frasco, a ruptura da monocamada OP9-DL4 durante este processo é mínima, e as células OP9-DL4 que se soltam serão filtradas com o filtro celular de 40 μm, uma vez que as células OP9-DL4 são muito maiores do que os timócitos primários. Se as células OP9-DL4 ainda estiverem 80%–90% confluentes, remova os timócitos e plaqueie-os novamente no mesmo frasco OP9-DL4. Alternativamente, um novo frasco OP9-DL4 deve ser preparado. - Centrifugar os timócitos no filtrado a partir do passo 8.3 a 300 × g durante 5 min. Descarte o sobrenadante.
- No tubo de 50 ml, ressuspender os timócitos em 0,5–1 ml de meio OP9 + citocinas (ver passo 7.1). Adicionar 1–2 mL de meio RPC contendo o vírus (duas vezes mais meio RPC que o meio timócito). Adicionar brometo de hexametrina (concentração de estoque de 10 μg/μL) para produzir 8 μg de brometo de hexametrina/mL de suspensão celular total.
- Espinocular centrifugando as células a 850 × g durante 1 h à temperatura ambiente.
- Ressuspender as células em 6 ml de meio OP9 + citocinas por frasco (ver passo 7.1) e adicionar a suspensão novamente à monocamada celular OP9-DL4.
OBS: Alternativamente, ter novos frascos OP9-DL4 prontos para receber os timócitos transduzidos. Se os timócitos forem colocados de volta em um frasco OP9-DL4 usado, certifique-se de adicionar meio OP9 fresco às células OP9-DL4 durante a 1 h de espinoculação do timócito para manter as células saudáveis e garantir 80%-90% de confluência. - Incubar a 37 °C durante a noite.
- Repita as etapas 8.2–8.7 usando um novo poço do sobrenadante celular contendo retrovírus.
9. Manter timócitos transduzidos em cultura OP9-DL4 por 2 a 5 dias ou congelar conforme necessário (Dia 9+)
- Avaliar a diferenciação timocitária por citometria de fluxo corando timócitos para marcadores de desenvolvimento de células T como CD4, CD8, CD25 e CD4423. Veja os resultados típicos da diferenciação dos timócitos na Figura 5.
Resultados
A eficiência da depleção pode ser avaliada citometria de fluxo marcando a fração celular magneticamente não marcada para CD4 e CD8 após a separação imunomagnética de células (MACS) e analisando-a em um dot-plot bivariado bidimensional (Figura 3). Um bom rendimento de células duplamente negativas (CD4−, CD8−) é de 95% ou mais, como representado na Figura 3. Duas das causas mais comuns de menor rendimento são o erro de cálculo das microesferas com base no número de células e no número de células marcadas que excedem a capacidade da coluna. Recomenda-se escolher o número correto de colunas MACS de acordo com o número de células marcadas. Quando se trabalha com timócitos, o número de células marcadas (células DP e SP) é quase igual ao número de células totais (mais de 96%). A contagem de células pode ser feita em um contador automático de células, uma câmara de Neubauer ou com qualquer método alternativo. Como resultado, os volumes alocados para a contagem de células podem precisar ser ajustados dependendo da máquina específica e do método de contagem escolhido.
É importante determinar o número de células presentes antes e depois da depleção. Essas contagens de células são necessárias para calcular o número de colunas de depleção de DL necessárias e distribuir uniformemente os timócitos de DN no número apropriado de frascos OP9-DL4. Os controles para citometria de fluxo (células não marcadas, células marcadas para CD4 e células marcadas para CD8) podem ser realizados com a amostra pré-depleção reservada, pois esta contém mais células. No entanto, como se espera que a maioria das células seja retida dentro da coluna, a amostra pós-esgotamento a ser marcada exigirá um volume maior. Consequentemente, ajustes podem precisar ser feitos de acordo com o protocolo de rotulagem selecionado.
Ao utilizar vetores que expressam marcadores rastreáveis, como um gene de fluorescência, a transfecção e a transdução podem ser avaliadas grosseira e empiricamente por microscopia de fluorescência (Figura 2). A eficiência da transdução pode ser analisada pela coleta do timócito da monocamada OP9-DL4, conforme descrito na etapa 8.3, e pela observação da expressão de um gene de fluorescência por citometria de fluxo. A eficiência da transdução usando um vetor retroviral vazio com GFP como gene repórter foi de 84,2% (Figura 4).
A diferenciação de células T em células OP9-DL4 pode ser observada 4 dias após a transdução. A citometria de fluxo é geralmente realizada para avaliar a diferenciação celular induzida pelo co-cultivo em células OP9-DL4 e/ou a expressão transgênica, como representado na Figura 5, onde as células foram marcadas para CD4, CD8, CD44 e CD25. Existem várias combinações possíveis de marcação de moléculas de superfície celular que têm se mostrado úteis para investigar os mecanismos moleculares e celulares do desenvolvimento de células T em camundongos24,25,26,27. Portanto, os painéis de anticorpos fluorescentes podem variar de acordo com a questão de interesse que está sendo abordada. A transdução dos timócitos DN com o vetor retroviral vazio pMIG, mostrada no painel B, apresentou aproximadamente as mesmas proporções de simples positivos (CD4+ ou CD8+), duplos positivos (CD4+/CD8+), duplos negativos (CD4−/CD8−) e seus subestágios duplo-negativos 1–4 (DN1–DN4) que os timócitos não transduzidos, mostrados no painel A, indicando que o desenvolvimento das células T não foi afetado pelo processo de transdução.
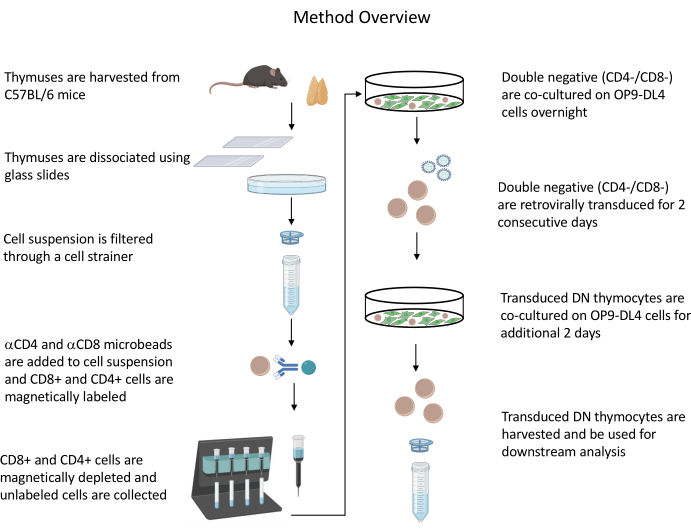
Figura 1: Diagrama das etapas de isolamento, transdução e co-cultivo de timócitos. Abreviação: DN = duplo-negativo. Clique aqui para ver uma versão maior desta figura.
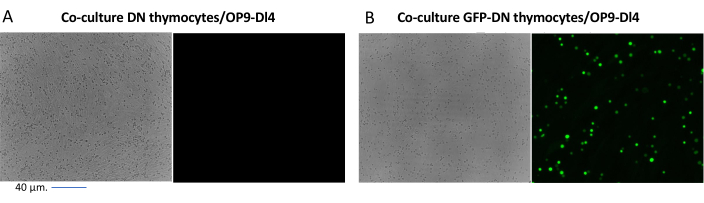
Figura 2: Microscopia de fluorescência em co-cultura de timócitos transduzidos por GFP ou não transduzidos e células OP9-DL4. (A) Timócitos não transduzidos e (B) timócitos murinos estáveis expressando GFP em OP9-DL4 no dia 3 após a segunda transdução. Foi utilizado microscópio de fluorescência Olympus-IX71 com lente de 40x e filtro 480/30 adequado para detecção de GFP. Barra de escala = 40 μm. Abreviações: GFP = proteína fluorescente verde; ND = duplo-negativo. Clique aqui para ver uma versão maior desta figura.
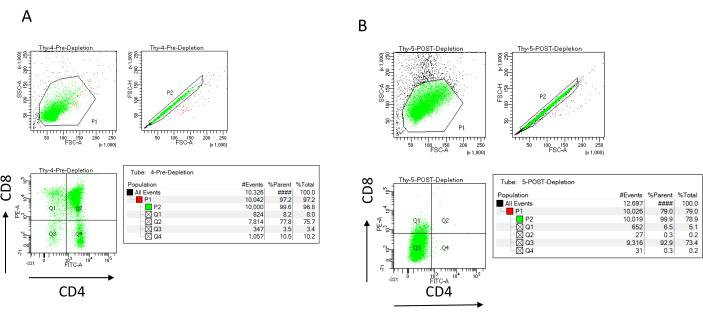
Figura 3: Citometria de fluxo da depleção timocitária. Gráficos representativos de citometria de fluxo analisando a expressão de CD4 e CD8 em timócitos obtidos de camundongos fêmeas C57BL/6J com 7-8 semanas de idade (A) pré-depleção e (B) pós-depleção de CD4+ e CD8+ usando microesferas e colunas LD de acordo com as instruções do fabricante. Os gráficos de pontos à esquerda mostram portões com base no tamanho e complexidade do evento (FCS-A e SSC-A, respectivamente). O painel do meio mostra FSC-H versus FSC-A para portar células únicas e excluir duplos. Nos gráficos à direita, as células foram definidas como CD4 e CD8 da porta unicelular. Abreviações: FSC-A = área do pico de dispersão anterior; SSC-A = área do pico de dispersão lateral; FSC-H = altura do pico de dispersão anterior; FITC = isotiocianato de fluoresceína; PE = ficoeritrina. Clique aqui para ver uma versão maior desta figura.
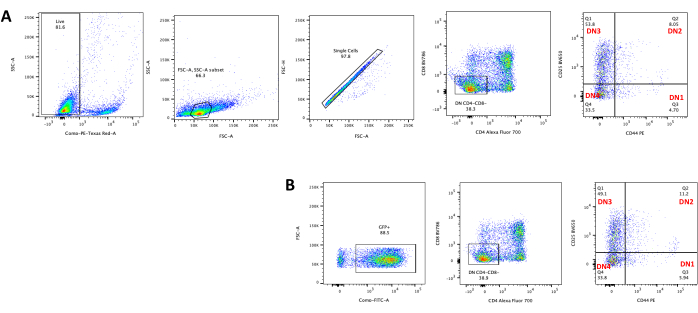
Figura 4: Eficiência da transdução retroviral por citometria de fluxo do timócito. (A) Timócitos não transduzidos co-cultivados em OP9-DL4; (B) timócitos transduzidos retroviricamente em OP9-DL4 no dia 3 pós-transdução. Abreviações: FSC-A = área do pico de dispersão anterior; SSC-A = área do pico de dispersão lateral; FSC-H = altura do pico de dispersão anterior; FITC = isotiocianato de fluoresceína; PE = ficoeritrina; ND = duplo-negativo. Clique aqui para ver uma versão maior desta figura.
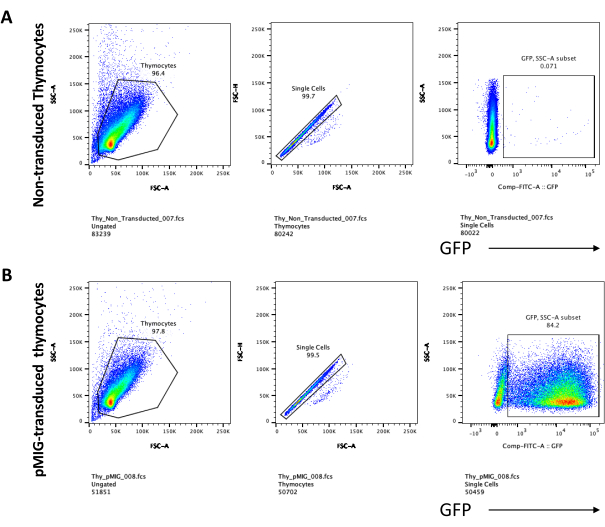
Figura 5: Citometria de fluxo dos timócitos transduzidos após 4 dias de co-cultura OP9-DL4. (A) Não transduzido e (B) transduzido com retrovírus pMIG. As células foram primeiro confinadas em células vivas e, em seguida, fechadas com base no tamanho e complexidade (FCS-A e SSC-A, respectivamente), seguido pela plotagem de FSC-H versus FSC-A para portar em células únicas e excluir duplos. Para as células não transduzidas, foi utilizada a seguinte estratégia de portão. A partir da porta unicelular, as células foram definidas como positivas simples (CD4+ ou CD8+), duplas positivas (CD4+/CD8+) e duplas negativas (CD4−/CD8−). Em seguida, a partir da porta dos duplos negativos (CD4−/CD8−), as células foram definidas como CD44+, CD25+, CD44+/CD25+ e CD44−/CD25−, como mostrado no painel A. Para as células transduzidas por pMIG, mostradas no painel B, a partir da porta unicelular, as células foram primeiramente definidas como GFP+/GFP− e, em seguida, a partir de células GFP+, a distribuição da população celular nos principais estágios de desenvolvimento de células T, definida pela expressão de CD4, CD8, CD44 e CD25, foi determinada usando a mesma estratégia de porta que para as células não traduzidas. Abreviações: FSC-A = área do pico de dispersão anterior; SSC-A = área do pico de dispersão lateral; FSC-H = altura do pico de dispersão anterior; FITC = isotiocianato de fluoresceína; GFP = proteína fluorescente verde. Clique aqui para ver uma versão maior desta figura.
Discussão
O protocolo aqui descrito foi desenvolvido especificamente para estudos de linfócitos T derivados do timo (CD4−/CD8−) com transfecção retroviral seguida de um modelo de diferenciação OP9-DL4. No entanto, é provável que as células-alvo que serão submetidas a este protocolo de transdução seguido de diferenciação celular tenham uma utilidade interdisciplinar mais ampla. Assim, além dos timócitos imaturos, células-tronco hematopoéticas, como células derivadas do fígado fetal ou da medula óssea, poderiam potencialmente ser utilizadas nesse protocolo.
O sistema OP9-DL4 tem se mostrado um modelo eficaz para estudar a função gênica em uma variedade de aspectos, incluindo diferenciação celular17 e oncogênese15. Enquanto a modificação retroviral de progenitores hematopoéticos é uma técnica bem estabelecida que permite a modificação genética estável, combinar a indução de diferenciação celular no sistema OP9-DL4 e transdução retroviral requer cuidado e habilidade. O aspecto crítico para o sucesso desse protocolo é garantir que todas as etapas sejam bem coordenadas, uma vez que o protocolo envolve a utilização de três tipos celulares diferentes que precisam ser mantidos saudáveis e na confluência ideal necessária para cada estágio específico. Com isso em mente, é importante realizar todas as análises de checkpoint de controle de qualidade após a execução de cada etapa antes de prosseguir para a próxima etapa. Isso garantirá que todas as etapas estejam funcionando. Portanto, a verificação das eficiências de depleção, transfecção e transdução é importante para a execução bem-sucedida desse protocolo (veja um resultado típico para eficiência de transdução na Figura 4). A boa eficiência da transdução de células primárias está ligada a um alto título viral. Normalmente, inserções maiores resultam em títulos virais mais baixos18. Para fins de treinamento, utilizamos um vetor vazio para representar os resultados que podem ser obtidos com este protocolo. Em nossa experiência, as eficiências de transdução e transfecção variam de acordo com o tamanho da inserção, especialmente considerando os genes repórteres que expressam a espinha dorsal viral, como GFP. Uma estratégia que pode ser usada ao estudar a interação de mais de um gene é clonar cada gene em um vetor de transferência diferente, seguido pela produção individual do vírus e, finalmente, a co-transdução da célula-alvo. Nesse caso, uma etapa de seleção pode ser aplicada para eliminar as células transduzidas isoladamente e reter apenas as células co-transduzidas.
É importante notar que a maioria daslinhagens celulares da camada alimentadora estromal OP9-DL1/DL4 foi geneticamente modificada para expressar GFP como parte dos construtos DL1 ou DL47. Nesse protocolo, utilizamos um vetor retroviral que também expressa a GFP; no entanto, é mais brilhante que a proteína GFP expressa pelas células OP9-DL4 e não interfere na inspeção da transdução ao visualizar a co-cultura ao microscópio de fluorescência.
As células OP9 diferenciam-se em adipócitos após muitas passagens, longos períodos em cultura ou sob condições de sobre-confluência19. Isso é evidenciado pelo desenvolvimento de grandes vacúolos. Assim, células OP9 que apresentem essas características não devem ser utilizadas neste protocolo. Transfectar células produtoras de retrovírus excessivamente confluentes resultará em um baixo título de vírus. De fato, o estágio subconfluente é quando as células são mais transfectáveis. Além disso, a transfecção de células produtoras de retrovírus de baixa confluência diminuirá o estresse celular no processo de transfecção e dará o maior título de vírus.
Embora, neste protocolo, não titulamos o sobrenadante do vírus, a titulação do sobrenadante do vírus deve ser considerada em alguns casos, como na ausência de um gene repórter no vetor retroviral, o que impediria a determinação indireta da produção viral, ou nos casos em que o desenho experimental requer um número mais preciso de cópias do transgene para ser integrado ao genoma da célula alvo. No entanto, é importante notar que o título do sobrenadante do vetor retroviral diminui significativamente quando armazenado a −80 °C ou 4 °C até que os resultados da titulação sejam obtidos. Portanto, o uso de sobrenadante viral recém-preparado para transdução produzirá melhor eficiência de transdução.
O timo contém um grande número de timócitos duplo-positivos (DP) (mais de 85%) e cerca de 10% de células15 simples-positivas (CD4 ou CD8), que são os timócitos pós-ND. As células DP não sobrevivem in vitro para manipulação retroviral, enquanto as células SP são intransdutíveis. Portanto, este protocolo pode ser aplicado para gerar timócitos de ND transdutíveis por vetor retroviral.
Divulgações
Os autores não têm conflitos de interesse a divulgar
Agradecimentos
Este trabalho foi apoiado pelo programa intramuros do Instituto Nacional do Câncer, projeto ZIABC009287. OP9-DL4 foi obtido do Dr. Juan Carlos Zúñiga-Pflücker (Sunnybrook Health Sciences Centre, Toronto, ON, Canadá). Os autores agradecem ao NCI-Frederick Laboratory Animal Sciences Program por sua assistência técnica contínua e aconselhamento e contribuição experimental, bem como Jeff Carrel, Megan Karwan e Kimberly Klarmann pela assistência em citometria de fluxo. Somos gratos a Howard Young pelos conselhos e contribuições críticas.
Materiais
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Sigma | M3148 | |
| ACK Lysis buffer | Lonza | 10-548E | |
| BSA | Cell Signaling Technology | 9998S | |
| CD4 Microbeads | Miltenyi | 130-049-201 | |
| CD8 Microbeads | Miltenyi | 130-049-401 | |
| Centrifuge 5910R | eppendorf | 5942IP802353 | |
| DMEM | Corning | 10-013-CV | |
| EDTA | Invitrogen | 15575-038 | |
| Fetal calf serum HyClone FBS | ThermoScientific | SH30910.03 | |
| LD columns | Miltenyi | 130-042-901 | |
| L-glutamine | Sigma | G7513 | Freeze glutamine in aliquots and use freshly-thawed glutamine |
| Lipofectamine 2000 | Invitrogen | P/N 52887 | |
| MEM-alpha Medium | Gibco | 12561-072 | |
| OPTI-MEM I Reducing Serum Medium | Invitrogen | 31985-062 | |
| PBS pH 7.2 | Corning | 21-040-CV | |
| pcL-Eco Plasmid | Addgene | 12371 | |
| penicillin/streptomycin | Gibco | 15140-122 | |
| pMIG Plasmid | Addgene | 6492 | |
| Polybrene | Chemicon | TR-1003-G | |
| Pre-Separation Filters | Miltenyi | 130-041-407 | |
| recombinant hFLT-3L | PeproTech | 300-19 | |
| recombinant IL-7 | Peprotech | 217-17 | |
| Retrovial packaging cell line Phoenix-Eco | Orbigen | RVC-10002 | |
| Syringe filter (0.45 µm) | Millipore | SLHV033RS |
Referências
- Zuniga-Pflucker, J. C. T-cell development made simple. Nature Reviews Immunology. 4 (1), 67 (2004).
- Kodama, H., Nose, M., Niida, S., Nishikawa, S., Nishikawa, S. Involvement of the c-kit receptor in the adhesion of hematopoietic stem cells to stromal cells. Experimental Hematology. 22 (10), 979-984 (1994).
- Nakano, T., Kodama, H., Honjo, T. Generation of lymphohematopoietic cells from embryonic stem cells in culture. Science. 265 (5175), 1098-1101 (1994).
- Nakano, T., Kodama, H., Honjo, T. In vitro development of primitive and definitive erythrocytes from different precursors. Science. 272 (5262), 722-724 (1996).
- Ueno, H., et al. A stromal cell-derived membrane protein that supports hematopoietic stem cells. Nature Immunology. 4 (5), 457-463 (2003).
- Jaleco, A. C., et al. Differential effects of Notch ligands Delta-1 and Jagged-1 in human lymphoid differentiation. Journal of Experimental Medicine. 194 (7), 991-1002 (2001).
- Mohtashami, M., et al. Direct comparison of Dll1- and Dll4-mediated Notch activation levels shows differential lymphomyeloid lineage commitment outcomes. Journal of Immunology. 185 (2), 867-876 (2010).
- Schmitt, T. M., Zuniga-Pflucker, J. C. Induction of T cell development from hematopoietic progenitor cells by delta-like-1 in vitro. Immunity. 17 (6), 749-756 (2002).
- Mazzucchelli, R., Durum, S. K. Interleukin-7 receptor expression: Intelligent design. Nature Reviews Immunology. 7 (2), 144-154 (2007).
- Robey, E. A., Bluestone, J. A. Notch signaling in lymphocyte development and function. Current Opinion in Immunology. 16 (3), 360-366 (2004).
- Lavaert, M., et al. Integrated scRNA-Seq identifies human postnatal thymus seeding progenitors and regulatory dynamics of differentiating immature thymocytes. Immunity. 52 (6), 1088-1104 (2020).
- Roh, K. H., Roy, K. Engineering approaches for regeneration of T lymphopoiesis. Biomaterials Research. 20 (20), (2016).
- Zlotoff, D. A., et al. CCR7 and CCR9 together recruit hematopoietic progenitors to the adult thymus. Blood. 115 (10), 1897-1905 (2010).
- Holmes, R., Zuniga-Pflucker, J. C. The OP9-DL1 system: Generation of T-lymphocytes from embryonic or hematopoietic stem cells in vitro. Cold Spring Harbor Protocols. 2009 (2), (2009).
- Cramer, S. D., et al. Mutant IL-7Ralpha and mutant NRas are sufficient to induce murine T cell acute lymphoblastic leukemia. Leukemia. 32 (8), 1795-1882 (2018).
- Treanor, L. M., et al. Interleukin-7 receptor mutants initiate early T cell precursor leukemia in murine thymocyte progenitors with multipotent potential. Journal of Experimental Medicine. 211 (4), 701-713 (2014).
- Yokoyama, K., et al. In vivo leukemogenic potential of an interleukin 7 receptor alpha chain mutant in hematopoietic stem and progenitor cells. Blood. 122 (26), 4259-4263 (2013).
- Simmons, A., Alberola-Ila, J. Retroviral transduction of T cells and T cell precursors. Methods in Molecular Biology. 1323, 99-108 (2016).
- . Retroviral systems Available from: https://web.stanford.edu/group/nolan/_OldWebsite/retroviral_systems/retsys.html (2022)
- Xing, Y., Hogquist, K. A. Isolation, identification, and purification of murine thymic epithelial cells. Journal of Visualized Experiments. (90), e51780 (2014).
- Gray, D. H., et al. Developmental kinetics, turnover, and stimulatory capacity of thymic epithelial cells. Blood. 108 (12), 3777-3785 (2006).
- Godfrey, D. I., Kennedy, J., Suda, T., Zlotnik, A. A developmental pathway involving four phenotypically and functionally distinct subsets of CD3-CD4-CD8- triple-negative adult mouse thymocytes defined by CD44 and CD25 expression. Journal of Immunology. 150 (10), 4244-4252 (1993).
- Wang, Y. B., Edinger, M., Mittar, D., McIntyre, C. Studying mouse thymocyte development using multiparametric flow cytometry: An efficient method to improve an 8-color panel on the BD FACSVerse™ system. BD Biosciences–Application Note. , (2012).
- Sakaguchi, N., et al. Altered thymic T-cell selection due to a mutation of the ZAP-70 gene causes autoimmune arthritis in mice. Nature. 426 (6965), 454-460 (2003).
- He, X., Park, K., Kappes, D. J. The role of ThPOK in control of CD4/CD8 lineage commitment. Annual Review of Immunology. 28, 295-320 (2010).
- Wang, Y., et al. A conserved CXXC motif in CD3epsilon is critical for T cell development and TCR signaling. PLoS Biology. 7 (12), e1000253 (2009).
- Aliahmad, P., Kadavallore, A., de la Torre, B., Kappes, D., Kaye, J. TOX is required for development of the CD4 T cell lineage gene program. Journal of Immunology. 187 (11), 5931-5940 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados