Method Article
Co-coltura e trasduzione di timociti murini su cellule stromali esprimenti delta-like 4 per studiare oncogeni nella leucemia a cellule T
In questo articolo
Riepilogo
Questo protocollo descrive l'isolamento dei timociti doppi negativi dal timo di topo seguito da trasduzione retrovirale e co-coltura sul sistema di co-coltura della linea cellulare stromale del midollo osseo a 4 esprimenti delta (OP9-DL4) per ulteriori analisi funzionali.
Abstract
I timociti immaturi di topo trasdotti possono essere differenziati in cellule T in vitro utilizzando il sistema di co-coltura della linea cellulare stromale del midollo osseo simile a 4 delta (OP9-DL4). Poiché la trasduzione retrovirale richiede la divisione delle cellule per l'integrazione del transgene, OP9-DL4 fornisce un ambiente in vitro adatto per la coltivazione di cellule progenitrici ematopoietiche. Ciò è particolarmente vantaggioso quando si studiano gli effetti dell'espressione di un gene specifico durante il normale sviluppo delle cellule T e la leucemogenesi, in quanto consente ai ricercatori di aggirare il processo dispendioso in termini di tempo di generazione di topi transgenici. Per ottenere risultati positivi, è necessario eseguire attentamente una serie di passaggi coordinati che comportano la manipolazione simultanea di diversi tipi di cellule. Sebbene si tratti di procedure molto consolidate, la mancanza di una fonte comune in letteratura spesso richiede una serie di ottimizzazioni, che possono richiedere molto tempo. Questo protocollo si è dimostrato efficace nella trasduzione dei timociti primari seguita dalla differenziazione sulle cellule OP9-DL4. Dettagliato qui è un protocollo che può servire come guida rapida e ottimizzata per la co-coltura di timociti trasdotti retroviralmente su cellule stromali OP9-DL4.
Introduzione
La linea cellulare stromale OP9 del midollo osseo fornisce un utile sistema in vitro per l'induzione della linfopoiesi da diverse fonti di progenitori1. I primi studi che hanno utilizzato cellule OP9 hanno dimostrato che la mancanza di espressione del fattore stimolante le colonie di macrofagi (MCSF) ha contribuito alla capacità della linea cellulare OP9 di supportare l'emopoiesi e la differenziazione delle cellule B dalle cellule staminali ematopoietiche derivate dal midollo osseo (HSC), come è stato successivamente dimostrato anche per le cellule staminali embrionali (ESC)2,3,4,5 . In studi precedenti, la generazione di cellule OP9 che esprimono 1/4 delta-like (OP9-DL1/OP9-DL4) ha permesso l'induzione dell'impegno della linea di lenza T6 e ha dimostrato la capacità di ricapitolare con successo la maturazione timica 7,8. In breve, lo sviluppo delle cellule T è stato descritto dall'espressione sequenziale di molecole CD4 e CD8. I timociti immaturi sono "doppio-negativi" (DN, CD4− CD8−) e possono essere ulteriormente suddivisi in base all'espressione superficiale di CD44 e CD25. I timociti DN si differenziano attraverso lo stadio immaturo single-positivo (ISP), caratterizzato dall'espressione di CD8 nei topi e CD4 nell'uomo, seguito dallo stadio double-positive (DP), caratterizzato dalla co-espressione di CD4 e CD8 e, infine, dallo stadio maturo single-positivo, caratterizzato dall'espressione di CD4 o CD89. Le HSC esprimono il recettore Notch1, che normalmente interagisce con il delta-like 4 (DL4) espresso sulle cellule epiteliali timiche per indurre la differenziazione della linea T10. Pertanto, l'interesse per il modello OP9-DL1/4 è progressivamente aumentato, portando all'uso estensivo di questo approccio in un'ampia varietà di applicazioni negli ultimi due decenni 8,11,12,13. Sebbene DL1 e DL4 siano entrambi in grado di supportare la differenziazione delle cellule T in vitro, mostrano requisiti differenziali in vivo e alcuni studi hanno suggerito che OP9_DL4 è più efficiente di OP9_DL1 nel ricapitolare l'ambiente timico del topo 7,14.
Tra le potenziali applicazioni del sistema OP9-DL1/4, vi è un particolare interesse per la combinazione di questo sistema con la trasduzione di cellule DN o HSC con vettori retrovirali. Questa combinazione è un modo efficace per manipolare l'espressione genica durante il normale sviluppo delle cellule T e la leucemogenesi e ha dimostrato di essere un metodo efficace per indurre o inibire la funzione di un gene di interesse15,16,17. Questo modello è stato utilizzato con particolare successo per studiare la collaborazione tra gli oncogeni che guidano la leucemia15 in quanto è flessibile e consente l'esame degli effetti di più combinazioni geniche in un tempo ragionevole, in contrasto con la generazione di topi transgenici. Inoltre, modelli simili sono stati utilizzati in precedenza per valutare gli effetti dell'introduzione di oncogeni nelle cellule normali15,16,17. Inoltre, la trasduzione retrovirale richiede la divisione delle cellule per l'integrazione del transgene18, e mentre la trasduzione lentivirale supererebbe questa limitazione eliminando la necessità di dividere le cellule per l'integrazione del transgene, non siamo stati in grado di ottenere la trasduzione di successo dei timociti DN usando vettori lentivirali. Pertanto, OP9-DL1 / DL4 è uno strumento adatto per la crescita di cellule progenitrici ematopoietiche.
Il protocollo standard per la linfopoiesi dei timociti trasdotti su OP9-DL4 prevede una serie di passaggi coordinati che devono essere eseguiti con attenzione per ottenere un risultato positivo. Sebbene queste siano tecniche che hanno servito bene la comunità per molti anni, spesso i protocolli disponibili in letteratura sono frammentati. Di conseguenza, ogni laboratorio è costretto ad adattare e ottimizzare le diverse fasi della procedura, il che può richiedere molto tempo. Qui, questo protocollo descrive l'isolamento dei timociti DN dal timo di topo, seguito da trasduzione retrovirale e co-coltura su cellule stromali OP9-DL4 per ulteriori analisi funzionali. Questo protocollo stabilito ha dimostrato di essere efficiente e riproducibile nella trasduzione dei timociti primari, seguita dalla differenziazione sulle cellule OP9-DL4 o dall'induzione della leucemia linfoblastica acuta delle cellule T15.
Protocollo
Tutti gli esperimenti sugli animali descritti sono stati approvati dal NIH Institutional Biosafety Committee (IBC) e dall'Animal Care and Use Committee (ACUC). Vedere la tabella dei materiali per i dettagli relativi a tutti i reagenti e materiali utilizzati in questo protocollo. Fare riferimento alle linee guida pubblicate19 per maggiori dettagli sulla coltura cellulare produttrice di retrovirus e sulle procedure di mantenimento. Vedere la Figura 1 per una panoramica del protocollo.
1. Avvio della coltura di cellule OP9-DL4 (Giorno 1)
- Scongelare rapidamente le cellule OP9-DL4 tenendo il flaconcino e agitando delicatamente a bagnomaria a 37 °C. Trasferire immediatamente le cellule in una provetta da centrifuga contenente 5 mL di terreno OP9 (terreno MEM-alfa, 10% FBS, 50 U/mL di penicillina/streptomicina, 55 μM di 2-mercaptoetanolo, 2 mM di L-glutammina) per rimuovere gli agenti crioprotettivi.
- Centrifugare a 300 × g per 5 minuti a temperatura ambiente. Eliminare il surnatante e risospendere le cellule in 1 mL di terreno OP9.
- Introdurre 5 mL di terreno OP9 in un matraccio T25 e aggiungere le celle risospese a questo pallone. Coltura a 37 °C e 5% di CO2.
- Dopo 2-3 giorni, sottocolture le cellule usando una divisione 1: 3 (passaggio delle cellule ogni lunedì, mercoledì e venerdì).
NOTA: dividere le celle OP9-DL4 prima che raggiungano la confluenza. Il matraccio OP9-DL4 deve essere confluente all'80%-90% nei giorni 6-7 e nei giorni 8-9+ per la cocoltura con timociti. Quindi, è importante pianificare quante celle OP9-DL4 saranno necessarie in quei giorni mentre si dividono le celle OP9-DL4.- Scartare il mezzo dal pallone e lavare il monostrato con 1x PBS. Scartare il PBS, aggiungere 1 mL di tripsina allo 0,25% e incubare per 1-5 minuti a 37 °C o fino a quando le cellule non si sono staccate dal pallone. Battere il pallone per rimuovere le cellule.
NOTA: (Opzionale) L'andamento della dissociazione cellulare può essere controllato al microscopio. - Aggiungere 5 mL di terreno OP9 per inattivare la tripsina e risospendere le cellule risciacquando la superficie del pallone ricoperta di cellule durante la risospensione.
- Centrifugare la sospensione cellulare a 300 × g per 5 minuti a temperatura ambiente e risospendere in 3 ml di terreno OP9 (per una divisione 1:3).
- Introdurre 1 mL della sospensione cellulare in un matraccio T25 che contenga già 5 mL di terreno OP9 fresco. Congelare/scartare le celle rimanenti.
- Per congelare le cellule OP9-DL4, risospendere le cellule in 90% FBS/10% DMSO con un rapporto di 1 mL di mezzo di congelamento per un flaconcino di cellule.
- Congelare in 1 mL di crioviali a -80 °C o in azoto liquido, a seconda delle esigenze future. Conservare in azoto liquido per una conservazione prolungata.
- Scartare il mezzo dal pallone e lavare il monostrato con 1x PBS. Scartare il PBS, aggiungere 1 mL di tripsina allo 0,25% e incubare per 1-5 minuti a 37 °C o fino a quando le cellule non si sono staccate dal pallone. Battere il pallone per rimuovere le cellule.
2. Avvio della coltura della linea cellulare produttrice di retrovirus (Giorno 1)
- Scongelare rapidamente la linea cellulare produttrice di retrovirus tenendo il flaconcino e agitando delicatamente a bagnomaria a 37 °C. Trasferire immediatamente le cellule in una provetta da centrifuga contenente 5 ml di terreno cellulare produttore di retrovirus (RPC: DMEM, 10% FBS) per rimuovere l'agente crioprotettivo.
- Centrifugare a 300 × g per 5 minuti a temperatura ambiente. Eliminare il surnatante e risospendere le cellule in 1 mL di terreno RPC.
- Introdurre 5 mL di terreno RPC in un matraccio T25 e aggiungere le celle risospese a questo pallone. Coltura a 37 °C e 5% di CO2.
- Passare le celle ogni 2-3 giorni con una divisione da 1:5 a 1:8.
- Colpire il pallone per rimuovere le cellule e risospendere le cellule pipettando in modo aggressivo.
NOTA: Queste cellule possono essere rimosse dalla superficie del pallone mediante pipettaggio aggressivo senza tripsina. In alternativa, tripsinizzare (0,05% tripsina / 0,53 mM EDTA) fino a quando le cellule si staccano facilmente e possono essere facilmente pipettate in una sospensione a singola cellula.
- Colpire il pallone per rimuovere le cellule e risospendere le cellule pipettando in modo aggressivo.
- Centrifugare la sospensione cellulare a 300 × g per 5 minuti e risospendere in 5 ml di mezzo RPC (per una divisione 1:5).
- Introdurre 1 mL della sospensione cellulare in un matraccio T25 che contenga già 5 mL di terreno RPC fresco. Congelare/scartare le celle rimanenti. Congelare le cellule produttrici di retrovirus nello stesso modo descritto per le cellule OP9-DL4 (passaggi 1.4.4.1–1.4.4.2).
3. Inizio della coltura delle cellule produttrici di retrovirus in piastre a 6 pozzetti (giorni 4-5)
- Seminare le cellule produttrici di retrovirus 18-24 ore prima della trasfezione al 70%-90% di confluenza in ciascun pozzetto di una piastra di coltura tissutale a 6 pozzetti in 2 ml di terreno RPC e incubare a 37 °C e 5% di CO2.
NOTA: Trasfettare due pozzetti delle cellule produttrici di retrovirus per ogni 2-5 × 105 a 1 × 106 timociti che verranno trasdotti.
4. Trasfettare le cellule produttrici di retrovirus per generare il retrovirus contenente i geni di interesse (Giorni 5-6)
- Sostituire il terreno con un mezzo RPC fresco 1 ora prima della trasfezione.
- Preparare miscele di lipofezione (per ogni pozzetto di una piastra a 6 pozzetti, scalabili secondo necessità): diluire 4 μg di DNA (2 μg di plasmide helper (pCL-Eco) e 2 μg di plasmide di trasferimento (pMIG) in 250 μL di terreno a siero ridotto. Mescolare delicatamente.
- Mescolare 10 μL di reagente di trasfezione con 250 uL del mezzo a siero ridotto dal punto 4.2. Incubare per 5 minuti a temperatura ambiente.
- Dopo 5 minuti di incubazione, combinare il DNA diluito con un reagente di trasfezione diluito (volume totale = 500 μL). Mescolare delicatamente e incubare per 20-25 minuti a temperatura ambiente.
- Aggiungere delicatamente i 500 μL di miscele di reagenti DNA/trasfezione al pozzetto contenente le cellule produttrici di retrovirus facendo cadere sulle cellule con un movimento circolare. Mescolare delicatamente facendo oscillare la piastra avanti e indietro e incubare la piastra in un'incubatrice a 37 °C per 16-24 ore.
NOTA: Le cellule subconfluenti (80% -90%) sono più adatte per la trasfezione e potenzialmente generano il titolo virale più alto. Poiché le cellule produttrici di retrovirus si staccano molto facilmente dal pallone, evitare movimenti improvvisi quando si maneggiano queste cellule.
5. Modifica del mezzo cellulare produttore di retrovirus (giorni 6-7)
- Sostituire il vecchio mezzo circa 16 ore dopo la trasfezione con 2 ml di terreno RPC fresco. Continuare ad incubare le cellule a 37 °C per 20-24 ore.
- Valutare l'efficienza di trasfezione al microscopio a fluorescenza (opzionale).
NOTA: In questo protocollo, le cellule GFP-positive sono le cellule di interesse (vedere Figura 2). Questo passaggio dipende dal vettore retrovirus selezionato per l'uso in questo protocollo (se un gene reporter è presente nella spina dorsale).
6. Preparazione dei timociti e deplezione delle cellule CD4+ e CD8+ (giorni 6-7)
- Raccogli il timo da un topo C57BL / 6 di 4-6 settimane. Per maggiori dettagli sulla raccolta del timo, fare riferimento a Xing e Hogquist20.
NOTA: I topi sono stati eutanizzati per inalazione di CO2 seguita da lussazione cervicale. - Preparare una sospensione monocellulare di timociti posizionando il timo in 5 ml di PBS in una capsula di Petri. Utilizzando vetrini sterili, posizionare il timo tra le superfici smerigliate dei vetrini e strofinare delicatamente i vetrini insieme, facendo rotolare il timo tra i due vetrini. Risciacquare i vetrini per raccogliere le cellule e scartare il tessuto stromale timico residuo.
NOTA: La resa stimata da un timo è 90 × 10 6-100 × 106 cellule e circa l'1% delle cellule rimarrà dopo la deplezione di CD4 e CD8. Utilizzare il tessuto del timo dei topi più giovani per una migliore resa delle cellule, poiché la cellularità del timo del topo nelle prime settimane dopo la nascita aumenta rapidamente, raggiunge un plateau a 4-6 settimane di età e progressivamente evolve successivamente21. Contare le cellule utilizzando un contatore automatico delle cellule del sangue o qualsiasi metodo alternativo. - Filtrare la sospensione timica attraverso un filtro da 30 μm o 40 μm pipettando 5 mL della sospensione timica attraverso un filtro cellulare. Centrifugare le cellule a 300 × g per 10 minuti.
- Rimuovere il surnatante e lisare i globuli rossi con tampone di lisi ACK aggiungendo (a seconda della dimensione del campione) 1 ml di tampone per tubo per 1 minuto. Aggiungere 5 mL di tampone di deplezione cellulare (500 mL di PBS [pH 7,2], 0,5% BSA, 2 mM 0,5M EDTA, pH 8,0) per disattivare il tampone di lisi ACK, centrifugare a 300 × g per 10 minuti e risospendere in 1-5 mL di tampone di deplezione cellulare per il conteggio.
NOTA: Mettere da parte quanto segue sul ghiaccio prima della deplezione (pre-deplezione ): 70 μL di sospensione cellulare da contare (variare a seconda del metodo di conteggio delle cellule scelto) e 200 μL di sospensione cellulare per determinare l'efficienza della deplezione mediante colorazione per CD4 e CD8 e analisi mediante citometria a flusso22,23. - Contare le cellule e centrifugare a 300 × g per 10 minuti. Risospendere le cellule a 1 × 107/80 μL di tampone di deplezione cellulare. Aggiungere 10 μL ciascuno di microsfere CD4 e CD8 per 1 × 107 cellule. Mescolare bene e incubare per 15 minuti al buio in frigorifero (2-8 °C).
- Preparare una colonna di esaurimento sciacquandola con 2 ml di tampone di deplezione ed eliminando il flowthrough.
- Lavare le cellule dal punto 6.6 aggiungendo 1-2 ml di tampone di deplezione per 1 × 107 cellule e centrifugare a 300 × g per 10 minuti. Rimuovere e scartare il surnatante.
- Risospendere fino a 1,25 × 108 celle in 500 μL di tampone di deplezione Applicare la sospensione cellulare alla colonna e raccogliere il flusso attraverso (celle non etichettate). Lavare la colonna 2x con 1 mL di tampone e raccogliere il flusso continuo.
NOTA: aggiungere un nuovo buffer solo quando il serbatoio della colonna è vuoto. Mettere da parte quanto segue sul ghiaccio dopo l'esaurimento (controllo post-esaurimento): 70 μL di sospensione cellulare da contare (variare a seconda del metodo di conteggio delle cellule scelto) e 1.000 μL di sospensione cellulare per determinare l'efficienza della deplezione mediante colorazione per CD4 e CD8 e analisi mediante citometria a flusso22,23. - Colorare i controlli dell'efficienza di deplezione dividendo i 200 μL di cellule raccolte prima dell'esaurimento in quattro provette FACS: non colorato, CD4 monocolorato, CD8 monocolorato e CD4/CD8 a doppia colorazione (utilizzare i campioni non colorati e monocolorati per impostare i parametri di citometria a flusso). Utilizzare i 1.000 μL di cellule raccolte dopo l'esaurimento per colorare CD4 e CD8 e confrontarli con il campione a doppia colorazione raccolto prima dell'esaurimento (vedere i risultati tipici della deplezione in Figura 3).
NOTA: Regolare i volumi per la colorazione con citometria a flusso secondo le raccomandazioni del produttore dell'anticorpo (ad esempio, 1 μL di anti-CD4-FITC + 0,5 μL di anti-CD8-PE per 1 x 106 cellule in 50 μL di tampone).
7. Coltura di timociti su cellule OP9-DL4 (giorni 6-7)
- Posizionare 2-5 × 10da 5 a 1 × 106 timociti post-deplezione in un matraccio T25 di cellule OP9-DL4 confluenti all'80%-90% in mezzo OP9 contenente citochine (IL-7 ricombinante 10 ng/ml e hFLT-3 ricombinante). Coltura a 37 °C e 5% di CO2. Crescere per circa 24 ore su cellule OP9-DL4.
NOTA: Questa durata è necessaria per rendere trasducibili le cellule T (vedere un risultato tipico nella Figura 4). Riservare un matraccio T25 della cocoltura di timociti e cellule OP9-DL4 da utilizzare come controllo (non trasdotto). I timociti non trasdotti saranno colorati insieme ai timociti trasdotti (passo 9.1) come controllo negativo per valutare l'efficienza di trasduzione. I timociti non trasdotti possono anche essere usati per misurare l'effetto dell'espressione del transgene sulla differenziazione cellulare. Eliminare il mezzo contenente citochine dopo 1 mese.
8. Raccolta del retrovirus dal surnatante e utilizzo per trasdurre i timociti (giorni 8-9)
- Raccogliere il surnatante contenente i retrovirus dalle cellule trasfettate inclinando la piastra a 6 pozzetti e posizionando una siringa da 3-5 ml nella parte inferiore della piastra mentre si tira lo stantuffo per aspirare il surnatante. Sostituire il fluido con 2 mL di terreno RPC fresco. Continuare a incubare le cellule a 37 °C per 20-24 ore per la seconda trasduzione al punto 8.9.
- Filtrare il retrovirus surnatante attraverso un filtro a siringa da 0,45 μm e raccogliere il filtrato in una provetta da 50 ml.
NOTA: Non congelare il surnatante retrovirale. Utilizzare una preparazione virale appena fatta per la trasduzione. - Raccogliere i timociti dalla coltura OP9-DL4 mediante pipettaggio aggressivo per rimuovere i timociti e le cellule OP9-DL4 dalla superficie del pallone. Filtrare la sospensione cellulare attraverso un filtro cellulare da 40 μm per rimuovere la maggior parte delle cellule OP9-DL4 e raccogliere il filtrato in un tubo da 50 ml.
NOTA: le celle OP9 sono molto aderenti. Sebbene il pipettaggio aggressivo possa rimuovere alcune delle cellule OP9-DL4 dal pallone, la rottura del monostrato OP9-DL4 durante questo processo è minima e le cellule OP9-DL4 che si staccano verranno filtrate con il filtro cellulare da 40 μm, poiché le cellule OP9-DL4 sono molto più grandi dei timociti primari. Se le cellule OP9-DL4 sono ancora confluenti all'80%-90%, rimuovere i timociti e riplaccarli nello stesso matraccio OP9-DL4. In alternativa, deve essere preparato un nuovo matraccio OP9-DL4. - Centrifugare i timociti nel filtrato dal punto 8.3 a 300 × g per 5 minuti. Scartare il surnatante.
- Nel tubo da 50 ml, risospendere i timociti in 0,5-1 mL di mezzo OP9 + citochine (vedere punto 7.1). Aggiungere 1-2 mL di terreno RPC contenente il virus (il doppio del mezzo RPC rispetto al mezzo di timocita). Aggiungere bromuro di esametrina (concentrazione madre di 10 μg/μL) per ottenere 8 μg di bromuro di esametrina/ml di sospensione cellulare totale.
- Spinoculare centrifugando le cellule a 850 × g per 1 h a temperatura ambiente.
- Risospendere le cellule in 6 mL di mezzo OP9 + citochine per pallone (vedere punto 7.1) e aggiungere nuovamente la sospensione sul monostrato cellulare OP9-DL4.
NOTA: In alternativa, avere nuovi palloni OP9-DL4 pronti a ricevere i timociti trasdotti. Se i timociti devono essere rimessi in un matraccio OP9-DL4 usato, assicurarsi di aggiungere un nuovo mezzo OP9 alle cellule OP9-DL4 durante 1 ora di spinoculazione dei timociti per mantenere le cellule sane e garantire una confluenza dell'80% -90%. - Incubare a 37 °C durante la notte.
- Ripetere i passaggi 8.2-8.7 usando un nuovo pozzetto del supernatante cellulare contenente retrovirus.
9. Mantenimento dei timociti trasdotti su coltura OP9-DL4 per 2-5 giorni o congelamento secondo necessità (Giorno 9+)
- Valutare la differenziazione dei timociti mediante citometria a flusso colorando i timociti per i marcatori di sviluppo delle cellule T come CD4, CD8, CD25 e CD4423. Vedere i risultati tipici della differenziazione dei timociti nella Figura 5.
Risultati
L'efficienza di deplezione può essere valutata citometricamente a flusso marcando la frazione cellulare magneticamente non marcata per CD4 e CD8 dopo separazione cellulare immunomagnetica (MACS) e analizzandola su un dot-plot bivariato bidimensionale (Figura 3). Una buona resa di cellule doppie negative (CD4−, CD8−) è pari o superiore al 95%, come rappresentato nella Figura 3. Due delle cause più comuni di minore resa sono l'errore di calcolo delle microsfere in base al numero di celle e al numero di celle marcate che superano la capacità della colonna. Si consiglia di scegliere il numero corretto di colonne MACS in base al numero di celle etichettate. Quando si lavora con i timociti, il numero di cellule marcate (cellule DP e SP) è quasi uguale al numero di cellule totali (oltre il 96%). Il conteggio delle cellule può essere effettuato in un contatore automatico delle celle, in una camera Neubauer o con qualsiasi metodo alternativo. Di conseguenza, potrebbe essere necessario regolare i volumi allocati per il conteggio delle celle a seconda della macchina specifica e del metodo di conteggio scelto.
È importante determinare il numero di cellule presenti prima e dopo l'esaurimento. Questi conteggi cellulari sono necessari per calcolare il numero di colonne di deplezione LD necessarie e per distribuire uniformemente i timociti DN nel numero appropriato di palloni OP9-DL4. I controlli per la citometria a flusso (cellule non etichettate, cellule marcate per CD4 e cellule marcate per CD8) possono essere eseguiti con il campione pre-deplezione riservato, poiché questo contiene più cellule. Tuttavia, poiché la maggior parte delle celle dovrebbe essere conservata all'interno della colonna, il campione post-esaurimento da etichettare richiederà un volume maggiore. Di conseguenza, potrebbe essere necessario apportare modifiche in base al protocollo di etichettatura selezionato.
Quando si utilizzano vettori che esprimono marcatori schermabili, come un gene di fluorescenza, la trasfezione e la trasduzione possono essere valutate approssimativamente ed empiricamente mediante microscopia a fluorescenza (Figura 2). L'efficienza di trasduzione può essere analizzata prelevando il timocita dal monostrato OP9-DL4, come descritto nella fase 8.3, e osservando l'espressione di un gene di fluorescenza mediante citometria a flusso. L'efficienza della trasduzione utilizzando un vettore retrovirale vuoto con GFP come gene reporter è stata dell'84,2% (Figura 4).
La differenziazione delle cellule T sulle cellule OP9-DL4 può essere osservata 4 giorni dopo la trasduzione. La citometria a flusso viene solitamente eseguita per valutare la differenziazione cellulare indotta dalla co-coltura su cellule OP9-DL4 e/o l'espressione del transgene, come rappresentato nella Figura 5, dove le cellule sono state marcate per CD4, CD8, CD44 e CD25. Esistono diverse possibili combinazioni di marcatura delle molecole di superficie cellulare che si sono dimostrate utili per studiare i meccanismi molecolari e cellulari dello sviluppo delle cellule T nei topi24,25,26,27. Pertanto, i pannelli di anticorpi fluorescenti possono variare a seconda della questione di interesse affrontata. La trasduzione dei timociti DN con il vettore retrovirale vuoto pMIG, mostrato nel pannello B, presentava approssimativamente le stesse proporzioni di singoli positivi (CD4+ o CD8+), doppi positivi (CD4+/CD8+), doppi negativi (CD4−/CD8−), e i suoi sottostadi doppio-negativi 1-4 (DN1-DN4) dei timociti non trasdotti, mostrati nel pannello A, indicando che lo sviluppo delle cellule T non era influenzato dal processo di trasduzione.
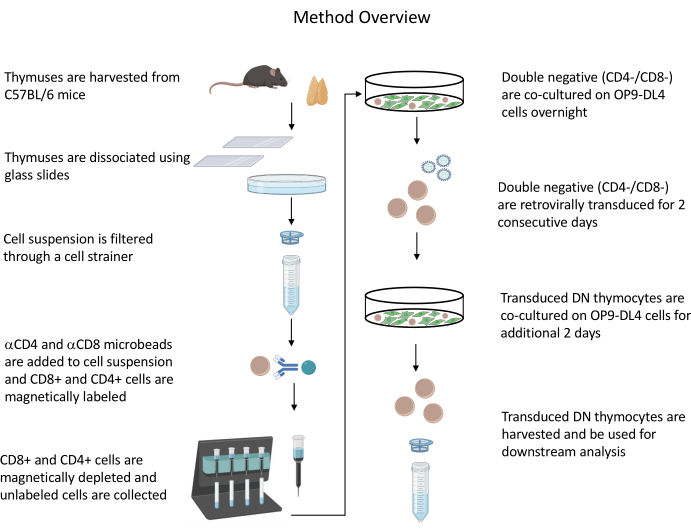
Figura 1: Diagramma delle fasi di isolamento, trasduzione e co-coltura dei timociti. Abbreviazione: DN = doppio negativo. Fare clic qui per visualizzare una versione ingrandita di questa figura.
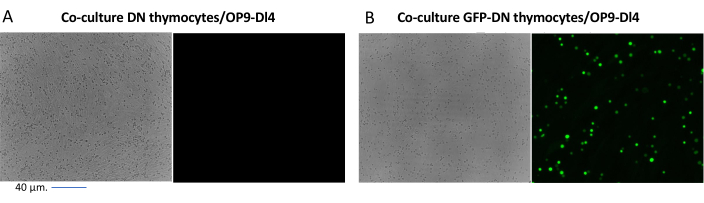
Figura 2: Microscopia a fluorescenza in co-coltura di timociti trasdotti da GFP o timociti non trasdotti e cellule OP9-DL4. (A) timociti non trasdotti e (B) timociti murini stabili che esprimono GFP su OP9-DL4 al giorno 3 dopo la seconda trasduzione. È stato utilizzato un microscopio a fluorescenza Olympus-IX71 con una lente 40x e un filtro 480/30 adatto per la rilevazione GFP. Barra di scala = 40 μm. Abbreviazioni: GFP = proteina fluorescente verde; DN = doppio negativo. Fare clic qui per visualizzare una versione ingrandita di questa figura.
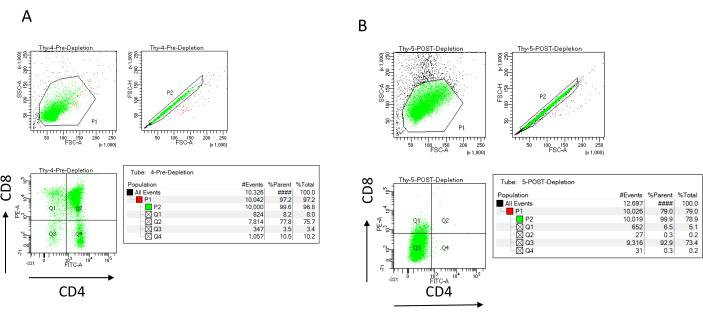
Figura 3: Citometria a flusso della deplezione dei timociti. Grafici rappresentativi di citometria a flusso che analizzano l'espressione di CD4 e CD8 sui timociti ottenuti da topi femmina C57BL/6J di 7-8 settimane (A) pre-deplezione e (B) post-deplezione di CD4+ e CD8+ utilizzando microsfere e colonne LD secondo le istruzioni del produttore. I grafici a punti a sinistra mostrano le porte in base alle dimensioni e alla complessità dell'evento (FCS-A e SSC-A, rispettivamente). Il pannello centrale mostra FSC-H contro FSC-A per eliminare singole celle ed escludere doppietti. Nei grafici a destra, le cellule sono state definite come CD4 e CD8 dalla porta a cella singola. Abbreviazioni: FSC-A = area di picco di dispersione in avanti; SSC-A = area laterale del picco di dispersione; FSC-H = altezza del picco di dispersione in avanti; FITC = isotiocianato di fluoresceina; PE = ficoeritrina. Fare clic qui per visualizzare una versione ingrandita di questa figura.
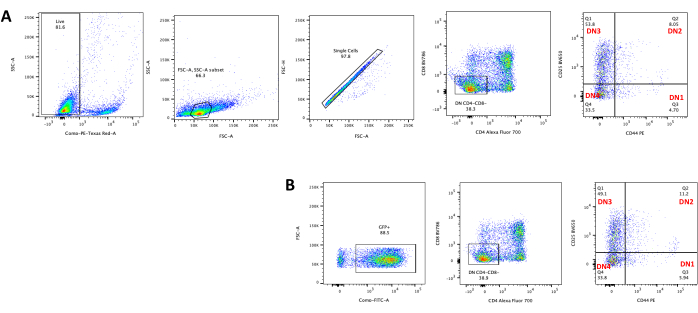
Figura 4: Citometria a flusso dell'efficienza di trasduzione retrovirale dei timociti. (A) timociti non trasdotti co-coltivati su OP9-DL4; (B) timociti trasdotti troviralmente su OP9-DL4 al giorno 3 dopo la trasduzione. Abbreviazioni: FSC-A = area di picco di dispersione in avanti; SSC-A = area laterale del picco di dispersione; FSC-H = altezza del picco di dispersione in avanti; FITC = isotiocianato di fluoresceina; PE = ficoeritrina; DN = doppio negativo. Fare clic qui per visualizzare una versione ingrandita di questa figura.
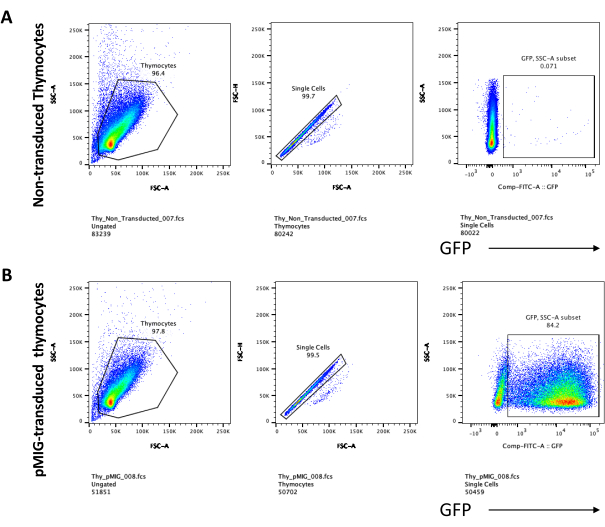
Figura 5: Citometria a flusso di timociti trasdotti dopo 4 giorni di co-coltura OP9-DL4. (A) Non trasdotto e (B) trasdotto con retrovirus pMIG. Le cellule sono state prima recintate su cellule vive e poi controllate in base alle dimensioni e alla complessità (FCS-A e SSC-A, rispettivamente), seguite dal tracciamento di FSC-H rispetto a FSC-A per il gate su singole celle ed escludere doppietti. Per le celle non trasdotte, abbiamo usato la seguente strategia di gate. Dalla porta a singola cellula, le cellule sono state definite come singoli positivi (CD4 + o CD8 +), doppi positivi (CD4 + / CD8 +) e doppi negativi (CD4 - / CD8 - ). Quindi, dal gate doppio negativo (CD4−/CD8−), le cellule sono state definite come CD44+, CD25+, CD44+/CD25+ e CD44−/CD25−, come mostrato nel pannello A. Per le cellule trasdotte da pMIG, mostrate nel pannello B, dalla porta a singola cellula, le cellule sono state prima definite come GFP+/GFP−, e poi dalle cellule GFP+, la distribuzione della popolazione cellulare nelle principali fasi di sviluppo delle cellule T, come definito dall'espressione di CD4, CD8, CD44 e CD25, è stata determinata utilizzando la stessa strategia di gate delle cellule non tradotte. Abbreviazioni: FSC-A = area di picco di dispersione in avanti; SSC-A = area laterale del picco di dispersione; FSC-H = altezza del picco di dispersione in avanti; FITC = isotiocianato di fluoresceina; GFP = proteina fluorescente verde. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Discussione
Il protocollo qui descritto è stato sviluppato specificamente per studi sulle cellule T DN derivate dal timo (CD4−/CD8−) con trasfezione retrovirale seguita da un modello di differenziazione OP9-DL4. Tuttavia, è probabile che le cellule bersaglio che saranno sottoposte a questo protocollo di trasduzione seguito da differenziamento cellulare avranno una più ampia utilità interdisciplinare. Pertanto, oltre ai timociti immaturi, le cellule staminali ematopoietiche, come le cellule derivate dal fegato fetale o dal midollo osseo, potrebbero potenzialmente essere utilizzate in questo protocollo.
Il sistema OP9-DL4 ha dimostrato di essere un modello efficace per studiare la funzione genica in una varietà di aspetti, tra cui la differenziazione cellulare17 e l'oncogenesi15. Mentre la modificazione retrovirale dei progenitori ematopoietici è una tecnica consolidata che consente una modificazione genetica stabile, combinare l'induzione della differenziazione cellulare sul sistema OP9-DL4 e la trasduzione retrovirale richiede cura e abilità. L'aspetto critico per raggiungere il successo con questo protocollo è garantire che tutti i passaggi siano ben coordinati, poiché il protocollo prevede l'utilizzo di tre diversi tipi di cellule che devono essere mantenute sane e alla confluenza ideale richiesta per ogni fase specifica. Con questo in mente, è importante eseguire tutte le analisi dei punti di controllo qualità dopo l'esecuzione di ogni fase prima di procedere alla fase successiva. Ciò garantirà che tutti i passaggi funzionino. Pertanto, il controllo dell'efficienza di deplezione e trasduzione è importante per la corretta esecuzione di questo protocollo (vedere un risultato tipico per l'efficienza di trasduzione nella Figura 4). Una buona efficienza di trasduzione cellulare primaria è legata ad un elevato titolo virale. In genere, inserti più grandi comportano titoli virali più bassi18. Ai fini dell'allenamento, utilizziamo un vettore vuoto per rappresentare i risultati che possono essere ottenuti con questo protocollo. Nella nostra esperienza, le efficienze di trasduzione e trasfezione variano a seconda delle dimensioni dell'inserto, specialmente considerando i geni reporter che esprimono la spina dorsale virale, come la GFP. Una strategia che può essere utilizzata quando si studia l'interazione di più di un gene è quella di clonare ciascun gene in un diverso vettore di trasferimento, seguito dalla produzione di virus individuale e, infine, dalla co-trasduzione della cellula bersaglio. In tal caso, può essere applicata una fase di selezione per eliminare le cellule trasdotte singolarmente e mantenere solo le cellule co-trasdotte.
Vale la pena notare che la maggior parte delle linee cellulari dello strato di alimentazione stromale OP9-DL1 / DL4 sono state geneticamente modificate per esprimere GFP come parte dei costrutti DL1 o DL47. In questo protocollo, abbiamo utilizzato un vettore retrovirale che esprime anche GFP; tuttavia, è più luminoso della proteina GFP espressa dalle cellule OP9-DL4 e non interferisce con l'ispezione di trasduzione quando visualizza la co-coltura al microscopio a fluorescenza.
Le cellule OP9 si differenziano in adipociti dopo molti passaggi, lunghi periodi in coltura o in condizioni di sovraconfluenza19. Ciò è evidenziato dallo sviluppo di grandi vacuoli. Pertanto, le cellule OP9 che presentano queste caratteristiche non dovrebbero essere utilizzate in questo protocollo. La trasfezione di cellule produttrici di retrovirus eccessivamente confluenti si tradurrà in un basso titolo virale. In effetti, la fase subconfluente è quando le cellule sono più trasfettabili. Inoltre, la trasfezione di cellule produttrici di retrovirus a bassa confluenza ridurrà lo stress cellulare nel processo di trasfezione e darà il titolo virale più alto.
Mentre, in questo protocollo, non titoliamo il virus supernatante, la titolazione del virus supernatante deve essere considerata in alcuni casi, come in assenza di un gene reporter nel vettore retrovirale, che impedirebbe la determinazione indiretta della produzione virale, o nei casi in cui il disegno sperimentale richiede un numero più accurato di copie del transgene da integrare nel genoma della cellula bersaglio. Tuttavia, è importante notare che il titolo del vettore retrovirale surnatante diminuisce significativamente se conservato a -80 °C o 4 °C fino a quando non si ottengono i risultati della titolazione. Pertanto, l'uso di supernatante virale appena preparato per la trasduzione produrrà una migliore efficienza di trasduzione.
Il timo contiene un gran numero di timociti doppi positivi (DP) (oltre l'85%) e circa il 10% di15 cellule single-positive (CD4 o CD8), che sono i timociti post-DN. Le cellule DP non possono sopravvivere in vitro per la manipolazione retrovirale, mentre le cellule SP non sono trasducibili. Pertanto, questo protocollo può essere applicato per generare timociti DN transducibili vettori retrovirali.
Divulgazioni
Gli autori non hanno conflitti di interesse da divulgare
Riconoscimenti
Questo lavoro è stato sostenuto dal programma intramurale del National Cancer Institute, progetto ZIABC009287. OP9-DL4 è stato ottenuto dal Dr. Juan Carlos Zúñiga-Pflücker (Sunnybrook Health Sciences Centre, Toronto, ON, Canada). Gli autori ringraziano il NCI-Frederick Laboratory Animal Sciences Program per la loro continua assistenza tecnica e consulenza e input sperimentale, così come Jeff Carrel, Megan Karwan e Kimberly Klarmann per l'assistenza alla citometria a flusso. Siamo grati a Howard Young per i consigli critici e il contributo.
Materiali
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Sigma | M3148 | |
| ACK Lysis buffer | Lonza | 10-548E | |
| BSA | Cell Signaling Technology | 9998S | |
| CD4 Microbeads | Miltenyi | 130-049-201 | |
| CD8 Microbeads | Miltenyi | 130-049-401 | |
| Centrifuge 5910R | eppendorf | 5942IP802353 | |
| DMEM | Corning | 10-013-CV | |
| EDTA | Invitrogen | 15575-038 | |
| Fetal calf serum HyClone FBS | ThermoScientific | SH30910.03 | |
| LD columns | Miltenyi | 130-042-901 | |
| L-glutamine | Sigma | G7513 | Freeze glutamine in aliquots and use freshly-thawed glutamine |
| Lipofectamine 2000 | Invitrogen | P/N 52887 | |
| MEM-alpha Medium | Gibco | 12561-072 | |
| OPTI-MEM I Reducing Serum Medium | Invitrogen | 31985-062 | |
| PBS pH 7.2 | Corning | 21-040-CV | |
| pcL-Eco Plasmid | Addgene | 12371 | |
| penicillin/streptomycin | Gibco | 15140-122 | |
| pMIG Plasmid | Addgene | 6492 | |
| Polybrene | Chemicon | TR-1003-G | |
| Pre-Separation Filters | Miltenyi | 130-041-407 | |
| recombinant hFLT-3L | PeproTech | 300-19 | |
| recombinant IL-7 | Peprotech | 217-17 | |
| Retrovial packaging cell line Phoenix-Eco | Orbigen | RVC-10002 | |
| Syringe filter (0.45 µm) | Millipore | SLHV033RS |
Riferimenti
- Zuniga-Pflucker, J. C. T-cell development made simple. Nature Reviews Immunology. 4 (1), 67 (2004).
- Kodama, H., Nose, M., Niida, S., Nishikawa, S., Nishikawa, S. Involvement of the c-kit receptor in the adhesion of hematopoietic stem cells to stromal cells. Experimental Hematology. 22 (10), 979-984 (1994).
- Nakano, T., Kodama, H., Honjo, T. Generation of lymphohematopoietic cells from embryonic stem cells in culture. Science. 265 (5175), 1098-1101 (1994).
- Nakano, T., Kodama, H., Honjo, T. In vitro development of primitive and definitive erythrocytes from different precursors. Science. 272 (5262), 722-724 (1996).
- Ueno, H., et al. A stromal cell-derived membrane protein that supports hematopoietic stem cells. Nature Immunology. 4 (5), 457-463 (2003).
- Jaleco, A. C., et al. Differential effects of Notch ligands Delta-1 and Jagged-1 in human lymphoid differentiation. Journal of Experimental Medicine. 194 (7), 991-1002 (2001).
- Mohtashami, M., et al. Direct comparison of Dll1- and Dll4-mediated Notch activation levels shows differential lymphomyeloid lineage commitment outcomes. Journal of Immunology. 185 (2), 867-876 (2010).
- Schmitt, T. M., Zuniga-Pflucker, J. C. Induction of T cell development from hematopoietic progenitor cells by delta-like-1 in vitro. Immunity. 17 (6), 749-756 (2002).
- Mazzucchelli, R., Durum, S. K. Interleukin-7 receptor expression: Intelligent design. Nature Reviews Immunology. 7 (2), 144-154 (2007).
- Robey, E. A., Bluestone, J. A. Notch signaling in lymphocyte development and function. Current Opinion in Immunology. 16 (3), 360-366 (2004).
- Lavaert, M., et al. Integrated scRNA-Seq identifies human postnatal thymus seeding progenitors and regulatory dynamics of differentiating immature thymocytes. Immunity. 52 (6), 1088-1104 (2020).
- Roh, K. H., Roy, K. Engineering approaches for regeneration of T lymphopoiesis. Biomaterials Research. 20 (20), (2016).
- Zlotoff, D. A., et al. CCR7 and CCR9 together recruit hematopoietic progenitors to the adult thymus. Blood. 115 (10), 1897-1905 (2010).
- Holmes, R., Zuniga-Pflucker, J. C. The OP9-DL1 system: Generation of T-lymphocytes from embryonic or hematopoietic stem cells in vitro. Cold Spring Harbor Protocols. 2009 (2), (2009).
- Cramer, S. D., et al. Mutant IL-7Ralpha and mutant NRas are sufficient to induce murine T cell acute lymphoblastic leukemia. Leukemia. 32 (8), 1795-1882 (2018).
- Treanor, L. M., et al. Interleukin-7 receptor mutants initiate early T cell precursor leukemia in murine thymocyte progenitors with multipotent potential. Journal of Experimental Medicine. 211 (4), 701-713 (2014).
- Yokoyama, K., et al. In vivo leukemogenic potential of an interleukin 7 receptor alpha chain mutant in hematopoietic stem and progenitor cells. Blood. 122 (26), 4259-4263 (2013).
- Simmons, A., Alberola-Ila, J. Retroviral transduction of T cells and T cell precursors. Methods in Molecular Biology. 1323, 99-108 (2016).
- . Retroviral systems Available from: https://web.stanford.edu/group/nolan/_OldWebsite/retroviral_systems/retsys.html (2022)
- Xing, Y., Hogquist, K. A. Isolation, identification, and purification of murine thymic epithelial cells. Journal of Visualized Experiments. (90), e51780 (2014).
- Gray, D. H., et al. Developmental kinetics, turnover, and stimulatory capacity of thymic epithelial cells. Blood. 108 (12), 3777-3785 (2006).
- Godfrey, D. I., Kennedy, J., Suda, T., Zlotnik, A. A developmental pathway involving four phenotypically and functionally distinct subsets of CD3-CD4-CD8- triple-negative adult mouse thymocytes defined by CD44 and CD25 expression. Journal of Immunology. 150 (10), 4244-4252 (1993).
- Wang, Y. B., Edinger, M., Mittar, D., McIntyre, C. Studying mouse thymocyte development using multiparametric flow cytometry: An efficient method to improve an 8-color panel on the BD FACSVerse™ system. BD Biosciences–Application Note. , (2012).
- Sakaguchi, N., et al. Altered thymic T-cell selection due to a mutation of the ZAP-70 gene causes autoimmune arthritis in mice. Nature. 426 (6965), 454-460 (2003).
- He, X., Park, K., Kappes, D. J. The role of ThPOK in control of CD4/CD8 lineage commitment. Annual Review of Immunology. 28, 295-320 (2010).
- Wang, Y., et al. A conserved CXXC motif in CD3epsilon is critical for T cell development and TCR signaling. PLoS Biology. 7 (12), e1000253 (2009).
- Aliahmad, P., Kadavallore, A., de la Torre, B., Kappes, D., Kaye, J. TOX is required for development of the CD4 T cell lineage gene program. Journal of Immunology. 187 (11), 5931-5940 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon