Method Article
Imagem longitudinal intravital da dinâmica vascular na medula óssea da calvária
Neste Artigo
Resumo
A microscopia intravital permite o estudo de processos biológicos dinâmicos, como regeneração tecidual e desenvolvimento tumoral. A medula óssea da calvária, um tecido altamente dinâmico, oferece informações sobre hematopoiese e função vascular. O uso de um implante de fixação de cabeça impresso em 3D biocompatível permite imagens longitudinais repetitivas, aprimorando nossa compreensão da dinâmica do tecido e do microambiente tumoral.
Resumo
A microscopia de fluorescência longitudinal intravital surgiu como uma técnica crucial para estudar processos biológicos dinâmicos, principalmente no contexto da regeneração tecidual, desenvolvimento de tumores e respostas terapêuticas. Particularmente, a medula óssea da calvária é um tecido altamente dinâmico, onde o destino hematopoiético está interligado com o microambiente circundante, com vasos especializados que respondem à hematopoiese normal e patológica. A imagem tradicional de tecidos fixos oferece informações estáticas, muitas vezes limitando uma compreensão abrangente desses processos.
A integração de animais transgênicos que expressam marcadores específicos de células, traçadores de células vivas, avanços em equipamentos de imagem e o uso de câmaras especializadas elevaram a microscopia intravital a uma ferramenta fundamental para obter insights sobre fenômenos biológicos dinâmicos. Uma aplicação da imagem intravital é a investigação do comportamento dos vasos tumorais e dos efeitos terapêuticos. Um implante de fixação de cabeça de titânio impresso em 3D recém-projetado pode ser conectado de forma estável ao crânio do camundongo e é adequado para imagens longitudinais durante várias sessões.
O protocolo proposto permite o exame espacial e temporal da dinâmica vascular na medula óssea da calvária, incluindo visualização e quantificação da heterogeneidade vascular, interação com células estromais e hematopoiéticas e mensuração de parâmetros funcionais vasculares. Além disso, a técnica permite a visualização de leitos vasculares estabelecidos e o monitoramento dos efeitos terapêuticos, mobilização de células-tronco e localização de compostos quimioterápicos ao longo do tempo usando microscopia de dois fótons. No geral, este protocolo de imagem longitudinal intravital fornece uma plataforma abrangente para investigar o comportamento dos vasos tumorais e a dinâmica das células hematopoiéticas, oferecendo informações valiosas sobre os intrincados processos que regem esses fenômenos biológicos.
Introdução
A microscopia intravital da medula óssea (MO) da calvária serve como uma técnica poderosa e indispensável para investigar os processos dinâmicos de hematopoiese, regulação do microambiente tecidual e dinâmica vascular dentro do nicho da MO. O objetivo principal desta abordagem metodológica é permitir a visualização e análise em tempo real de comportamentos celulares, interações e organização espacial dentro do microambiente BM in vivo. Ao observar diretamente a calvária da MO usando técnicas avançadas de imagem juntamente com a marcação fluorescente, os pesquisadores podem elucidar a complexa interação entre células-tronco hematopoiéticas (HSCs), células estromais e a vasculatura circundante, fornecendo informações cruciais sobre a regulação da hematopoiese e respostas imunes.
O desenvolvimento e a utilização de células-tronco de imagem de microscopia intravital a partir das limitações dos métodos tradicionais de imagem histológica e ex vivo, que muitas vezes não conseguem capturar a natureza dinâmica dos comportamentos celulares e interações de um tecido. Ao contrário das técnicas de imagem estática, a microscopia intravital permite que os pesquisadores observem a dinâmica celular em tempo real, permitindo o estudo de processos longitudinais, como migração, proliferação e diferenciação celular dentro de seu nicho nativo, sem sacrificar animais experimentais. Além disso, a microscopia intravital oferece a vantagem única de estudar comportamentos funcionais in vivo, como funcionalidade vascular (por exemplo, perfusão, permeabilidade, hipóxia), preservando assim a relevância fisiológica e evitando artefatos associados à fixação e processamento de tecidos. Estudos pioneiros na área demonstraram as tremendas vantagens dessa abordagem 1,2, e seus achados foram corroborados e expandidos por abordagens refinadas mais recentemente 3,4,5 que utilizaram microscopia intravital para rastrear a localização, migração e interações endógenas do HSC com a vasculatura dentro do nicho de MO. Além disso, a microscopia intravital tem sido fundamental para elucidar os mecanismos subjacentes aos distúrbios hematopoiéticos, como leucemia e síndromes de falência da MO, oferecendo novos insights sobre a motilidade celular leucêmica 6,7, implicações vasculares associadas à doença8 e resposta a drogas9.
Existem várias vantagens da microscopia intravital da MO da calvária cobrir locais ósseos alternativos. Primeiro, o MO contido no osso do crânio fornece fácil acessibilidade para imagens intravitais em comparação com ossos mais profundos, como o fêmur ou a tíbia. Essa acessibilidade facilita a observação direta do microambiente tecidual, incluindo o próprio osso, por meio da visualização da geração de segundo harmônico (SHG)10, sem a necessidade de procedimentos cirúrgicos invasivos. Em segundo lugar, o crânio é relativamente fino e translúcido, permitindo uma melhor visualização da dinâmica celular dentro do nicho de MO. Essa transparência facilita a imagem de alta resolução com microscopia de dois fótons, microscopia confocal e microscopia de folha de luz, sem a necessidade de técnicas de afinamento ou limpeza óssea. O osso da calvária oferece uma plataforma estável, plana e rígida para experimentos de imagem intravitais, minimizando artefatos de movimento do tecido e garantindo condições de imagem consistentes durante períodos prolongados de observação. Essa estabilidade é particularmente vantajosa para estudos longitudinais que rastreiam comportamentos e respostas celulares ao longo do tempo. A reprodutibilidade é outra grande vantagem, dada a estrutura relativamente pequena e espacialmente definida do osso da calvária em animais experimentais. Essa uniformidade facilita a comparação entre diferentes grupos experimentais e permite uma análise estatística robusta dos dados de imagem.
Aqui, descrevemos um método para obter imagens da MO da calvária de camundongo por meio de microscopia intravital de dois fótons, introduzindo um implante de fixação de cabeça11 recém-desenvolvido, impresso em 3D usando liga de titânio biocompatível de grau 23 (Ti6Al4V), que apresenta uma capa dura dedicada e facilmente posicionada feita da mesma liga de titânio, permitindo o fechamento seguro da ferida para evitar infecções ou danos ao local da cirurgia. O implante fixa a cabeça do mouse de forma firme e estável ao estágio do microscópio por meio de um suporte de implante, minimizando os artefatos respiratórios e permitindo imagens longitudinais das mesmas áreas ao longo do tempo. Alguns exemplos são fornecidos de imagens multicoloridas representando células e estruturas do microambiente BM (superfície óssea SHG+; células mesenquimais nestina-GFP+; cdh5-DSRED+, cdh5-GFP+ ou pdgfb-GFP+ células endoteliais) e o compartimento hematopoiético maligno (células tdTOMATO+ AML), bem como agentes de contraste fluorescentes representando o lúmen dos vasos (dextrano-TRITC). Parâmetros vasculares medidos ao longo de horas ou dias, incluindo comprimento, retidão e diâmetro do vaso, bem como permeabilidade em diferentes regiões vasculares, podem fornecer informações importantes sobre o comportamento e a saúde do tecido.
Protocolo
Todos os experimentos em animais foram realizados sob o acordo ético APAFIS#27215-2020041513522374 v6, aprovado pelo "Ministère de l'enseignement supérieur, de la recherche et de l'innovation" francês.
1. Projeto de um implante de fixação de cabeça de titânio biocompatível para impressão 3D
NOTA: Projetamos um implante de fixação de cabeça biocompatível usando um software de design assistido por computador (CAD) de modelagem paramétrica com recursos integrados de Análise de Elementos Finitos (FEA) (consulte a Tabela de Materiais). As principais entradas para o processo de design incluem um modelo anatômico de alta resolução do crânio do camundongo, um modelo da objetiva do microscópio e um modelo do sistema de fixação, que juntos informam as dimensões e a configuração do implante. O resultado final é um arquivo de malha independente de plataforma, normalmente no formato STL ou STEP, compatível com a maioria dos softwares de impressão 3D. Este formato de arquivo garante uma transferência perfeita para a impressora 3D para uma fabricação precisa do implante.
- Preparação do modelo 3D do mouse
- Posicionar o crânio do rato, preparado ou in vivo, no scanner de micro-CT.
- Defina os parâmetros de digitalização para uma resolução ideal de detalhes anatômicos finos: abra o Painel de Controle e, na seção Condições de Digitalização , escolha um Campo de View (FOV). Use o LiveView para definir o volume de digitalização.
NOTA: Através da resolução do detector, o FOV determinará o tamanho do voxel com base na resolução do detector. Em máquinas de micro-CT mais recentes, um FOV de 5 mm normalmente corresponde a um tamanho de voxel de 5 μm, FOV de 10 mm a um tamanho de voxel de 10 μm, FOV de 25 mm a um tamanho de voxel de 21 μm. O FOV limitado nesta resolução pode exigir várias varreduras para cobrir todo o crânio. Várias varreduras exigem sobreposição entre as varreduras. - Junte várias varreduras de alta resolução em um software de imagem para criar um modelo único e abrangente do crânio.
- Importe arquivos DICOM micro-CT para o software de segmentação (por exemplo, 3D Slicer, Amira, Avizo ou Mimics) para isolar o crânio.
- Execute a segmentação baseada em limiar para separar o tecido ósseo das estruturas circundantes e faça ajustes manuais para refinar regiões complexas ou ruidosas.
- No 3D Slicer, primeiro importe arquivos DICOM e carregue o conjunto de dados e observe os cortes de TC nas visualizações de corte axial, sagital e coronal .
- Abra o Segment Editor, clique em Adicionar para criar uma nova segmentação | Adicionar para criar um novo segmento, que deve ser limitado.
- No painel Editor de segmentos , clique no efeito Limiar . Ajuste os controles deslizantes de limite inferior e superior (ou digite valores numéricos) para que a região a ser segmentada seja destacada em vermelho nas exibições de fatia. Um segmento 3D com limite está pronto.
- Para exportar o crânio segmentado como um arquivo STL, mude para o Módulo de segmentação. Certifique-se de que o segmento com limite esteja selecionado. Na seção Exportar/Importar Modelos e Mapas de Rótulos , defina Tipo de exportação como Modelos. e Formato de arquivo: STL. Clique no botão Exportar .
NOTA: Os arquivos STL usam um formato de malha triangular, representando a superfície do crânio com milhares de triângulos, capturando a forma anatômica em detalhes.- Se necessário, faça a costura no MeshLab da seguinte maneira:
- Importe malhas clicando em Arquivo | Importe a malha e selecione uma como uma verificação de referência.
- Alinhe outras digitalizações usando a ferramenta Alinhar (Filtros | Inscrição | Alinhar) marcando os pontos correspondentes e usando Processo.
- Mescle as varreduras alinhadas clicando em Filtros | Camada de malha | Achatar camadas visíveis com a opção Mesclar somente camadas visíveis .
- Se necessário, faça a costura no MeshLab da seguinte maneira:
- Limpe e repare a malha, eliminando vértices e triângulos duplicados da seguinte forma, para garantir a remoção de triângulos induzidos por ruído e o preenchimento de quaisquer orifícios não intencionais, para obter uma superfície lisa e contínua:
- Clique em Filtros | Limpeza e Reparação | Remova os vértices duplicados.
- Clique em Filtros | Limpeza e Reparação | Remova faces duplicadas.
- Clique em Filtros | Limpeza e Reparação | Remova peças isoladas (por número de face). Especifique um limite para o número mínimo de faces a serem retidas (por exemplo, defina esse limite baixo para remover pequenos aglomerados ou ruído).
- Clique em Filtros | Remalhamento, simplificação e reconstrução | Feche os buracos.
- Execute a simplificação da malha para ~ 10.000-30.000 triângulos, pois os arquivos STL iniciais do micro-CT contêm contagens de triângulos muito altas que não são compatíveis com o software CAD. Em áreas de alto detalhe, mantenha triângulos menores na parte superior do crânio para garantir o ajuste preciso do implante. Em regiões mais grosseiras, simplifique regiões menos críticas com triângulos maiores para reduzir o tamanho do arquivo e otimizar a eficiência do processamento.
NOTA: A simplificação da malha é feita por áreas e detalhada em 1.1.8.1-1.1.8.3.- Use a ferramenta de seleção Selecionar componentes conectados em uma região para selecionar áreas para simplificação.
- Use a dizimação de recolhimento de aresta quadrática nas áreas selecionadas com parâmetros. Clique em Filtros | Remalhamento, Simplificação e Reconstrução | Simplificação: Dizimação de colapso de aresta quádrica. Defina o Limite de qualidade como 0,8, Preserve o limite da malha como YES, Preserve a topologia como YES, Número de destino de faces: defina um número de destino correspondente à resolução necessária.
- Itere conforme necessário até que o número total de triângulos seja reduzido ao nível desejado. Em seguida, salve o modelo simplificado em um arquivo STL.
- Preparação do modelo de crânio no modelo CAD
- Importe o modelo de crânio simplificado no formato STL para o software CAD como uma nova peça ou modelo. Certifique-se de que o arquivo importado seja convertido corretamente em um corpo sólido ou de superfície, se necessário, dependendo dos recursos do software. Salve o modelo no formato de arquivo CAD nativo para permitir modificações adicionais e garantir a compatibilidade com as etapas subsequentes do projeto.
- Coloque pontos de referência em Bregma e Lambda e crie o eixo Bregma-Lambda neste modelo.
- Crie o plano mediano, incluindo o eixo Bregma-Lambda.
- no plano mediano, crie um eixo tangencial à calvária e, em seguida, salve o crânio preparado (Figura Suplementar S1).
- Crie uma nova montagem e insira o crânio preparado de forma que Bregma coincida com a origem, o plano mediano com o plano direito e o eixo da calvária seja horizontal.
- O implante de fixação da cabeça será projetado diretamente dentro do conjunto para garantir um ajuste perfeito com o modelo de crânio preparado. Para conseguir isso, crie o implante como uma nova peça dentro da montagem. Comece navegando até Inserir | Componente | Nova peça no software CAD. Quando solicitado, selecione a superfície da calvária ou o plano superior da montagem como referência para posicionar e ancorar a nova peça.
- Crie um conjunto de planos igualmente espaçados (espaçamento de 2 mm) ao longo do crânio. Em cada plano, crie um esboço e, em cada esboço, desenhe uma curva projetada de deslocamento de 0,2 mm na forma de uma spline simétrica com nove pontos, seguindo a seção da superfície superior do crânio 0,2 mm acima do crânio (Figura Suplementar S2).
- Crie uma superfície varrida usando essas estrias - a Superfície de Limite do Crânio - garantindo que essa superfície seja lisa sem nenhum defeito, para ser usada na construção das características do implante.
- Definição da superfície observável no crânio
NOTA: Com base no modelo 3D, a região mais plana do osso da calvária ideal para observação 3D é identificada. Esta região é cuidadosamente escolhida para garantir o acesso ideal para a objetiva do microscópio de dois fótons, considerando a integridade biológica e estrutural do crânio. A definição pode usar coordenadas estereotáxicas onde o ponto Bregma serve como origem do sistema de coordenadas.- Crie um esboço no plano da calvária e crie uma spline em forma de pêra de AP +6,5 a -2, 6 mm de largura em AP 0,0. Este será o Contorno de Superfície Observável (Figura Suplementar S3).
NOTA: A objetiva do microscópio está virtualmente posicionada em relação à superfície observável previamente definida no crânio. Esta etapa envolve determinar as restrições espaciais impostas pelo tamanho e forma da objetiva, garantindo que o projeto do implante acomode os requisitos do microscópio. - Insira o modelo de objetiva do microscópio na montagem verticalmente. Restrinja seu ponto de foco em um plano paralelo ao plano da calvária no osso da calvária e defina o volume delimitador da objetiva do microscópio.
- Crie um volume varrendo a objetiva seguindo o Contorno da superfície observável.
- Crie uma janela de observação ao redor da superfície observável definida, garantindo acesso desobstruído para a objetiva do microscópio.
NOTA: A janela de observação é uma estrutura em forma de anel com funções secundárias, como retenção de líquido de imersão e estrutura sólida para o fechamento correto da tampa de proteção. - Crie um esboço no plano da calvária e copie o contorno da superfície observável.
- Crie um contorno paralelo de 0,5 mm de espessura. Faça a extrusão deste contorno duplo para cima 0,9 mm e para baixo até a superfície Superfície limite do crânio com um ângulo de inclinação de 4°.
- Crie um esboço no plano da calvária e crie uma spline em forma de pêra de AP +6,5 a -2, 6 mm de largura em AP 0,0. Este será o Contorno de Superfície Observável (Figura Suplementar S3).
- Definição das estruturas de cimentação, cauda e cauda de andorinha.
- Crie um esboço no plano da calvária e desenhe uma forma de C de 0,5 mm de espessura conectando-se à Janela de Observação e extrude 2 mm de altura e para baixo até a superfície da Superfície Limite do Crânio com um ângulo de inclinação de 4 ° (Figura Suplementar S4).
- Crie um esboço no plano da calvária e preencha esse espaço com retângulos de 0,4 mm de espessura colocados radialmente com 1 mm de espaço entre eles.
- Faça a extrusão de forma semelhante à janela de observação.
- Crie a cauda do implante contando com o crânio para a fixação, respeitando o volume disponível.
- Crie um plano transversal tangencialmente à estrutura de cimentação e outro 12,5 mm para trás.
- Esboce retângulos nesses planos. Varra um corpo piramidal entre eles, definindo os ângulos aproximadamente 20 ° e 37 ° da horizontal.
- Adicione um orifício M1.6 rosqueado na cauda perto da estrutura de cimentação.
- Crie um esboço na extremidade da cauda e desenhe o perfil de cauda de andorinha.
- Extrudar este perfil em 8 mm.
- Verificação de não interferência com o objetivo.
- Examine todo o projeto do implante, incluindo a janela de observação e a cauda de andorinha, para garantir que não haja interferência mecânica com a objetiva do microscópio. Verifique se há interferências com o volume criado na etapa 1.3.3.
NOTA: Esta etapa de verificação é fundamental para garantir que a objetiva possa ser posicionada e movida conforme necessário, sem obstrução.
- Examine todo o projeto do implante, incluindo a janela de observação e a cauda de andorinha, para garantir que não haja interferência mecânica com a objetiva do microscópio. Verifique se há interferências com o volume criado na etapa 1.3.3.
- Remova o material desnecessário da cauda do implante para minimizar o peso e reduzir o desconforto potencial para o animal.
- Crie um esboço no plano mediano e desenhe recortes triangulares nele para formar uma estrutura de treliça quando extraído por extrusão do corpo da cauda.
- Repita no plano perpendicular.
- Verifique o deslocamento máximo por simulação do Método dos Elementos Finitos (FEM) para analisar as propriedades mecânicas do implante sob várias condições.
NOTA: Esta simulação ajuda a prever o deslocamento máximo e as tensões dentro do implante durante o uso, garantindo sua confiabilidade.- Ative o suplemento de simulação de software CAD.
- Na barra de ferramentas Simulação, crie um novo estudo estático. Navegue até a guia Simulação (se visível) ou vá para Simulação | Estude. Na caixa de diálogo Estudo , escolha Estático como o tipo de estudo. Clique em OK para criar o novo estudo (Figura Suplementar S5).
NOTA: Este tipo de estudo é adequado para analisar deformações e tensões sob cargas estáticas, conforme esperado no uso do implante. - Escolha um material para o implante que corresponda à composição real do implante (Ti6Al4V).
- Defina restrições fixas para simular os pontos de fixação do implante onde ele entraria em contato com a fixação em cauda de andorinha. Defina a superfície superior da cauda de andorinha como fixa.
- Defina a carga vertical na superfície inferior do implante. Conjunto de 1 N de força vertical.
- Gere uma malha. Refine a malha em áreas onde se espera alta tensão ou deformação (Figura Suplementar S6). Para fazer isso, clique com o botão direito do mouse no elemento de malha da árvore de estudo e selecione Criar malha... Na caixa de diálogo Malha , ajuste a Densidade da malha com o controle deslizante ou ativando a caixa Controle de malha para definir os tamanhos máximo e mínimo dos elementos de forma independente. Clique em OK para gerar a malha.
NOTA: O software gera automaticamente uma malha inicial para o estudo, mas para resultados mais precisos, a malha pode ser refinada para selecionar uma malha mais fina, particularmente em regiões críticas para a estabilidade do implante (como a janela de observação). A malha mais fina nessas regiões melhora a precisão da simulação, capturando as concentrações de tensão locais. - Inicie a análise estática para calcular as distribuições de deslocamento, tensão e deformação no implante.
- Analise os resultados. Examine o gráfico de deslocamento para identificar áreas de deformação máxima. Este gráfico mostra o quanto o implante se deformaria sob as cargas aplicadas, destacando quaisquer áreas com risco de movimento excessivo. Certifique-se de que a deformação dentro da janela de observação não exceda 3 mícrons.
- Por fim, projete uma capa protetora para proteger a janela de observação, quando não estiver em uso. Esta tampa protege o osso exposto de contaminantes ambientais e danos físicos.
2. Tratamentos com camundongos, anestesia e implante cirúrgico do implante de cabeça
NOTA: Aqui, machos ou fêmeas, C57BL / 6 de 7 a 12 semanas de idade ou camundongos transgênicos podem ser usados, conforme ilustrado. Para induzir a colonização leucêmica da MO, as células leucêmicas geradas, conforme descrito por Horton et al.12, são administradas por via intravenosa 2-3 semanas antes da imagem. Para garantir a saúde da ferida, técnicas estéreis devem ser usadas.
- Sob o capô, monte ferramentas cirúrgicas estéreis (tesouras e pinças rombas), aparador e consumíveis, incluindo gel para os olhos, soro fisiológico, desinfetante, cimento dental, cotonetes absorventes e lenços de limpeza. Ligue o esterilizador de contas.
- Encha a câmara de indução anestésica com isoflurano a 4% e oxigênio atmosférico e coloque o camundongo na câmara. Quando o animal estiver totalmente anestesiado (verifique se há perda do reflexo de endireitamento e padrão respiratório mais profundo e lento), mude o fluxo de isoflurano para o cone nasal da máscara anestésica e diminua a concentração de isoflurano para 2%.
- Coloque o mouse em uma almofada de aquecimento a 37 °C e monitore visualmente a frequência respiratória. Opcional: Para camundongos com fenótipo patológico, adicione um cobertor de sobrevivência para preservar a temperatura correta durante toda a anestesia.
- Administre analgésicos por via subcutânea (Buprecare, 0,01 mg/kg) 30 minutos antes da cirurgia.
- Raspe a cabeça do mouse com um barbeador elétrico.
- Adicione uma gota de gel oftálmico nos olhos do camundongo para evitar o ressecamento durante a anestesia, certificando-se de que o gel não pingue no couro cabeludo.
- Limpe o mouse para remover quaisquer pêlos residuais da área cirúrgica. Limpe a parte superior do couro cabeludo com desinfetante em um lenço de algodão. Certifique-se de remover todos os pelos para evitar artefatos de imagem, bem como o risco de infecção da ferida.
- Usando pinças e tesouras estéreis (após 30 s no esterilizador), faça uma pequena incisão na porção central do couro cabeludo para expor a cicatriz óssea central. Siga-o para definir a área de imagem de comprimento e largura correta. Remova cuidadosamente o pedaço do couro cabeludo, levante a pele entre as orelhas usando uma pinça e faça uma pequena incisão na parte de trás da cabeça. Segurando a pele, passe a tesoura sob a pele e corte cuidadosamente fora da área de imagem.
- Remova o tecido conjuntivo entre o crânio e o couro cabeludo. Limpe o osso exposto com PBS estéril em um cotonete. Prossiga rapidamente para a fixação do implante de cabeça ao crânio do camundongo para evitar a exposição prolongada do osso do crânio (<10 min).
- Faça uma pasta com cimento dentário suficiente em uma placa de Petri seguindo as instruções do fabricante e aplique-a rapidamente na parte inferior do implante de fixação da cabeça.
- Sem colocar nenhum cimento dental dentro da área de imagem, coloque o suporte da cabeça no crânio exposto do mouse e espere que ele endureça.
- Uma vez definido (normalmente dentro de 3-5 min), adicione algumas gotas de PBS a 37 ° C na câmara de imagem para manter o crânio hidratado.
3. Imagem usando um microscópio de dois fótons
- Antes de iniciar a cirurgia, ligue o laser e deixe-o aquecer para estabilizar. Ligue o microscópio e inicie o software de aquisição. Selecione a objetiva apropriada (imersão em ÁGUA 25x/0,95 para este experimento).
- Use um suporte de microscópio vertical de estágio fixo que tenha espaço suficiente para colocar uma placa de ensaio veterinária com uma almofada térmica, tudo para ser movido integralmente com o estágio xy. Use uma placa de ensaio óptica de alumínio sólido como plataforma para prender o implante da cabeça e a máscara estereotáxica. Conecte as mangueiras flexíveis e seus conectores associados à injeção e extração de isoflurano à máscara na gaiola do microscópio.
- Defina as configurações de aquisição para digitalizar rapidamente: formato 512 x 512 pixels; velocidade 600 Hz; e fator de zoom 1.
- Ajuste a linha de laser em 880 nm para poder detectar o SHG, GFP e tdTOMATO com o mesmo comprimento de onda de excitação.
- Desligue a luz da sala e feche a caixa ao redor do suporte do microscópio corretamente antes de ativar os detectores NDD e defina as portas de detecção espectral: PMT1, para detectar SHG [388 - 431 nm]; HyD2, para GFP [485 - 548 nm] e HyD3 para tdTOMATO [551 - 645 nm]. Deixe o deslocamento em 0.
- Crie um novo conjunto de dados de projeto e renomeie-o de acordo com o experimento específico.
- Uma vez que o implante de fixação da cabeça esteja preso ao crânio do camundongo (confirmado pelo endurecimento do cimento), ligue o isoflurano em direção à máscara estereotáxica do microscópio.
- Traga o mouse rapidamente para o microscópio. Insira cuidadosamente os dentes do rato na máscara para permitir a penetração do isoflurano pelo nariz do rato, levantando o nariz com uma mão.
- Enquanto segura o mouse com uma mão, deslize suavemente a cauda de andorinha do implante de fixação da cabeça no suporte de fixação com a outra mão e prenda-o com meia volta do botão do parafuso.
- Introduza uma sonda retal pré-embutida com um gel à base de água para monitoramento de temperatura e adicione gotas oftálmicas aos olhos do camundongo.
- Encha a câmara de imagem do cabeçote com uma grande quantidade de gel à base de água ou PBS e abaixe a objetiva de imersão em água para mergulhá-la completamente para obter excitação e detecção ideais dos sinais gerados pela excitação de dois fótons.
- Mova o estágio x-y e a unidade z para focar no osso central na cicatriz do andaime.
NOTA: É conveniente usar as oculares do microscópio e uma lâmpada do tipo iodetos metálicos como fonte de luz, filtrada por um filtro de emissão de excitação tri-band. A falta de seccionamento óptico impossibilita a obtenção de uma imagem nítida; Mas com a prática, a área central do crânio é facilmente reconhecível. - Depois de identificar o tecido, marcado por uma superfície óssea e veia central, desligue a lâmpada de iodetos metálicos e feche as janelas da gaiola do microscópio para proteger os detectores HyD e permitir a passagem do laser infravermelho (IR).
- Defina o ganho PMT/SHG para 850 V (sua faixa de operação linear) e defina os ganhos HyD em 100%. Aumente a potência do laser IR até que uma imagem com uma faixa dinâmica de 200 níveis de cinza (com detectores dinâmicos de 8 bits) seja obtida no mais baixo dos três canais. Reduza os ganhos dos outros detectores se as diferenças de emissão forem muito importantes ou se houver saturação usando o modo "abaixo da tabela de pesquisa inferior" (LUT).
NOTA: No nosso caso, uma potência de laser de 30% foi suficiente para obter contraste em todos os canais. - No software de aquisição, procure uma região composta por bolsas de MO (GFP+, tdTOMATO+) envolta nas superfícies ósseas (SHG).
- Para encontrar diferentes ROIs, ative o LAS NAVIGATOR e crie uma visão geral de toda a região usando o modo espiral. Pare a aquisição quando a superfície adquirida for grande o suficiente. Salve a visão geral, mescle-a e renomeie-a: esta será a imagem de referência no caso de imagens longitudinais.
NOTA: Tenha cuidado para não gerar uma espiral muito grande, pois isso pode fazer com que o suporte do dispositivo entre em contato com a objetiva. - Registre algumas posições de ROI clicando no ícone de imagem única e renomeie cada posição na lista de tarefas. Faça uma captura de tela para ter a posição relativa dos vários ROIs sobreposta à visão geral salva anteriormente.
NOTA: Esta captura de tela será essencial para substituir as ROIs em imagens longitudinais, pois o software de aquisição não permite que esse grupo de posições seja recuperado na versão utilizada para este experimento. - Para adquirir um volume de pilha z, selecione o modo de pilha z, defina o tamanho da etapa em 3 μm e desmarque a opção de tamanho de pilha igual para todas as regiões . Selecione a primeira posição, defina as posições superior e inferior , verifique o tamanho do passo z e clique em redefinir pilha. Um ícone de cubo aparece ao lado do nome da posição na lista de posições. Repita esta etapa para todas as posições dos diferentes ROIs.
- Pressione adquirir e salve as imagens (três canais z-stacks com ~50 planos) na pasta apropriada. Esta etapa marca Tctrl do experimento; Verifique se os parâmetros de aquisição estão corretos (obtenha a faixa dinâmica correta nos três canais, evite a saturação do sinal o máximo possível e verifique o tempo de aquisição para um ciclo completo).
- Para medir uma característica dinâmica (permeabilidade vascular neste caso), adquira um lapso de tempo.
- Nesta configuração, os três detectores permitem imagens simultâneas de três canais; se um quarto canal for necessário para o experimento (monitoramento de Dextran neste caso), ative o modo sequencial e adicione uma segunda sequência de aquisição. Ajuste a linha de laser IR em 820 nm para poder excitar o fluoróforo Dextran Cy5. Altere a faixa de detecção de Hyd2 para [650 - 744 nm] e desative os outros detectores. Selecione entre o modo de aquisição de pilhas .
NOTA: Por padrão, a segunda sequência de aquisição tem os mesmos parâmetros que a sequência 1. - Antes de iniciar o timelapse, inicie a aquisição de todas as posições para garantir que o tempo de gravação não exceda 3 min (levando em consideração a adição do modo sequencial), o tempo mínimo necessário para detectar o vazamento de dextrano.
- Antes da injeção de Dextrana, altere o modo de aquisição para xyzt. No módulo t, ajuste o intervalo de tempo para 3 min e a duração para 1 h.
- Nesta configuração, os três detectores permitem imagens simultâneas de três canais; se um quarto canal for necessário para o experimento (monitoramento de Dextran neste caso), ative o modo sequencial e adicione uma segunda sequência de aquisição. Ajuste a linha de laser IR em 820 nm para poder excitar o fluoróforo Dextran Cy5. Altere a faixa de detecção de Hyd2 para [650 - 744 nm] e desative os outros detectores. Selecione entre o modo de aquisição de pilhas .
- Injete 100 μL de Dextran 70 KDa-TRITC (ou 500 KDa-Cy5) a 3 mg / camundongo por via intravenosa e inicie a aquisição.
- Após a aquisição, salve o conjunto de dados na pasta apropriada.
NOTA: Durante essas várias etapas, monitore a frequência e a temperatura respiratórias do mouse com frequência e ajuste o fluxo de isoflurano, se necessário. Idealmente, temperatura, ECG e frequência respiratória poderiam ser registrados e incluídos como metadados do experimento.
4. Recuperação do mouse
- Quando a sessão de imagem estiver concluída, desative o detector HyDs no software de aquisição e ligue a caixa de aquecimento do mouse (37 °C). Abra a gaiola do microscópio, levante a objetiva e remova a sonda retal do mouse. Em seguida, deslize suavemente a cauda de andorinha do implante de fixação da cabeça para fora do suporte de fixação e mova o mouse da máscara estereotáxica do microscópio para a máscara de cirurgia na almofada térmica.
- Desligue a injeção de isoflurano sob o microscópio e desligue a almofada térmica.
- Remova suavemente o gel à base de água ou PBS do crânio do camundongo com um cotonete estéril.
- Adicionar gel intra-sítio para manter a umidade do crânio entre as sessões de imagem, conforme descrito anteriormente13.
- Feche a área de imagem do implante de cabeça com a capa específica e prenda-a cuidadosamente com o parafuso.
- Coloque o mouse na caixa de aquecimento a 37 °C e espere até que ele acorde. Traga o rato para o biotério e aloje-o em uma gaiola limpa com hidrogel e enriquecimento. Monitore os animais todos os dias para detectar quaisquer sinais de dor ou infecção, caso em que administre Buprecare diariamente. Para preservar a umidade da calvária, troque o gel intrassítio 2x por semana.
NOTA: Escolha uma gaiola alta com abrigos de papelão e material de nidificação e certifique-se de colocar os ratos em gaiolas individuais.
5. Aquisições longitudinais
NOTA: O mouse pode ser fotografado novamente durante os dias seguintes. No entanto, certifique-se de não repetir mais de três sessões de imagem por semana para evitar efeitos indesejáveis de anestesia repetida, como secura ocular ou fadiga excessiva, bem como dificuldade respiratória e hipotermia.
- Para recriar a imagem de um mouse com um suporte de cabeça já instalado, siga a etapa 2.2 e coloque uma pequena gota de gel oftálmico nos olhos do mouse para evitar o ressecamento durante o procedimento.
- Siga a seção 3 até a etapa 3.16 para obter a imagem de visão geral.
NOTA: Usar o implante de titânio da cabeça sem ter que alterar a altura ou o ângulo da posição do suporte torna muito fácil encontrar a área geral de interesse. - Se necessário, realinhe as imagens anteriores e novas usando o módulo abrir imagem e alinhar.
- Abra a imagem de captura de tela da etapa 3.17 para marcar as posições em seu lugar original.
NOTA: O uso do contraste SHG é muito útil, especialmente quando se espera uma remodelação dos vasos sanguíneos e células associadas que expressam GFP. - Adquira z-stacks e timelapses da mesma maneira descrita nas etapas 3.18-3.22.
6. Quantificação de parâmetros vasculares
- Parâmetros vasculares
- Para abrir as imagens geradas com o software (arquivos .lif) no IMARIS, primeiro converta-as para o formato ".ims" com o File Converter. Abra o arquivo ".ims" com IMARIS, verifique os metadados (escalas X, Y, Z) e selecione o módulo Filament . Selecione ignorar criação automática, editar manualmente.
- Para uma melhor visualização durante a geração do filamento, navegue até Configurações (símbolo de folha), selecione Linha no estilo e deixe o número de pixels no valor padrão (1). Marque as caixas Mostrar dendritos, incluindo Ponto inicial, Pontos de ramificação e Pontos terminais. Desmarque Mostrar lombada.
- Em desenhar (símbolo de pincel), selecione Método AutoPath; 5 μm como diâmetro e a fonte do canal associada ao vaso (TRITC-Dextran neste caso). Finalmente, marque o centro automático e o diamete automáticor.
- Comece a desenhar os filamentos usando shift + clique com o botão direito do mouse para escolher um ponto inicial de ramificação e shift + clique esquerdo para escolher um ponto final de uma ramificação.
NOTA: O ponto inicial está em azul claro, os pontos de ramificação estão em vermelho ou azul escuro e os pontos terminais estão em verde. - Para definir o diâmetro correto do filamento durante o desenho, use o botão de rolagem do mouse.
- Para unir dois filamentos com pontos terminais distintos, crie um novo ponto de partida entre eles e conecte-o em cada ponto final.
NOTA: Assim, os pontos terminais verdes aparecem nos pontos de ramificação. - Depois que a árvore vascular for criada, volte para as configurações e certifique-se de que os números de pontos iniciais, ramificados e terminais estejam corretos. Selecione o cone para visualizar os diâmetros e verificar sua aparência.
- Se um diâmetro de filamento (ou conexão) parecer incorreto, navegue até Editar (símbolo de lápis), selecione o filamento a ser removido com um clique com o botão direito do mouse e clique em Excluir. Redesenhe o filamento ajustando o diâmetro do filamento com o botão de rolagem do mouse.
- Para extrair todos os dados estatísticos gerados e salvá-los, clique no ícone Estatística | exportar todas as estatísticas para arquivo-(ícone de vários disquetes na parte inferior da tela).
NOTA: Para evitar quaisquer filamentos potencialmente aberrantes que não correspondam aos vasos, apenas filamentos com mais de 20 μm foram incluídos na análise estatística.
- Quantificação de vazamento
NOTA: Para abrir as imagens geradas com o software (arquivo .lif) no IMARIS, primeiro converta-as para o formato ".ims" com o File Converter.- Para mesclar imagens tiradas antes e depois da injeção de dextrano na mesma série temporal, vá para o modo Superar, clique na primeira imagem (Tctrl , antes da injeção de dextrana) e na imagem de lapso de tempo (tempo 1, 60 min) para abri-las e combine-as clicando em Editar e adicione Pontos de Tempo.
NOTA: Para corrigir eventuais desvios 3D, siga as etapas 6.2.2-6.2.7 usando a função de ponte entre Fiji e IMARIS. Instale os seguintes plugins em Fiji: "IMARIS_Bridge96.jar" e "IMARISBridgeUtils.jar". O registro de desvio 3D é realizado usando "StackReg Pluging" (já disponível em Fiji). Se não for necessário, vá para a etapa 6.2.8 - Para fazer a correção de desvio 3D, abra Fiji para conectar imagens do IMARIS clicando em Plugins | IMARIS | Imagem do IMARIS.
- Em Fiji, selecione Plug-ins | Inscrição | Corrigir desvio 3D.
- Nas novas janelas exibidas, escolha o canal apropriado para o registro.
NOTA: Foi Nestin-GFP neste caso, mas pode ser qualquer canal destacando objetos imóveis e alta relação sinal-fundo. - Para aprimorar a detecção de pequenos desvios, selecione Computação de escala de tempo múltipla, Correção de desvio de subpixel e Imagens aprimoradas de borda e deixe as outras configurações como padrão.
- Para fazer a ponte entre a imagem corrigida e o IMARIS, selecione-a e clique em Plugins | IMARIS | Imagem para IMARIS.
- Salve-o na pasta apropriada clicando em Arquivo | Salvar como.
- Para visualizar todos os canais adquiridos, clique em Editar | Mostrar ajuste de exibição. Selecione o canal de dextrano e o tempo 1 (quadro 2 na parte inferior da tela).
- Crie uma nova superfície definindo o lúmen vascular no ponto de tempo 1 marcando a caixa de seleção adicionar nova superfície (objeto azul)
- No Surface 1, aceda a Propriedades e desmarque todos os parâmetros em Definições de Algoritmo. Clique na próxima etapa (seta azul)
- Selecione o canal vascular (dextrano, neste caso) em canal de origem e marque Subtração suave e de fundo. Deixe todos os outros parâmetros com valores padrão. Clique na próxima etapa (seta azul).
- Ajuste o valor do limite de acordo com a aparência da superfície em termos de espessura e cobertura de todas as áreas positivas. Clique na próxima etapa (seta azul).
- Exclua superfícies não específicas (principalmente pontos pequenos) usando o filtro por padrão Número de Voxels. Clique na etapa final (seta verde).
- Se necessário, continue ajustando a superfície manualmente clicando em editar (ícone de lápis) em Parâmetros de configuração. Para selecionar um objeto a ser removido, clique com o botão esquerdo nele e exclua selecionando excluir. Se o objeto a ser excluído for agrupado em outro, desconecte as duas partes separadas da superfície clicando em shift + clique à esquerda entre as duas e selecionando Superfície de corte. Repita esta etapa quantas vezes forem necessárias.
- Por fim, combine todos os segmentos usando o modo de seleção (ícone de destino à direita) e pressione control + scroll para recuperar todos os objetos. Quando todos estiverem selecionados, clique com o botão esquerdo do mouse e clique em unificar.
- Para gerar a superfície de referência, copie essa superfície para os outros pontos de tempo. Selecione a superfície global e duplique-a clicando em Duplicar e renomeie-a como Intravascular.
- Selecione a superfície Intravascular e clique em Duplicar para todos os pontos de tempo.
- Divida o sinal de dextrana para distinguir as áreas intra (IN) e extravascular (OUT). Para criar um canal IN para todos os pontos de tempo, clique na superfície Intravascular | Editar (botão de lápis) | Mascarar tudo.
- Em Seleção de canal, selecione o canal apropriado e marque o canal duplicado antes de aplicar a máscara. Em Configurações de máscara, marque apenas Constante dentro/fora com voxels definidos fora da superfície como 0. Por fim, marque também Aplicar a todos os pontos de tempo.
- Em Ajuste de exibição, um novo canal é criado por padrão com o nome mascarado. Renomeie-o como IN.
- Para criar o canal OUT para todos os pontos de tempo, repita as etapas 6.2.18-6.2.20, mas selecione Definir voxels dentro da superfície como 0. Renomeie este novo canal como OUT.
- Para extrair dados estatísticos, clique no ícone Estatística | Exportar todas as estatísticas para o arquivo (ícone de vários disquetes na parte inferior da tela)
- Para analisar o vazamento, divida o canal de soma de intensidade externo pelo canal de soma de intensidade interno para cada ponto de tempo.
- Para mesclar imagens tiradas antes e depois da injeção de dextrano na mesma série temporal, vá para o modo Superar, clique na primeira imagem (Tctrl , antes da injeção de dextrana) e na imagem de lapso de tempo (tempo 1, 60 min) para abri-las e combine-as clicando em Editar e adicione Pontos de Tempo.
Resultados
Na Figura 1 e na Figura 2, é mostrado o modelo CAD de um implante de fixação de cabeça de titânio posicionado em um crânio de camundongo digitalizado, projetado para seguir a estrutura anatômica do crânio e fornecer um dispositivo leve e biocompatível capaz de segurar firmemente o estágio do microscópio, garantindo a estabilidade do nível celular. Seguindo este protocolo passo a passo, o implante é fixado de forma estável ao crânio do camundongo e pode ser firmemente preso ao suporte do microscópio por sua cauda de andorinha, permitindo uma área de imagem plana para retenção de líquidos e observação intravital ao longo do tempo. Pode ser fechado com uma tampa para minimizar qualquer dano ou infecção da ferida, permitindo imagens repetidas da mesma área de tecido ao longo de semanas. Uma vez acordado, o camundongo que usa um implante de cabeça pode andar livremente, se alimentar e ter uma rotina regular.
A Figura 3 mostra uma visão de varredura em bloco da vasculatura da MO da calvária feita de capilares heterogêneos, incluindo arteríolas, capilares de transição e sinusóides. Os vasos são incorporados em um microambiente de tecido complexo em contato próximo com a superfície óssea e as células mesenquimais perivasculares. Durante o desenvolvimento da leucemia, células leucêmicas isoladas podem ser detectadas no microambiente da MO nas proximidades dos vasos, e seu enxerto aumenta com o tempo, preenchendo a calvária nos estágios finais da doença.
A Figura 4 mostra como as imagens obtidas com esse protocolo podem fornecer dados quantitativos, que podem ser analisados com métodos estatísticos. Mostramos como segmentar vasos com a ferramenta de filamento IMARIS e medir o comprimento e o diâmetro dos fragmentos vasculares, bem como sua retidão. A correlação desses parâmetros também pode ser avaliada.
A Figura 5 mostra a aquisição de imagens longitudinais de duas posições diferentes da MO da calvária durante a progressão da LMA nos dias 4, 7 e 10, com o dia 10 sendo associado a um enxerto de ~ 50% da MO com células leucêmicas, conforme medido por citometria de fluxo (não mostrado). Podemos observar um importante remodelamento do tamanho dos vasos preexistentes, bem como a formação de novos vasos em áreas específicas associadas à perda óssea local.
Finalmente, na Figura 6, mostramos como a permeabilidade vascular pode ser medida como um parâmetro dinâmico com imagens de lapso de tempo mostrando a capacidade de diferentes barreiras vasculares de reter um corante fluorescente ao longo do tempo.
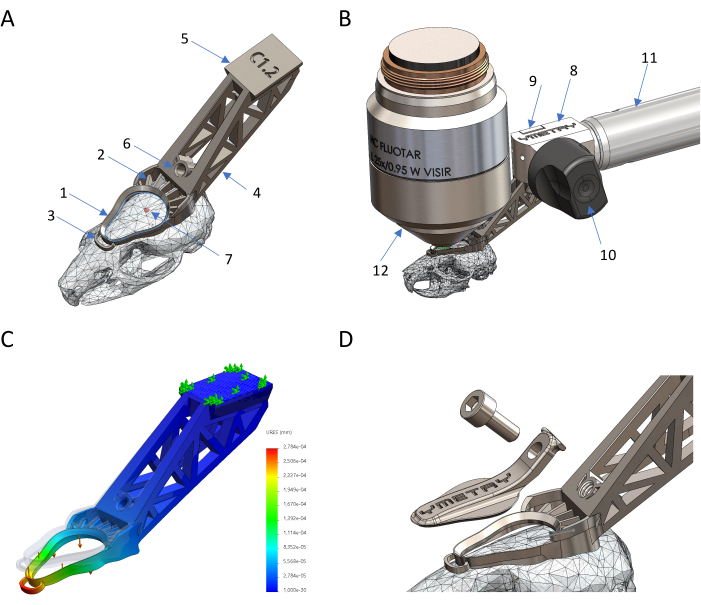
Figura 1: Projeto e produção de um suporte de cabeça biocompatível à base de titânio. (A) Partes do implante in situ: 1 anel de observação, 2 características de cimentação, 3 âncoras estabilizadoras, 4 caudas, 5 caudas de andorinha, 6 furos roscados, 7 Bregma. (B) Conexão do implante de cabeça ao suporte: 8 corpo de fixação, 9 braçadeira, 10 alavanca excêntrica, 11 estrutura, 12 objetiva de microscópio. (C) Deformação do implante contra carga por simulação FEM, onde o deslocamento máximo é de 0,23 μm contra 0,04 N de força. (D) Tampa de proteção e seu parafuso. Clique aqui para ver uma versão maior desta figura.

Figura 2: Preparação do camundongo para imagens intravitais. (A) Vista do implante de cabeça e da área de imagem exposta cirurgicamente antes da imagem. (B) Implante de cabeça firmemente preso ao crânio do camundongo. (C) Rato acordado na gaiola de recuperação com a tampa fechada no implante da cabeça. Clique aqui para ver uma versão maior desta figura.
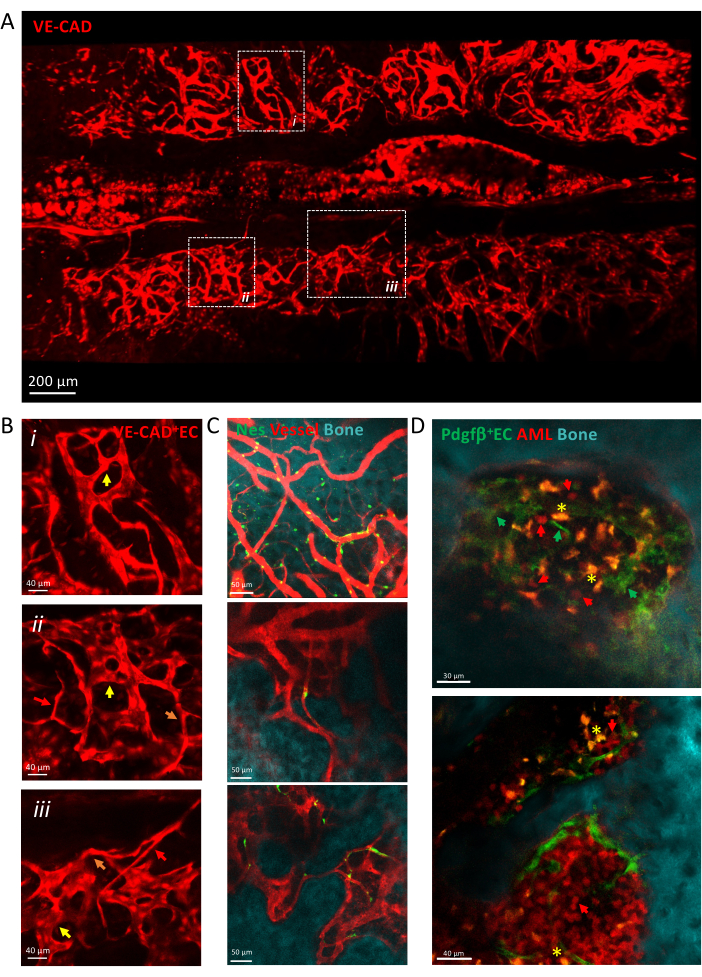
Figura 3: Imagem intravital da vasculatura da calvária. (A) projeção z da visão de varredura em bloco da vasculatura da calvária marcada por cdh5-DSRED. (B) Amplie as áreas representadas para descrever diferentes tipos de vasos representados por setas, arteríolas por setas vermelhas, capilares de transição por setas laranja, sinusóides por setas amarelas. (i) e (ii) projeções z de tecido X μm; (iii) fatia única. (C) Cortes únicos de vários campos de visão dos vasos da MO, mostrando a superfície óssea (SHG), as células perivasculares (nes-GFP+) e o lúmen vascular (dextrana-TRITC). (D) Nicho vascular associado à progressão da LMA. Fatias representativas dos pontos de tempo iniciais (superiores) e tardios (inferiores) do desenvolvimento da LMA. A leucemia MLL-AF9 é marcada com tdTOMATO (setas vermelhas), enquanto os vasos são marcados com pdgfb-GFP (setas verdes), a superfície óssea com SHG e os macrófagos em amarelo (autofluorescência, asterisco amarelo). Barras de escala = 200 μm (A), 40 μm (B,D-painel inferior), 50 μm (C). Abreviaturas: MO = medula óssea; GFP = proteína fluorescente verde; LMA = leucemia mieloide aguda; CE = Células endoteliais. Clique aqui para ver uma versão maior desta figura.
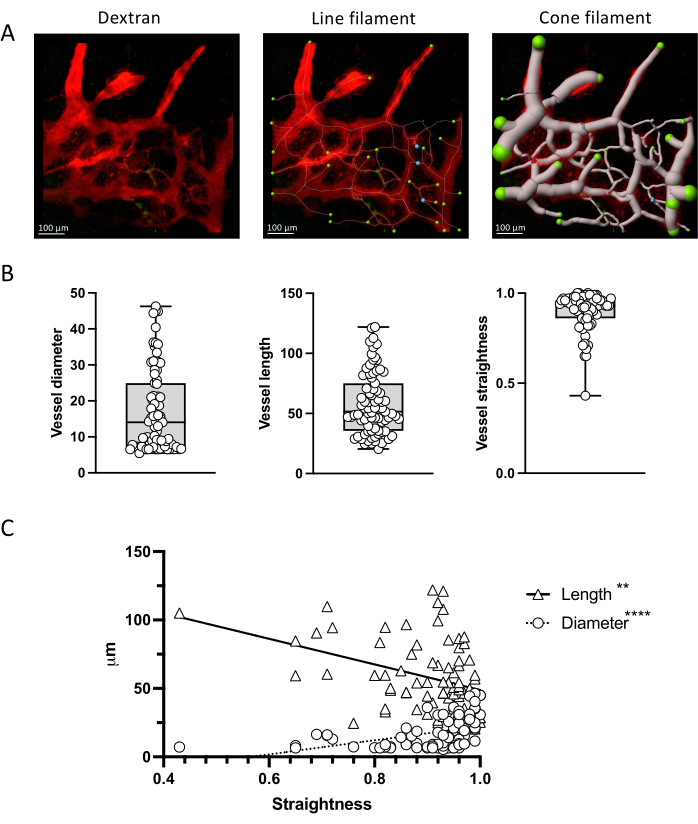
Figura 4: Quantificação de parâmetros vasculares. (A) Medição de parâmetros vasculares via ferramenta de filamento IMARIS em uma projeção z representativa de vasos da medula óssea marcados com TRITC-dextrano. As representações de linha e cone são mostradas. (B) Quantificação dos parâmetros do vaso na imagem mostrada em A. (C) Correlação entre os parâmetros vasculares mostrando uma correlação oposta entre a retidão e o comprimento do vaso (negativo, Spearman r = -3523; p < 0,0001; R2 = 0,2102) vs diâmetro (positivo; Spearman r = 0,4110; p < 0,0001; R2 = 0,1299). Barras de escala = 100 μm. Clique aqui para ver uma versão maior desta figura.
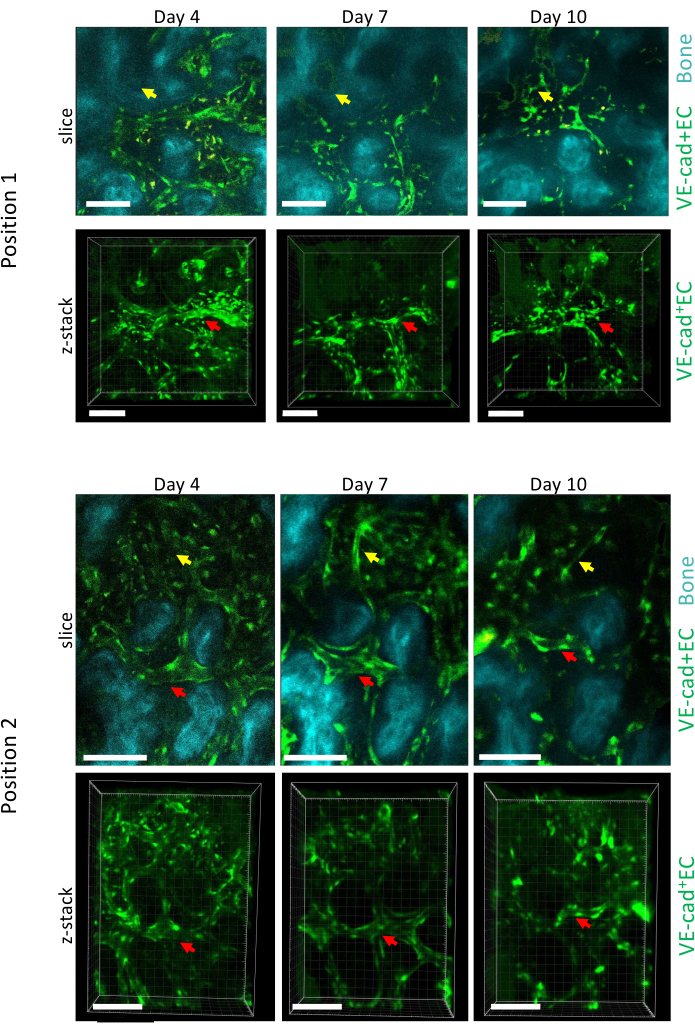
Figura 5: Imagem longitudinal de duas posições diferentes da calvária MO sobre o desenvolvimento da LMA. As células endoteliais que revestem os vasos são marcadas com cdh5-GFP, a superfície óssea com SHG e os macrófagos em amarelo (autofluorescência). Remodelação de vasos preexistentes (setas vermelhas) e formação de novos vasos (setas amarelas) são mostrados. Barras de escala = 100 μm. Clique aqui para ver uma versão maior desta figura.
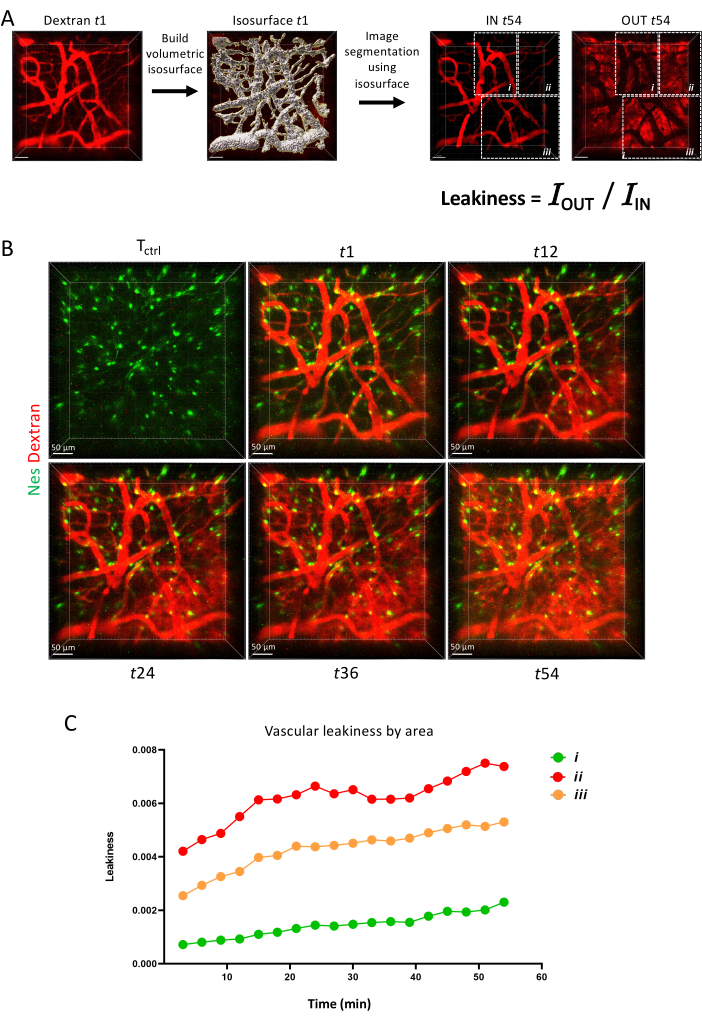
Figura 6: Permeabilidade vascular. (A) Esquemas de medição da permeabilidade vascular via ferramenta de superfície IMARIS. (B) Projeção Z da mesma área fotografada longitudinalmente ao longo de 1 h. (C) Quantificação da permeabilidade vascular dentro das áreas conforme descrito em A. Clique aqui para ver uma versão maior desta figura.
Figura Suplementar S1: Crânio preparado. No plano mediano, crie um eixo tangencial à calvária e, em seguida, salve o crânio preparado. Clique aqui para baixar este arquivo.
Figura suplementar S2: Planos espaçados de 2 mm. Como criar um conjunto de planos igualmente espaçados (espaçamento de 2 mm) ao longo do crânio. Clique aqui para baixar este arquivo.
Figura Suplementar S3: Contorno de superfície observável. Crie um esboço no plano da calvária e crie uma spline em forma de pêra de AP +6,5 a -2, 6 mm de largura em AP 0,0. Clique aqui para baixar este arquivo.
Figura Suplementar S4: Observação vindow. Crie um esboço no plano da calvária e desenhe uma forma C de 0,5 mm de espessura conectando-se à janela de observação . Clique aqui para baixar este arquivo.
Figura Suplementar S5: Clique em novo estudo. Navegue até a guia Simulação (se visível) ou vá para Simulação | Estude. Na caixa de diálogo Estudo , escolha Estático como o tipo de estudo. Clique em OK para criar o novo estudo. Clique aqui para baixar este arquivo.
Figura Suplementar S6: Clique com o botão direito do mouse para criar a malha. Refine a malha em áreas onde é esperada alta tensão ou deformação. Clique aqui para baixar este arquivo.
Discussão
A principal força da microscopia intravital é sua capacidade de capturar processos celulares dinâmicos em tempo real em seu ambiente nativo, superando as limitações dos métodos tradicionais de imagem histológica e ex vivo. Ao observar diretamente a MO da calvária usando as técnicas avançadas de imagem juntamente com a marcação fluorescente, conforme descrito neste protocolo, os pesquisadores podem estudar não apenas os parâmetros funcionais vasculares 8,14, mas também os processos longitudinais, como enxerto e migração de células leucêmicas 6,15, proliferação e atividade metabólica16,17 preservando a relevância fisiológica e evitando artefatos associados à fixação e processamento de tecidos.
As vantagens da imagem intravital da calvária BM sobre locais ósseos alternativos incluem fácil acessibilidade, transparência do osso do crânio permitindo melhor visualização sem danos ósseos, estabilidade do osso da calvária minimizando artefatos de movimento do tecido e reprodutibilidade entre animais experimentais facilitando a comparação entre diferentes grupos experimentais. A possibilidade de recuperar os animais e realizar imagens longitudinais permite também reduzir o número de animais utilizados em um estudo. Vale ressaltar que, como foram observadas diferenças específicas da cepa nos padrões de crescimento dos vasos cranianos e consequente osteogênese18, é importante levar isso em consideração ao projetar o implante de imagem específico para a cepa de camundongo desejada para permitir perfeita compatibilidade anatômica e acesso ao local de imagem desejado.
Outro ponto importante a ser mencionado são as possíveis diferenças nas propriedades vasculares e hematopoiéticas dentro da MO da calvária em comparação com outros ossos, uma questão pouco explorada até os últimos anos. Novos estudos sugerem funções localizadas para diferentes ossos, com a MO da calvária diferindo de outros ossos em termos de hematopoiese, estrutura e função óssea e vascular 19,20,21,22,23, bem como resposta a patologias neurológicas 24. Essas diferenças precisam ser mais exploradas e levadas em consideração antes de generalizar os achados específicos da calvária.
O projeto e a construção de um implante de imagem é uma etapa fundamental para essa configuração experimental, a partir da escolha do material. Os implantes biocompatíveis desempenham um papel fundamental na pesquisa biomédica, facilitando uma ampla gama de aplicações, desde engenharia de tecidos e medicina regenerativa até sistemas de administração de medicamentos e dispositivos de monitoramento in vivo . A escolha do material para um implante de cabeça adaptado para imagens intravitais é crítica. Idealmente, o material deve apresentar excelente biocompatibilidade, propriedades mecânicas adequadas para garantir flexibilidade mínima e estabilidade geral e, finalmente, a capacidade de se integrar ao crânio sem induzir inflamação. O titânio é ideal por sua boa tolerabilidade dentro do corpo do animal, sem indução de reações adversas em contato com tecidos biológicos, bem como por sua resistência à corrosão, garantindo estabilidade a longo prazo mesmo se exposto a fluidos corporais ou soluções experimentais. Além disso, sua resistência mecânica o torna resistente à deformação e fratura. Apesar de sua resistência, o titânio tem uma densidade relativamente baixa, resultando em dispositivos de imagem intravitais leves que minimizam a carga sobre animais experimentais e pesquisadores. Finalmente, sua versatilidade na fabricação permite a personalização de dispositivos de imagem intravitais para atender a requisitos experimentais específicos, como tamanho, forma e funcionalidade.
Aqui, projetamos um implante de fixação de cabeça biocompatível usando software CAD de modelagem paramétrica com recursos integrados de Análise de Elementos Finitos (FEA), especificamente o SolidWorks. Essa abordagem permite ajustes precisos e iterativos nos requisitos estruturais e espaciais do implante, garantindo compatibilidade anatômica e resiliência mecânica. Alternativas gratuitas, como o FreeCAD, oferecem funcionalidades semelhantes de modelagem e simulação. As principais entradas para o processo de design incluem um modelo anatômico de alta resolução do crânio do camundongo, um modelo da objetiva do microscópio e um modelo do sistema de fixação, que juntos informam as dimensões e a configuração do implante. O resultado final é um arquivo de malha independente de plataforma, normalmente no formato STL ou STEP, compatível com a maioria dos softwares de impressão 3D.
A etapa inicial envolve a captura de características anatômicas detalhadas da cabeça do camundongo usando técnicas de imagem de alta resolução, como microtomografia computadorizada in vivo . Essa abordagem fornece os mais altos detalhes anatômicos e precisão, capturando as microestruturas do crânio. A digitalização 3D de um crânio preparado e nu também pode ser uma opção. Este método, usando scanners a laser ou de luz estruturada, é comumente empregado em um crânio preparado e fornece contornos de superfície precisos, embora com menos detalhes internos em comparação com a micro-CT. Caso contrário, os modelos de anatomia podem ser baixados de publicações e bancos de dados de código aberto25 ou DigiMorph {https://www.digimorph.org/specimens/Mus_musculus/}. Embora convenientes, esses modelos podem não ter detalhes específicos da amostra, portanto, ajustes são frequentemente necessários para o animal específico no estudo, como escalar para a distância real de Bregma-Lambda. Os dados adquiridos são então usados para criar um modelo 3D preciso do crânio do camundongo, servindo como modelo fundamental para o projeto do implante.
Para fixar o implante no crânio, uma estrutura de cimentação é projetada para cobrir a superfície restante do crânio não ocupada pela janela de observação. Essa estrutura deve fornecer pontos de fixação robustos, evitando características anatômicas críticas. A estrutura de cimentação tem várias aberturas para garantir a polimerização ideal do cimento sob o implante por meio de difusão. Além disso, as paredes da estrutura de cimentação têm um pequeno ângulo de inclinação, o que permite que o cimento se ancore com segurança contra essas paredes angulares. A cauda do implante, que se estende do corpo principal ao sistema de fixação, é projetada. Este componente é crucial para alinhar e estabilizar o implante durante a observação, e seu design deve considerar o espaço disponível e as restrições anatômicas da cabeça do camundongo. Finalmente, um mecanismo de cauda de andorinha é integrado ao design do implante para facilitar a fixação e descolamento do implante do sistema de fixação. Esse recurso aumenta a praticidade e a usabilidade do implante durante observações repetidas. O mecanismo de cauda de andorinha fornece fixação repetível para fácil recuperação de tecidos observados para sessões de imagem repetidas.
Os leitores que consideram a adoção de imagens de microscopia intravital da MO da calvária devem avaliar cuidadosamente seus objetivos de pesquisa e requisitos experimentais para determinar se esse método é apropriado para seus estudos. Embora a microscopia intravital ofereça insights incomparáveis sobre hematopoiese, regulação do microambiente tecidual e dinâmica vascular in vivo, ela também apresenta certos desafios e limitações técnicas. Os pesquisadores devem estar preparados para enfrentar esses desafios por meio de um projeto experimental cuidadoso, otimização dos parâmetros de imagem e utilização de controles apropriados. Além disso, os pesquisadores devem considerar a disponibilidade de equipamentos de imagem especializados, experiência em técnicas de rotulagem fluorescente e recursos computacionais para análise de imagens. No geral, a imagem de microscopia intravital da MO da calvária tem um tremendo potencial para avançar nossa compreensão da hematopoiese e da biologia vascular, oferecendo uma janela única para os processos dinâmicos que ocorrem no microambiente da MO.
Divulgações
Jozsua Fodor é o fundador da empresa YMETRY (registro nº 888312352). Os demais autores não têm conflitos de interesse.
Agradecimentos
Os autores gostariam de agradecer a todos os funcionários do IMAG'IC e das instalações de animais do Institut Cochin por seu apoio com experimentos de microscopia e alojamento de camundongos. Os camundongos Tg(Nes-EGFP)33Enik e Tg(Pdgfb-icre/ERT2)1Frut foram um presente gentil do Dr. Bonnet (The Francis Crick Institute, Londres). Tg(Cdh5-cre/ERT2)1Rha e B6. Os camundongos Cg-Gt (ROSA) 26Sortm9 (CAG-tdTomato) Hze / J foram um presente gentil do Dr. Rafii (Weill Cornell Medicine, Nova York). O trabalho descrito foi apoiado pelo CNRS, INSERM e Université de Paris Cite, e bolsas da ATIP-AVENIR, Fondation ARC pour la recherche sur le cancer (R19084KS - RSE20008KSA), Ville de Paris "Emergence" (R20192KK - RPH20192KKA), Laurette Fugain (R23197KK), Cancéropôle IDF (RPH23177KKA), INCA PLBIO (RPH21162KKA), Fondation de France (RAF23152KKA), Ligue contre le cancer (282273/807251), Institut du cancer Paris Carpem, Associação Europeia de Hematologia (RAK23130KKA) e Conselho Europeu de Pesquisa ERC-STG (EEA24092KKA). A instalação central do IMAG'IC é apoiada pela National Infrastructure France BioImaging (concessão ANR-10-INBS-04). O laboratório Passaro é afiliado ao "Institut Hors Murs des Sciences Cardiovasculaires" e ao "Leukemia Institute Paris Saint-Louis.
Materiais
| Name | Company | Catalog Number | Comments |
| Amira | Thermo ScientificTM | ||
| Anesthesia | Isoflurane 2% to 3% | ||
| Anesthesic mask (animal detection) | Minerve | ||
| Anesthesic unit | Minerve | ||
| B6.Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze | Jackson laboratories | MGI:3809523 | |
| CalvariaVue | Ymetry | https://ymetry.com/site/head-fixation-implants/44-mouse-head-fixation-implant-for-calvaria.html | |
| Dental cement | Kemdent | SUN527 | |
| Dextran 500 kDa-Cy5 | Tebu-bio | DX500-S5-1 | 3 mg/mouse |
| Dextran 70 kDa-TRITC | Sigma | T1162 | 3 mg/mouse |
| Disinfectant | MP-Labo | Dermidine-60ml | |
| Electric razor | Aescular | Isis | |
| Eye gel | Ocry-gel | 10g | |
| Fiji | https://imagej.net/software/fiji/downloads | v 17 May 30 | |
| Fiji plugings to bridge with Imaris | https://imagej.net/software/fiji/downloads | Imaris_Bridge96.jar ; ImarisBridgeUtils.jar | |
| Heating box | Datesand | Thermacage | |
| Heating pad for surgery | Minerve | ||
| Imaging heating pad & rectal probe | F. Haer | ||
| Imaris v9.6.0 | Oxford instruments | ||
| Intrasite gel | Chinoxia | 2390766 | |
| LAS AF Software | Leica | LAS X 3.5.7.23225 | |
| Medication | Buprecare, 0.01 mg/kg | ||
| Objective HCX IRAPO L 25x/0.95 WATER | Leica | 506374 | |
| Saline buffer (PBS 1x) | Sigma | P4417 | Sterilize by autoclave |
| SP8 DIVE FALCON Multiphoton Microscope | Leica | ||
| Stereotoxic mask | Minerve | 1201261 | |
| Sterilizator beads | Sigma | Z742555 | |
| Surgery tools | Moria | 4877A; 2183 | |
| Survival blanket | SECURIMED | 11006 | |
| Swabs / Tissues | Sterilize by autoclave | ||
| Syringe 1 mL 26 G | BD Plastipak | 305501 | |
| Temperature controller | F. Haer | 40-90-5D-02 | |
| Tg(Nes-EGFP)33Enik mice | Jackson laboratories | MGI:5523870 | |
| Tg(Pdgfb-icre/ERT2)1Frut mice | Jackson laboratories | MGI:3793852 | |
| Tg(Cdh5-cre/ERT2)1Rha mice | Jackson laboratories | MGI:3848982 | |
| Ultrasound gel | Parker laboratories | Aquasonic 100 |
Referências
- Lo Celso, C., et al. Live-animal tracking of individual haematopoietic stem/progenitor cells in their niche. Nature. 457 (7225), 92-96 (2009).
- Pittet, M. J., Weissleder, R. Intravital imaging. Cell. 147 (5), 983-991 (2011).
- Itkin, T., et al. Distinct bone marrow blood vessels differentially regulate haematopoiesis. Nature. 532 (7599), 323-328 (2016).
- Christodoulou, C., et al. Live-animal imaging of native haematopoietic stem and progenitor cells. Nature. 578 (7794), 278-283 (2020).
- Upadhaya, S., et al. Intravital imaging reveals motility of adult hematopoietic stem cells in the bone marrow niche. Cell Stem Cell. 27 (2), 336-345.e4 (2020).
- Hawkins, E. D., et al. T-cell acute leukaemia exhibits dynamic interactions with bone marrow microenvironments. Nature. 538 (7626), 518-522 (2016).
- Duarte, D., et al. Defining the in vivo characteristics of acute myeloid leukemia cells behavior by intravital imaging. Immunol Cell Biol. 97 (2), 229-235 (2019).
- Passaro, D., et al. Increased vascular permeability in the bone marrow microenvironment contributes to disease progression and drug response in acute myeloid leukemia. Cancer Cell. 32 (3), 324-341.e6 (2017).
- Jia, Y., et al. FLT3 inhibitors upregulate CXCR4 and E-selectin ligands via ERK suppression in AML cells and CXCR4/E-selectin inhibition enhances anti-leukemia efficacy of FLT3-targeted therapy in AML. Leukemia. 37 (6), 1379-1383 (2023).
- Pendleton, E. G., Tehrani, K. F., Barrow, R. P., Mortensen, L. J. Second harmonic generation characterization of collagen in whole bone. Biomed Opt Express. 11 (8), 4379-4396 (2020).
- Dispositif de liaison d'un animal de laboratoire à au moins un système d'expérimentation, et procédé de fixation d'un tel dispositif. France patent. , WO/2021/123449 (2021).
- Horton, S. J., et al. Acute myeloid leukemia induced by MLL-ENL is cured by oncogene ablation despite acquisition of complex genetic abnormalities. Blood. 113 (20), 4922-4929 (2009).
- Scott, M. K., Akinduro, O., Lo Celso, C. In vivo 4-dimensional tracking of hematopoietic stem and progenitor cells in adult mouse calvarial bone marrow. J Vis Exp. (91), e51683(2014).
- Jung, Y., et al. Intravital imaging of mouse bone marrow: Hemodynamics and vascular permeability. Methods Mol Biol. 1763, 11-22 (2018).
- Le, V. H., et al. In vivo longitudinal visualization of bone marrow engraftment process in mouse calvaria using two-photon microscopy. Sci Rep. 7, 44097(2017).
- Spencer, J. A., et al. Direct measurement of local oxygen concentration in the bone marrow of live animals. Nature. 508 (7495), 269-273 (2014).
- Yang, M., Mahanty, A., Jin, C., Wong, A. N. N., Yoo, J. S. Label-free metabolic imaging for sensitive and robust monitoring of anti-CD47 immunotherapy response in triple-negative breast cancer. J Immunother Cancer. 10 (9), e005199(2022).
- Li, W., et al. Tracking strain-specific morphogenesis and angiogenesis of murine calvaria with large-scale optoacoustic and Ultrasound Microscopy. J Bone Miner Res. 37 (5), 1032-1043 (2022).
- Lassailly, F., Foster, K., Lopez-Onieva, L., Currie, E., Bonnet, D. Multimodal imaging reveals structural and functional heterogeneity in different bone marrow compartments: functional implications on hematopoietic stem cells. Blood. 122 (10), 1730-1740 (2013).
- Rindone, A. N., et al. Quantitative 3D imaging of the cranial microvascular environment at single-cell resolution. Nat Commun. 12 (1), 6219(2021).
- Mills, W. A. 3rd, Coburn, M. A., Eyo, U. B. The emergence of the calvarial hematopoietic niche in health and disease. Immunol Rev. 311 (1), 26-38 (2022).
- Bixel, M. G., et al. Angiogenesis is uncoupled from osteogenesis during calvarial bone regeneration. Nat Commun. 15 (1), 4575(2024).
- Koh, B. I., et al. Adult skull bone marrow is an expanding and resilient haematopoietic reservoir. Nature. 636 (8041), 172-181 (2024).
- Kolabas, Z. I., et al. Distinct molecular profiles of skull bone marrow in health and neurological disorders. Cell. 186 (17), 3706-3725.e9 (2023).
- Rosenhain, S., et al. A preclinical micro-computed tomography database including 3D whole body organ segmentations. Sci Data. 5, 180294(2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados