Method Article
Обнаружение пищевых бактериальных патогенов из отдельных Filth мух
В этой статье
Резюме
A PCR-based protocol was adapted to detect Cronobacter spp., Salmonella enterica, and Listeria monocytogenes from body surfaces and alimentary canals of individual wild-caught flies. The goal of this protocol is to detect and isolate bacterial pathogens from individual insects collected as part of an environmental sampling program during foodborne outbreak investigations.
Аннотация
There is unanimous consensus that insects are important vectors of foodborne pathogens. However, linking insects as vectors of the pathogen causing a particular foodborne illness outbreak has been challenging. This is because insects are not being aseptically collected as part of an environmental sampling program during foodborne outbreak investigations and because there is not a standardized method to detect foodborne bacteria from individual insects. To take a step towards solving this problem, we adapted a protocol from a commercially available PCR-based system that detects foodborne pathogens from food and environmental samples, to detect foodborne pathogens from individual flies.Using this standardized protocol, we surveyed 100 wild-caught flies for the presence of Cronobacter spp., Salmonella enterica, and Listeria monocytogenes and demonstrated that it was possible to detect and further isolate these pathogens from the body surface and the alimentary canal of a single fly. Twenty-two percent of the alimentary canals and 8% of the body surfaces from collected wild flies were positive for at least one of the three foodborne pathogens. The prevalence of Cronobacter spp. on either body part of the flies was statistically higher (19%) than the prevalence of S. enterica (7%) and L.monocytogenes (4%). No false positives were observed when detecting S. enterica and L. monocytogenes using this PCR-based system because pure bacterial cultures were obtained from all PCR-positive results. However, pure Cronobacter colonies were not obtained from about 50% of PCR-positive samples, suggesting that the PCR-based detection system for this pathogen cross-reacts with other Enterobacteriaceae present among the highly complex microbiota carried by wild flies. The standardized protocol presented here will allow laboratories to detect bacterial foodborne pathogens from aseptically collected insects, thereby giving public health officials another line of evidence to find out how the food was contaminated when performing foodborne outbreak investigations.
Введение
Насекомые играют важную роль в передаче болезней, связанных с питанием, потому что они могут распространяться патогены пищевых продуктов или контактных поверхностей и посуды 1. Среди насекомых, мух, тараканов, и муравьи проявляют поведение, которые способствуют распространению возбудителей пищевого происхождения. Такое поведение включает ассоциацию с распадающейся материи, отказаться и фекалии, endophily (ввод здания), и synanthropy (сожительство с человека) 2. .. Пищевых патогенов, таких как сальмонелла, листерий, Campylobacter SPP, кишечная палочка O157: H7, и члены рода Cronobacter (ранее Enterobacter sakazakii), как сообщается, передаваемых насекомыми 3-5. Синантропных грязь летит механически распространять пищевых бактерий путем передачи патогенов из своих загрязненных поверхностей тела. Тем не менее, наличие пищевых патогенов в желудочно-кишечном тракте мух может быть до трех разбольше, чем это наблюдалось на их поверхности тела (тело, голова, ноги и крылья) 5. Пищевые патогены могут также оставаться в желудочно-кишечном тракте мухи для большей продолжительности, чем на поверхности тела 6,7, а в некоторых случаях они способны размножаться, колонизации желудочно-кишечного 4,8,9 мухи путей. Это увеличивает векторный потенциал мух так как они могут дальнейшего распространения пищевых патогенов через дефекации и регургитации 10,11.
В настоящее время существует улучшать системы эпиднадзора, которые способны выявлять вспышки болезней пищевого происхождения более быстрыми темпами. При выполнении пищевого происхождения при расследовании вспышек эпидемий, чиновники системы здравоохранения искать пищу, которая может быть источником (ы) или носитель (ы) инфекции. Следователи также может выполнять экологическую оценку объекта (или объектов), участвующих чтобы узнать, как еда была загрязнена и может собирать образцы в рамках расследования 12. DespITE огромное количество научной литературы, касающейся насекомых в качестве носителей возбудителей пищевого происхождения, соединяющей насекомых в качестве переносчиков возбудителя конкретная вспышка болезни пищевого происхождения был сложным. Это главным образом потому насекомые не стерильно, собранные в рамках программ отбора проб окружающей среды во время пищевого происхождения расследования вспышек. Чтобы включить насекомых, особенно тех, которые обладают поведения, которые способствуют распространению возбудителей пищевого происхождения, как часть процедуры отбора проб окружающей среды, стандартизации, быстрых, чувствительных и надежных протокола обнаружения пищевых патогенов из одного насекомого должен быть на месте.
Традиционные методы металлизации для обнаружения пищевых патогенов от насекомых являются трудоемкими и зависят от конкурентной роста бактерий-мишеней в различных питательных сред для преодоления быстрого роста врожденной комменсальной микрофлоры насекомого. Большинство исследований, которые, связанных с насекомыми с баcterial патогены увеличили чувствительность метода путем объединения вместе несколько насекомых, а не установления присутствия патогенов на на индивидуальной основе. Таким образом, эти исследования не отличить части тела насекомого где патогены были найдены 13-18. Способность определить, является ли пищевых патогенов расположены на поверхности тела или в желудочно-кишечном тракте индивидуального насекомого очень важно, поскольку это может иметь эпидемиологические последствия и может привести к различным стратегиям по смягчению последствий. Как механическими переносчиками, летит эту землю на продукты питания в течение короткого времени может передавать только низкие уровни бактерий с их поверхности тела, в то время как те мухи, что пересказывают и дефекации на еду увеличить вероятность передачи патогенных микроорганизмов на потенциально более высокие уровни инфекции. Следовательно, важно оценить распространённость пищевого возбудителя по индивидуальному насекомых и дифференцировать часть тела этого насекомого, где бактериальная р athogen находится.
Даже если использование культуральных-независимых методов для обнаружения пищевых патогенов все чаще реализуется, они не были коммерчески используется для обнаружения пищевых патогенов из одного насекомого. В настоящее время проверяются молекулярные протоколов, которые имеются в продаже для быстрого обнаружения пищевых патогенов из продуктов, которые в настоящее время используется в промышленности и регулирующих органов. Эти методы включают в себя системы на основе ДНК для обнаружения возбудителей в различных образцах пищевых продуктов. Хотя молекулярные протоколы быстрее, чем традиционные методы металлизации, обогащение образца по-прежнему требуется, чтобы получить уровень чувствительности 10 2 колониеобразующих единиц (КОЕ) в бактериальным патогеном, необходимого в полимеразной цепной реакции (ПЦР) основе методов 19. Кроме того, выделение чистых бактериальных колоний от ПЦР-положительных образцов, чтобы подтвердить возбудителя с помощью соответствующих методов.
Содержание "> Целью этого протокола является стандартизация имеющихся в продаже, основанные на ПЦР систему, используемую для определения возбудителей от пищевых продуктов и проб окружающей среды для выявления пищевых бактерий с поверхности тела и желудочно-кишечного тракта одной лету и еще больше изолировать тех, патогены из чувствительности samples.The протокола, описанного здесь впервые откалиброван с лабораторными выращенных взрослых домашние мухи (Муха Доместикой), которые были экспериментально кормили с серийными разведениями каждого бактериального патогена. стандартизированный протокол был впоследствии использован для обследования 100 пойманных летит на наличие пищевых патогенов с их поверхностей тела и / или пищевых каналов. Это стандартизированный протокол позволит лаборатории общественного здравоохранения для выявления угроз для здоровья, связанных с насекомыми, допуская возможность сбора их как часть программы отбора проб окружающей среды при выполнении пищевого происхождения Исследования вспышек.протокол
1. Сбор мух
- Сбор отдельных мух с использованием стерильных энтомологические сети развертки. Положите сети в холодильнике и передавать их в лабораторию.
2. Препарирование мух
- Зафиксировать собрана с соблюдением асептики мух, помещая их при температуре -20 ° С в течение 5 - 7 мин.
- Использование стерильного пинцета поместите одну муху в стерильную пробирку 2 мл, содержащую 1 мл подогретого (37 ° C) в буфер пептонной воды (BPW). Смешайте трубку осторожно инверсии в течение 2 мин. Очень важно, чтобы все тело лету быть в контакте со средствами массовой информации, так что микрофлора присутствует на поверхности тела (S) мухи будут переданы на BPW (BPW-S). Этикетка трубки с номером и части тела мухи (то есть, 1С).
ПРИМЕЧАНИЕ: Пожалуйста, смотрите таблицу специфических реагентов / Оборудование для подробного описания материалов и реагентов, указанных в настоящем Протоколе. - Использование стерильного пинцета удалить муху из средств массовой информации и передачи BPW-Sее в пустую и чистую пробирку 2 мл на поверхность дезинфицируют муху. Выдержите трубки, содержащей СМИ BPW-х годов в 37 ° C во время выполнения протокола дезинфекции и рассечение.
- Поверхностно-дезинфекции муху при погружении его в 1 мл 70% этанола в течение 1 мин, после чего промыть стерильной дистиллированной воды перед погружением его в 1 мл свежеприготовленного 0,05% (объем / объем) раствор отбеливателя. Промыть 3 раза стерильной дистиллированной воды. Переброска воды из последнего полоскания к обработке в автоклаве 2 мл пробирку.
ПРИМЕЧАНИЕ: жидкость удалите каждый раз при использовании 1000 мкл микропипетки или путем обращения трубку, убедившись, что муха остается внутри трубки. Аккуратно перемешайте, переворачивая на каждом этапе процесса поверхность дезинфекции. - Чтобы оценить эффективность процесса дезинфекции, передавать 100 мкл воды от последнего полоскания в Триптиказно соевого агара (TSA) пластины и распространение с использованием стерильной L-образный распределитель одноразовый. Инкубируйте планшет при 37 ° С в течение 24 ч. После того как вcubation, зарегистрируйтесь присутствие каких-либо бактериальных колоний.
ПРИМЕЧАНИЕ: Наличие бактериальных колоний на пластинах АСП указывает на неэффективный процесс поверхностного обеззараживания. Если это происходит, наличие пищевых патогенов должны быть представлены только на поверхности тела мухи, потому что перекрестное загрязнение между поверхностью тела и желудочно-кишечного тракта не может быть исключена.
- Поверхностно-дезинфекции муху при погружении его в 1 мл 70% этанола в течение 1 мин, после чего промыть стерильной дистиллированной воды перед погружением его в 1 мл свежеприготовленного 0,05% (объем / объем) раствор отбеливателя. Промыть 3 раза стерильной дистиллированной воды. Переброска воды из последнего полоскания к обработке в автоклаве 2 мл пробирку.
- После поверхностного обеззараживания муху, перенести его на части автоклавного бумажным полотенцем, чтобы удалить лишнюю воду, а затем в стерильную 60 мм одноразовой чашке Петри.
- Поместите чашку Петри под рассекает рамки и определить летать на видовом уровне, используя дихотомических ключей для двукрылых семейств 20,21.
- Использование автоклавного тонкий наконечник щипцов осторожно потяните анус и весь пищеварительный тракт (A) из мухи и асептически перенести его в другую стерильную пробирку 2 мл, содержащую 1 мл подогретого (37 ° C) BPW с 0,5 мм циркониевый / кремнезема бусы (BPW-). Этикетка-йэлектронной трубки с таким же числом, выбранным для индивидуального лету и части тела мухи (т.е., 1A).
- Смешайте трубки, содержащей BPW-A тщательно для 5 - 10 мин с помощью мобильного дизраптор. Инкубировать при 36 ± 1 ° С во время выполнения остальной части протокола.
- Для ваучер и / или хранения образцов для долгосрочной перспективе, поместите остаток лету в чистую пробирку 2 мл и добавить 1 - 2 мл 95% этанола.
3. Первичный и вторичный Обогащение
- Этикетка все трубы первичного и вторичного обогащения, содержащие носитель в соответствии с количеством образца и части тела мухи.
- Под стерильных капот, трансфер 300 мкл BPW-S (поверхность) в стерильные 2 мл пробирки, содержащие следующие носители:
- Для Salmonella, используйте 1 мл подогретого (42 ° C) BPW. Инкубируют на водяной бане при рециркуляции 42,5 ° С в течение 22 - 24 ч. Для вторичного обогащения, трансфер 100 мкл обогащенного BPW до 400 & #181; л предварительно нагретом (37 ° С) сердечно-мозговым экстрактом (BHI) бульоне, ранее помещенных в стерильные пробирки кластера. Инкубируют при 37 ° С в течение 3 ч.
- Для Cronobacter, используйте 1 мл подогретого (37 ° C) BPW с новобиоцина (10 мг / л; Wallace, М., личное сообщение). Кроме того, использование 1 мл R & F Enterobacter sakazakii обогащения бульоне с дополнением (ванкомицин и цефсулодин) в качестве первичного обогащения. Выдержите при 37 ° С в течение 22 - 26 часов. Для вторичного обогащения, трансфер 100 мкл обогащенного BPW с новобиоцина 400 мкл подогретого (37 ° C) BHI бульона ранее размещенных в стерильных кластера труб. Инкубируют при 37 ° С в течение 3 ч.
- Для L. моноцитогенес, используйте 1 мл свежеприготовленного RT 24 Listeria бульона обогащения (24 LEB) с селективной добавки. Инкубируют при 37 ° С в течение 44 ± 5 ч. Нет вторичного обогащения не требуется для обнаружения L. моноцитогенес.
- Повторите шаги 3.2.1 - 3.2.3 выше, с использованием трубки помечены как BPW-A.
4. Подготовка ПЦР-системы на базе для усиления и обнаружения цели пищевого происхождения возбудителя
Шаги 4-8 использование коммерческого ПЦР велосипедист / детектор системы, компьютерной рабочей станции, и готовы к использованию наборы для выявления сальмонеллы (Salmonella 2 Стандартный набор для анализа), виды Cronobacter (Е. sakazakii стандарт набора для анализа), и листерий (L. Monocytogenes 24E набора для анализа). Стандартные анализы использовать ПЦР определения конечной точки. Каждый комплект содержит ПЦР-готовые таблетки с интеркалирующего красителя, который испускает флуоресцентный сигнал при связывании с двухцепочечной ДНК. Сигнал улавливается на этапе определения программы системы ПЦР, создавая кривую плавления, которая интерпретируется программным обеспечением как положительным, так и отрицательным.
- Подготовка реагентов и оборудования, как указано в производства фирмыПротокол RER на душу каждого целевого пищевого возбудителя.
ПРИМЕЧАНИЕ: Протоколы для обнаружения сальмонеллы и Cronobacter требуют процедуры лизиса один шаг в то время как протокол для обнаружения L. моноцитогенес требует процедуры лизиса двухступенчатый (см разделов 5 и 6 соответственно). - Включите автоматизированной нагревательного блока выбора конкретной программе целевого патогенного микроорганизма. Кроме того, если нагревательные блоки руководство, установите температуру до 37 ° C (для Salmonella, Cronobacter SPP, Л. Monocytogenes.) Или 55 ± 2 ° С (для Л. Monocytogenes часть 2 лизиса - см шаг 6,2) и 95 ± 3 ° С.
- Убедитесь в том, что охлаждающие блоки были в холодильнике O / N, в противном случае охладить их в 2 - 8 ° С в течение по крайней мере 2 часа.
- Использование программного обеспечения в ПЦР-системы обнаружения, создать стойку файл в соответствии с инструкциями изготовителя.
- Этикетка и организовать кластерПробирки, содержащие лизирующий реагент в стойке, в соответствии с файлом стойки.
- Инициализация ПЦР-систему на основе обнаружения инструмент.
5. Выполните лизиса для обнаружения сальмонеллы и Cronobacter
- Подготовка лизирующий реагент добавлением 150 мкл протеиназы, чтобы один 12 мл флакон буфера для лизиса.
- Передача 200 мкл лизирующего реагента на каждый из ранее обозначенных кластера труб.
Примечание: Кластер Пробирки, содержащие лизирующий реагент можно хранить при температуре 2 - 8 ° С в течение 2 недель. - Использование длинных наконечников, передача 20 мкл вторичных обогащенных образцов (см шаги 3.2.1 и 3.2.2) в соответствующий кластер пробирки, содержащие 200 мкл реактива для лизиса. Используйте новые наконечники для каждого образца.
ПРИМЕЧАНИЕ: Держите трубы из первичного и вторичного обогащения в холодильнике (Salmonella) или при комнатной температуре (Cronobacter) для дальнейшего анализа подтверждения ПЦР-положительных / отрицательных образцов. - Подготовка отрицательного контроля, добавив 20 мкл стерильной BHI СМИ кластерных пробирки, содержащие 200 мкл реактива для лизиса.
- Подготовка положительного контроля, добавив 20 мкл O / N бактериальных культур (выращенный в BHI) любого известного Salmonella или деформации Cronobacter кассетных пробирки, содержащие 200 мкл реактива для лизиса.
- Cap кластера трубки и закрепите плотно с помощью укупорки инструмент.
- Поместите стойку кластера труб в автоматизированной нагревательного блока после выбора конкретной программы для целевой возбудителя. Кроме того, инкубировать кластера трубы при 37 ± 2 ° С в течение 20 мин с последующей инкубацией при 95 ± 3 ° С в течение 10 мин. И, наконец, перевести кластера трубки с охлаждающей блоков (2 - 8 ° С) в течение 5 мин.
Примечание: Кластер пробирки, содержащие лизата можно хранить при -20 ° С в течение 2 недель.
6. Выполните лизиса для обнаружения L. моноцитогенес
- Выполните первую частьлизиса следующим образом:
- Добавить 1,8 мл стерильной деионизированной воды в бутылке полностью талой раствор ющим средством 1.
ПРИМЕЧАНИЕ: Хранить не лизиса агента 1 на 2 - 8 ° С до готовности к использованию. После открытия и разбавления, хранить при комнатной температуре (20 - 30 ° С) в течение до 6 месяцев. - Комбинат лизиса агентам 1 и 2 в соотношении 4: 1 (40 мкл разбавленного лизирующим агента 1 и 10 мкл лизирующего агента 2 на каждый образец). Передача 50 мкл объединенных лизиса агентов для кластеризации трубки. Используйте смесь в течение 4 ч.
- Добавить 500 мкл первичного обогащенного образца (см шаг 3.2.3) к кластерной пробирку, содержащую 50 мкл объединенных лизиса агентов.
- Приготовьте отрицательный контроль путем добавления 500 мкл стерильной 24 LEB к 50 мкл объединенных лизиса агентов.
- Подготовка положительного контроля добавлением 500 мкл O / N Л. моноцитогенес культуры выращивают в 24 LEB к 50 мкл объединенных лизиса агентов.
- Крышка кластера трубы, аккуратно перемешать и местов нагревательном блоке при 37 ± 1 ° С в течение 30 мин.
Примечание: имейте трубы из первичного обогащения в холодильник для последующего анализа подтверждения ПЦР-положительных / отрицательных образцов.
- Добавить 1,8 мл стерильной деионизированной воды в бутылке полностью талой раствор ющим средством 1.
- Выполнение часть 2 лизиса следующим образом:
- Подготовка лизирующий реагент, как указано в пунктах 5.1 и 5.2.
- Использование длинных наконечники передать 20 мкл части первой лизата кластерных пробирки, содержащие 200 мкл реактива для лизиса. Используйте новые наконечники для каждого образца.
- Cap кластера трубки и закрепите плотно с помощью укупорки инструмент.
- Место кластера трубы в автоматизированной нагревательного блока выборе конкретной программе L. моноцитогенес. Кроме того, инкубировать кластера трубы на 55 ± 2 ° С в течение 30 мин с последующей инкубацией при 95 ± 3 ° С в течение 10 мин. И, наконец, перевести кластера трубки с охлаждающей блоков (2 - 8 ° С) в течение 5 мин.
Примечание: Кластер пробирки, содержащие лизата можно хранить при -20 ° С в течение 2 недель.
7. Сода ПЦР-готовых таблеток
- Выбор охлажденной (4 ° С) ПЦР блок охлаждения и поместить трубку стойки ПЦР на вставку.
- Поместите соответствующий ПЦР пробирки с ПЦР-готовые таблетки (включены в каждый набор) для цели пищевого возбудителя в держателе, в соответствии с файлом стойки.
- Использование decapping инструмент, аккуратно удалите колпачки с ПЦР-пробирки. Откажитесь от крышки и убедитесь, что каждая трубка содержит таблетку.
- Передача 50 мкл (для сальмонеллы и Cronobacter) или 30 мкл (для L. моноцитогенес) лизата к конкретным ПЦР пробирок. Используйте новые оптические шапки и закрепите плотно на пробирки для ПЦР с использованием укупорки инструмент.
Примечание: После добавления лизата с ПЦР-готовые таблетки, образцы должны оставаться охлажденным до 2 - 8 ° С до тех пор, пока загружен в ПЦР-системы детекции. ПЦР пробирки можно центрифугировали при 2500 х г в течение нескольких секунд, чтобы гарантировать, что полный объем в нижней тон трубка. - Загрузите пробирки для ПЦР в циклере / системы обнаружения инструмента PCR, открыв инструмент ящик.
- Поместите стойку пробирок для ПЦР в лунки в ящик и убедитесь, что трубы установлены правильно.
- Закройте ящик и запустить программу, как описано в соответствии с протоколом производителя.
ПРИМЕЧАНИЕ: Прибор на основе ПЦР имеет предустановленные параметры велосипедные для каждого пищевого возбудителя. - Убедитесь, что в строке состояния на велосипеде ПЦР показывает синюю полосу, указывающую, что усиление часть программы выполняется.
ПРИМЕЧАНИЕ: Для стандартных ПЦР-анализов, время обработки по полной программе (амплификации и детекции) занимает примерно 3 - 3,5 ч, чтобы закончить.
8. Обзор Результаты
- После завершения обработки, следуйте подсказкам на экране, от ПЦР-системы инструмента для удаления образцов и результаты обзора.
- Если цель пищевого возбудитель присутствует в образце (илиповерхность или пищеварительный тракт мухи) и красный с надписью «плюс» (плюс). Если возбудитель отсутствует, а зеленый со знаком «минус» (отрицательной).
- Если же желтый с красной полосой по центру, это указывает на ошибку сигнала.
9. Выделение бактериальных патогенов методом ПЦР положительные результаты
- Выберите трубки из основной (для L. моноцитогенес) или среднее (для сальмонеллы и Cronobacter) обогащения тех образцов, которые были ПЦР-позитивными. Кроме того, случайным образом выбирать 3 - 5% проб, которые были ПЦР-отрицательными и действуйте следующим образом:
- Для Salmonella:
- Добавить 100 мкл вторичного СМИ обогащения до 10 мл Раппапорт-Vassiliadis (RV) среды и 1 мл тетратионата (TT) бульона. Выдержите трубки 42,5 ° С на водяной бане рециркуляции для 22 - 24 часов.
- После инкубации полоска 3 мм петлей (10 мкл) каждого, Р. апd TT СМИ на висмут сульфит (BS) агар, ксилоза лизин дезоксихолат (XLD) агар, и Hektoen кишечно (HE) агар. Планшеты инкубируют при 35 ± 1 ° С в течение 22 - 24 часов.
- После инкубации пластин для изучения присутствии типичных колоний сальмонелл на каждой среды. Если нет изолированных колоний не могут быть получены после нескольких суб-культивирования шагов, рассмотрим пример негативной и сообщить, как ложное срабатывание для системы ПЦР-.
ПРИМЕЧАНИЕ: Для типичных колоний сальмонелл на конкретных средствах массовой информации смотри 22. Выберите пять предполагаемые типичные колонии Salmonella и субкультуры их на BS, XLD, либо он пока чистые культуры изолированных / отдельные колонии пока не будут получены. - Выберите один чистый колонии и определить предполагаемый сальмонеллы с помощью биохимических коммерческих тестов, таких как удостоверения личности VITEK 2 или API биохимические системы идентификации, следуя инструкциям изготовителя.
- Для Cronobacter: <ол>
- Серия 3 мм петлю (10 мкл) вторичного сред обогащения на двух пластинах хромогенных культуральных сред, таких как R & F Enterobacter sakazakii (Cronobacter) хромогенного гальванического среды, и / или ChromID Sakazakii агаре. Планшеты инкубируют при 35 ° С в течение 22 - 24 ч.
- После инкубации, изучить пластины на наличие типичных колоний Cronobacter (сине-черного до серо-голубой). Выберите 5 предполагаемых колоний Cronobacter и не субкультура их на R & F Enterobacter sakazakii (Cronobacter) Хромогенный покрытия среды, ChromID Sakazakii агар или АСП, пока чистые культуры изолированных / отдельных колоний получены.
ПРИМЕЧАНИЕ: Если нет изолированных колоний не могут быть получены после нескольких суб-культивирования шагов, рассмотрим пример негативной и сообщить, как ложное срабатывание для ПЦР-системы обнаружения. - Выберите один чистый колонии и определить предполагаемый Cronobacter с помощью биохимического Commerвые тесты, такие как удостоверения личности VITEK 2 или API 20E биохимические системы идентификации в соответствии с инструкциями изготовителя.
- Серия 3 мм петлю (10 мкл) первичного сред обогащения на двух пластинах агара Блеск Listeria (BLA). Планшеты инкубируют в 36 ± 1 ° С в течение 22 - 26 часов.
- После инкубации, изучить пластины на наличие предполагаемого L. моноцитогенес (сине-зеленый) колонии. Выберите 5 предполагаемый Л. моноцитогенес колонии и не субкультура их на BLA до получения чистых культур изолированных / отдельных колоний. Повторное инкубировать отрицательные пластины на 36 ± 1 ° С в течение дополнительных 22 - 26 ч.
- Выберите один чистый колонии и определить предполагаемый L. моноцитогенес с помощью коммерческого биохимические тесты, такие как удостоверения личности VITEK 2 или API Listeria биохимические системы идентификации, после ввода завода-изготовителяnstructions.
Результаты
Этот протокол был первым откалиброван на множестве лабораторных выращенных домашних мух, которые были экспериментально, подаваемого в течение 24 ч с жидкой пищи летучей (2% сухого молока), содержащего дублированные серийные разведения (10 2 - 10 8 КОЕ / мл) С sakazakii, С. enterica, Л. моноцитогенес или C. jejuni (п = 21 для каждого бактериальным патогеном). Обогащение СМИ, а также время инкубации и температуры были скорректированы для каждого пищевого возбудителя, пока система на основе ПЦР не удалось обнаружить самые низкие уровни бактерий (10 2 КОЕ / мл) с поверхности тела и желудочно-кишечного тракта в единый экспериментально подается летать. Использование средств массовой информации по обогащению и условия, описанные в разделе протокола, система на основе ПЦР обнаружены C. sakazakii, С. enterica, Л. моноцитогенес от поверхности тела 100% от мух, которых кормили бактериальных инокулята> 10 3 КОЕ / мл (рис 1a). Когда мухи питались 102 КОЕ / мл, процент обнаружения С sakazakii, С. enterica, Л. моноцитогенес с их поверхности тела был 100%, 66% и 33%, соответственно (рис 1а). Система на основе ПЦР обнаружены также эти три пищевых патогенов из желудочно-кишечного тракта мух, которых кормили всех бактериальных концентраций, при процентном ≥33% (рис 1б). Тем не менее, обнаружение С jejuni была достигнута только тогда, когда лабораторные выращенных мухи были экспериментально подают жидкий пищевой продукт, содержащий высокий бактериального инокулята (10 8 КОЕ / мл). Таким образом, C. jejuni был исключен из группы пищевых патогенов, которые могли бы быть проверены от индивидуального синантропных грязи мухи с помощью этой ПЦР-систему обнаружения.
С этого стандартизированного протокола, мы смогли определить распространенность Cronobacter SPP., С. enterica, Л. моноцитогенес от поверхности тела и / или ме желудочно-кишечного тракта 100 диких мух, которые были индивидуально и в асептических условиях, пойманной в районе корзины десяти городских ресторанов, расположенных в столичном районе Вашингтона, округ Колумбия 5 Сборник Filth мухи представитель по крайней мере, шесть видов, включая М. Domestica (47%), Lucilia cuprina (33%), Л. sericata (14%), Cochliomyia macellaria (2%), Sarcophaga haemorrhoidalis (2%), а Ophyra leucostoma (1%). Один муха была определена только уровне семьи (Anthomyiidae; 1%). Протокол поверхностно-дезинфекции была эффективна на предотвращение перекрестного загрязнения между частями тела лету, потому что нет роста бактерий не наблюдалось на TSA пластин для воды от последнего полоскания дезинфекции каждого отдельного лету. Таким образом, можно провести различие между пищевыми бактерий, присутствующих на частях тела каждого лету.
Нет ложных срабатываний не было обнаружено в образцах поверхности тела и пищеварительного трактаОтдельные мухи при использовании этого коммерческую систему на основе ПЦР для обнаружения S. enterica и Л. моноцитогенес и подтверждение жизнеспособных возбудителей на чашках с агаром был в согласии с ПЦР-положительных результатов. Однако, это не удалось выделить чистые культуры Cronobacter SPP. от всех ПЦР-положительных образцов. Таким образом, обнаружение этого патогена с помощью ПЦР на основе система показала ложных срабатываний от поверхности тела (50%; 9/18) и желудочно-кишечного тракта (48%; 16/33) отдельных пойманных мух. Произвольно выбранные ПЦР-отрицательных образцов, что высевали на конкретных СМИ, подтвердил отсутствие пищевых патогенов. Таким образом, нет ложноотрицательных не было обнаружено в любом из образцов при использовании этого коммерческую систему на основе ПЦР для обнаружения Cronobacter SPP., С. enterica или L. моноцитогенес.
Только те ПЦР-положительные образцы, где возбудитель был выделен и подтвержденные были рассмотрены положительнои включены для статистического анализа. Общая наличие пищевых патогенов в желудочно-кишечном тракте диких поймали Filth мухи было значительно выше, чем на поверхности тела (χ 2 = 6,8772, DF = 1, р = 0,0087). 22% из пищевых каналов и 8% поверхности тела, собранных диких мух были положительными, по крайней мере, один из трех пищевых патогенов (фиг.2). В целом, распространенность Cronobacter SPP. либо на поверхности тела или пищевых каналов собранных мух была выше (19%; точный критерий р Фишера = 0,0165), чем распространенность S. enterica (7%) и L. моноцитогенес (4%). Тем не менее, никаких статистических различий не наблюдалось при выполнении парных сравнений между частями тела мух для каждого бактериального патогена (рис 3; точный критерий р Фишера = 0,1464, р = 0,1184 и р = 0,6212 для CronobacterSPP., С. enterica, Л. моноцитогенес, соответственно). Ни один из мух не были положительными для всех трех возбудителей оценку. Тем не менее, три из мух (два Л. cuprina и один Л. sericata) осуществляется сальмонелла. и Л. моноцитогенес на поверхности или в желудочно-кишечном тракте.
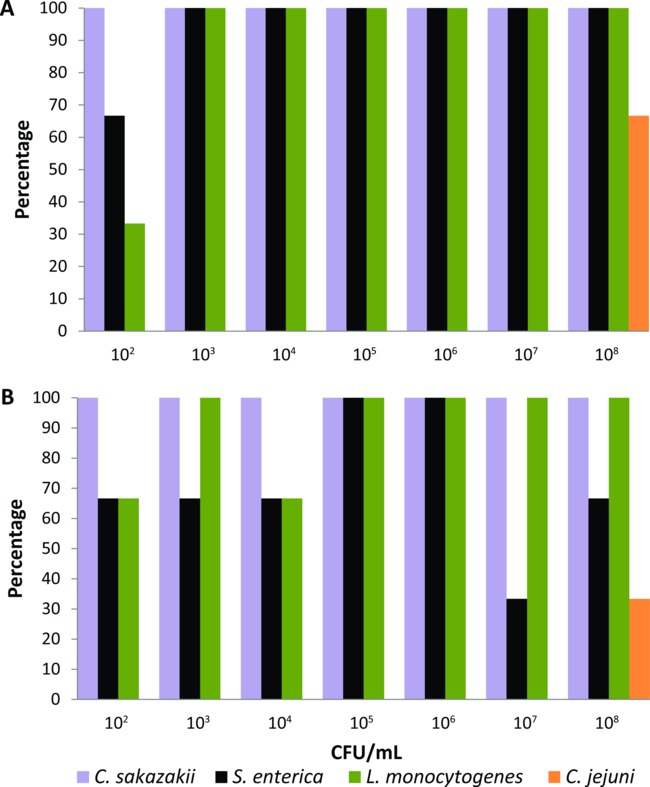
Рисунок 1. уровень обнаружения Cronobacter sakazakii, Сальмонелла энтерика, листерий и Campylobacter jejuni из (а) поверхности тела и (б) желудочно-кишечного тракта отдельных лабораторных выращенных дома мух кормили жидкой пищей, содержащей различных бактериальных инокулята (N = 21 для каждого бактериальным патогеном, п = 3 в каждой бактериальной концентрации).

Рисунок 2. Доля поверхности тела и пищевых каналов отдельных мух нашли положительный любой из целевых пищевых патогенов.
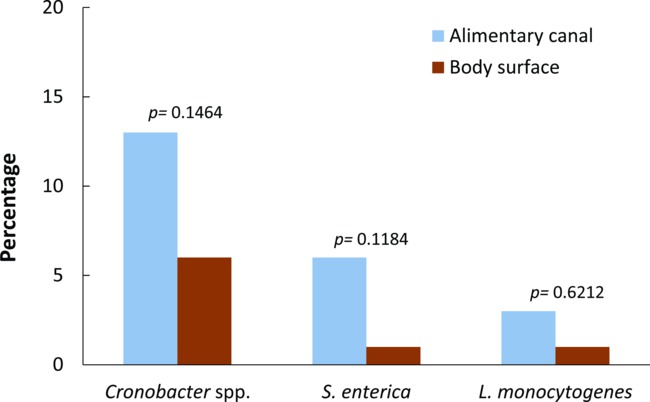
Рисунок 3. Распространенность Cronobacter SPP., Сальмонелла энтерика и листерий от поверхности тела и желудочно-кишечного тракта синантропных пойманных мух. Значения р сообщили бывают из парных сравнений между поверхностью тела и желудочно-кишечного тракта для каждого бактериального патогена (точный критерий Фишера, р значение <0,05 Статистическая значимость). Copyright © Американское общество по микробиологии, Журнал прикладной и экологической микробиологии 78 (22): 7891-902, 2012. DOI: 10,1128 / AEM.02195-12.
Обсуждение
Предыдущие исследования, которые выявляют пищевых патогенов от диких насекомых использовали большое разнообразие протоколов, которые не могут включать информацию, необходимую для точной оценки связанных с пищевыми продуктами риск наличия одного лету в пище или связанных с пищевыми продуктами условиях 13,15, 23,24. Здесь мы показали, что с помощью этого стандартизированного протокола, то можно обнаружить и изолировать Cronobacter SPP., С. enterica, Л. Monocytogenes с поверхности тела и желудочно-кишечного тракта отдельных мух, пойманных в дикой природе. Потому что насекомые могут нести низкие числа целевой пищевого возбудителя и большое число других коренных микрофлоры 25,26, этот протокол требует первичный (а иногда и вторичного) обогащение образцов в конкретных питательных сред для повышения чувствительности обнаружения целевой пищевого возбудителя , Результаты системы обнаружения на основе ПЦР были получены в течение примерно 30 ч (за DETEие из Cronobacter SPP. и С. enterica) и 48 ч (для обнаружения L. моноцитогенес) после первоначально обработки образцов. Таким образом, этот протокол является надежным, а также быстрое и достаточно чувствителен для выявления одного муху на наличие пищевых патогенов.
Подтверждение ПЦР-положительных результатов и изоляция жизнеспособных бактерий является частью стандартной операционной процедуры многих лабораториях. Кроме того, для целей эпидемиологии, чистые бактериальные культуры из ПЦР-положительных образцов должны дополнительно подтвердить и серотипа в пищевого происхождения патогена с использованием биохимических, иммунологических или генетических методов. Хотя не наблюдалось ложных срабатываний при обнаружении S. enterica и Л. моноцитогенес из частей тела отдельных пойманных мух, используя этот протокол, мы нашли до скорости ложных срабатываний для Cronobacter SPP 50%. Это говорит о том, что ПЦР-система обнаружения рода Cronobactэ может перекрестно реагировать с другими энтеробактерий присутствует среди весьма сложной микрофлоры осуществляется мухами. Таким образом, выделение и очистка из чистых колоний рода Cronobacter из ПЦР-положительных образцов требуют более селективное покрытие по сравнению с другими патогенами оцененных.
Данный протокол, прежде всего, были стандартизированы на экран отдельных пойманных мух на наличие Cronobacter SPP., С. enterica, Л. моноцитогенес используя коммерческую систему обнаружения на основе ПЦР. Тем не менее, этот протокол также легко адаптировать к экрану части тела отдельных мух на наличие других пищевых патогенов, таких как Энтерогеморрагическая кишечной палочки O157: H7 (либо с помощью кишечной палочки O157: H7 MP стандарт набора для анализа или кишечная палочка O157: H7 набор для анализа в режиме реального времени) и Шига-токсигенные E. палочки (STEC) группа (с использованием в режиме реального времени Стек Люкс), получение чувствительность> 80% (Отменить публикациюДанные ред). Кроме того, этот протокол потенциально могут быть приспособлены для обнаружения пищевых патогенов от других насекомых, которые, как известно, переносчики болезней (тараканов и муравьев), но больше исследований в этой области не требуется.
Болезни пищевого происхождения Исследования вспышек очень динамичны и включают в себя многоступенчатый процесс, который может меняться в зависимости от конкретной ситуации и локальное окружение расследуется 12,27. Эти исследования важны, потому что они обеспечивают мгновенную защиту общественного здравоохранения путем предотвращения будущих заболеваний. Кроме того, эти исследования могут пролить свет новые механизмы, с помощью которых распространяются пищевые микроорганизмы и поднимают важные вопросы, которые приводят к новым областям исследований 28. Методы расследования, а также стандартные, быстрые и чувствительные протоколы необходимы для обнаружения пищевых патогенов из отдельных насекомых. Это стандартизированный протокол открывает возможность асептически собирать насекомых, таких как мухи, WHICH могут вектора пищевых бактериальных патогенов, в рамках экологической программы отбора проб. Данные эпидемиологических исследований, которые могут быть получены из этого мог бы использоваться в построении точную картину о механизмах передачи пищевых патогенов насекомыми (например, длина Выдержка: муха на посадку по сравнению с мухами посадки, дефекации, и извергает).
Наконец, несмотря на то, коммерческая ПЦР-система обнаружения, описанный здесь, практичны в использовании и упрощает ПЦР-амплификации и визуализации родовой уровня ампликоне, это ни в коем случае не единственный соответствующую систему. Лизат из обогащенных образцов в качестве альтернативы может быть использован для скрининга на наличие пищевых патогенов с помощью общедоступных пары праймеров видоспецифической. Тем не менее, чувствительность обнаружения должна быть продемонстрирована до их использования.
Раскрытие информации
The use of specified instrumentation is not an endorsement by the U.S. Food and Drug Administration. The authors certify that there is no conflict of interest with any financial organization regarding the material discussed in this article.
Благодарности
Thanks to Ben D. Tall, Yi Chen, and Thomas Hammak from the U.S. Food and Drug Administration (FDA), Center for Food Safety and Applied Nutrition (CFSAN) for critically reviewing the manuscript. The authors also thank Hannah Lee (research internship program, Joint Institute for Food Safety and Applied Nutrition (JIFSAN), University of Maryland) for laboratory assistance and David Weingaertner (FDA, CFSAN) for preparing the figure of the schematic overview shown in the video.
Материалы
| Name | Company | Catalog Number | Comments |
| Bismuth sulfite (BS) agar | Fisher Scientific | R452402 | *Multiple suppliers. |
| Brain heart infusion (BHI) broth | Becton, Dickson and Company | 299070 | *Pre-warmed to 37°C. Multiple suppliers. |
| Brilliance Listeria agar (BLA) | Fisher Scientific | CM1080B | *Multiple suppliers. |
| Buffered peptone water (BPW) | Becton, Dickson and Company | 212367 | *Pre-warmed to 37°C or 42°C. Multiple suppliers. |
| Brilliance Cronobacter agar (Druggan-Forsythe-Iversen formulation/DFI) | Fisher Scientific | CM1055B | *Multiple suppliers. |
| chromID Sakazakii Agar | bioMérieux | 43741 | *Call for information: 800.682.2666 |
| R & F Enterobacter sakazakii (Cronobacter) Chromogenic Plating Medium | R & F Laboratories | Various | *Call for information: +1.630.969.530 |
| R & F Enterobacter sakazakiii Enrichment Broth and supplement | R & F Laboratories | Various | *Call for information: +1.630.969.530 |
| Hektoen enteric (HE) agar | Fisher Scientific | OXCM0419B | *Multiple suppliers. |
| 24 Listeria enrichment broth (24LEB) | Oxoid | CM1107 | *Freshly prepared at room-temperature. Multiple suppliers. |
| Listeria selective enrichment supplement | Oxoid | SR0243 | *Multiple suppliers. |
| Novobiocin | Fisher Scientific | OXSR0181E | *Multiple suppliers. Store at 2-8 °C |
| Vancomycin hydrochloride hydrate | Sigma Aldrich | 861987 | Store at 2-8 °C |
| Cefsulodin sodium salt hydrate | Sigma Aldrich | C8145 | Store at 2-8 °C |
| Rappaport-Vassiliadis (RV) medium | Fisher Scientific | CM0669B | *Multiple suppliers. |
| Tetrathionate (TT) Broth | Becton, Dickson and Company | 249120 | *Multiple suppliers. |
| Trypticase soy agar (TSA) | Becton, Dickson and Company | 236930 | *Multiple suppliers. |
| Xylose lysine desoxycholate (XLD) agar | Becton, Dickson and Company | 221284 | *Multiple suppliers. |
| API Biochemical identification system | bioMérieux | Various | *Call for information: +1.800.682.2666 |
| VITEK 2: Product Safety | bioMérieux | Various | *Call for information: +1.800.682.2667 |
| BAX System Q7 | DuPont | ||
| BAX E. sakazakii Standard assay kit | DuPont | D11801836 | |
| BAX L. monocytogenes 24E assay kit | DuPont | D13608125 | |
| BAX Salmonella 2 Standard assay kit | DuPont | D14368501 | |
| Capping tool | DuPont | D11677028 | |
| Decapping tool | DuPont | D11134095 | |
| PCR tube rack/holder | DuPont | D12701663 | |
| Featherweight forceps, wide tip | BioQuip | 4750 | Sterilize before use. Multiple suppliers. |
| Fine point, straight tip forceps | BioQuip | 4731 | Sterilize before use. Multiple suppliers. |
| Zirconia/silica beads, 0.5 mm | Bio Spec Products, Inc. | 11079105z | Multiple suppliers. |
| Petri dishes - 60X15mm | Fisher Scientific | 08-772B | Multiple suppliers. |
| Disposable inoculating loops, 10µL | Fisher Scientific | 22-363-606 | Multiple suppliers. |
| L-shaped cell spreaders | Fisher Scientific | 14-665-230 | Multiple suppliers. |
| Microcentrifuge tubes, 2 ml | Fisher Scientific | Various | Sterilize before use when needed. Secure lid is preferred. Multiple suppliers. |
| Cluster tubes | Fisher Scientific | 05-500-13 | |
| Cluster tubes caps | Fisher Scientific | 05-500-23 | |
| Sodium hypochlorite (Liquid chlorine bleach) | *Dilute to 0.05% with water. Multiple suppliers. | ||
| Sterile deionized water | Multiple suppliers. | ||
| Sterile distilled water | Multiple suppliers. | ||
| Ethyl alcohol 190 proof | *Dilute to 70% with water when needed. Multiple suppliers. | ||
| Genie cell disruptor, 120V - for 1.5ml and 2.0ml microtubes | Scientific Industries, Inc. | SI-D238 | Multiple suppliers. |
| Heating block | Multiple suppliers. | ||
| Cooling block | Multiple suppliers. | ||
| Recirculating water bath | Multiple suppliers. | ||
| Stereo microscope | Multiple suppliers. | ||
| Centrifuge | Multiple suppliers. | ||
| Incubator | Multiple suppliers. |
Ссылки
- Zurek, L., Gorham, J. R., Voeller, J. G. . Wiley Handbook of Science and Technology for Homeland Security. , 1-16 (2008).
- Olsen, A. R., Gecan, J. S., Ziobro, G. C., Bryce, J. R. Regulatory action criteria for filth and other extraneous materials V. Strategy for evaluating hazardous and nonhazardous filth. Regulatory Toxicology and Pharmacology. 33 (3), 363-392 (2001).
- Hald, B., Skovgård, H., Pedersen, K., Bunkenborg, H. Influxed insects as vectors for Campylobacter jejuni and Campylobacter coli in Danish broiler houses. Poultry Science. 87 (7), 1428-1434 (2008).
- Kobayashi, M., et al. Houseflies: Not simple mechanical vectors of enterohemorrhagic Escherichia coli O157:H7. American Journal of Tropical Medicine and Hygiene. 61 (4), 625-629 (1999).
- Pava-Ripoll, M., Pearson, R. E. G., Miller, A. K., Ziobro, G. C. Prevalence and relative risk of Cronobacter spp., Salmonella spp., and Listeria monocytogenes associated with the body surfaces and guts of individual filth flies. Applied and Environmental Microbiology. 78 (22), 7891-7902 (2012).
- Hewitt, C. G. . Houseflies and how they spread disease. , (1912).
- Rochon, K., Lysyk, T. J., Selinger, L. B. Retention of Escherichia coli by house fly and stable fly (Diptera:Muscidae) during pupal metamorphosis and eclosion. Journal of Medical Entomology. 42 (3), 397-403 (2005).
- Greenberg, B., Kowalski, J. A., Klowden, M. J. Factors affecting the transmission of Salmonella by flies: natural resistance to colonization and bacterial interference. Infection and Immunity. 2 (6), 800-809 (1970).
- Mramba, F., Broce, A. B., Zurek, L. Vector competence of stable flies, Stomoxys calcitrans L. (Diptera:Muscidae), for Enterobacter sakazakii. Journal of Vector Ecology. 32 (1), 134-139 (2007).
- Ekdahl, K., Normann, B., Andersson, Y. Could flies explain the elusive epidemiology of campylobacteriosis. Bmc Infectious Diseases. 5 (11), 11 (2005).
- Nayduch, D., Noblet, G. P., Stutzenberger, F. J. Vector potential of houseflies for the bacterium Aeromonas caviae. Medical and Veterinary Entomology. 16 (2), 193-198 (2002).
- Sievert, K., Messler, S., Klimpel, S., Feffer, K. Comprehensive study on the occurrence and distribution of pathogenic microorganisms carried by synanthropic flies caught at different rural locations in Germany. Journal of Medical Entomology. 46 (5), 1164-1166 (2009).
- Hernández-Escareño, J. J., et al. Presence of Enterobacteriaceae, Listeria spp., Vibrio spp. and Staphylococcus spp. in house fly (Musca domestica L.), collected and macerated from different sites in contact with a few animals species. Revista Cientifica-Facultad De Ciencias Veterinarias. 22 (2), 128-134 (2012).
- Mian, L. S., Maag, H., Tacal, J. V. Isolation of Salmonella from muscoid flies at commercial animal establishments in San Bernardino county, California. Journal of Vector Ecology. 27 (1), 82-85 (2002).
- Olsen, A. R., Hammack, T. S. Isolation of Salmonella spp. from the housefly, Musca domestica L., and the dump fly, Hydrotaea aenescens (Wiedemann) (Diptera:Muscidae), at caged-layer houses. Journal of Food Protection. 63 (7), 958-960 (2000).
- Holt, P. S., Geden, C. J., Moore, R. W., Gast, R. K. Isolation of Salmonella enterica serovar Enteritidis from houseflies (Musca domestica) found in rooms containing Salmonella serovar Enteritidis-challenged hens. Applied and Environmental Microbiology. 73 (19), 6030-6035 (2007).
- Mramba, F., Broce, A., Zurek, L. Isolation of Enterobacter sakazakii from stable flies, Stomoxys calcitrans L. (Diptera:Muscidae). Journal of Food Protection. 69 (3), 671-673 (2006).
- Koyuncu, S., Andersson, M. G., Haggblom, P. Accuracy and Sensitivity of Commercial PCR-Based Methods for Detection of Salmonella enterica in Feed. Applied and Environmental Microbiology. 76 (9), 2815-2822 (2010).
- Gagné, R. Insect and mite pests in food, an illustrated. 1, 269-296 (1991).
- Greenberg, B. . Flies and disease, Vol I. Ecology, classification and biotic associations. , (1971).
- Barro, N., Aly, S., Tidiane, O. C. A., Sababenedjo, T. A. Carriage of bacteria by proboscises, legs, and feces of two species of flies in street food vending sites in Ouagadougou, Burkina Faso. Journal of Food Protection. 69 (8), 2007-2010 (2006).
- Ugbogu, O. C., Nwachukwu, N. C., Ogbuagu, U. N. Isolation of Salmonella and Shigella species from house flies (Musca domestica L.) in Uturu, Nigeria. African Journal of Biotechnology. 5 (11), 1090-1091 (2006).
- Brownlie, J. C., Johnson, K. N. Symbiont-mediated protection in insect hosts. Trends in Microbiology. 17 (8), 348-354 (2009).
- Feldhaar, H. Bacterial symbionts as mediators of ecologically important traits of insect hosts. Ecological Entomology. 36 (5), 533-543 (2011).
- Wright, A. P., Gould, L. H., Mahon, B., Sotir, M. J., Tauxe, R. V. Overview of the Impact of Epidemic-Assistance Investigations of Foodborne and Other Enteric Disease Outbreaks, 1946-2005. American Journal of Epidemiology. 174, S23-S35 (2011).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены