Method Article
С открытым исходным кодом Анализ Одночастичного для супер-разрешение микроскопии с VirusMapper
В этой статье
Резюме
Эта рукопись использует Фиджи на основе открытого исходного пакета программного обеспечения VirusMapper для применения анализа одночастичных образами микроскопии сверхвысокого разрешения для того, чтобы генерировать точные модели наноразмерной структуры.
Аннотация
Супер-разрешение флуоресцентная микроскопия в настоящее время реконструирует исследования клеточной биологии. Его способность сломать предел разрешения около 300 нм позволяет для рутинного визуализации наноразмерных биологических комплексов и процессов. Это увеличение разрешения также означает, что методы популярные в электронной микроскопии, такие как анализ одночастичного, могут быть легко применены к супер-разрешение флуоресцентной микроскопии. Комбинируя этот аналитический подход с оптической визуализацией сверхвысокого разрешения, становится возможным воспользоваться молекулами конкретной маркировки емкости флуоресцентной микроскопии для генерации структурных карт молекулярных элементов внутри метастабильной структуры. С этой целью мы разработали новый алгоритм - VirusMapper - упакованное в качестве простого в использовании, высокая производительность, и высокой пропускной способностью ImageJ плагин. Эта статья представляет собой подробное руководство для этого программного обеспечения, демонстрируя свою способность раскрыть новые структурные особенности в биологических мolecular комплексы. Здесь мы представляем, как собрать совместимые данные и предоставлять протокол шаг за шагом о том, как использовать этот алгоритм, чтобы применить анализ одночастичную к изображениям супер-разрешением.
Введение
Супер-разрешение (SR) микроскопия оказала большое влияние на клеточной биологии, предоставляя возможность для изображения основных молекулярных процессов наряду с молекулярной конкретной маркировкой решающей значения для их понимания. SR теперь позволяет световой микроскопии , чтобы приблизиться к разрешения (20-150 нм) ранее только достижимые с электронной микроскопии (ЭМ), сохраняя при этом основные преимущества световой микроскопии, такие как потенциал для изображения живых клеток 1, 2. Кроме того, структурное сохранение найти на наноуровне позволяет применение анализа одночастичного (SPA) -SR данных, концепция широко используется в электронной микроскопии 3. Используя SPA, многие высоко консервативны копии структуры могут быть отображены и в среднем вместе, чтобы улучшить разрешение, точность, или отношение сигнал-шум визуализируемого объекта. При использовании в сочетании с SR, СП был продемонстрирован, чтобы быть мощным инструментом для высокого рrecision отображение компонентов комплекса пор ядерной 4, 5, 6, центросомах и вирусы, такие как ВИЧ , 7 и HSV-1 8.
Однако, процедура комбинированное применение SR и SPA была поставлена под сомнение из-за отсутствия доступного программного обеспечения. По этой причине мы разработали VirusMapper, плагин для популярного программного обеспечения для обработки изображений ImageJ / Фиджи 9. Это первый свободно доступный пакет программного обеспечения для обобщенной SPA с флуоресцентными изображениями 10 , предназначенных для обеспечения быстрого, удобный, многоканальное наивное усреднения структур изображаемых с SR микроскопией. Несмотря на то, предназначены для вирусов, он может быть применен к любому макромолекулярному комплексу, в котором различные молекулярные частицы могут быть отображены, идентифицированный и локализованным.
VirusMapper может быть использован для получения высокой точности молекулярныхмодели любой известной структуры, что позволяет для расчета средних размеров и других параметров. Дизайн алгоритма делает его особенно полезным для разделения популяций структур, обеспечивающее для определения различных ориентаций или различных морфологических состояний. Кроме того, многоканальное формирование изображение может быть использовано для использовать опорный канал в тех случаях, когда основная структура хорошо известно, тем самым позволяя для обнаружения опорной на основе структуры. Инструкции по загрузке и установке программного обеспечения предоставляются на https://bitbucket.org/rhenriqueslab/nanoj-virusmapper . Пример данных также можно найти там, и пользователям рекомендуется практиковать с помощью программного обеспечения на примере данных, прежде чем пытаться применить его к своей собственной.
Здесь шаги для использования этого плагина для создания SPA модели из исходных данных описаны. Программное обеспечение принимает сырые изображения, содержащих одинарные Oг мульти-меченые структуры в качестве входных данных. Он возвращается, с учетом ряда параметров, которые настраиваются как программное обеспечение запускается, модели SPA, показывающие средние распределения меченых компонентов в пределах изображаемых структур.
Целью данного протокола является получение точных SPA модели , дающей средние локализаций компонентов внутри отображенных структур в соответствии с трубопроводом , описанным на фиг.1. Как показано на рисунке 1, рабочий процесс программного обеспечения целесообразно разделить на три этапа. Первая стадия заключается в сегменте больших изображений, что приводит к стопкам частиц для каждого канала. Эти частицы являются единицами, которые будут усреднены для создания моделей и для производства семян для генерации модели. Второй этап заключается в создании семенных изображений, которые используются, чтобы зарегистрировать весь набор частиц в конечной стадии. Это делается путем выбора опорного канала и ручной выбор частиц в этом канале, что будет способствовать сeeds. Семена выбираются в этом опорном канале, но могут быть сгенерированы для всех каналов. Частицы первоначально перестроены путем установки 2D гауссово в этом канале. Все частицы, которые были выбраны и пересмотренные затем усредняются для получения семян. Для каждой общей структуры видели в данных, которые должны быть смоделированы, частицы должны быть выбраны в качестве семян, которые четко и точно представляют эту структуру. Интерфейс на этом этапе также полезен для сканирования данных для таких структур.
На заключительном этапе для создания моделей с помощью сопоставления шаблона. Это достигается за счет регистрации частиц, первоначально экстрагированных к семенам изображений, полученных в предыдущем разделе по кросс-корреляции. Подмножество зарегистрированных частиц в среднем вместе, и процесс далее итерация, чтобы уменьшить модель среднеквадратичной ошибки, если это желательно. Это подмножество определяется путем установки минимального сходства против семени, которое должно быть выполнено. При создании моделиев одновременно в нескольких каналах, совместное сходство, или среднее значение сходства для каждого канала, используются. Полученные модели и зарегистрированные частицы, которые способствовали их могут быть затем дополнительно проанализированы.
протокол
Примечание: Этот протокол и видео дополняют оригинальный документ , описывающий 10 пакет программного обеспечения более подробно. Читателям рекомендуется ознакомиться с этим внимательно дополнительными указаниями относительно использования программного обеспечения. Существуют три основные этапа: извлечение частиц, какие сегменты больших изображений на отдельные частицы; Выбор семян, где общие структуры определены в данном и выровнены для получения семян, которые используются в завершающей стадии; и генерация модели, где соответствующий шаблон на основе этих семян выравнивает Извлеченный частиц и усредняет подмножество для производства моделей SPA.
1. Установка Перед Запуск программного пакета
- Подготовка образцов структуры при исследовании на покровном или в соответствующих экспериментальных условиях.
- Изображение образцы с супер-разрешением флуоресцентной микроскопии, такие как структурированного освещения микроскопии (SIM) или 11 StimulaИстощение Ted выбросов (STED) 12 микроскопии.
ПРИМЕЧАНИЕ: Точные детали того, как подготовить и образцы изображений в значительной степени зависит от характера исследуемой структуры, поэтому соответствующая литературе следует проводить консультации. В качестве примера, точный способ получения и отображения образцов вируса осповакцины, таких как те, которые используются здесь, описан в разделе результатов репрезентативного. - Создание образов нескольких полей зрения, показывающих большое количество отдельных копий структуры или частиц, предпочтительно тысячи. Частицы изображения, которые так же, отделенные друг от друга, как это возможно, и обеспечить, чтобы изображения были свободны от грязи или других флуоресцентных структур, которые не интересно.
- Открыть все изображения, содержащие частицы в Фиджи путем перетаскивания файлов в панели инструментов Фиджи или выбрав «Файл»> «Открыть».
- Выберите «Изображение»> «Стек»> «Инструменты»> «Объединить» в сроститьизображения в один стек. Затем, если полученное изображение является HyperStack, превратить его в стек, выбрав «Image»> «Hyperstacks»> «HyperStack в стек».
Примечание: Окончательный стек должен иметь интеркалированные каналы. Если имеются два канала, первый срез в стеке должен быть канал 1 с первого изображения, второй срез должен быть соответствующий канал 2, третий срез должен быть канал 1 из второго изображения, и так далее.
2. Извлеките Particles
- Выберите изображение для сегмента и выберите «Извлечь Вирусные Structures». Выберите место для сохранения извлекаемых частиц и просмотра «Extract Вирусных Structures» Диалоговое окно (Рисунок 2).
- Назначение параметров экстракции с первоначальными оценками, введя их в «Извлечь Вирусные Structures» диалог следующим образом. Тонкая настройка этих параметров после просмотра сегментации изображения.
- Установите онемелиэр каналов в наборе данных , как количество различных каналов флуоресценции , которые были изображаемого (например, 2).
- Установите опорный канал, из которого частицы извлекаются путем обнаружения пиков в этом канале путем ввода номера выбора канала. Выберите наиболее последовательный канал; то есть, канал, в котором большинство частиц имеют одинаковый внешний вид.
Примечание: Если возможно, частицы в этом канале будут иметь центральный максимум. - Выберите, следует ли применять предварительно обнаружения Гаусс. Установка предварительного обнаружения Гаусса до 0 применять не размытость; если это значение увеличивается, гауссово размывание фильтр заданного радиуса применяется до локального обнаружения максимумов. Используйте эту функцию , если опорный канал не имеет центральный максимум (например, кольцо формы); размывание вызывает появление одного.
Примечание: сегментированные области интереса (трансформирования) не имеют гауссово размытие применительно к ним, так как эта функция только использованиед в положение трансформирования в опорном канале. - Установите радиус ROI (который будет установлен вокруг каждого локального максимума) в пикселях. Выберите значение таким образом, что , что трансформирования немного больше , чем крупные частицы, такие , как на фиг.3. Например, если появляются самые крупные частицы имеют диаметр около 30 пикселей (по оценкам примерно на глаз), а затем установить радиус ROI до 20 пикселей.
- Установить количество трансформирования использовать для каждого кадра на начальное, относительно небольшое значение ниже 100.
- Установите максимальный ROI перекрытия. Если частицы хорошо разделены, держать этот маленький; если частицы группируются, увеличить это позволить трансформирования перекрываться.
- Выберите «показать превью».
Примечание: При выборе этой опции, экстрагированные трансформировании для опорного канала, связанный с текущим кадром будут отображаться. - Регулировка радиуса ROI, количество трансформирования и максимальный ROI перекрытие, чтобы иметь трансформирование подходящего размера примерно столько же частицы, как это возможно, Как показано на рисунке 3.
- Выберите «OK» для запуска сегментации. Закройте изображение и менеджер ROI.
Примечание: Не изменяйте имена файлов наборов частиц. Эти имена должны быть в формате «вирусных частиц - channelX» для следующих разделов.
3. Выберите Семена
- Выберите «Создать Seeds», выберите папку , в которой сохраняются извлеченные частицы, а также просматривать «Сформировать Seeds» диалогового окна и (Рисунок 4).
- Назначают начальные параметры семян отбора путем их ввода в диалоге «Создать Seeds», следующим образом.
- Установите опорный канал, который будет устанавливаться для выравнивания и центра всех каналов. Выберите канал, в котором большинство частиц имеют одинаковый внешний вид и имеет центральный максимум, если это возможно.
- Установите флажки для всех каналов, для которых семена должны быть сгенерированы.
- Выберите, если семена должны быть rotatред на 90 °. Используйте эту функцию, чтобы иметь выравнивание в соответствии с другими моделями
- Выберите, следует ли применять предварительно выравнивания Гаусса. Установите радиус размытия для каждого канала 0 применять не размытости. Увеличьте это значение, чтобы применить Гауссово размывание фильтра заданного радиуса перед тем перестройкой. Используйте эту функцию, если частицы не имеют центральный максимум; размывание вызывает появление одного.
Примечание: Семена не будут иметь пятна применяется, так как эта функция только для получения последовательного выравнивания. - Выберите, следует ли использовать коррекцию смещения отдельно центрирования семян не эталонные каналы, хотя и не вращать их, с помощью флажков «коррекция сдвига» коробок для каждого канала.
Примечание: Используйте это, если каналы не хорошо согласованы друг с другом; он также может быть полезен для выравнивания других каналов исключительно по ссылке, без коррекции сдвига.
- Выберите частицы для использования в качестве семян. Поиск по тон частица последовательности, чтобы найти частицу, которая напоминает структуру, которая должна быть смоделирована и записать номер кадра.
- Введите номер в «Frames использовать» окно, и появится больше окон (рисунок 5). Введите несколько номеров кадров, разделенных запятыми.
- Просмотр семенных кадров и результирующие средних семян в окнах, которые появляются.
Примечание: Журнал предложит семена, похожие на среднее. - Регулировка опорного канала, радиус гауссова размытия и параметры коррекции сдвига, чтобы оптимизировать процесс отбора семян, так что количество затравочных кадров, обнаруженных имеют схожий внешний вид. Продолжайте добавлять семена, пока средние семена не будут созданы, удовлетворительно напоминают структуру видели в данных, которые должны быть смоделированы.
- Назовите семена и где они будут сохранены и выберите «OK», чтобы сохранить конечные семенные изображения для последующего использования в производстве модели.
4. Сформировать модели
- SeLect «Генерация моделей на основе семян», выберите папку , в которой сохраняются извлеченные частицы, а также просматривать «Сформировать модели» диалоговое окно (Рисунок 6).
- Загрузите семена для каждого канала с помощью флажков.
Примечание: Семена будут сохранены в папке, которая была названа в «Generate» Семена диалога. По умолчанию используется имя «Анализ». Позаботьтесь , чтобы выбрать семенную средние, а не рамы, которые также сохраняются для справки. - Назначают начальные параметры генерации модели, введя их в диалог «Создать модели», следующим образом. Тонкая настройка этих параметров позже.
- Выберите, следует ли использовать опорный канал для выравнивания, который вычисляет переводы и вращение только из опорного канала и применяет их ко всем каналам. При использовании этой опции, выберите канал для использования в качестве ссылки.
Примечание: Этот параметр следует выбирать только специально использовать один канал в качестве refereсть, например, если это открытие ссылочной на основе структуры. В противном случае, это приведет к снижению точности моделей. Каналы должны быть хорошо выровнены при использовании этой опции. - Выберите ли квадрат интенсивности изображения во время согласования шаблона. Это подчеркнет небольшие различия, поэтому использовать это при создании модели, которая имеет особенно тонкие черты.
- Выберите минимальное сходство в отношении семян с помощью «Минимального сходства против семени» ползунка или введите число в поле.
Примечание: только частицы с сходством семян больше, чем будет использоваться этим отсечка. 60-80%, как правило, хороший выбор, чтобы начать с. - Установите максимальное число итераций 1, оптимизировать минимальное сходство в отношении семян, а также увеличить его позже, по мере необходимости.
- Установите флажки для выбора элементов процесса создания модели, которая появится.
Примечание: «Показать семена» покажет семена, которые были загружены с Boxes в верхней части диалогового окна. «Показать модели» будут отображать все итерации модели, которые создаются при усреднении подмножества частиц, которое отвечает минимальное сходство от семени. «Показать MSE» покажет средний квадрат ошибки (MSE) изображение, которое выдвигает на первый план области модели, которые являются наиболее изменчивы. «Показать частицы» будут отображать подмножество частиц, которые используются для создания моделей, зарегистрированное в соответствии с высочайшей итерацией модели, которая отображается.
- Выберите, следует ли использовать опорный канал для выравнивания, который вычисляет переводы и вращение только из опорного канала и применяет их ко всем каналам. При использовании этой опции, выберите канал для использования в качестве ссылки.
- Выберите «Показать карту» для создания моделей просмотра и просмотра результатов.
Примечание: Это наиболее интенсивных вычислений шаг процесса. Время работы для набора нескольких тысяч частиц с диаметром в несколько десятков пикселей на настольном компьютере должна быть около 10 мин. Если время вычисления является проблемой, пользователи должны сначала попробовать алгоритм на меньшее подмножество данных или использовать меньший радиус ROI в шаге 2.2.4, если это возможно. - Просмотр ГенерироватьD модели и оптимизации параметров, в частности минимальное сходство в отношении семян. Увеличение минимального сходства против семени, пока только истинные частицы моделируемой морфологии не включены в модель.
- Увеличение максимального числа итераций с помощью «максимального числа итераций» ползунка или путем ввода числа в поле и позволяют процесс генерации модели для итерации. Используйте значение около 10 для максимальной итерации.
- Название модели и выберите «OK» для сохранения модели стеки эволюции, которые содержат все итерации конечного процесса генерации модели.
Примечание: Если минимальное сходство с семенами настолько высоко, что частицы не имеют это сходство, ничего не будет обновляться. Если плагин, кажется, замораживать, рассмотреть возможность того, что минимальное сходство слишком высоко.
Результаты
Здесь мы демонстрируем программное обеспечение на модель поксвирусной, вирус коровьей оспы. Одним из наиболее сложных вирусов млекопитающих, коровьих пакетов около 80 различных белков в пределах 350 х 270 х 250 нм 3 кирпич-образной форме частицы 13, 14. Три подструктур различимы с помощью электронной микроскопии: центральное ядро, которое содержит геном днДНКа; две белковые структуры, называемые боковые тела, которые фланкируют ядро; и один протеолипидный бислой конверт 15. Большой размер, сложная структура, и аменабельность с рекомбинантным белком флуоресцентного мечения сделать осповакцины отличной систему, чтобы продемонстрировать рабочий процесс VirusMapper.
Используя программное обеспечение, как описано здесь, распределение различных белков на коровьем вирионе может быть смоделировано. Белок был маркирован и отображается, возможно, в комбинации с другим белком, известным распределением в качестве эталона, и программное обеспечение было использовано, как описано для получения средних моделей локализации этого белка на частицу. В этом примере два белка были смоделирована, внутренний L4 ядра белка, и основной компонент F17 бокового тела.
Рекомбинантный вирус коровьей оспы , который имеет F17 с меткой GFP и L4 с меткой была использована mCherry 16. Очищенный вирус разводили в 1 мМ Трис, рН 9, и связан с покровными промывают, высокопроизводительного путем покрытия их в течение 30 мин при комнатной температуре. Затем образцы фиксировали с применением 4% формальдегида в PBS в течение 20 мин. Покровные были немедленно установлены на слайдах antifade монтажной среды. Визуализация проводили с помощью SIM-карты на коммерческую SIM микроскопе. Поле зрения был выбран, содержащий сотни вирусов и изображения были получены с использованием 5 фазовых сдвигов и 3 сетки вращения с 561 нм (32 мкм решетки PeRIOD) и 488 нм (32 мкм периода решетки) лазеров. Изображения были получены с помощью камеры sCMOS и обрабатываются с помощью программного обеспечения микроскопа. Каналы были выровнены на основе разноцветного бисера слайда изображаемого с теми же параметрами захвата изображений. После реконструкции и выравнивания канала изображения SIM были открыты в Фиджи и объединены в один стек изображения.
Вирусные частицы были извлечены из изображений с использованием канала L4 в качестве эталона и без применения какого-либо Гауссово размывания, так как эти частицы имеют центральный максимум. Около 15000 частиц были извлечены в этом эксперименте.
Из-за геометрии коровьей оспы, боковые тела имеют отчетливо различный внешний вид, основанный на ориентации вируса. Мы визуализировали две ориентации, в которой один или два боковых тела можно было различить. Мы ссылались на эти ориентации как фронтальные и сагиттальные, по отношениюively.
Отдельные семена для фронтальной и сагиттальной ориентации были выбраны путем поиска списка частиц на стадии «Генерировать Семена» (фиг.4 и 5); Частицы, которые явно находились в одной ориентации или другие были выбраны. Канал L4, был использован в качестве опорного канала для выравнивания семян друга с другом. Опять же, не Гауссово размывание не было необходимости. 5 частиц для каждой ориентации были выбраны и были усреднены для получения семян.
Модели были созданы для каждой ориентации на основе этих семян. были использованы Ни опорный канал, ни квадратов значений интенсивности. Максимальное число итераций было установлено изначально на 1, а минимальное сходство было установлено, чтобы включать в себя около 1000 частиц в каждом случае, что дало последовательное возникновение для каждой ориентации. Максимальное число итераций затем увеличивали допозволяют сходимости модели. Модели были , таким образом , генерируется для двух ориентаций в двух каналах (рисунок 7).
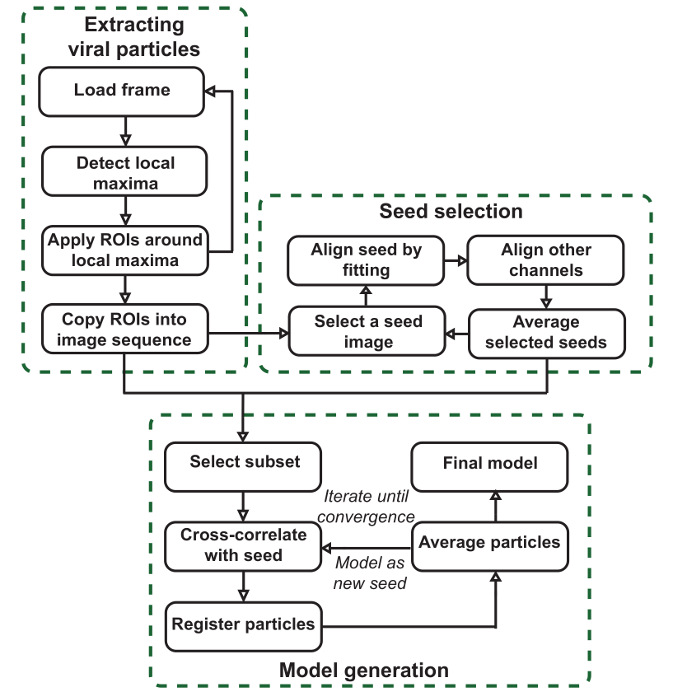
Рисунок 1: VirusMapper рабочий процесс. Плагин состоит из трех основных этапов. Вирусные частицы извлекаются из больших изображений, шаблонов изображений или семена выбирают полу-вручную из данных, и конечные модели SPA генерируются из данных со ссылкой на семена. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
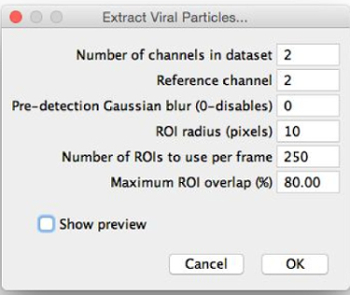
Рисунок 2: «Извлечение Вирусные структур» диалог. При выборе «Extract Вирусный Структур эс», это диалоговое окно появится. Эти параметры должны быть заполнены с начальными оценками для оптимальной сегментации.„Показать предварительный просмотр“ , то можно выбрать, позволяя трансформирование быть просмотрено и параметры , чтобы быть доработаны. Пожалуйста , нажмите здесь для просмотра большая версия этой фигуры.

Рисунок 3: Установка параметров экстракции. После предварительного просмотра трансформирования, которые будут извлечены, радиус ROI, количество трансформирования, и максимальный ROI перекрытия регулируют так, чтобы достичь подобной ситуации. Трансформирования немного больше, чем частицы, все частицы включены в ROI и трансформировании могут перекрываться в достаточной степени, чтобы позволить частицы кластерных быть разделены.апк "> Пожалуйста, нажмите здесь, чтобы посмотреть увеличенную версию этой фигуры.
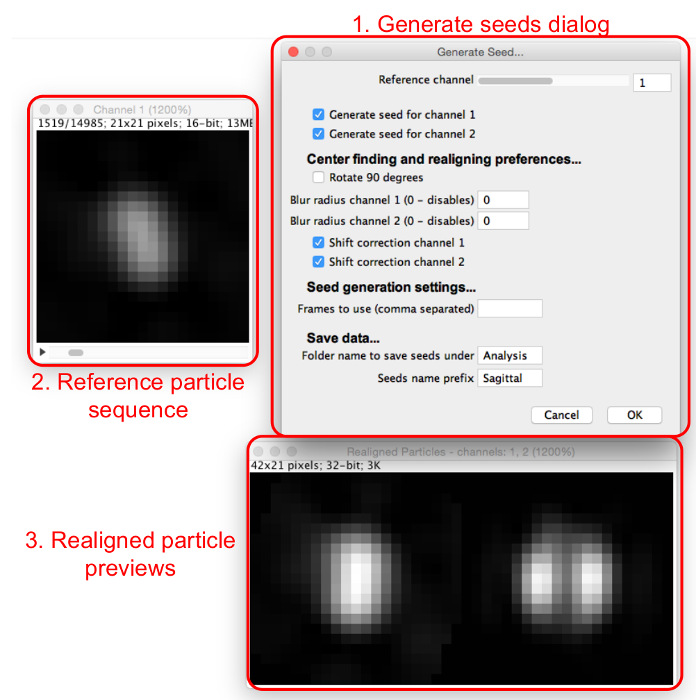
Рисунок 4: Создание семян , соответствующих шаблонов. «Создать Seeds» Диалог (1) устанавливает параметры, которые будут назначены. Последовательность опорных частиц (2) позволяет пользователю сканировать через частицы в опорном канале. Когда частица рассматриваются в последовательности опорных частиц перераспределяются частицы для всех каналов можно рассматривать в перераспределяемых предварительных просмотрах частиц (3). Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
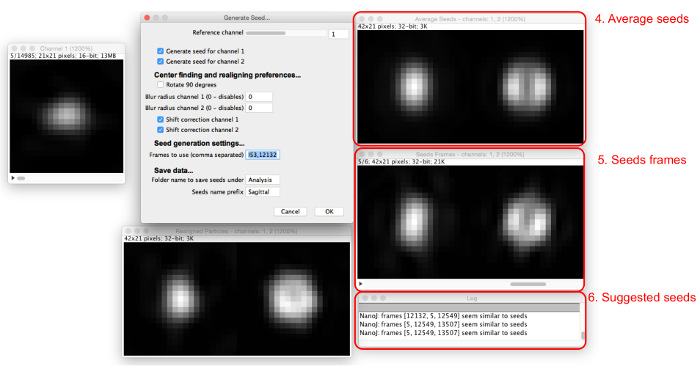
Рисунок 5: Добавление семян изображений. Как семена добавляют к «Рамки использовать» окно, (5) отображаются среднее значение всех семян (4) и кадры участвуют. Частицы, которые похожи на нынешние средние семена предлагаются в диалоговом окне (6). Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
Рисунок 6: «Сформировать модели» диалоговое окно. При выборе «Сформировать модели на основе семян» появится это диалоговое окно. Параметры должны быть заполнены начальными оценками для выработки оптимальной модели, и элементы процедуры генерации модели, которые будут показаны во время расчета должны быть выбраны. «Показать предварительный просмотр», то может быть выбран, позволяя процесс генерации модели для запуска и параметры, чтобы быть доработаны.ftp_upload / 55471 / 55471fig6large.jpg»целевых =„_blank“> Пожалуйста, нажмите здесь, чтобы посмотреть увеличенную версию этой фигуры.
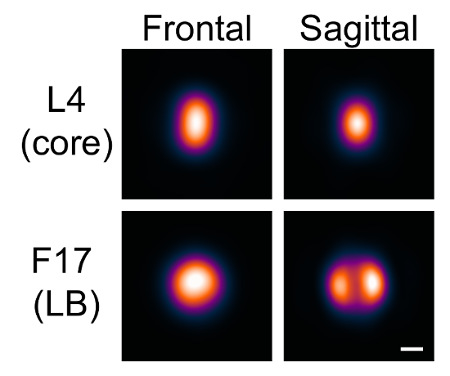
Рисунок 7: Модели , созданные с VirusMapper. Коровья оспа вирионы с коровым белком L4 меченого с mCherry и белком тела F17 бокового помеченный EGFP были обследованы с помощью SIM-карты. Модели были затем генерируется с помощью программного обеспечения, как описано в протоколе. Две ориентации, фронтальная и сагиттальная, отличаются по внешнему виду боковых тел. Шкала бар = 100 нм. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
Обсуждение
С помощью этого метода исследователи оснащены, чтобы объединить мощь SPA и SR микроскопии с целью получения высокой точности, многоканальные 2D модели архитектуры белков вирусов и других макромолекулярных комплексов. Тем не менее, некоторые важные соображения должны быть приняты во внимание.
Семена должны быть выбраны, чтобы представить структуру, которая последовательно видел. Таким образом, исходные данные должны быть проверены тщательно перед выбраны семенами. Это важно для предотвращения предвзятых моделей. Выбор может быть подтвержден рассмотрением минимального пороговых значений подобия, необходимых для включения определенного количества частиц в моделях. Ясно, что при выборе семян, тем выше этот порог должен быть для заданного числа частиц, тем более, что структура является очевидной в данных.
Концепция согласования шаблона является особенно полезной, когда существует гетерогенность в данных. Все различные структуры, которые VIsible должна быть определена и различные модели созданы для каждого случая. При разделении гетерогенных структур в одном канале, но одновременно создавая модели во втором канале, узоры могут возникнуть, что бы не было сразу видно.
Еще одно соображения необходимо учитывать при использовании этого алгоритма является то, что итерационная процедура будет максимально стохастическая асимметрия. Например, при моделировании структуры с двумя симметричными максимумами, все небольшие асимметрии между максимумами будут выровнены друг с другом во время итерации, и окончательная модель будет, таким образом, максимально асимметрична. Если это не отражает известную симметрию в структуре моделируемого, то это должно быть принято во внимание. В настоящее время единственный способ избежать этого максимизации, чтобы ограничить число итераций 1, хотя потенциал развитие будет для VirusMapper включать ось симметрии в процесс генерации модели. Любые новые версии VirusMapper будет AvaiКонтрактное на который ссылается веб - сайт (см Материалы таблицу). Кроме того, пользователи найдут FAQ здесь, чтобы ответить на любые общие запросы.
Программное обеспечение, как описано применимо к любой структуре, которая может быть отображена с достаточным разрешением, чтобы визуализировать функции, которые пользователь желает, чтобы смоделировать. Хотя SPA может улучшить разрешение, это явно не улучшит видимость компонентов, которые в противном случае не видны. Этот протокол не является, следовательно, способом улучшения качества данных. Как и с любой техникой, тщательной подготовки проб и оптимизации стратегии формирования изображения обеспечит чистейшие данные и лучшие модели результирующие.
Выбор метода визуализации SR также имеет важное значение и, в целом, будет зависеть от образца под рукой. VirusMapper была подтверждена хорошо работать с SIM - картой и STED 10, и она также может быть использована с данными локализации микроскопии высокого качества, но следует соблюдать осторожность в этом случае,как разреженная маркировка может вызвать проблемы, аналогичные асимметрии максимизации.
В настоящее время VirusMapper является единственным в свободном доступе алгоритма для анализа одночастичного флуоресцентных изображений и только общего назначение 2D SPA усреднения программного обеспечения. Другие исследования , которые сделали использование одних и тех же принципов , 4, 6, 8 использовали специальное программное обеспечение специализированное для каждого конкретного исследования. Алгоритмы общего назначения для реконструкции 3D данных были опубликованы 5, 18, хотя программное обеспечение не было.
При использовании, как описаны в этой статье, VirusMapper может быть использован для получения точных, точные и надежных моделей макромолекулы архитектуры белков вирусов и других комплексов. С помощью этих моделей исследователи могут принимать точные измерения средних размеров структурыУРЭС изучаемые, потенциально позволяя им достичь биологические выводы, которые иначе не могли бы было возможно.
Кроме того, с возможностями многоканальных этой техники, можно сопоставить неограниченное количество белков и компонентов внутри комплексов и обнаружить новую организацию белка. Изучение изменений в наноразмерных структурах в различных биологически соответствующих условиях, например, разные стадиях жизненного цикла вируса, имеет потенциал, чтобы предложить ценную информацию в биологию.
Раскрытие информации
Авторы не имеют ничего раскрывать.
Благодарности
Мы хотели бы поблагодарить Корина Бирли Джерзи Самолеж, Педро Матос Pereira, Кристофер Блек и Катрин Шерер за их вклад в первоначальную разработку и валидацию VirusMapper. Мы хотели бы также поблагодарить Артур Якимович за критическое прочтение рукописи. Эта работа финансируется за счет грантов от биотехнологии и биологических наук Исследовательского Совета (BB / M022374 / 1) (RH); Основное финансирование в лаборатории MRC для молекулярной биологии клетки, University College London (JM); Исследовательский Совет Европы (649101-UbiProPox) (JM); и Совет по медицинским исследованиям (MR / K015826 / 1) (RH и JM). RG финансируется инженерных и физических наук Исследовательского Совета (EP / M506448 / 1).
Материалы
| Name | Company | Catalog Number | Comments |
| Fiji | Open-source image analysis software | ||
| NanoJ-VirusMapper | developed by the Henriques lab | Open source-Fiji plugin (https://bitbucket.org/rhenriqueslab/nanoj-virusmapper) | |
| VectaShield antifade mounting medium | Vector Labs | H-100 | |
| Elyra PS1 | Zeiss | ||
| ZEN BLACK | Zeiss | Image processing software for SIM | |
| High performance coverslip | Zeiss | 474030-9000-000 | |
| TetraSpeck beads | ThermoFisher | T7279 |
Ссылки
- Henriques, R., Griffiths, C., Rego, E. H., Mhlanga, M. M. PALM and STORM: Unlocking live-cell super-resolution. Biopolymers. 95 (5), 322-331 (2011).
- Gustafsson, N., Culley, S., et al. Fast live-cell conventional fluorophore nanoscopy with ImageJ through super-resolution radial fluctuations. Nat Commun. 7, 12471 (2016).
- Cheng, Y., Grigorieff, N., Penczek, P. A., Walz, T. A Primer to Single-Particle Cryo-Electron Microscopy. Cell. 161 (3), 438-449 (2015).
- Szymborska, A., de Marco, A., Daigle, N., Cordes, V. C., Briggs, J. A. G., Ellenberg, J. Nuclear pore scaffold structure analyzed by super-resolution microscopy and particle averaging. Science. 341 (6146), 655-658 (2013).
- Broeken, J., Johnson, H., et al. Resolution improvement by 3D particle averaging in localization microscopy. Methods Appl Fluoresc. 3 (1), 14003 (2015).
- Burns, S., Avena, J., Unruh, J., Yu, Z. Structured illumination with particle averaging reveals novel roles for yeast centrosome components during duplication. Elife. , (2015).
- Lelek, M., Di Nunzio, F., Henriques, R., Charneau, P., Arhel, N., Zimmer, C. Superresolution imaging of HIV in infected cells with FlAsH-PALM. Proc Nat Acad Sci U S A. 109 (22), 8564-8569 (2012).
- Laine, R. F., Albecka, A., Svan de Linde, ., Rees, E. J., Crump, C. M., Kaminski, C. F. Structural analysis of herpes simplex virus by optical super-resolution imaging. Nat Commun. 6, 5980 (2015).
- Schindelin, J., Arganda-Carreras, I., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Gray, R. D. M., Beerli, C. VirusMapper: open-source nanoscale mapping of viral architecture through super-resolution microscopy. Sci Rep. 6, 29132 (2016).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. J of Micros. 198 (2), 82-87 (2000).
- Hell, S. W., Wichmann, J. Breaking the diffraction resolution limit by stimulated emission: stimulated-emission-depletion fluorescence microscopy. Opt Let. 19 (11), 780 (1994).
- Chung, C. -. S., Chen, C. -. H., Ho, M. -. Y., Huang, C. -. Y., Liao, C. -. L., Chang, W. Vaccinia virus proteome: identification of proteins in vaccinia virus intracellular mature virion particles. J Virol. 80 (5), 2127-2140 (2006).
- Moss, B. Poxviridae: the viruses and their replication. Fields virology. , (2010).
- Condit, R. C., Moussatche, N., Traktman, P. In a nutshell: structure and assembly of the vaccinia virion. Adv Virus Res. 66 (6), 31-124 (2006).
- Schmidt, F. I., Bleck, C. K. E., et al. Vaccinia virus entry is followed by core activation and proteasome-mediated release of the immunomodulatory effector VH1 from lateral bodies. Cell Rep. 4 (3), 464-476 (2013).
- . Nanoj-virusmapper Available from: https://bitbucket.org/rhenriqueslab/nanoj-virusmapper (2016)
- Fortun, D., Guichard, P., Unser, M. Reconstruction From Multiple Poses in Fluorescence Imaging: Proof of Concept. IEEE J Sel Topics Signal Process. 10 (1), 61-70 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены