Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Объектив Бесплатные видео микроскопии для динамического и количественный анализ культуры адэрентных клеток
В этой статье
Резюме
Объектив Бесплатные видео микроскопии позволяет нам контролировать клеточных культур непосредственно внутри инкубатора. Здесь мы описываем полный протокол, используемый для приобретения и анализировать 2,7 дня приобретения культивируемых клеток HeLa, ведущих к dataset 2,2 х 106 измерения отдельных клеток морфологии и 10584 клеточного цикла треков.
Аннотация
Здесь мы показываем, что объектив Бесплатные видео микроскопии позволяет нам одновременно захватить кинетика тысяч клеток непосредственно внутри инкубатора, и что это возможно для мониторинга и количественной оценки одиночных клеток вдоль нескольких циклов клеток. Мы описываем полный протокол, используемый для мониторинга и количественной оценки культуре клеток HeLa 2.7 дней. Во-первых, приобретение культуры клеток производится с микроскопом объектив Бесплатные видео, и затем данные анализируются следующие четыре этапа: мульти волны голографической реконструкции, клетки слежения, клетки сегментации и деление клеток обнаружения алгоритмы. В результате мы покажем, что это возможно собрать dataset с участием более чем 10000 клеточного цикла треков и более чем 2 х 106 клеток морфологических измерений.
Введение
Мониторинга культивируемых клеток млекопитающих на протяжении нескольких циклов клеток и точного измерения ячеек размер и клетки сухой массы является сложной задачей. Несколько лейбл бесплатно оптические методы могут выполнять эту задачу1,2: фазосдвигающие интерферометрии3, голографические цифровой микроскопии (DHM)4,5,6, 7, quadriwave боковой наклон интерферометрии8,9 и количественную фазу томография10,11. Эти методы привели к многие новые идеи в понимании клеточного цикла mammalian клеток. Однако они редко связаны с автоматической ячейки отслеживания алгоритмов и их пропускная способность остается ограниченным, при измерении ячейки массового траекторий1 (N < 20 соответственно3,4,5 , 6). Следовательно, Роман оптический метод необходим для измерения массы клеток траекторий с большой статистики (N > 1000).
В этой статье мы продемонстрировать способность объектива Бесплатные видео микроскопии одновременно изображения тысячи клеток непосредственно внутри инкубатора, и затем определить метрики одну ячейку вдоль тысячи отдельных клеточного цикла треков. Объектив свободный микроскопии является количественных этап визуализации технику, которая позволяет приобретение фазы изображения плотно упакованных клеток через очень большое поле зрения (обычно несколько десятков мм2, здесь 29,4 мм2)12,13 ,14,15. Определяются несколько метрик на уровне отдельной ячейки, например, ячейка области, клетки сухой массы, клетки толщина, длина главной оси клеток и клеточной пропорции12,15, от каждого изображения. Затем применив алгоритм отслеживания клеток, эти особенности могут быть отображены для каждый single cell как функция время эксперимента,14,,15. Кроме того выявляя возникновение клеточных делений в ячейке треков, можно извлечь другие важные сведения, такие как первоначальная клеточная сухой массы (сразу после деления клетки), окончательный клеточной сухой массы (непосредственно перед деление клеток) и клетки цикла, длительность, то есть, время между двух последовательных подразделений15. Все эти измерения может быть вычислена с очень хорошей статистики (N > 1000) поскольку большое поле зрения обычно позволит анализ 200 до 10 000 ячеек в одной свободной объектив приобретения.
Для того, чтобы объяснить эту методологию, основанную на объектив Бесплатные видео микроскопии, мы описываем протокола для мониторинга и количественной оценки культуре клеток HeLa 2.7 дней. Анализ данных является 4 ступенчатый процесс, основанный на мульти волны голографической реконструкции, отслеживания клеток, клеток сегментации и деление клеток алгоритмов. Здесь показано, что пространственное разрешение и относительно высокой частоте кадров (одно приобретение каждые 10 минут), полученные с этой установки объектива Бесплатные видео микроскопии совместим с стандартных алгоритмов клеток отслеживания. Полный анализ этого набора данных приводит к измерение 10,584 клеток треков на полных цикла клетки.
Подводя итог, объектив Бесплатные видео микроскопии является мощным инструментом автоматически контролировать тысячи неподписанных, несинхронизированные и неизмененной клетки за эксперимент; Каждая ячейка, отслеживается в течение нескольких циклов клеток. Таким образом, наши измерения обеспечивают среднее значение нескольких параметров клеток, но что более важно, между клеток изменчивость над большой популяции клеток.
протокол
1. клеточная культура мониторинг приобретения
- Рост клеток HeLa в среде DMEM + глютамин (например, GlutaMAX) средний, дополненная 10% (v/v) тепло инактивированная плода теленка сыворотки и 1% пенициллина и стрептомицина.
- Покройте 6-ну дно культуры стекла с фибронектина (25 мкг/мл) за 1 ч. Затем семена 2 х 104 клетки на хорошо.
- В процессе приобретения измените носитель каждые 3 дня.
- Промежуток времени приобретения используйте видео объектив свободный Микроскоп (коммерчески доступных).
Примечание: Это на основе изображений, объектив свободной вычислительной технике как описано Ozcan и др. 16 , который был изменен для выполнения постоянного мониторинга в инкубаторе при контролируемой температуре 37 ° C12,15. К услугам гостей комплементарный металло-оксидный полупроводник (CMOS) сенсор с пикселей 1,67 микрон и площадью изображений 6.4 x 4,6 мм2. Несколько волн освещение обеспечивается multichip света Светоиспускающий диод (СИД) устройство, которое обеспечивает освещение красного, зелёного и синего. Длины волн центрируются, 636, 521 и 452 Нм соответственно с спектральный 25, 45 и 25 Нм соответственно. Красный зеленый синий (RGB) светодиоды расположены выше 150 мкм обскуры на расстоянии приблизительно в 5 см от клеток. Силы света, измеренная рядом светодиод как низко как 10 мкВт на каждой длине волны и время освещения только одну секунду на приобретение. Следовательно никаких проблем фото токсичность, как ожидается, когда клеточных культур наблюдаются под микроскопом объектив Бесплатные видео. - Поместите контейнер культуры клеток, положить в контакте с CMOS-датчик (рис. 1). Используйте контейнер с стекла coverslip внизу, которая имеет важное значение для качества приобретения объектив бесплатно. Можно также использовать пластиковые контейнеры, но приобретения свободной объектив будет ухудшаться вследствие плохой оптическое качество этих контейнеров.
- Управление видео объектив свободный Микроскоп с приобретения программного обеспечения (коммерчески доступных), который выполняет промежуток времени приобретения и голографической реконструкции. Параметры, которые должны вводиться пользователем в интерфейсе программного обеспечения являются частота кадров, продолжительность эксперимента и тип клеточной культуры, т.е. адэрентных клеток или плавающей клетки.
- Входных параметров, приобретение программного обеспечения. Задайте частоту кадров присвоено одно приобретение каждые 10 минут и установить «приверженца» тип культуры клеток.
Примечание: Частота кадров одного приобретения каждые 10 минут представляет собой хороший компромисс, поскольку она обеспечивает надежный ячейки отслеживания в то время как размер результирующего набора данных для анализа по-прежнему разумным. Быстрый частоты кадров достижимые с этой установки объектива бесплатно микроскопии — одно приобретение каждые 5 минут. Дальнейшее увеличение частоты кадров приводит к чрезмерного нагрева CMOS сенсор, который может влиять на жизнеспособность клеток в культуре. - Запустите промежуток времени приобретения, и голографической реконструкции алгоритм (коммерчески доступных) будет автоматически обрабатывать свободной объектив приобретения для получения изображения этапа культуры клеток. Голографические реконструкции алгоритм основывается на нескольких волны фазы поиска алгоритм17 и обеспечивает реконструированный фазы изображения RGB в диапазоне [-π, + π] для каждой одной ячейки над большое поле зрения 29,4 мм2. Принцип свободной объектив в линии голографии является освещать тонкий образец (z = 0) с плоской волны (амплитуды 1 после нормализации) и для обнаружения на CMOS-датчик изображения интенсивность последующих дифракции на короткое расстояние z = Z, обычно в 1 мм после Пример. В нашей установки, освещение последовательно переключается на 3 длинах волн (λ1= 0,450 мкм, λ2= 0.540 мкм, λ3= 0.647 мкм) для измерения три модели дифракции той же пробы.
2. клетки культуры анализ данных
- Для отслеживания ячейки, используйте Trackmate алгоритм, открытым исходным кодом Фиджи плагин для автоматизированного отслеживания одной частицы18. На первых, загрузить полный промежуток времени приобретения в Фиджи (нагрузки изображений последовательности команд). Ввиду большого размера наборов данных, который имеет обычно 400 RGB кадры 29,7 МБ, это быстрее, чтобы загрузить их в 8-битном формате. Далее модуль Trackmate Фиджи проводит пользователя через несколько этапов алгоритма отслеживания клеток, а именно стадии обнаружения клетки, стадии трекера ячейки и несколько фильтров, применяемых к обнаружений клеток и вычисляемые треков. На каждом этапе настройки алгоритмов и отображать результаты сразу так, что, при необходимости, одно могут легко перемещаться вперед и назад перенастраивать параметры. Различные меню интерфейса Trackmate показаны на рисунке 2 с фактические параметры, используемые для эксперимента клетки HeLa.
- Задайте входные параметры приобретения программного обеспечения как после (см. Рисунок 2). Установите оценкам blob диаметром до 15 пикселей, детектор порог до 0,25, увязки максимальное расстояние до 15 пикселей, разрыв закрытие Макс расстояние до 15 пикселей и количество мест в треки 3.5.
Примечание: Эти параметры будут явно отличаются от одного эксперимента на другой, особенно «оценкам blob диаметр» которая соответствует размер ячейки (приведены в пикселях 1,67 мкм). То же замечание относится к «ссылок Макс расстояние», которых приходится максимальное расстояние, пройденное ячейки в течение двух последовательных кадров (приведены в пикселях 1,67 мкм). Его оптимальное значение будет зависеть от подвижности клеток, но также на плотность клеток. - В конце процесса отслеживания клеток, генерировать результаты в виде трех текстовых файлов («Анализа» кнопку, см. Рисунок 2). Наиболее полезным файл, названный «Места в статистике треки», состоит из таблица с перечнем всех обнаруженных клетки с их соответствующие (x, y) позиции по приобретению, их номер кадра и их номер трека. На основе этих данных выполняют клетки сегментации и выявления клеточных делений, с помощью специальных алгоритмов (см. файлы дополнительного кода). Два других файлов названы «Ссылки в статистике треки» и «Отслеживать статистику» и включают все результаты, касающиеся треков, например трек длительностей, количество обнаруженных недостатков, отслеживать начальный кадр, и т.д.
- Использовать алгоритм сегментации клеток (см. дополнительный код файл) автоматически извлечь несколько метрик, описанием морфологии клеток из реконструированных фазы изображения. Эти метрики являются площадь поверхности клетки (S), клетки главной оси Длина (L), клетки малая ось Длина (l) и пропорции клеток (L/l). Фаза, оправился от свободной объектив микроскопии пропорциональна плотности и толщины слоя образца, как обсуждалось ранее,15.
- Помимо морфологических особенностей ячейки экстракт количественных клетки сухой массы (МЧР) измерения из реконструированных этап13,19,20
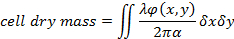 Уравнение (1)
Уравнение (1)
где φ(x,y) это реконструированный фазы сдвиг19, длина волны λ и α = 1.8 x 10-4 м3/кг конкретных преломления, который касается изменения показателя преломления для сухой массы1, 19. - Оцените толщину ячейки с помощью оценки разницы между клеток преломления и что средства массовой информации культуры. Связь между ячейки толщиной h(x,y) и измеренных фазовый сдвиг задается20:
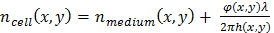 Уравнение (2.1)
Уравнение (2.1) Уравнение (2.2)
Уравнение (2.2)
с Δn(x,y) = nклеток (x,y) - nсреднего (x,y), преломления разница между ячейкой и культуры средств массовой информации. В следующем, предположим постоянное значение Δn = 0,025, который оценивается от преломления, измеренная в ядрах клеток HeLa (n = 1.35511) и что фосфат амортизированное saline (PBS) культуры средств массовой информации (n = 1,33). Хотя толщина значение ячейки является лишь ориентировочной, поскольку она основана на предположении, его относительные различия имеют смысл и может быть использована для выявления деления клеток. - Используйте специальный алгоритм (см. файл дополнительного кода) обнаружить возникновение клеточных делений и затем извлечь клетки треков, которые связаны с обнаруженного клеточных делений, чтобы обнаружить деления клеток, клеток с измеренной толщины, больше, чем 8 мкм сначала определяются, а затем проверить ли новая ячейка появляется тесно в пространстве и времени. Таким образом обнаружения клеточных делений опирается на двух надежных критериев. 21,22,на lensfree в промежуток времени приобретения23могут применяться альтернативные и более сложные методы обнаружения деление клеток.
Результаты
Для процесса голографической реконструкции, светлое поле описывается скалярным полем A (где 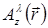 комплекс значение A на плоскости на расстоянии z из образца и боковой позиции
комплекс значение A на плоскости на расстоянии z из образца и боковой позиции 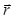 и на длине ...
и на длине ...
Обсуждение
В этой статье мы покажем, что объектив Бесплатные видео микроскопии может использоваться внутри инкубатор для захвата кинетика тысяч клеток. Для того чтобы описать общую методологию, мы объяснили, как промежуток времени приобретения 2.7 день клеток HeLa в культуре могут быть проанализиро...
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Авторы не имеют ничего подтвердить.
Материалы
| Name | Company | Catalog Number | Comments |
| Cytonote lens-free video microscope | Iprasense | ||
| Horus acquisition software | Iprasense | ||
| 6-well glass bottom culture plates | MatTek corporation | Part No: P06G-0-14-F | |
| DMEM + GlutaMAX medium | Gibco | ||
| heat-inactivated fetal calf serum | Eurobio | ||
| penicillin and streptomycin | Gibco | ||
| Fibronectin | Sigma Aldrich | ||
| Matlab, image processing toolbox | Mathworks |
Ссылки
- Zangle, T. A., Teitell, M. A. Live-cell mass profiling: an emerging approach in quantitative biophysics. Nat Methods. 11 (12), 1221-1228 (2014).
- Popescu, G., Park, K., Mir, M., Bashir, R. New technologies for measuring single cell mass. Lab Chip. 14 (4), 646-652 (2014).
- Reed, J., et al. Rapid, massively parallel single-cell drug response measurements via live cell interferometry. Biophys J. 101 (5), 1025-1031 (2011).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci. USA. 108 (32), 13124-13129 (2011).
- Girshovitz, P., Shaked, N. T. Generalized cell morphological parameters based on interferometric phase microscopy and their application to cell life cycle characterization. Biomed Opt Express. 3 (8), 1757-1773 (2012).
- Kemper, B., Bauwens, A., Vollmer, A., Ketelhut, S., Langehanenberg, P. Label-free quantitative cell division monitoring of endothelial cells by digital holographic microscopy. J Biomed Opt. 15 (3), (2010).
- Mir, M., Bergamaschi, A., Katzenellenbogen, B. S., Popescu, G. Highly sensitive quantitative imaging for monitoring single cancer cell growth kinetics and drug response. PLoS One. 9 (2), 1-8 (2014).
- Bon, P., Savatier, J., Merlin, M., Wattellier, B., Monneret, S. Optical detection and measurement of living cell morphometric features with single-shot quantitative phase microscopy. J Biomed Opt. 17 (7), (2012).
- Aknoun, S., et al. Living cell dry mass measurement usinq quantitative phase imaging with quadriwave lateral shearing interferometry: an accuracy and sensitivity discussion. J Biomed Opt. 20 (1), 1-4 (2015).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Choi, W., et al. Tomographic phase microscopy. Nat Methods. 4 (9), 717-719 (2007).
- Kesavan, S. V., et al. High-throughput monitoring of major cell functions by means of lensfree video microscopy. Sci Rep. 4, 1-11 (2014).
- Zheng, G., Lee, S. A., Antebi, Y., Elowitz, M. B., Yang, C. The ePetri dish, an on-chip cell imaging platform based on subpixel perspective sweeping microscopy (SPSM). Proc Natl Acad Sci USA. 108 (41), 16889-16894 (2011).
- Pushkarsky, I., et al. Automated single-cell motility analysis on a chip using lensfree microscopy. Sci Rep. 4, 4717 (2014).
- Allier, C., et al. Imaging of dense cell cultures by multiwavelength lens-free video microscopy. Cytom Part A. 91 (5), 1-10 (2017).
- Su, T. -. W., Seo, S., Erlinger, A., Ozcan, A. High-throughput lensfree imaging and characterization of a heterogeneous cell solution on a chip. Biotechnol Bioeng. 102 (3), 856-868 (2009).
- Delacroix, R., et al. Cerebrospinal fluid lens-free microscopy: a new tool for the laboratory diagnosis of meningitis. Sci Rep. 7, 39893 (2017).
- Tinevez, J. -. Y., et al. TrackMate: an open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2016).
- Popescu, G. Optical imaging of cell mass and growth dynamics. Am J Physiol Physiol. 295 (2), 538-544 (2008).
- Liu, P. Y., et al. Cell refractive index for cell biology and disease diagnosis: past, present and future. Lab Chip. 16, 634-644 (2016).
- Rapoport, D. H., Becker, T., Mamlouk, A. M., Schicktanz, S., Kruse, C. A novel validation algorithm allows for automated cell tracking and the extraction of biologically meaningful parameters. PLoS One. 6 (11), e27315 (2011).
- Al-Kofahi, O., et al. Automated cell lineage construction: A rapid method to analyze clonal development established with murine neural progenitor cells. Cell Cycle. 5 (3), 327-335 (2006).
- Meijering, E., Dzyubachyk, O., Smal, I., van Cappellen, W. A. Tracking in cell and developmental biology. Semin Cell Dev Biol. 20 (8), 894-902 (2009).
- Posakony, J. W., England, J. M., Attardi, G. Mitochondrial growth and division during the cell cycle in HeLa cells. J Cell Biol. 74 (2), 468-491 (1977).
- Zocchi, E., et al. Expression of CD38 Increases Intracellular Calcium Concentration and Reduces Doubling Time in HeLa and 3T3 Cells. J Biol Chem. 273 (14), 8017-8024 (1979).
- Reitzer, L. J., Wice, B. M., Kennell, D. Evidence that glutamine, not sugar, is the major energy source for cultured HeLa cells. J Biol Chem. 254 (8), 2669-2676 (1979).
- Benedetti, A. D. E., Joshi-barve, S., Rinker-Schaeffer, C., Rhoads, R. E. Expression of Antisense RNA against Initiation Factor eIF-4E mRNA in HeLa Cells Results in Lengthened Cell Division Times, Diminished Translation Rates, and Reduced Levels of Both eIF-4E and the p220 Component of eIF-4F. Mol Cell Biol. 11 (11), 5435-5445 (1991).
- Kumei, Y., Nakajima, T., Sato, A., Kamata, N., Enomoto, S. Reduction of G1 phase duration and enhancement of c-myc gene expression in HeLa cells at hypergravity. J Cell Sci. 93 (2), 221-226 (1989).
- Ginzberg, M. B., Kafri, R., Kirschner, M. On being the right (cell) size. Science. 348 (6236), 1245075 (2015).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены